基于COI基因的摇蚊亚科部分属的系统发育分析(双翅目:摇蚊科)
2015-11-01王婷婷王新华闫春财
姜 丽,闫 娇,王婷婷,郭 琴,王新华,闫春财
(1.天津师范大学a.生命科学学院,b.天津市动植物抗性重点实验室,天津300387;2.南开大学 生命科学学院,天津300071)
摇蚊科隶属双翅目长角亚目(Diptera:Nematocera),摇蚊科种类约占淡水湖沼生物区系的25%,其幼虫生物量约占底栖动物总量的70%~80%.由于摇蚊科昆虫具有个体相对较大、易获得、生活习性多样、区域性强、底栖生活等优点,因此是监测水体环境和污染状况的优良指示生物[1].
摇蚊亚科是摇蚊科中仅次于直突摇蚊亚科的第2大亚科,目前对摇蚊亚科的研究相对较少.摇蚊亚科有3个族:摇蚊族(Chironomini,至少 69个属)、长跗摇蚊族(Tanytarsini,至少16属)和伪摇蚊族(Pseudochironomini,至少5属).由于形态学和核型特征的不同造成了复杂的物种多样性和较小的属间差异,出现了基因重组和趋同进化,导致物种鉴定的难度加大和摇蚊物种分类地位的不明确.为解决这一问题,近年来分子系统学利用标记基因在物种分类方面取得了一些进展.这些标记基因的特点是多态性高、共显性遗传、选择中性、容易获得、容易操作及重复性好.线粒体细胞色素氧化酶I基因(COI)自2004年以来被广泛应用于昆虫的条形码研究[2].迄今,一个包含超过8万种昆虫信息的数据库已经形成[3].广泛多态性和丰富的可用数据使COI基因成为一个能够鉴定物种多样性的分子系统学工具.Guryev[4]第一次尝试基于COI的核苷酸序列和线粒体DNA的细胞色素b基因(Cytb)研究摇蚊科的系统发育.然而,Guryev仅利用了几个属(Chironomus、Kiefferulus、Sergentia、Polypedilum和Axarus)建立系统发育树来反映亚科的系统发育关系.此后,Sharma[5]基于rDNA的ITS1和ITS2建立系统发育树,确定了摇蚊族9个属的物种地位.Ekrem等[6]基于线粒体COI基因的核苷酸序列获得了长跗摇蚊族(Tanytarsini)5个属的亲缘关系,包括2个核基因(FE1和CAD)及3个线粒体基因(COI、COⅡ和16s RNA)的分析[7].关于Chironomus和Sergentia的属内关系[8-11]也有少量报道.迄今为止,所有的摇蚊亚科系统发育重建均包含数目有限的属和种,属间系统发育关系仍然不明确.
本研究基于摇蚊亚科39属48种的COI基因序列,拟重建摇蚊亚科属级之间的系统发育关系.
1 材料和方法
1.1 样品来源
实验所用的摇蚊样品主要来自野外采集,采集方式包括网捕和灯诱.标本放置于-20℃的冰箱中保存.经形态学初步鉴定,供系统发育研究的摇蚊样本详细信息如表1所示,在GenBank下载的25种摇蚊COI基因的相关序列信息如表2所示.

表1 实验样品来源Tab.1 Sourceof samples
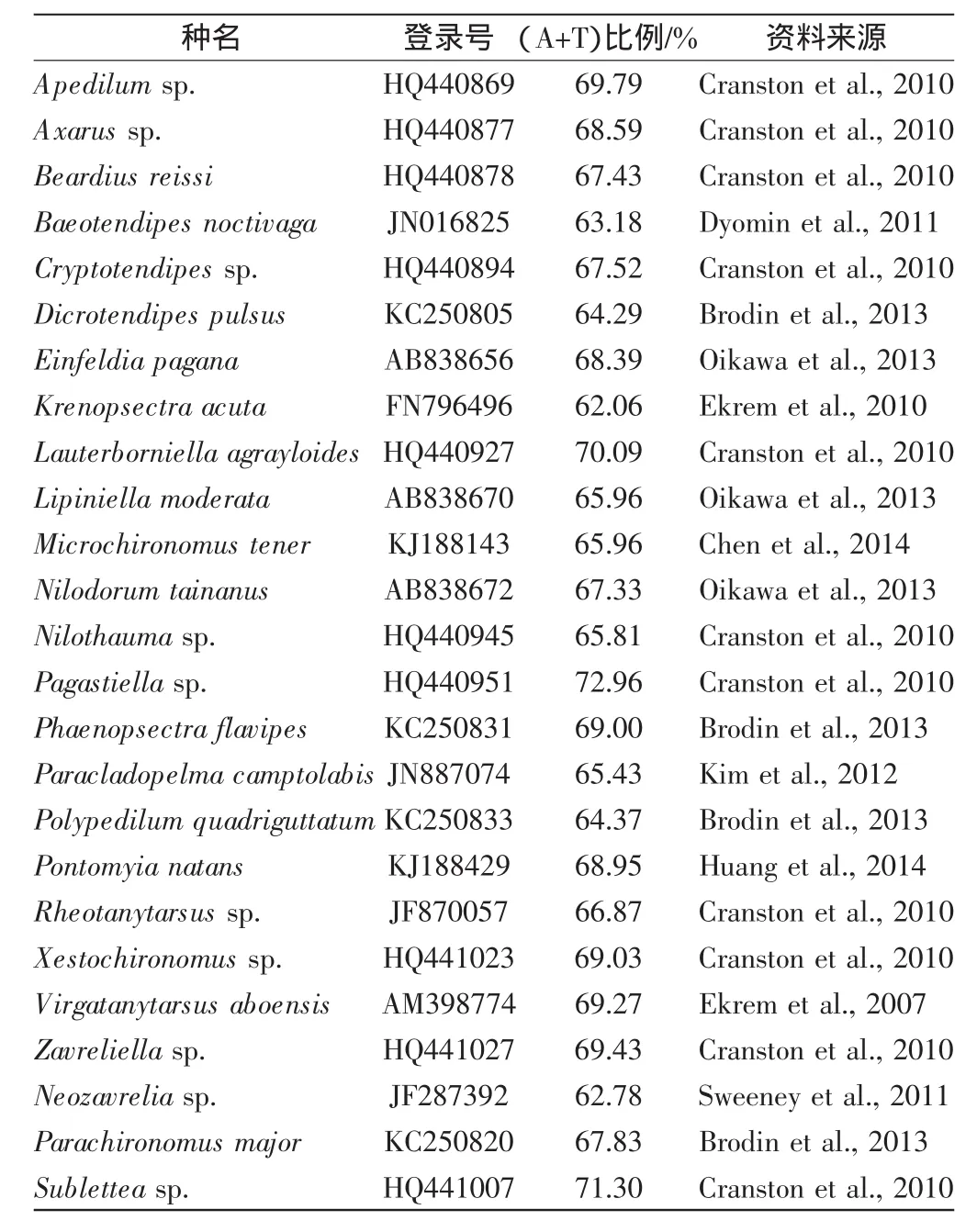
表2 25种摇蚊COI基因的相关信息Tab.2 Information of COI genesof 25 species
1.2 DNA的提取
取头部、触角、翅、足、腹部和生殖节,用加拿大树胶做永久装片,为种类鉴定提供依据,剩下的胸部用于DNA提取.使用天根生化科技有限公司生产的血液/细胞/组织/基因组DNA提取试剂盒,按照使用说明书,微调部分操作步骤进行DNA提取.
1.3 PCR扩增
对COI基因进行扩增使用的是通用引物LCO1490(5′-GGTCAACAAATCATAAAGATATTGG-3′)和 HCO-2198(5′-TAAACTTCAGGGTGACCAAAAAAT-CA-3′).这2个引物组合能够扩增长度大约为1 kbp的片段.大多数个体都能用这段引物扩增得到相应序列.
PCR反应体系为25μL,包括:ddH2O 16.2μL,10×PCR Buffer 2.5 μL,dNTPs(2.5 mmol/L)2 μL,上游和下游引物(10μmol/L)各 1μL,Taq酶(500 U,2.5 U/μL)0.3μL,DNA 模板2μL.
PCR反应条件:95℃预变性5 min;95℃变性40 s,56℃退火40 s,72℃延伸1 min,32个循环,最后72℃延伸5 min,温度降低到4℃终止扩增.得到的PCR产物用质量分数1%的琼脂糖凝胶进行电泳检测,经检测符合要求的产物送北京华大基因研究中心测序.
1.4 实验数据的处理和分析
所测得序列先进行校对,然后在NCBI进行BLAST比对,得到的同源序列绝大多数来自昆虫纲线粒体基因组细胞色素氧化酶 I(Cytochrome oxidase I,COI)的目标DNA片段,在排除了外源基因污染后,经过比对再将确认无误后的序列采用BioEdit 7.01软件进行比对,去掉两端不整齐的序列.
2 基于COI基因序列的系统发育分析
2.1 COI基因序列的碱基组成
利用MEGA5.1软件对摇蚊亚科48种摇蚊的基因序列进行分析,结果如表3所示.序列总长度平均为698 bp,序列中各碱基含量的平均值是:A=29.3%,T=37.7%,G=14.7%,C=18.3%,(C+G)含量(33%)远低于(A+T)含量(67%),表现出明显的碱基偏倚性,这与绝大多数昆虫线粒体的基因具有一致性.
在比对的序列中,C(保守位点数)占69.6%(486/698),V(变异位点数)占24.6%(172/698),Pi(简约信息位点数)占 16.1%(113/698),S(自裔位点数)占5.73%(40/698).密码子第2位点较为保守,其保守位点数占全部保守位点数的49.8%(242/486).密码子第3位点可变数占全部可变位点数的84.8%(146/172),而且简约信息位点也集中在第3位点上,其比例为62.5%,由此可见密码子第3位点上的碱基进化速率较快.
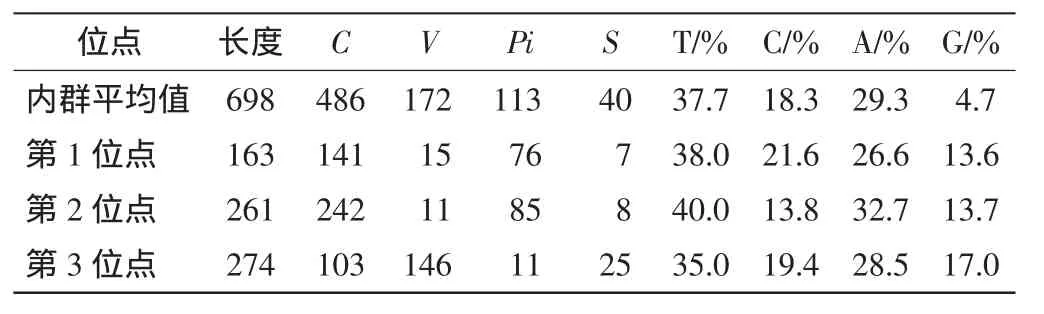
表3 摇蚊亚科48种摇蚊COI基因序列信息Tab.3 Information of COI gene sequencesof 48 species from Chironom inae
2.2 密码子氨基酸组成分析
对摇蚊亚科48种摇蚊的密码子使用频率进行统计,结果如表4所示.该序列编码了212种氨基酸,富含AT的密码子包括:亮氨酸(Leu L)的使用频率(CU)和相对使用频率(RSCU)值范围分别为2.9~9.0和0.64~1.98;甲硫氨酸(Met M)的CU和RSCU值范围分别为0.0~1.8和0.00~1.00.富含GC的密码子包括:甘氨酸(Gly G)的CU和RSCU值范围分别为0.3~1.6和0.22~1.27;脯氨酸(Pro P)的CU和RSCU值范围分别为 0.6~3.1和 0.32~1.56;精氨酸(Arg R)的 CU 值和RSCU值范围分别为0.2~0.6和0.27~0.71.苯丙氨酸(F)使用量最高;其次是异亮氨酸(I);再次是酪氨酸(Y).因此COI基因表达产生的氨基酸在组成上具有一定的偏向性,AT密码子的相对使用频率大于GC密码子的相对使用频率.
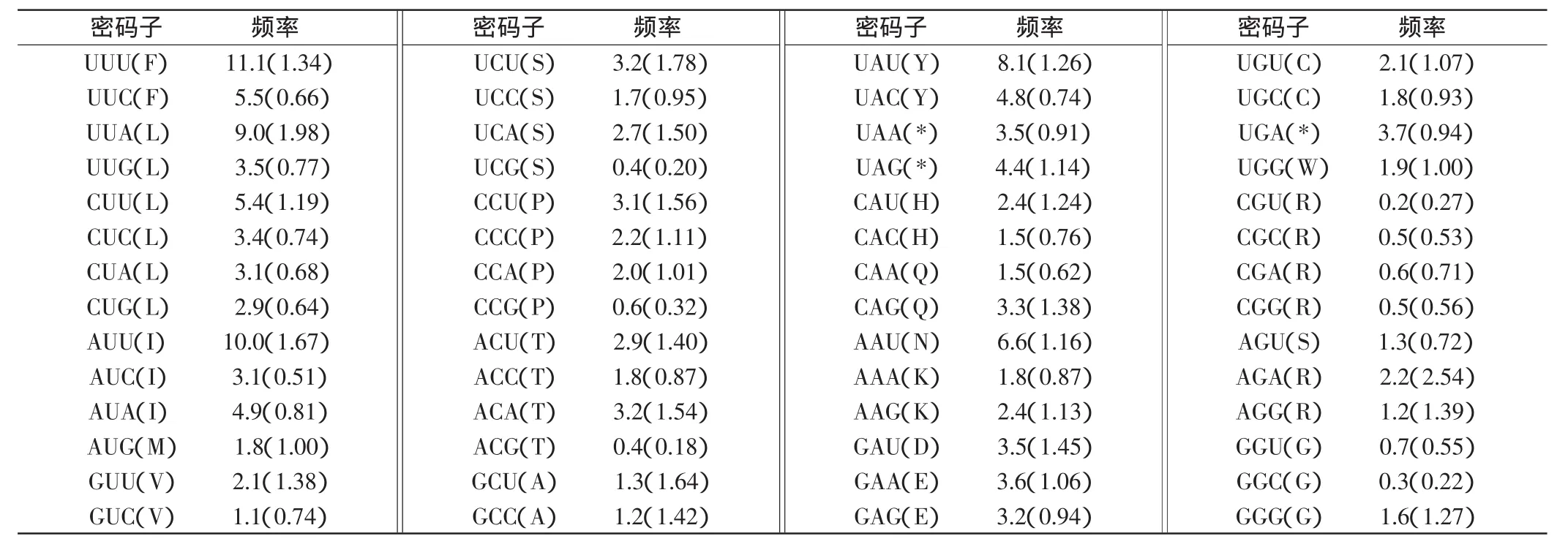
表4 摇蚊亚科48种摇蚊的密码子使用频率Tab.4 Codon usage frequency of 48 species in Chironom inae
2.3 碱基替换分析
对摇蚊亚科48种摇蚊的COI基因进行碱基替换分析,结果如表5所示.总转换数为84,总颠换数为105,转换略低于颠换.不同密码子之间的碱基替换具有差异性.密码子第3位点上出现了较为明显的碱基替换,替换比例达到7%(70/189),其中转换占全部转换数的 34.5%(29/84),颠换占 39%(41/105).第 2 点位点最为保守,只有26.9%(51/189)的替换率,分别占转换发生数的32.1%(27/84),颠换发生数的22.8%(24/105).内群平均转换比颠换(R 值)=0.8,小于 1,表明碱基替代未达到饱和.
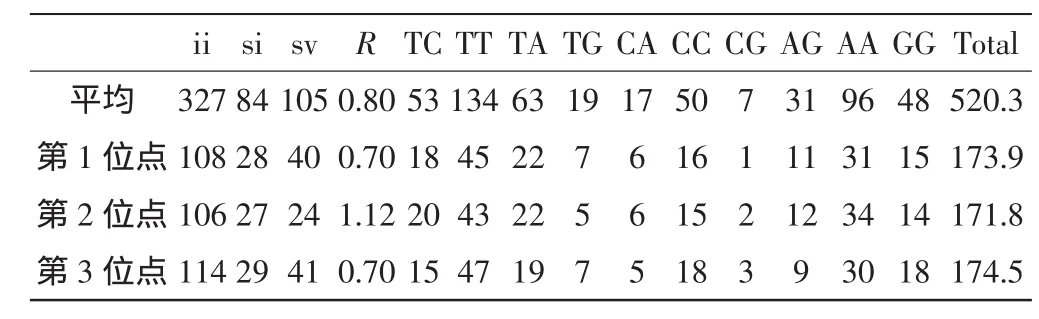
表5 摇蚊亚科48种摇蚊COI基因序列基因碱基替换Tab.5 Nucleotidessubstitution of COI gene sequencesof 48 species in Chironom inae
2.4 COI基因序列碱基替换饱和度分析结果
利用MEGA 5.1软件对摇蚊亚科48种摇蚊COI基因进行碱基替换饱和度分析,结果如图1所示.
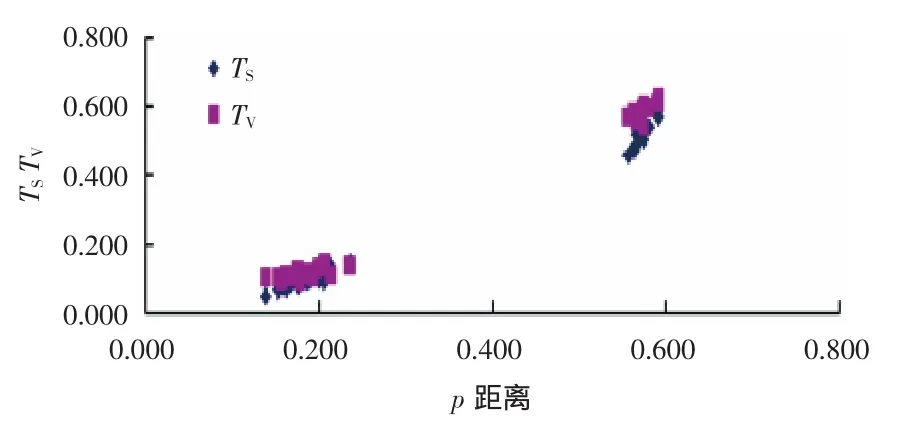
图1 COI基因转换颠换数与p距离的关系Fig.1 Relationship between COI gene conversion and transversion with p-distance
从图1可以看出,转换数(Ts)和颠换数(Tv)大部分重合,呈直线分布,随着p距离的增大而增多,颠换略高于转换,但两者没有明显的饱和趋势.同Tv相比,Ts与p距离的线性关系更强,碱基替换未达到饱和.推测颠换数高于转换数的原因与COI序列中(A+T)含量较高有关,因为高(A+T)含量增加了A-T颠换的几率.
2.5 遗传距离分析
计算48种摇蚊之间未校正的p距离,结果显示:摇蚊亚科内各属间p距离的变化范围为0.061~0.183;而外群环足摇蚊属与摇蚊亚科间的p距离的变化范围则为0.013~0.156,所有序列的平均p距离为0.201.
2.6 系统发育关系的构建
以环足摇蚊属的5个种作为外群,使用MEGA5.1软件构建基于Kimura-2-parameter模型的邻接法(NJ)和最大简约法(MP)的48种摇蚊的系统发育关系,结果如图2和图3所示.以重复抽样分析(Bootstrap test)1 000次来检验分子系统树各分支的置信度.2种建树方法得到的拓扑结构基本一致.
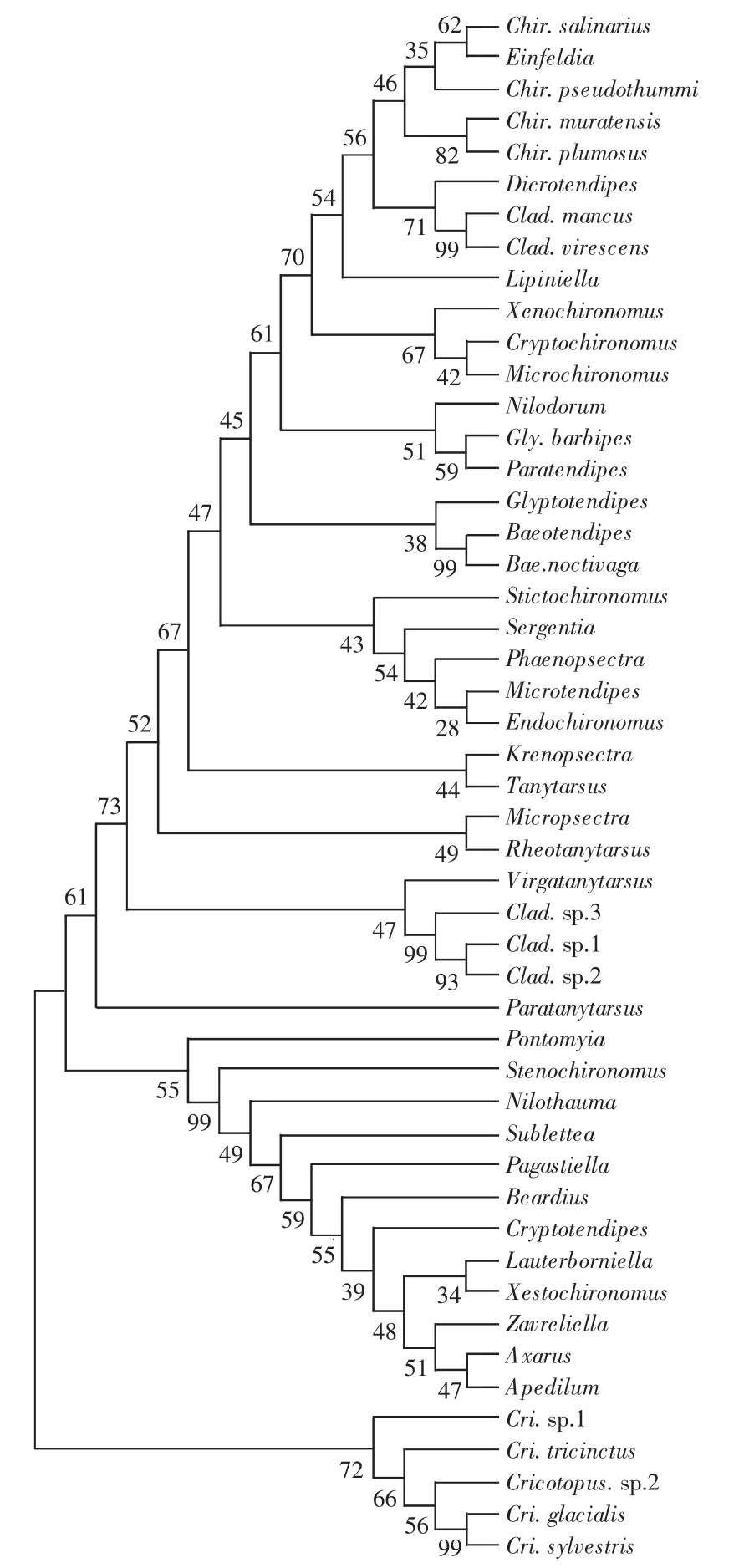
图2 基于COI基因序列构建的48种摇蚊的NJ树Fig.2 NJ tree of 48 speciesof Chironom inae based on COI gene sequences
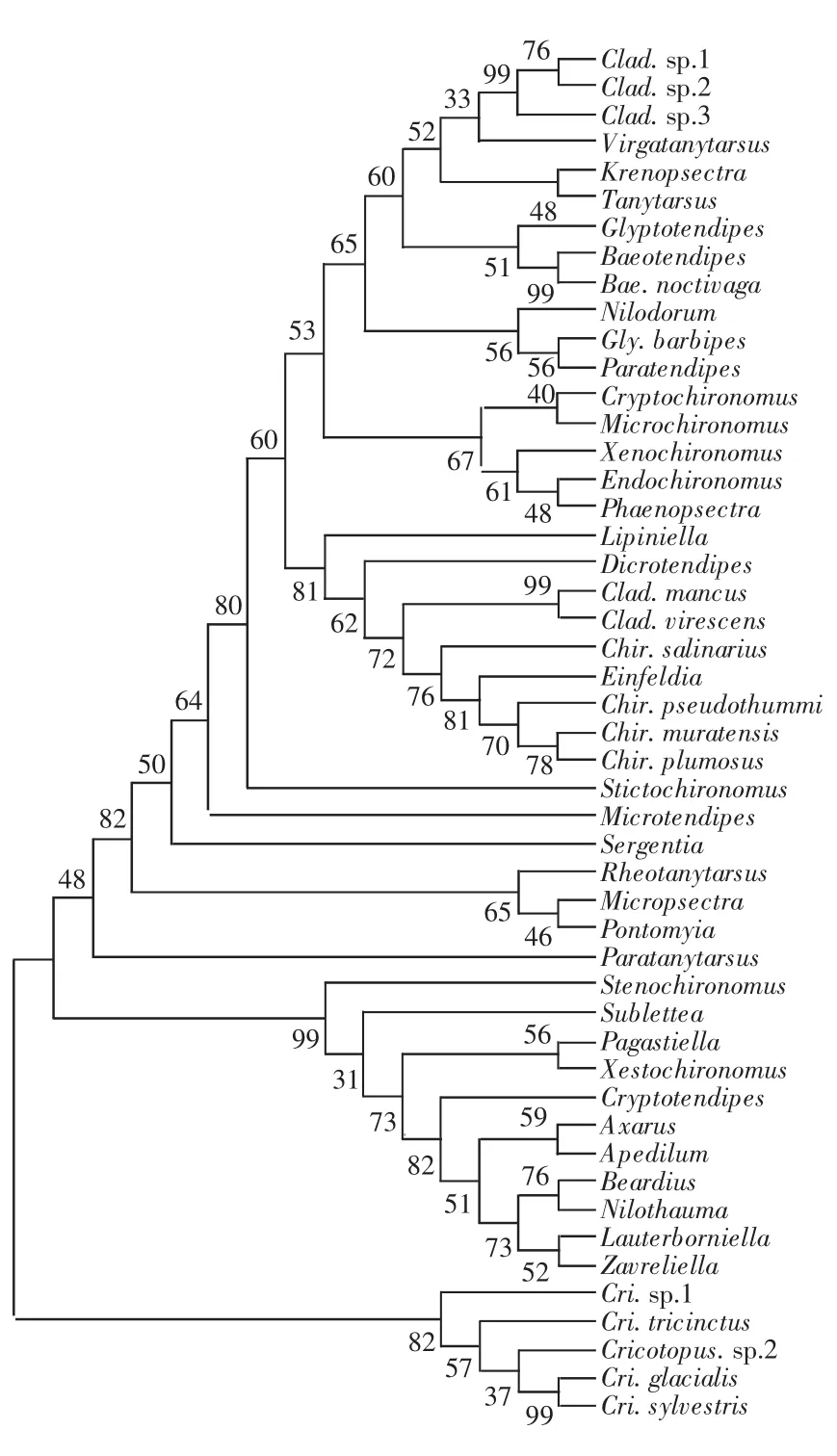
图3 基于COI基因序列构建的48种摇蚊的MP树Fig.3 MP tree of 48 species of Chironom inae based on COI gene sequences
3 讨论
本研究中NJ树和MP树形成的拓扑结构基本相似,所反映的部分属间关系情况大致相同.摇蚊亚科的2个属群的进化基本得到了解决,出现了2个明显的进化枝.总体上系统发育关系支持了形态学上鉴定的属和大部分种的地位.摇蚊亚科包括两大有区别的集群:摇蚊族和长跗摇蚊族,它们在约8 500万年以前开始分化,这些族的代表属在形态学特征上有显著不同.目前分类学家分辨出的Pseudochironomini的代表属在Dillon等[11]构建的系统发育图中没有形成一个分支,Pseudochironomus sp.和Chironomini的属在一个进化枝上,其分类地位还不明确.摇蚊亚科和直突摇蚊亚科的代表种形成约在104万年以前,与古生物学家鉴定的白垩纪摇蚊[12]的形态相一致.105万年以前的一个摇蚊亚科的代表—狭摇蚊属,从摇蚊亚科和直突摇蚊亚科之间单独形成一支.在本研究中,多足摇蚊属群中的内摇蚊属(Endochironomus Kieffer)、倒毛摇蚊属(Microtendipes Kieffer)、明摇蚊属(Phaenopsectra Kieffer)和斑摇蚊属(Stictochironomus Kieffer)很好地聚集在一起,有很近的亲缘关系,与形态学的分类一致.分类学家依据一些相似的特征(如触角13鞭节,下唇须5节,中足具一内一外2根短胫距及偶尔外胫距退化,上、下附器、肛尖形状变异情况以及有或无中附器等)将11个近缘的属聚为多足摇蚊属群.长跗摇蚊属群中杆长跗摇蚊属(Virgatanytarsus)和长跗摇蚊属(Tanytarsus),小突摇蚊属(Micropsectra)和流长跗摇蚊属(Rheotanytarsus)很好地聚集在了一起,与形态学的分类一致,还有一些同属群的物种个体并没有聚类在一起.Cranston[13]在1989年编制的属级鉴别特征,依据非常长的中附器、V型的臀板带、肛尖顶尖、指附器未伸出上附器内缘等相似特征将31个近缘属聚为长跗摇蚊属群,但是其中泉摇蚊属(Krenopsectra)和长跗摇蚊属(Tanytarsus)聚为姐妹群,这与形态学分类有明显差异.多数基于形态学研究的系统发育关系显示:狭摇蚊属(Stenochironomus)和明摇蚊属(Phaenopsectra)互为姐妹群,但是在本研究中这2个类群并没有聚合在一起形成姐妹群关系.在本文中显示较高支持率的姐妹群为:阿克西摇蚊属(Axarus)和异足摇蚊属(Apedilum)、二叉摇蚊属(Dicrotendipes)和枝角摇蚊属(Cladopelma).由于目前尚无分子系统学和形态学研究的相关报道,本文结果有待进一步验证,在今后研究中需要增加更多的基因序列进行联合分析.
[1]SAETHEROA.Femalegenitaliain Chironomidaeand other Nematocera:morphology,phylogenies,keys[J].Bulletin of the Fisheries Research Board of Canada,1977,23:197-211.
[2]RATNASINGHAMS,HEBERTPDN.Thebarcodeof lifedata system[J].Molecular Ecology Notes,2007,7(3):355-364.
[3]ANDERSEN T,CRANSTONPS,EPLER JH.Insect Systematics and Evolution Supplements(formerly Entomologica Scandinavica Supplement)Volume:66[M].Sweden:Entomological Society of Lund,1989.
[4]GURYEV V,MAKAREVICH I,BLINOV A,et al.Phylogeny of the genus Chironomus(Diptera)inferred from DNA sequences of mitochondrial Cytochrome b and cytochrome oxidase I[J].Molecular Biology and Evolution,2001,19(1):9-21.
[5]SHARMA M.Molecular Identification of Chironomid Species Based On ITS-1and ITS-2 Regions of Master of rDNA[D].Dayton:Wright State Univ,2007.
[6]EKREM T,WILLASSENE,STURE.A comprehensive DNA sequence library is essential for identification with DNA barcodes[J].Molecular Phylogeneticsand Evolcution,2007,43(2):530-542.
[7]EKREM T,WILLASSEN E,STURE,et al.Phylogenetic utility of five genes for Dipteran phylogeny:a test case in the chironomidae leads to generic synonymies[J].Molecular Phylogenetics and Evolcution,2010,57(2):561-571.
[8]MAKAREVICH IF,BEREZIKOVEV,GURYEVVP,etal.Molecular phylogeny of the Chironomus genus deduced from nucleotide sequences of twonuclear genes[J].Mol Biol(Moscow),2000,34(4):606-712.
[9]PAPOUCHEVA E,PROVIZ V,LAMBKIN C,et al.Phylogeny of the endemic Baikalian Sergentia (Chironomidae,Diptera)[J].Molecular Biology and Evolution ,2003,18(2):112-113.
[10]PFENNINGER M,NOWAK C,KLEY C,et al.Utility of DNA taxonomy and barcoding for the inference of larval community structure in morphologically cryptic Chironomus(Diptera)species[J].Molecular Ecology,2007,16(9):1957-1968.
[11]DILLON M,PINDER L,REISS F,et al.Keys and diagnoses of the adultmalesof thesubfamily Chironominae(Diptera,Chironomidae)[J].Entomologica Scadninavica,1989,11(34):353-502.
[12]FOLMERO,BLACKM,HOEHW,etal.DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ifrom diverse metazoan invertebrates[J].Molecular Marine Biology and Biotechnology,1994,3(5):294-299.
[13]CRANSTON P S,DILLON ME,PINDER L C V,et al.Keys and diagnoses of the adult males of the subfamily Chironominae(Diptera,Chironomidae)[J].Entomologica Scadninavica,1989,34:353-502.
