华根霉脂肪酶在黑曲霉中的重组表达研究
2015-10-26张谦王剑英林智贾佳郭宏涛
张谦王剑英林智贾佳郭宏涛
(1.深圳技师学院,深圳 518116;2.深圳市绿微康生物工程有限公司,深圳 518055)
华根霉脂肪酶在黑曲霉中的重组表达研究
张谦1王剑英2林智2贾佳2郭宏涛2
(1.深圳技师学院,深圳 518116;2.深圳市绿微康生物工程有限公司,深圳 518055)
将华根霉脂肪酶基因RCL在黑曲霉中进行了重组表达。通过PCR技术分别获得黑曲霉Pgpd启动子和RCL-TtrpC融合片段并克隆至质粒pCHAMBIA230,构建出RCL超表达载体pCHAMBIA2302∷Pgpd-RCL-TtrpC。将该载体通过冻融法转化农杆菌EHA105,进而通过农杆菌介导转化法转化黑曲霉,随机挑选7株转化子,进行PCR和Southern杂交鉴定,均为阳性。对这7株转化子进行发酵和脂肪酶活力测定,结果显示,所有转化子均能表达RCL,其中T6转化子的脂肪酶活力最高,达到71 U/mL。SDS-PAGE分析表明,重组表达的RCL的分子量约为37 kD。
重组表达;华根霉;脂肪酶;黑曲霉;根癌农杆菌
华根霉脂肪酶(Rhizopus chinensis lipase,RCL)是丝状真菌华根霉(R. chinensis)产生的脂肪酶,其全长ORF由389个氨基酸残基构成,包含26个氨基酸的信号肽,94个氨基酸的前导肽和269个氨基酸的成熟肽[1]。研究表明,RCL在食品加工领域有重要应用价值,它能够显著增加面包的比容、改善面包结构、延缓面包老化,与国内同类产品相比,RCL在改善面包和馒头的硬度、弹性、胶着性和咀嚼性方面效果更佳,能够替代传统的化学改良剂[2,3]。但是,在食品安全方面,华根霉不是一般认为安全(general recognized as safe,GRAS)菌株,因此,它所分泌的脂肪酶不能直接作为食品添加剂。
随着分子生物学的发展,RCL的基因被克隆,研究人员在毕赤酵母中实现了RCL的重组表达[1,4],但是毕赤酵母发酵过程需要添加甲醇,甲醇是一种剧毒化学物质,微量的甲醇就会导致中枢神经损害、眼部损害及代谢性酸中毒[5],因此以甲醇作为诱导剂生产的RCL仍然不能作为食品添加剂。为了实现RCL在食品加工领域中的广泛应用,必须选用食品安全菌株来表达RCL。
黑曲霉(Aspergillus niger)是一种重要的工业微生物,在工业酶制剂方面应用广泛,可以产生脂肪酶、淀粉酶、酸性蛋白酶、纤维素酶、果胶酶等30多种酶制剂[6]。此外,黑曲霉用于食品酿造和加工方面已经有上百年的历史,因此被美国FDA认定为GRAS菌株,可以用于食品和药品级酶制剂的生产[7]。由于黑曲霉具备出色的蛋白质分泌能力、高度的安全性、成熟的发酵及后处理工艺,因此,常被作为宿主菌用以表达异源酶基因[8-10]。然而,目前尚没有黑曲霉表达RCL的研究报道。
本研究构建了黑曲霉甘油醛-3-磷酸脱氢酶基因启动子(Pgpd)驱动的RCL超表达载体,并利用农杆菌介导转化法(Agrobacterium tumefaciens-mediated transformation,ATMT)转化黑曲霉,探索了RCL在黑曲霉中的重组表达方法,研究结果对促进RCL在食品加工领域的应用有重要意义。
1 材料与方法
1.1 材料
黑曲霉(Aspergillus niger,CICC 2475)购至中国菌种保藏中心;大肠杆菌(Escherichia coli)DH5α和根癌农杆菌(Agrobacterium tumefaciens)EHA105均为深圳市绿微康生物工程有限公司保存;载体pCHAMBIA2302,由上海交通大学唐克轩教授惠赠;华根霉基因已构建在pMD18-T(simple)上,命名为pMD18-T∷RCL由本公司保存;Hybond-N+尼龙膜和Southern杂交试剂盒购至GE公司;限制性内切酶、DNA聚合酶、连接酶、pMD18-T 载体等均购自TaKaRa公司;其余所有化学试剂均为分析纯;所用引物由Invitrogen公司合成。
PCR 仪购至Bio-Rad公司;5417R 型台式冷冻离心机购至德国Eppendorf 公司;Southern杂交管和杂交炉购至Thermo Fisher公司。
1.2 方法
1.2.1 载体构建 以黑曲霉基因组DNA为模板,利用引物Pgpd-F/ Pgpd-R扩增黑曲霉的甘油醛三磷酸脱氢酶启动子(Pgpd);片段经Sal I/Pme I消化克隆至二元表达载体pCHAMBIA2302的相应位点。
以质粒pAN7-1为模板,以TtrpC-F/ TtrpC-R为引物扩增构巢曲霉色氨酸合成酶终止子;以pMD18-T∷RCL为模板,以RCL-F/RCL-R为引物扩增华根霉编码序列RCL;然后以RCL-F/TtrpC-R为引物,利用重叠延伸PCR,构建出RCL-TtrpC融合片段;片段经Spe I/Pme I消化后克隆至二元表达载体pCHAMBIA2302的相应位点;构建出最终的RCL超表达载体pCHAMBIA2302∷Pgpd-RCL-TtrpC,并经测序确认正确。所有引物序列,见表1。
表1 PCR引物序列
1.2.2 根癌农杆菌介导的黑曲霉遗传转化 利用冻融法将构建的pCHAMBIA2302∷Pgpd-RCL-TtrpC表达载体转化根癌农杆菌EHA105。黑曲霉的转化方法参照de Groot等[11]报道的方法,有部分步骤改进。首先,将含有RCL超表达载体的EHA105接种于MM培养基(含卡拉霉素100 μg/mL,链霉素100 μg/mL)过夜培养;吸取适量的培养物离心去上清,并用IM液体培养基稀释至OD600=0.15左右,然后再培养至OD600=0.5-0.6。制备新鲜的黑曲霉孢子悬液,并稀释成106个/mL。随后取上述孢子悬液与EHA105等体积混合,均匀涂布到铺有玻璃纸的CM平板上。共培养60 h后,转移到PDA筛选培养基(含有潮霉素100 μg/mL,头孢菌素300 μg/mL)上,等待转化子长出。将抗性转化子挑出,接种到新的PDA筛选培养基,如此连续传代3次;如稳定传代,则初步认定为黑曲霉转化子。
1.2.3 转化子的鉴定 随机挑选7株黑曲霉转化子进行扩大培养,抽提基因组DNA,利用引物RCLF/TtrpC-R对转化子进行PCR鉴定;对PCR鉴定为阳性的转化子,进行Southern杂交分析,Southern杂交的具体步骤参见GE公司的Southern杂交试剂盒说明书(RPN3680)。
1.2.4 转化子的发酵 使用YPD培养基,在25℃、250 r/min条件下,分别对出发菌CICC 2475和鉴定为阳性的黑曲霉转化子发酵3 d。然后对发酵产物离心收集上清,以橄榄油为底物,采用NaOH滴定法测定脂肪酶活力[12]。酶活力定义为每分钟分解底物生成1 μmol脂肪酸的酶量为一个活力单位。
1.2.5 SDS-PAGE分析 样品的浓缩:取发酵液200 μL,冰浴10 min,加入三氯乙酸(TCA,50%)50 μL,冰浴30 min,然后10 000 r/min离心15 min,弃上清液,沉淀加入0.2 mL预冷的丙酮洗涤,然后离心,弃上清,并置于真空泵抽中干燥10 min;备用。电泳:浓缩后的样品溶解于25 μL的Tris缓冲液中(100 mmol/L,pH6.8),然后加入6 μL Loading Buffer(5×),进行SDS-PAGE电泳分析,电泳完毕后用考马斯亮蓝R250染色过夜,然后脱色至获得清晰的电泳条带为止。
2 结果
2.1 RCL超表达载体的构建
以黑曲霉基因组为模板,PCR获得黑曲霉Pgpd启动子(图1-A),克隆至pCHAMBIA2302,构建出中间载体pCHAMBIA2302∷Pgpd。然后通过重叠延伸PCR获得RCL-TrpC融合片段(图1-B),克隆至pCHAMBIA2302∷Pgpd,构建出RCL的超表达载体pCHAMBIA2302∷Pgpd-RCL-TtrpC(图1-C)。表达载体经测序确认正确后转化农杆菌EHA105,获得了含RCL超表达载体的工程农杆菌EHA105。
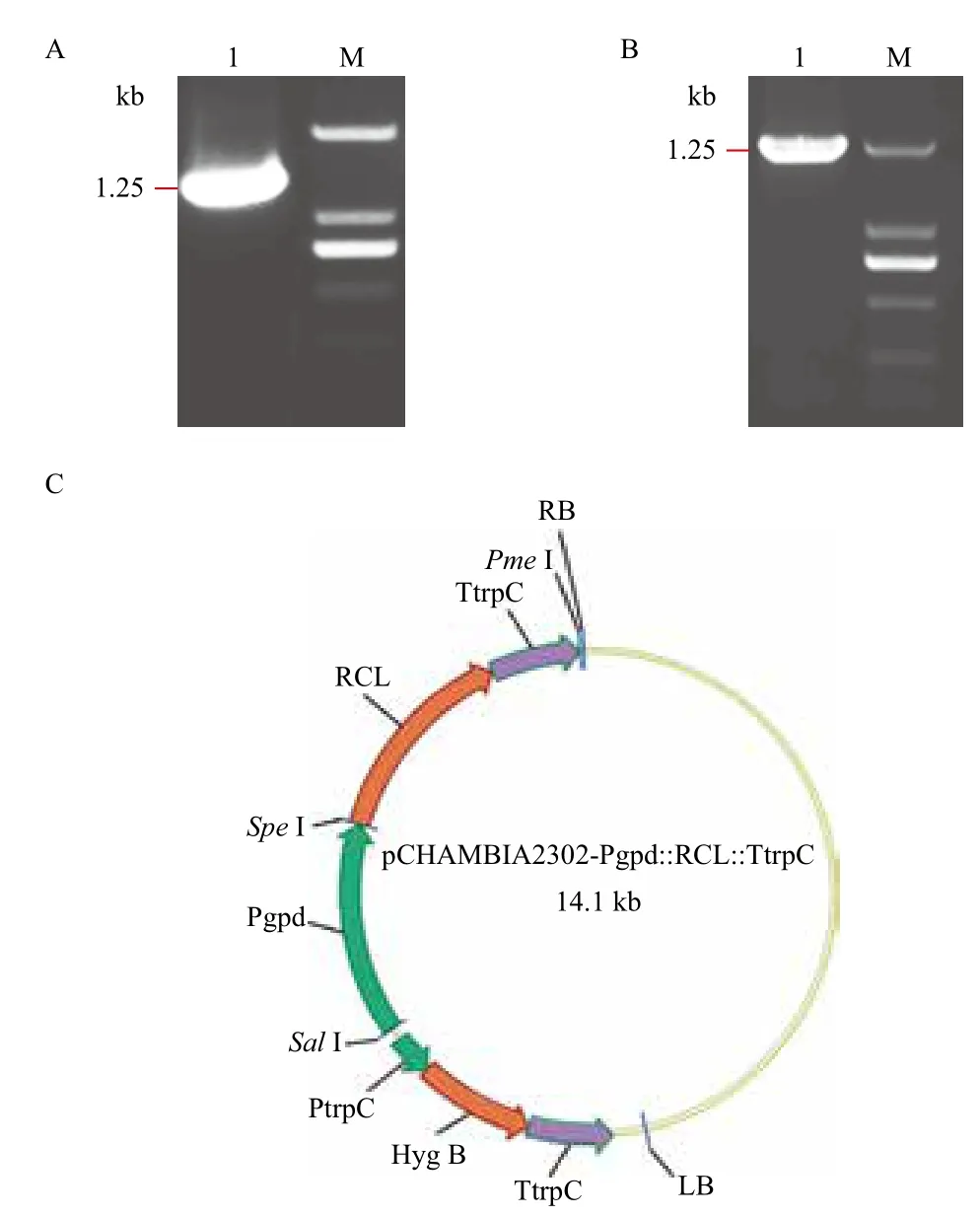
图1 RCL超表达载体的构建
2.2 黑曲霉转化子的鉴定
用含有载体pCHAMBIA2302∷Pgpd-RCL-TtrpC的农杆菌EHA105侵染黑曲霉,获得了黑曲霉转化子,对转化子进行鉴定。随机挑选7株转化子,进行DNA的抽提,以转化子的基因组为模板以出发菌CICC 2475(未转化的黑曲霉菌株)为对照,进行PCR鉴定。结果(图2-B)显示,所有转化子均出现大小为1.9 kb的扩增产物,与阳性对照一致;而未转化的黑曲霉菌株没有出现任何条带,初步证实所挑选转化子均为阳性转化子。对扩增产物进行测序,测序结果与目标序列一致,进一步证实所得转化子的为阳性克隆。
为了分析所得转化子中的目标基因拷贝数和整合位点,利用Southern杂交技术对上述阳性转化子进行进一步研究。Southern杂交结果(图2-C)显示,所有转化子均为单拷贝,7株转化子的杂交片段大小各不相同,表明目标基因的插入位点是随机的。
2.3 黑曲霉转化子的发酵
将上述7株转化子进行发酵,发酵完毕后离心,取上清液进行脂肪酶活力测定。脂肪酶活力测定采用NaOH滴定法,测定结果如图3。
测定结果显示,各转化子的发酵液中均检测到了较强的脂肪酶活力,而对照菌(未转化的黑曲霉菌株)的发酵液中却几乎检测不到脂肪酶活力,说明RCL在黑曲霉中得以重组表达,其中产量最高的T6转化子可以达到71 U/mL。

图2 黑曲霉转化子的鉴定
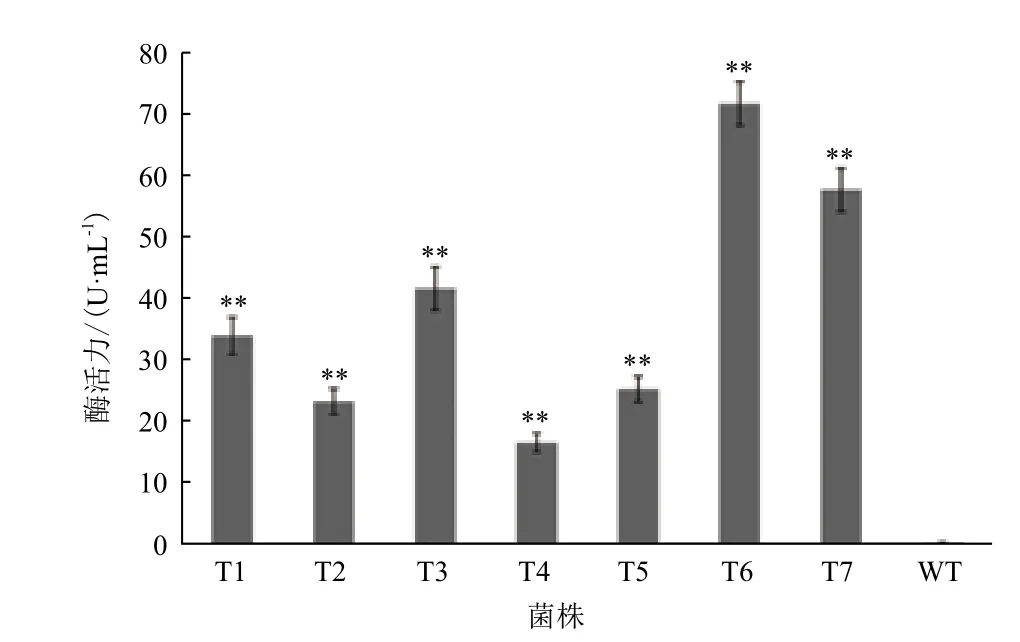
图3 黑曲霉转化子的发酵酶活力测定
2.4 发酵液的电泳分析
以未转化的黑曲霉为对照,对产量最高的T6转化子的发酵液进行SDS-PAGE分析,结果(图4)显示在箭头所指位置出现了明显的蛋白条带,而未转化的黑曲霉发酵液中没有,说明这一条带即为重组表达的RCL。根据软件Gel-pro 3.0计算,该条带的分子量约为37.5 kD。

图4 T6发酵液的SDS-PAGE分析
3 讨论
RCL虽然在食品加工中有重要应用价值,但是一直没有一种符合食品安全标准的RCL生产菌株,严重限制了RCL在食品加工中的应用。针对这一问题,本研究探索了RCL在黑曲霉中重组表达的可能性,获得了表达RCL的黑曲霉基因工程菌,建立了RCL在黑曲霉中的重组表达方法,研究结果对构建RCL的黑曲霉工业生产菌,促进RCL在食品工业中的应用有重要意义。
本研究选用了黑曲霉自身的强启动子Pgpd作为RCL的启动子,这有利于实现RCL的高效表达。甘油醛-3-磷酸脱氢酶基因(gpd)是很多生物的管家基因,其表达水平较高,有的可以达到胞内可溶蛋白的5%[13],因此其启动子(Pgpd)是较强的启动子,经常用来启动各种基因的表达[14,15]。黑曲霉自身的Pgpd启动子相较于其它异源的Pgpd启动子更容易与黑曲霉相关转录因子结合,实现RCL的高效表达。
黑曲霉的遗传转化可以采用PEG-介导的原生质体转化法(PMT)和农杆菌介导转化法(ATMT)。PMT需要制备原生质体,过程繁琐,转化效率低,转化子多不稳定;而ATMT不用制备原生质体,过程简便、高效,转化子稳定[16]。所以本研究采用了ATMT法转化黑曲霉,并获得了转化子。对随机挑选7株转化子进行鉴定,均为阳性,且为单拷贝,这与农杆菌介导转化其它真菌的特征一致[11,17],单拷贝插入是转化子稳定传代的重要原因。转入的RCL表达盒随机插入到黑曲霉基因组中,这也与之前的研究结果一致[18]。
转化子的发酵和酶活力分析结果证实了RCL的重组表达,不同转化子的酶活力有差异,这可能是表达盒插入的位点不同导致的[19]。T6转化子的发酵酶活力最高,达到71 U/mL,这与毕赤酵母的表达水平尚有一定差距[1],还需要进一步提高黑曲霉表达RCL的产量。进一步提高黑曲霉的RCL产量可从以下3方面改进:(1)根据黑曲霉偏爱密码子重新合成RCL基因并进行高效表达,偏好密码子对酶的表达量影响显著[20,21]。(2)对转化子的发酵条件进行优化,培养基配方对酶的发酵产量影响较大[22],可以采用RSM法、主成分分析法等优化培养基配方。(3)可以将RCL表达盒定点整合到黑曲霉基因组中的转录活跃区,整合位点对基因的表达量影响明显,当目标基因整合到异染色质区域时,转录量低,甚至不转录,因此重组酶产量低,甚至没有酶活力[23],可以利用同源重组技术将RCL整合到转录活跃区。最后,对T6转化子的发酵液进行SDS-PAGE分析,发现其重组表达的RCL分子量约为37.5 kD,与酵母重组表达的RCL分子量相近[1]。
4 结论
本研究以黑曲霉内源的Pgpd为启动子构建了RCL的超表达载体pCHAMBIA2300∷Pgpd-RCLTtrpC,并采用农杆菌介导转化法,将RCL表达盒插入到黑曲霉基因组中,转化子中RCL拷贝数多为单拷贝,整合方式为随机整合,转化子均能分泌RCL,其中T6转化子的脂肪酶活力最高,可以达到71 U/mL,重组表达的RCL分子量为37.5 kD。
[1]Yu XW, Wang LL, Xu Y. Rhizopus chinensis lipase:gene cloning,expression in Pichia pastoris and properties[J]. Journal of Molecular Catalysis B:Enzymatic, 2009, 57(1-4):304-311.
[2]张峦, 黄立群, 喻晓蔚, 等. 重组华根霉脂肪酶的酶学性质及其对面团热机械学和烘焙特性的影响[J]. 食品科学, 2010, 31(13):158-163.
[3]李珍妮, 王宏兹, 黄卫宁, 等. 重组华根霉脂肪酶协同转谷氨酰胺酶提高冷冻面团抗冻发酵特性的研究[J]. 食品科学, 2011,32(13):109-113.
[4]Sha C, Yu XW, Lin NX, et al. Enhancement of lipase r27RCL production in Pichia pastoris by regulating gene dosage and coexpression with chaperone protein disulfide isomerase[J]. Enzyme and Microbial Technology, 2013, 53(6-7):438-443.
[5]李洪钊, 李亮助, 孙强明, 等. 巴斯德毕赤酵母表达系统优化策略[J]. 微生物学报, 2003, 43(2):288-292.
[6]郭艳梅, 郑平, 孙际宾. 黑曲霉作为细胞工厂:知识准备于技术基础[J]. 生物工程学报, 2010, 26(10):1410-1418.
[7]Schuster E, Dunn-Coleman N, Frisvad JC, et al. On the safety of Aspergillus niger-a review[J]. Applied Microbiology and Biotechnology, 2002, 59(4-5):426-435.
[8]Mekmouche Y, Zhou S, Cusano AM, et al. Gram-scale production of a Basidiomycetous laccase in Aspergillus niger[J]. Journal of Bioscience and Bioengineering, 2014, 117(1):25-27.
[9]Fleissner A, Dersch P. Expression and export:recombinant protein production systems for Aspergillus[J]. Applied Microbiology and Biotechnology, 2010, 87(4):1255-1270.
[10]Prathumpai W, Flitter SJ, McIntyre M, et al. Lipase production by recombinant strains of Aspergillus niger expressing a lipaseencoding gene from Thermomyces lanuginosus[J]. Journal of Bioscience and Bioengineering, 2004, 65(6):714-719.
[11]de Groot MJ, Bundock P, Hooykaas PJ, et al. Agrobacterium tumefaciens-mediated transformation of filamentous fungi[J]. Nature Biotechnology, 1998, 16(9):839-842.
[12] Zhang T, Qi Z, Wang Y, et al. Agrobacterium tumefaciens-mediated transformation of Penicillium expansum PE-12 and its application in molecular breeding[J]. Research in Microbiology, 2013, 168(3):130-137.
[13] Piechaczyk M, Blanchard JM, Marty L, et al. Post-transcriptional regulation of glyceraldehyde-3-phosphate-dehydrogenase gene expression in rat tissues[J]. Nucleic Acids Research, 1984, 12(18):6951-6963.
[14]Li B, Rotsaert FAJ, Gold MH, et al. Homologous expression of recombinant cellobiose dehydrogenase in Phanerochaete chrysosporium[J]. Biochemical and Biophysical Research Communications, 2000, 270(1):141-146.
[15]Kajita S, Sugawara S, Miyazaki Y, et al. Overproduction of recombinant laccase using a homologous expression system in Coriolus versicolor[J]. Applied Microbiology and Biotechnology, 2004, 66(2):194-199.
[16] Michielse CB, Hooykaas PJ, van den Hondel CA, et al. Agrobacterium-mediated transformation as a tool for functional genomics in fungi[J]. Current Genetics, 2005, 48(1):1-17.
[17] Meyer V. Genetic engineering of filamentous fungi-Progress,obstacle and future trends[J]. Biotechnology Advances, 2008,26(2):177-185.
[18]黎明, 刘萌, 黄云雁, 等. 根癌农杆菌介导的黑曲霉遗传转化体系的建立及优化[J]. 中国生物工程杂志, 2012, 32(1):56-63.
[19]Verdoes JC, Punt PJ, van den Hondel CAMJJ. Molecular genetic strain improvement for the overproduction of fungal proteins by flamentous fungi[J]. Applied Microbiology and Biotechnology,1995, 43(2):195-205.
[20]张黎, 赵鹤云, 徐莉, 等. 解脂耶氏酵母脂肪酶基因lip1的克隆、密码子优化及功能表达[J]. 微生物学报, 2010, 50(7):969-974.
[21]杨江科, 严翔翔, 张正平, 等. 二步法黑曲霉脂肪酶基因lipA的全基因合成及其在毕赤酵母中的高效表达[J]. 生物工程学报, 2009, 25(3):381-387.
[22]Dai DZ, Xia LM. Enhanced production of Penicillium expansum PED-03 lipase through control of culture conditions and application of the crude enzyme in kinetic resolution of racemic allethrolone[J]. Biotechnology Progress, 2005, 21(4):1165-1168.
[23]张田, 唐克轩. 丝状真菌的遗传工程研究进展[J]. 上海交通大学学报:农业科学版, 2010, 28(5):480-486.
(责任编辑 李楠)
Recombinant Expression of Rhizopus chinensis Lipase in Aspergillus niger
Zhang Qian1Wang Jianying2Lin Zhi2Jia Jia2Guo Hongtao2
(1.Shenzhen Institute Technology,Shenzhen 518116;2 .Shenzhen Leveking Bio-engineening Co.,Ltd,Shenzhen 518055)
This article carried out the recombinant expression of Rhizopus chinensis lipase(RCL)gene in Aspergillus niger. Promoter of Pgpd from A. niger and RCL-TtrpC fusing fragment were respectively obtained by PCR and further cloned to plasmid pCHAMBIA2302 to generate the RCL over-expression vector pCHAMBIA2302∷Pgpd-RCL-TtrpC. The resulting vector was introduced into Agrobacterium tumefaciens EHA105, and then transformed into A. niger through the mediation of A. tumefaciens. Seven randomly selected transformants were analyzed by PCR and Southern blot. As a result, all the transformants were found to be positive. Then, the seven transformants were subjected to fermentation and lipase assay, and the results showed that all the transformants secreted recombinant RCL, among which transformant T6 produced the highest lipase specific activity, reaching up to approximately 71 U/mL. The fermentation broth was further analyzed by SDS-PAGE,which revealed the molecular weight of the recombinant RCL was 37 kD.
recombinant expression;Rhizopus chinensis;lipase;Aspergillus niger;Agrobacterium tumefaciens
10.13560/j.cnki.biotech.bull.1985.2015.04.024
2014-08-27
深圳市科技创新委员会技术创新计划项目(CXZZ20130425111508558)
张谦,男,高级工程师,硕士,研究方向:工业微生物及发酵工程;E-mail:qianz50@hotmail.com
