莱茵衣藻酰基辅酶A合成酶cDNA克隆及其酵母表达
2015-10-25宋燕子贾彬林柏成胡章立黄瑛
宋燕子 贾彬 林柏成 胡章立 黄瑛
(深圳市海洋生物资源与生态环境重点实验室 深圳市海洋藻类开发与应用工程实验室 深圳大学生命科学学院,深圳 518060)
莱茵衣藻酰基辅酶A合成酶cDNA克隆及其酵母表达
宋燕子 贾彬 林柏成 胡章立 黄瑛
(深圳市海洋生物资源与生态环境重点实验室深圳市海洋藻类开发与应用工程实验室深圳大学生命科学学院,深圳518060)
旨在预测并克隆莱茵衣藻酰基辅酶A合成酶cDNA(cracs),分析其在酵母中的功能。RT-PCR克隆cracs序列,ClustalW和MEGA6.0软件分别分析其编码蛋白保守序列和进化树,表达并分析其在酵母YB525中的底物偏好性。结果表明,首次在莱茵衣藻中克隆获得一个cracs,测序表明其序列大小为2 004 bp,编码667个氨基酸,编码蛋白crACS的预测分子量为72.3 kD,包含酰基辅酶A合成酶的两个保守区:AMP-binding区和FACS区。进化树比对显示,crACS与拟南芥的长链酰基辅酶A合成酶LACSs具有较高的同源性。酵母表达显示cracs编码蛋白能互补酵母YB525 LACS的缺陷表型,活化并优先利用C16∶1和C14∶0。莱茵衣藻cracs编码蛋白可活化外源脂肪酸,属于酰基辅酶A合成酶家族。
莱茵衣藻;酰基辅酶A合成酶;酵母YB525;进化树;脂肪酸
酰基辅酶A合成酶(acyl-CoA synthetase,ACS)也称为脂肪酸:CoA连接酶,一般情况下,ACS根据其脂肪酸底物的碳链长度而被分为短链ACS、中链ACS、长链ACS三类[1,2]。ACS广泛存在于真核和原核生物中,多含两个高度保守区域:AMP-binding domain和fatty acid acyl-coA synthetase(FACS)区。其中FACS区作为脂肪酸绑定位点,特异存在于中长链ACS中,对酶催化活性有重要影响[3,4]。ACS能将细胞内脂肪酸活化成与之对应的辅酶A硫酯底物,该底物能参与胞内脂肪酸碳链延长、三脂酰甘油合成、蛋白脂酰化、脂肪酸氧化降解等过程[5]。
莱茵衣藻作为微藻研究的模式生物,已被广泛地应用于微藻油脂合成研究[6-8],但其酰基辅酶A合成酶crACS却鲜有报道,在油脂合成与代谢中的功能尚不明确。本研究通过生物信息学方法预测一条莱茵衣藻酰基辅酶A合成酶基因,对其cDNA进行克隆,并通过酵母互补实验初步研究其生物学功能,以期为莱茵衣藻油脂合成与分解代谢研究提供依据。
1 材料与方法
1.1 材料
1.1.1 藻株和菌株 莱茵衣藻(Chlamydomonas reinhardtii CC849)由本实验室藻种库提供。酿酒酵母(Saccharomyces cerevisiae YB525)由中国海洋大学潘克厚教授慷慨提供[9]。
1.1.2 酶及试剂 RNA提取试剂盒TaKaRa RNAiso Plus、反转录试剂盒、酵母基因组提取试剂盒均购于TaKaRa生物工程(大连)有限公司。引物由上海生物工程有限公司合成。脂肪酸标准品购于上海安普生物科学仪器有限公司。尿嘧啶缺陷型培养基购于北京泛基诺科技有限公司。其他均为常规分子和化学分析纯试剂。
1.2 方法
1.2.1 培养方法 莱茵衣藻生长培养基为TAP,培养温度23℃,100 μE/m2/s,持续光照12 h/d;YB525生长培养基为YPD,筛选培养基为尿嘧啶缺陷型培养基添加2%(W/V)葡萄糖,诱导培养基为尿嘧啶缺陷型培养基添加2%半乳糖,功能分析培养基为诱导培养基添加不同碳链长度的脂肪酸(分别为C12∶0、C14∶0、C16∶0、C16∶1和C18∶0,浓度100 μmol/L),培养温度30℃,转速220 r/min。
1.2.2 总RNA的提取 取对数末期CC849藻液2 mL,去上清后用TaKaRa RNAiso Plus试剂盒提取总RNA,具体步骤参照试剂盒说明书。经Nanodrop ND2000和琼脂糖凝胶电泳分析,质量合格的RNA置-80℃保存备用。
1.2.3 cDNA克隆及序列分析 取1 μg CC849总RNA,用TaKaRa反转录试剂盒,合成cDNA第1条链。根据NCBI数据库中同源物种酰基辅酶A合成酶序列XM_001702895,设计引物crACSF和crACSR(表1),使用GC buffer I、LA-Taq酶扩增莱茵衣藻酰基辅酶A合成酶cDNA cracs。PCR条件为:94℃预变性5 min;94℃ 30 s,60℃ 30 s,72℃ 2 min,35个循环;72℃延伸10 min。PCR扩增产物切胶回收后连接到pEASY-T1 simple载体并转化感受态细胞,挑选阳性克隆测序,测序正确的质粒命名为cracs-T。
MEGA6.0分析基因家族的系统进化树,采用Bootstrap方法重复1 000次分析,Neighbor-Joining算法构建进化树。DNAMAN软件分析cracs编码蛋白的属性。ClustalW分析保守序列。

表1 引物序列
1.2.4 YB525表达载体构建 利用限制性内切酶Kpn I和 Xho I分别双酶切质粒pYES2和cracs-T,酶切产物pYES2和cracs回收后进行酶连,转化大肠杆菌,经过PCR、双酶切和测序验证正确的重组载体被命名为pYES2-cracs。
1.2.5 YB525转化子的筛选及鉴定 将重组载体pYES2-cracs和pYES2以电转化的方法[10]转入YB525,30℃培养一周,用筛选培养基筛选转化子。随机选取转化子并培养后提取基因组DNA,以表1引物VF/VR对其进行PCR鉴定和测序分析。pYES2和pYES2-cracs的酵母阳性转化子分别命名为YPES2和YPACS。
1.2.6 crACS在YB525中的表达与功能验证 筛选培养基30℃,220 r/min培养YPES2和YPACS至对数中后期,收集100 mL菌体,2 mol/L山梨醇洗两次,诱导培养基培养4 h。取100 μL培养液接种于10 mL功能分析培养基,培养240 h后测定其OD600值。
2 结果
2.1 cDNA克隆及序列分析
克隆cDNA序列片段及将其连接到pEASY-T1 simple载体后的双酶切结果如图1所示,测序显示其大小为2 004 bp,与NCBI数据库登录号为XM_001702895的序列一致。根据核苷酸序列测定结果和比较基因组学分析,推测出该基因包含16个外显子,15个内含子,内含子均以GT开始并以AG结尾,基本符合内含子的特征。该基因外显子与内含子的分布,见图2。DNAMAN分析表明,cracs编码的蛋白质含有667个氨基酸残基,推测其分子量为72.3 kD,等电点为6.87。
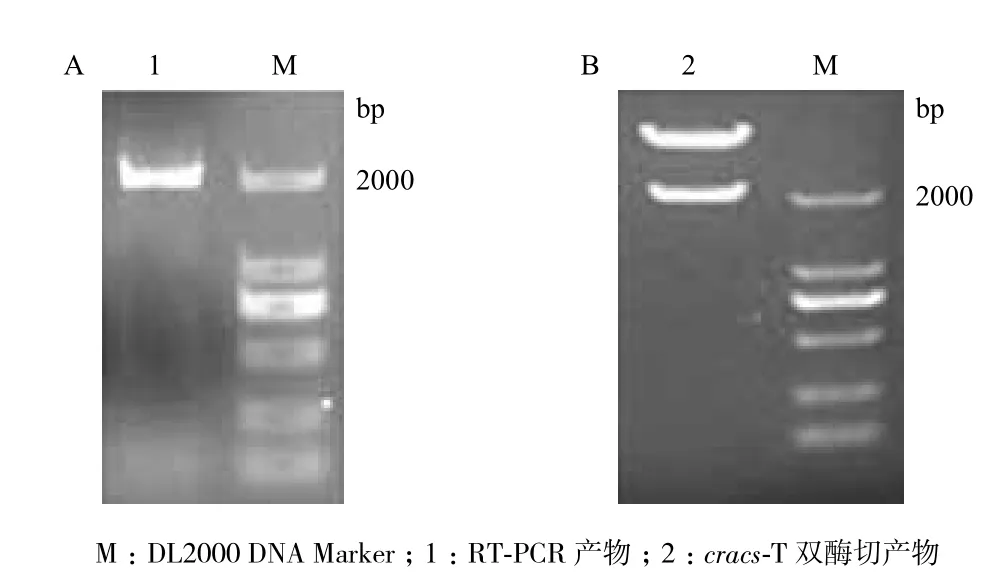
图1 莱茵衣藻cracs的RT-PCR产物(A)和cracs-T(B)双酶切产物凝胶电泳分析

图2 cracs基因示意图
用Bioedit软件比对crACS与拟南芥等9个物种的10条ACS氨基酸序列,结果(图3)显示crACS存在两段高度保守的区域。保守序列分别为“YTSGTTGDPK”和“DGXXHTGDXGXXXXXGXLK-IIDRKK”,前者为AMP-binding蛋白家族成员中高度保守的AMP-绑定位点,该序列普遍存在于所有已报道的酰基活化酶超家族成员中,后者恰为脂肪酸的绑定位点FACS区。这两个保守区基本存在于所有物种的中长链ACS中。因此,初步推测crACS为莱茵衣藻酰基辅酶A合成酶。

图3 莱茵衣藻crACS与其他物种的ACS比对的部分结果
2.2 进化树分析
基于crACS与拟南芥、三角褐指藻等9个物种的23条ACS蛋白序列绘制的进化树(图4)显示,所有的ACS都起源于同一个祖先。莱茵衣藻crACS与 拟 南 芥AtLACS1、AtLACS2、AtLACS3、AtLACS4、AtLACS5及甘蓝型油菜BnLACS同属一个进化分支,而与拟南芥AtLACS6、AtLACS67、AtLACS68、AtLACS9聚类于不同的进化分支。进化树还显示crACS与人、褐家鼠、铜绿假单胞菌的ACS的亲缘均较远。此外,两个超长链ACS AAC17118.1/ NP_003636处于进化分枝的最外围,与crACS的亲缘关系最远,据此推测crACS不属于超长链ACS。

图4 莱茵衣藻CC849 crACS与其他8个物种22条ACS的进化树
2.3 YB525转化子的鉴定
YPES2、YPACS 基因组PCR结果(图5)显示,分别得到约160 bp和约2 000 bp的基因片段,测序结果表明PCR结果与cracs序列完全一致。

图5 YB525转化子YPES2和YPACS的鉴定
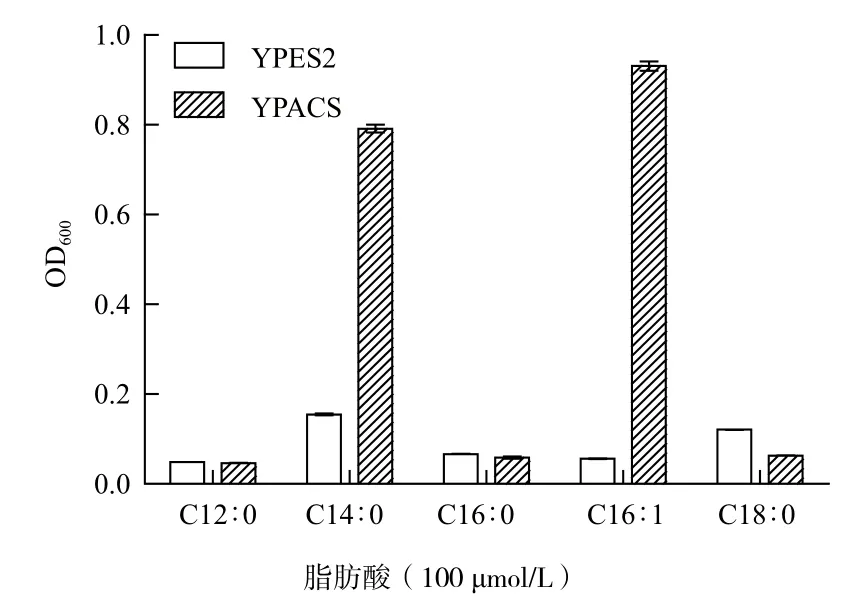
图6 培养10 d YPES2和YPACS的生长状况
2.4 莱茵衣藻crACS功能验证
YB525是ACS基因部分缺失的酵母,不能以脂肪酸为唯一碳源生长,转入具有ACS功能的外源基因后可恢复生长。YPES2、YPACS在YB525中的功能验证结果(图6)显示,YPACS在含有C14∶0和C16∶1的功能分析培养基上生长良好,而YPES2在所有的功能分析培养基中基本不生长,表明cracs转入后成功互补了YB525 ACS基因的缺陷。此外,YPACS在含有C12∶0、C16∶0或C18∶0的功能分析培养基中都不能正常生长,说明crACS具有底物偏好性。因此,莱茵衣藻crACS具有酰基辅酶A合成酶活性。
3 讨论
ACS广泛存在于真核和原核生物中,并在脂肪酸代谢过程中起重要作用,越来越多的研究表明,ACS与脂肪酸碳链延长、三脂酰甘油合成和脂肪酸β-氧化降解等途径紧密相关[5,12]。目前一些高等植物、细菌和微藻的LACS家族基因已经被克隆和分析[13-16],其中油菜LACS参与油脂的合成,铜绿假单胞菌LACS可以催化长链脂肪酸且参与脂肪酸β-氧化反应,假微型海链藻LACS与DHA、EPA的代谢关系密切。因此,克隆莱茵衣藻cracs及分析其编码蛋白的功能,有助于了解莱茵衣藻脂肪酸和油脂代谢调控机制。
莱茵衣藻crACS进化树比对发现,crACS与拟南芥AtLACS1-5在同一个进化分枝,而与AtLACS6 -7处于临近分枝。拟南芥的LACS家族成员各具不同的功能,其中AtLACS1-3不仅能利用酵母的脂肪酸还具有脂肪酸转运蛋白酶的功能[11,17]。Faldua[18]进一步研究表明AtLACS6和AtLACS7编码的过氧化酶体ACS蛋白参与脂肪酸β-氧化。因此,可推测crACS参与脂肪酸的合成途径。进化树还显示crACS与进化树中所有的长链ACS分布于一个大分枝,与进化树中两个超长链ACS(AAC17118.1/ NP_003636.2)亲缘关系较远,因此认为crACS不是超长链酰基辅酶A合成酶。
微 拟 球 藻 的LACS NOLACS偏 好C14∶0、C16∶0、C18∶0等脂肪酸底物,且优先利用长链脂肪酸[9]。YPES2和YPACS的生长状况所示的研究结果与此相似,crACS偏好利用C16∶1、C14∶0等长链脂肪酸作为底物,这些长链脂肪酸是莱茵衣藻脂肪酸的主要成分,说明crACS可能活化莱茵衣藻中的长链脂肪酸并参与其代谢反应。一般同一个物种中存在多个ACS,如拟南芥[11]和三角褐指藻[19]中分别分离鉴定了9个和5个ACS。目前本研究仅克隆并初步分析了莱茵衣藻的1个ACS,未来将进一步研究crACS的功能并寻找更多的crACS同工酶,同时将通过基因调控crACS,研究其对脂肪酸合成与代谢的影响。
4 结论
本研究运用生物信息学的方法首次从莱茵衣藻中预测并克隆一个酰基辅酶A合成酶的cDNA序列,酵母表达实验证实该基因能互补缺陷型酵母YB525,具有酰基辅酶A合成酶的活性,能活化外源脂肪酸。
[1]Steinberg SJ, Morgenthaler J, Heinzer AK, et al. Very longchain acyl-CoA synthetases:Human “bubblegum” represents a new family of proteins capable of activating very long-chain fatty acids[J]. Biological Chemistry, 2000, 275(45):35162-35169.
[2]Fulda M, Shockey J, Werber M, et al. Two long-chain acyl-CoA synthetases from Arabidopsis thaliana involved in peroxisomal fatty acid β-oxidation[J]. Plant, 2002, 32(1):93-103.
[3]Black PN, Dirusso CC, Sherin D, et al. Affinity labeling fatty acyl-CoA synthetase with 9-p-azidophenoxy nonanoic acid and the identification of the fatty acid-binding site[J]. Biological Chemistry, 2000, 275(49):38547-38553.
[4]Babbit PC, Kenyon GL. Ancestry of the 4-ch lorobenzoate dehalogenase:analysis of amino acid sequence identities among families of acy1:adenyl ligases, enoyl-CoA hydratases/isomerases,and acyl-CoA thioesterasest[J]. Biochemistry, 1992, 31(24):5594-5604.
[5]Pei Z, Oey NA, Zuidervaart MM, et al. The acyl-CoA Synthetase "Bubblegum"(Lipidosin):Furture characterization and role in neuronal fatty acid β-oxdation[J]. Biological Chemistry, 2003,278(47):47070-47078.
[6]Sato A, Matstmura R, Hoshino N, et al. Responsibility of regulatory gene expression and repressed protein synthesis for triacylglycerol accumulation on sulfur-starvation in Chlamydomonas reinhardtii[J]. Frontiers in Plant Science, 2014, 5(444):1-12.
[7]Ibáñez-Salazar A, Rosales-Mendoza S, Rocha-U-ribe A, et al. Overexpression of Dof-type transcription factor increases lipid production in Chlamydomonas reinhardtii[J]. Journal of Biotechnology,2014, 184:27-38.
[8]Deng X, Cai J, Li Y, et al. Expression and knockdown of the PEPC1 gene affect carbon flux in the biosynthesis of triacylglycerols by the green alga Chlamydomonas reinhardtii[J]. Biotechnology Letters,2014, 36(11):2199-2208.
[9]Zhang L, Ma X, Yang G, et al. Isolation and characterization of along-chain acyl-coenzyme A synthetase encoding gene from the marine microalga Nannochloropsis oculata[J]. Applied Phycology,2012, 24(4):873-880.
[10]Xu Z, Zhang Y, Wang Z, et al. A highly efficient protocol transformation of Saccharomyces cerevisiae and Pichia pastoris by electroporation[J]. Acta Scientiarum Naturealum Univeritatis Sunyatseni, 2010, 49(3):98-101.
[11]Shockey JM, Fulda MS, Browse JA. Arabidopsis contains nine longchain acyl-Coenzyme A synthetase genes that participate in fatty acid and glycerolipid metabolism[J]. Plant Physiology, 2002,129(4):1710-1722.
[12]Watkins PA. Fatty acid activation[J]. Progress in Lipid Researh,1997, 36(1):55-83.
[13]于莉莉, 谭小力, 侯文胜. 大豆长链脂酰辅酶A合成酶基因GmLACS在酵母中的表达[J]. 大豆科学, 2011, 30(5):719-722.
[14]朱福各, 谭小力, 崇保强, 等. 油菜脂酰CoA合成酶基因pXT166的鉴定和功能分析[J]. 中国油料作物学报, 2009,31(3):274-278.
[15]王建林, 邵新峰, 梁振普, 等. 铜绿假单胞菌长链脂酰辅酶A合成酶的克隆及活性鉴定[J]. 河南科学, 2014, 32(3):355-361.
[16]Tonon T, Qing R, Harvey D, et al. Identification of a long-chain polyunsaturated fatty acid acyl-Coenzyme A synthetase from the Diatom Thalassiosira pseudonana[J]. Plant Physiology, 2005,138(1):402-408.
[17]Lü S, Song T, Kosma DK, et al. Arabidopsis CER8 encodes longchain acyl-coA synthase 1(LACS1)that has overlapping functons with LACS2 in plant wax and cuitin synthesis[J]. Plant, 2009,59(4):553-564.
[18]Fulda M, Shockey J, Werber M, et al. Two long-chain acyl-CoA synthetases from Arabidopsis thaliana involved in peroxisomal fatty acid beta-oxidation[J]. Plant, 2002, 32(1):93-103.
[19]Guo X, Jiang M, Wan X, et al. Identification and biochemical characterization of five long-chain acyl-coenzyme A synthetases from the diatom Phaeodactylum tricornutum[J]. Plant Physiology and Biochemistry, 2014, 74:33-41.
(责任编辑 马鑫)
cDNA Cloning and Yeast Expression of Acyl-CoA Synthetase of Chlamydomonas reinhardtii
Song Yanzi Jia Bin Lin Baicheng Hu Zhangli Huang Ying
(Shenzhen Key Laboratory of Marine Bioresource and Eco-environmental Science,Shenzhen Engineering Laboratory of Marine Algae Biotechnology,College of Life Sciences,Shenzhen University,Shenzhen518060)
This work aims to predict and clone cDNA of Chlamydomonas reinhardtii a cyl-CoA synthetase(gene cracs), and analyze its function in yeast Saccharomyces cerevisiae YB525. The cracs sequence was cloned by RT-PCR, its conserved sequence of encoded protein and phylogenetic tree were analyzed with ClustalW and MEGA6.0, then the substrate specificity in YB525 of expressed gene was analyzed. As the results, a cracs was cloned for the first time with sequence of 2 004 bp and encoded a 72.3 kD protein crACS of 667 amino acids containing two conserved regions of including acyl-CoA synthetase:the AMP-binding domain and the FACS motif. The phylogenetic tree analysis indicated that crACS shared high homology with LACs of Arabidopsis thaliana. Yeast expression experiments showed that crACS restored acyl-CoA synthetase deficient phenotype of YB525 and assimilated foreign palmitoleic acid and myristic acid. Conclusively, cracs of C. reinhardtii can activate exogenous fatty acid and belongs to acyl-CoA synthetase family.
Chlamydomonas reinhardtii;acyl-CoA synthetase;yeast YB525;phylogenetic tree;fatty acid
10.13560/j.cnki.biotech.bull.1985.2015.09.016
2015-01-21
国家自然科学基金项目(31100582,31470431),中国博士后科学基金项目(2014M562199),深圳市科技计划项目(JCYJ20120613112512654)
宋燕子,女,硕士研究生,研究方向:海洋生物学;E-mail:swallowsong@163.com
黄瑛,女,博士,副教授,研究方向:藻类生物能源;E-mail:huangy@szu.edu.cn
