花生中一个NBS—LRR类基因的克隆和表达分析b
2015-10-21赵小波等
赵小波等


摘要:本研究利用同源克隆方法,根据已知植物抗病(R)基因保守结构域设计一对简并引物,采用RT-PCR方法对花生抗旱品种J11进行扩增,获得了一个通读的NBS-LRR类基因片段,命名为PDR1。PDR1长度为474 bp,核苷酸比较分析表明,PDR1与已克隆的大豆抗性基因(XM006573278)相似性为75%,与豌豆(AF123699)、苜蓿(XM003604518)抗性基因的相似性为74%;生物信息学预测其全长CDS编码框为5 838 bp,编码的蛋白质含1 945个氨基酸,含有NB-ARC等保守结构域。荧光定量PCR分析表明,PDR1基因在花生幼苗根、茎、叶中均有表达,相对表达量为叶>根>茎;干旱胁迫后PDR1的相对表达量在12 h达到顶峰,随后下降,趋于平稳。本研究从花生中成功获得了NBS-LRR类基因的同源序列,为筛选新的抗旱花生种质提供了理论依据。
关键词:花生; NBS-LRR;同源序列;基因克隆;表达分析
中图分类号:S565.2:Q781文献标识号:A文章编号:1001-4942(2015)05-0001-06
Cloning and Expression Analysis of a NBS-LRR
Resistance Gene in Peanut
Zhao Xiaobo1, Zhang Tingting1, Yan Caixia1, Zhang Hao1,2, Shan Shihua1*, Chen Dianxu1*
(1.Shandong Peanut Research Institute, Qingdao 266100, China; 2.Shandong Agricultural University, Taian 271018, China)
AbstractIn this study, one resistance gene homology fragment was isolated from peanut by homology cloning method. A pair of degenerate primers was designed according to the conserved domains of the known plant disease resistance genes, and then, the fragment with continuous open reading frame was obtained from the peanut variety J11 with drought tolerance by RT-PCR. The sequence was named PDR1. The length of PDR1 was 474 bp. The homology research results showed that the nucleotide of PDR1 was 75% identical to a cloned Glycine max resistance gene (XM006573278), and 74% identical to the sequences from Pisum sativum (AF123699) and Medicago truncatula(XM003604518), respectively. By bioinformatics method, it was predicted that the CDS of PDR1 was 5 838 bp, encoding 1 945 amino acids and containing the conserved domain of NB-ARC. The results of real-time fluorescence quantitative PCR showed that PDR1 expressed in peanut root, stem and leaves. The relative expression in different organs was leaves > root > stem. After drought stress, the relative expression of PDR1 in J11 reached the peak on the 12th hour, then decreased and moved towards stabilization. The obtain of this resistance homology sequence provided theoretical basis for the screening of drought resistant germplasms form peanut.
Key wordsPeanut; NBS-LRR; Homology sequences; Gene clone; Expression analysis
我国是世界花生生产大国,2007~2013连续6年总产量增加,达到1 700×104 t,占世界花生总产的40%以上,居世界第一位,2012年出口14.6万吨,出口金额一直保持在2亿美元以上。但是,花生产业的进一步发展受到旱灾的威胁,据统计我国每年因干旱引起的减产达30%~50%。除减产外,干旱还有可能导致花生黄曲霉毒素污染、品质下降等一系列后果。所以,选育抗旱、高产、优质花生品种是花生育种工作的重要方向之一。
研究花生的抗旱机制是花生抗旱育种的基础,目前一些NBS-LRR类基因被认为在花生抗旱过程中起到一定的作用。NBS-LRR类基因(nucleotide binding site-leucine-rich repeat)是R基因(抗病基因, resistance genes)中最大的类群,其蛋白产物含一个NBS和一个LRR结构域,NBS结构域具有4个高度保守的基序。目前,大多数NBS-LRR类基因是根据植物抗病基因保守序列结构特点设计引物扩增基因组DNA或cDNA获得的。仅国内,就已在水稻、拟南芥、毛果杨、大豆和小麦等植物中成功扩增了许多抗病基因同源片段。在花生NBS-LRR类基因研究领域,Yuksel等做了大量的工作,取得了重要的成果,他的团队设计了4对简并引物和2对特异引物,总计获得了234个具有开放阅读框的NBS-LRR类基因片段;晏立英等利用已知的NBS保守区域设计引物,从中花6号中获得了5条具有连续ORF的抗病基因序列。但是,相对于植物基因组中的NBS-LRR类基因数量,目前已知的该类基因数目仍然较少,需要设计新的引物或采用其他方法去获得。endprint
本研究根据已克隆的植物抗病基因NBS-LRR保守结构域,参考已有研究成果设计简并引物,从花生品种J11的cDNA中获得抗病基因同源序列,并进行同源性比较及功能验证,为今后花生抗旱种质的筛选奠定基础。
1材料与方法
1.1试验材料与处理
供试花生材料抗旱品种J11、不耐旱品种JH1012为山东省花生研究所提供,试验所用材料为当年田间种植样品。取饱满一致的高活力花生种子各10粒,放在灭菌培养皿中置于人工培养箱,使用Hoagland营养液。人工培养箱培养条件设置为:27℃,光照强度2 000 lx,光照周期为16L∶8D。培养10天后取新鲜叶片,用液氮速冻,并保存于-80℃冰箱待用。
对27℃正常培养的J11花生幼苗进行15% PEG 6000溶液模拟干旱处理,分别取处理前(0 h)及处理后6、12、18 h和24 h幼苗第1片复叶;同时采集正常状态J11花生幼苗的茎、根、叶,液氮速冻后于-80℃保存,用于荧光定量PCR分析。试验设3次重复。
1.2叶片总RNA的提取和cDNA第一链合成
总RNA提取采用杨晨等的方法,略有改进。用紫外分光光度计(AA-680,日本岛津) 检测RNA浓度并利用琼脂糖凝胶电泳检测RNA完整性。cDNA第一链的合成采用Vazyme的HiScript第一链cDNA合成试剂盒。
1.3NBS-LRR类基因片段获得
根据NBS类抗病基因编码的蛋白质保守区域P-loop和GLPL设计一对简并引物,引物序列见表1。以花生抗旱品种J11、不耐旱品种JH1012的cDNA第一链为模板进行PCR扩增。反应体系为20 μL,包含10×PCR buffer 2 μL,MgCl2 (25 mmol/L)1.5 μL,dNTPs(2.5 mmol/L)2 μL,Taq(0.5 U/μL)0.2 μL,引物(10 mmol/L)各0.5 μL,模板1 μL,ddH2O补足至20 μL。反应程序为:94℃预变性3 min;94℃变性1 min,50℃退火1 min,72℃延伸2 min,40个循环;最后72℃延伸10 min。
1.4测序与序列比较
测序委托南京金丝瑞生物技术公司完成。序列数据首先与GenBank中已有序列信息进行比对。然后利用ClustalW软件对测序数据进行人工对齐排序。选择Kimura 2-parameter公式计算遗传距离。并在MEGA6.0中采用邻近法(Neighbor-Joining, NJ)构建系统树,自举值Bootstrap为1 000次重复。
1.5荧光定量RT-PCR分析
根据扩增得到的序列,用Prime 5.0设计荧光定量PCR特异引物,分别为YG1: 5′-TCACGACAAGAGATAGAAG-3′; YG2: 5′-TCCAGCATAAGCAACTACAT-3′,以花生肌动蛋白基因(actin)为内参基因,引物序列为Actin-F: 5′-GAGGAGAAGCAGAAGCAAGTTG-3′;Actin-R: 5′-AGACAGCATATCGGCACTCATC-3′。在7500FAST荧光定量PCR仪(ABI公司)上对花生干旱胁迫的叶片,以及正常状态根、茎、叶目的基因表达情况进行分析。反应条件为 95℃ 10 s; 95℃ 5 s,60℃ 30 s,72℃ 10 s,40个循环;绘制溶解曲线,温度每10 s升高0.5℃。
2结果与分析
2.1花生NBS-LRR类基因片段分离与同源片段的聚类分析
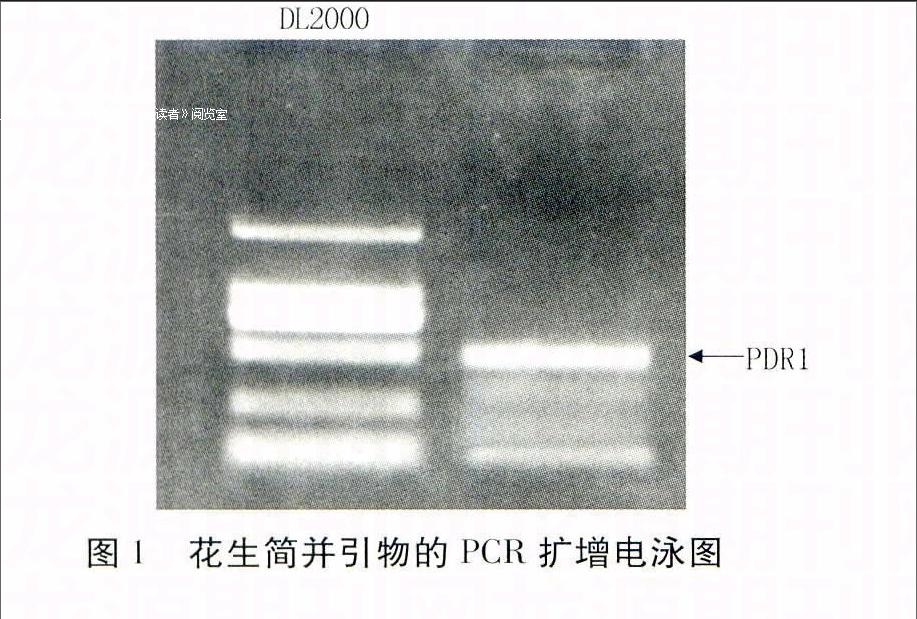
根据NBS-LRR类基因保守结构域设计一对简并引物S1、S2,对反转录合成的cDNA进行扩增,经1.5%琼脂糖凝胶电泳,在J11中获得部分条带(图1),参考已有研究经验,选取其中一条约500 bp的条带进行回收,克隆测序(在JH1012中未获得该片段)。经测序,该片段为一条通读序列,长度为474 bp,命名为PDR1。
2.2PDR1的生物信息学分析
利用花生全基因组相关数据(未公布)找到PDR1所在的一个基因组contig,将相关信息输入GENSCAN网站(http://genes.mit.edu/GENSCAN.html),预测其全长CDS编码框。预测结果显示,PDR1全长CDS编码框为5 838 bp,编码的蛋白质含1 945个氨基酸,其中丙氨酸87个,占4.47%;半胱氨酸28个,占1.44%;天冬氨酸116个,占5.96%;谷氨酸137个,占7.04%;苯丙氨酸85个,占4.37%;甘氨酸113个,占5.81%;组氨酸47个,占2.42;异亮氨酸141个,占7.25%;赖氨酸132个,占6.79;亮氨酸222个,占11.41%;蛋氨酸38个,占1.95;天冬酰胺84个,占4.32%;脯氨酸51个,占2.62%;谷氨酰胺79个,占4.06%;精氨酸111个,占5.71%;丝氨酸170个,占8.74%;苏氨酸82个,占4.22%;缬氨酸142个,占7.30%;色氨酸24个,占1.23%;酪氨酸56个,占2.88%。
通过DNAMAN软件预测出PDR1编码蛋白的分子量大小为221.248 kD、等电点为7.02。由TMpred程序预测PDR1编码蛋白质的跨膜结合区域,发现由内到外、由外到内的跨膜区域均为13个(图3)。根据NCBI上的Conserved Domain Search,发现该蛋白含有NB-ARC等保守结构域(图4)。
2.3PDR1基因表达分析
通过荧光定量PCR分别检测花生品种J11根、茎、叶以及干旱胁迫下叶中PDR1的表达情况。从图5中可以看出,正常情况下,PDR1基因在花生根、茎和叶中均有表达,其中叶中的相对表达量显著高于根(P<0.05),茎中的相对表达量最低。endprint
干旱胁迫之后叶中PDR1的表达情况见图6。0~12 h,基因的相对表达量处于上升阶段,与对照(0 h)相比,6 h的相对表达量达显著差异(P<0.05),12 h的表达量达极显著差异(P<0.01),在12 h达到顶峰,随后下降,18 h与24 h之间无显著差异(P>0.05),处于平稳表达阶段。
3讨论与结论
NBS-LRR类基因是目前发现的最重要的抗性基因,现有的工作已经证明根据保守结构域设计引物进行同源序列的克隆是可行的。本研究所获得的PDR1基因片段与已知NBS-LRR类基因具有较高的相似性,也证明了这一点。目前的许多工作是从基因组DNA中克隆该类基因,但是由于基因组DNA含有内含子,相对复杂,因此分离的NBS-LRR类基因可能有一些是不表达的假基因。魏芳等从小麦基因组中克隆了NBS-LRR类基因,经鉴定43个阳性重组子中有11个为假基因。Yuksel等利用花生基因组克隆NBS-LRR类基因,获得了1 028条克隆序列,但是其中只有234个通读序列。还有研究显示,拟南芥中发现的149个NBS-LRR类基因中12条是假基因;水稻中存在约500个NBS-LRR基因,其中超过100条被认为是假基因。因此,在今后的工作中,应使用RT-PCR的方法进行同源克隆,以避免假基因的干扰。
NBS-LRR类基因通常分为两大类,即TIR类和nonTIR类。TIR类N端为人白细胞介素1和果蝇的Toll样受体(即TIR)结构,如烟草的N抗性基因。nonTIR类N端为亮氨酸拉链结构,如番茄抗根结线虫病基因Mi。Meyers等认为,含有TIR的NBS-LRR类基因蛋白Kinase-2最后一个氨基酸残基通常为天冬氨酸(D),也可以是任意氨基酸;而nonTIR为色氨酸(W)。PDR1基因Kinase-2最后一个氨基酸为天冬氨酸(D),可认为是TIR类。本研究获得的PDR1基因全长是通过生物信息学方法预测得到的,而不是通过试验手段,如RACE。本研究过程中也曾试图利用RACE技术获得全长,然而却极其困难。同时,NCBI上相似度较高长度在4 500 bp以上的基因,除已经完成全基因组测序的苜蓿之外也全是预测序列,可见,较长序列的全长获得在世界范围内也是一个难题。PDR1基因全长的获得需要在接下来的工作中寻找新的方法,逐步获得。
我国有70%面积的花生受到不同程度的干旱危害,干旱胁迫可以显著影响花生出苗、生长、开花、产量及品质等。筛选花生抗旱重要基因,是通过基因工程技术培育高产抗逆花生种质的基础。虽然花生基因组测序已经取得重要进展,基因组已公布(http://peanutbase.org/genomes),但尚有大量工作未完成,已公布的数据无法直接应用,对于许多基因,研究者仍然知之甚少,因此同源基因的功能表达研究仍是花生逆境胁迫研究工作的重要组成部分。本研究对PDR1在干旱胁迫后不同时间点的表达情况进行了分析,总体趋势表现为先上升后下降,最后趋于稳定。PDR1是NBS-LRR类抗性基因,可能与花生的抗逆性相关。从本研究的结果来看,初始阶段,基因表达量上升,表明PDR1开始应答干旱环境,参与花生抗旱过程,随后下降并趋于稳定,但仍高于初始阶段相对表达量,表明该基因已适应干旱环境,平稳表达。因此推测,PDR1基因的功能可能与花生抗旱性相关。
本研究为发现新的NBS-LRR类基因提供了资源,并且为下一步克隆PDR1基因全长、筛选新抗旱花生种质提供了理论依据。
参考文献:
[1]万书波. 花生产业形势与对策[J]. 山东农业科学,2014, 46(10):128-132.
[2]孙学武,王才斌. 中国花生对外贸易概况及前景展望[J]. 山东农业科学, 2013, 45(11): 129-133.
[3]孙大容. 花生的抗旱育种[M].北京:中国农业出版社,1998:256-275.
[4]Wright G C. Alleviation of drought stress and aflatoxin incidence in peanut short maturing varieties [C]//Proceedings of the 9th Australian Agronomy Conference. Australian Society of Agronomy (Inc.),1998.
[5]Li C J, Liu Y, Zheng Y X, et al. Cloning and characterization of an NBS-LRR resistance gene from peanuts (Arachis hypogaea L.) [J]. Physiological and Molecular Plant Pathology, 2013, 84: 70-75.
[6]刘宇,李春娟,郑奕雄, 等. 苗期干旱对花生抗病相关基因PnAG3的表达及耐旱生理指标的影响[J]. 农业生物技术学报, 2012,20(6):642-648.
[7]Meyers B C, Kozik A, Griego A, et al. Genome-wide analysis of NBS-LRR -encoding genes in Arabidopsis [J]. Plant Cell, 2003, 15(4): 809-834.
[8]曲满义,查向东,王钰, 等. 甘薯NBS-LRR类抗病基因同源序列的克隆、分析及数目研究[J]. 热带作物学报, 2008, 29(5): 610-617.
[9]王世全, 张德春, 李平, 等. 水稻中一个NBS-LRR抗病同源基因家族的克隆和分析[J]. 遗传学报, 2005, 32(7): 704-711.endprint
[10]王岩, 李兆阳, 唐心龙, 等. 拟南芥基因组NBS-LRR类基因家族的生物信息学分析[J]. 中国农学通报, 2009, 25(15): 40-45.
[11]吴大强, 蔡诚, 魏国, 等. 毛果杨全基因组NBS类型抗病基因分析[J]. 林业科学, 2009, 45(2): 152-157.
[12]杨秀红, 陈庆山, 杨庆凯, 等. 大豆NBS类抗病相关基因的克隆与序列分析[J]. 高技术通讯,2005,15(2):71-78.
[13]王海燕, 杨文香, 刘大群. 小麦NBS-LRR类抗病基因同源序列的分离与鉴定[J]. 中国农业科学, 2006, 39(8): 1558-1564.
[14]Yuksel B, Estill J C, Schulze S R, et al. Organization and evolution of resistance gene analogs in peanut [J]. Mol. Gen. Genom., 2005, 274: 248-263.
[15]晏立英,廖伯寿,黄家权,等. 花生NBS类抗病基因类似物的分离与序列分析[C]//第五届全国花生学术研讨会论文集. 2007:244-248.
[16]杨晨, 徐凤花, 单世华, 等. 花生子叶RNA提取方法比较与分析[J]. 山东农业科学, 2011(1): 1-5.
[17]Kimura M. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences [J]. J. Mol. Evol., 1980, 16: 111-120.
[18]魏芳, 马鸿翔. 小麦抗赤霉病品种NBS同源序列克隆与分析[J]. 生物技术通报, 2012(2): 128-135.
[19]Monosi B, Wisser R J, Pennill L, et al. Full-genome analysis of resistance gene homologues in rice [J]. Theor. Appl. Genet., 2004,109(7):1434-1447.
[20]Whitham S, McCormick S, Baker B. The N gene of tobacco confers resistance to tobacco mosaic virus in transgenic tomato [J]. Proc. Natl. Acad. Sci. USA, 1996, 93:8776-8781.
[21]Milligan S B, Bodeau J, Yaghoobi J, et al. The root knot nematode resistance gene Mi from tomato is a member of the leucine zipper, nucleotide binding, leucine-rich repeat family of plant genes [J].Plant Cell, 1998, 10:1307-1319.
[22]Meyers B C, Dickerman A W, Michelmore R W, et al. Plant disease resistance genes encode members of an ancient and diverse protein family within the nucleotide-binding superfamily [J].The Plant Journal, 1999, 20(3): 317-322.endprint
