山东玫瑰花核糖体rDNA ITS序列分析初步探究*
2015-08-30李洪芹马昌豪彭艳丽
李洪芹,马昌豪,彭艳丽
山东玫瑰花核糖体rDNA ITS序列分析初步探究*
李洪芹1,马昌豪2,彭艳丽3
(1.菏泽学院,菏泽274015;2.菏泽市食品药品检验检测中心,菏泽274000;3.山东中医药大学,济南250355)
[目的]分析不同种质玫瑰花(Rosae Rugosae Flos)核糖体DNA的ITS序列,为其种质资源分子鉴别提供依据。[方法]采用聚合酶链反应(PCR)技术获得ITS基因,进行测序,经CLUSTALX(2.0)软件进行统计分析,计算各样本间的遗传距离。[结果]各样品间遗传距离范围为0.000~0.011,各样本不仅在非编码区的转录间隔区ITS1和ITS2存在多个变异位点,而且在保守的5.8S编码区也存在变异位点。[结论]ITS序列的测定为玫瑰花品种鉴别和种质资源优化提供分子生物学依据。
玫瑰花;rDNA;ITS序列
玫瑰花(Rosae Rugosae Flos)为蔷薇科蔷薇属多年生灌木玫瑰(Rosa rugosa Thunb.)的干燥花蕾,在中国有悠久的应用历史,具有理气解郁、活血止痛的功效[1]。作为一种药材,玫瑰花还有较高的经济价值,广泛应用于花卉观赏和精油、天然色素的提取等,具有重要开发研究价值。中国玫瑰种质资源十分丰富,栽培历史长达数百年,杂交品种较多,蔡芳[2]通过AFLP分子标记技术研究玫瑰种质资源的遗传多样性,亲缘关系及其系统关系,顾一珠等[3]对不同栽培品种玫瑰花药材进行聚丙烯酰胺凝胶电泳(PAGE)分析,目前还未见关于玫瑰花rDNA ITS序列分析的相关报道。
ITS是核糖体基因中,位于核糖体18S、5.8S和26S之间转录间隔区的DNA片段,被5.8S分割成2段,由于不加入成熟的核糖体,收到的选择压力较小,进化速度较快,但在被子植物中相对长度具有保守性,适合用于低分类群的系统发育分析[4-6],因而ITS序列广泛用于被子植物系统学的研究,尤其是近源属间和属内系统学的研究[7-13]。近年来ITS序列也作为鉴定性状,根据遗传物质DNA在不同生物个体的差异来鉴别生物物种[14-17],在川牛膝[18]、远志[19]、紫花地丁[20]等植物药中得到探讨和应用。
本实验对7个玫瑰花栽培品种进行rDNA ITS序列测定与分析,从分子水平比较不同栽培品种玫瑰花的差异,为玫瑰的种质资源的鉴定、质量控制以及玫瑰的产业化发展提供科学依据。
1 材料与方法
实验所用药材嫩叶均为2011年采自平阴玫瑰园,经山东中医药大学彭艳丽教授鉴定。1为北京妙峰山重瓣白玫瑰,2为北京妙峰山单瓣白玫瑰,3为传统玫瑰,4为保加利亚白玫瑰,7为苦水玫瑰,8为丰花一号,9为紫枝玫瑰。
主要实验仪器:聚合酶链反应(PCR)扩增仪(加拿大BBI公司),3730测序列分析仪(美国ABI公司),SW-CJ-1D洁净工作台(江苏苏洁净化设备厂),DK-8D型电热恒温水槽(上海森信实验仪器有限公司),DYY-8型稳压稳流电泳仪(上海琪特分析仪器有限公司),YXJ-2离心机(湘仪离心机仪器有限公司),H6-1微型电泳槽(上海精益有机玻璃制品仪器厂),凝胶成像系统(Gene Genius公司),U-3010紫外-可见分光光度计(Hitachi公司),移液器(范围100~1 000 mL,20~200 mL,0.5~10 mL)(加拿大BBI公司)。
1.1总DNA提取称取100 mg新鲜嫩叶,液氮中充分研磨成粉。然后按照上海生物工程技术服务有限公司生产的UNIQ-10柱式真菌基因组DNA抽提试剂盒提取,提取的DNA溶液置于-20℃保存。
1.2PCR扩增、纯化与测序
1.2.1PCR扩增体系PCR扩增反应在50 μL体系中完成,反应液含有Template(基因组)10 pmol,Primer up(10 μmol/L)1 μL,Primer down(10 μmol/L)1 μL,dNTP mix(10 mmol/L each)1 μL,10×Taq reaction Buffer 5 μL,Taq酶(5 U/μL)0.25 μL,加灭菌双蒸馏水至50 μL。
1.2.2 PCR程序设定扩增程序为:98℃预变性5min;循环95℃变性35 s,55℃复性35 s,72℃延伸40 s,35个循环后;72℃延伸8 min。
1.2.3引物的选择扩增ITS序列引物依据文献[21]设计,引物序列为:ITS1 5’TCCGTAGGTGAACCTGCGG 3‘ 19bp ITS4 5’TCCTCCGCTTATTGATATGC 3’20 bp扩增结果见图1,从扩增的结果看,7种玫瑰花都能得到一条清晰的PCR产物扩增带。
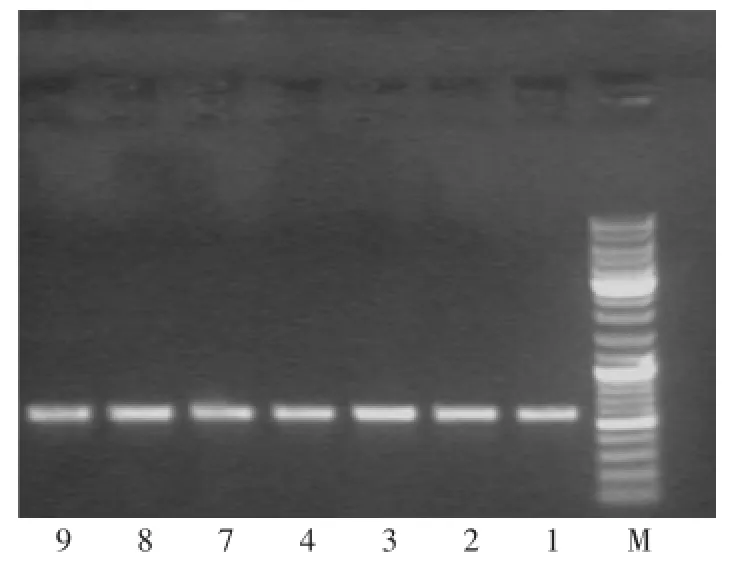
图1 PCR扩增后电泳图谱
1.2.4扩增产物的回收与纯化由PCR产物电泳结果切割所需DNA目的条带,采用UNIQ-10柱式胶回收试剂盒(SK1131)回收纯化。根据说明书操作,回收产物可立即使用或保存于-20℃备用。
1.2.5目的DNA片段的测序将获得的目的片段序列进行序列测定,用3730测序列分析仪进行测序,每个样品均采用双向测序,获得7个种的ITS序列信息。
1.2.6ITS序列分析用CLUSTALX(2.0)程序对位排列ITS序列,用系统发育分析软件MEGA4计算遗传距离标准方差。
2 结果与分析
2.1ITS长度与GC质量分数比较本实验测得的7个样品ITS(包括5.8S)序列的长度与G+C含量,见表1。

表1 样品序列长度及G+C含量
各样品ITS(包括 5.8S)G+C含量范围为57.25%~59.19%,所有种的5.8S编码区序列长度为163 bp。G+C含量差异较大,预示碱基位点发生某种程度的变化。
2.2基于ITS分析各样品间遗传距离各样品间遗传距离和标准方差,见表2,左下方为遗传距离,右上部为标准方差。
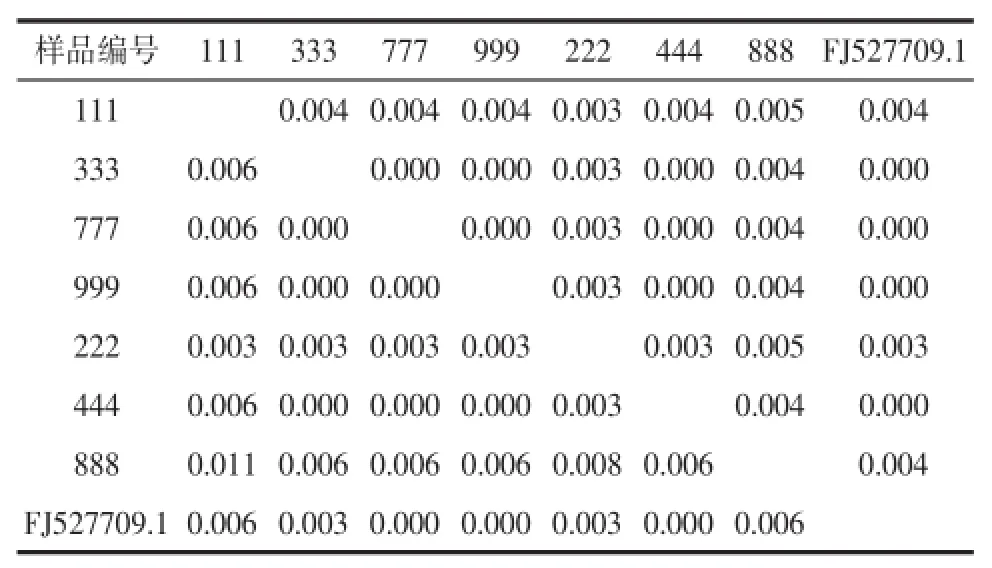
表2 各样品间遗传距离
通过对比发现,各样品间遗传距离均较小,范围为0.000~0.011,标准误差范围为0.003~0.005。
通过参照Genebank中玫瑰花ITS序列(登录号:FJ527709.1),确定 7个样品的 ITS区位置,FJ527709.1共测得ITS序列共696 bp,其中33~286为ITS 1序列,287~450为5.8S序列,451~658为ITS 2序列。从ITS整体变异情况看,各样本不仅在非编码区的转录间隔区ITS1和ITS2存在多个变异位点,而且在保守的5.8S编码区也存在变异位点。在5.8S区(163 bp)变异位点很少(变异位点序号均以FJ527709.1为标准),只有丰花一号(888号样品)在286位、409位上碱基与其他样品不同,在这两个位点丰花一号碱基为A,而其他样品为C,可以考虑将286和409位作为鉴别丰花一号的特异性分子标记,其可靠性还需进一步研究证实。在ITS1区中,苦水玫瑰(777号样品)可分析序列变异位点主要集中在137位(T-C)、155位(C-G)、174位(A-G)、229位(缺失);在ITS2区有较多的碱基缺失和调换,主要集中在466~470位:传统玫瑰(333号样品)在466位、467位、468位碱基缺失;北京妙峰山重瓣白玫瑰(111号样品)在466位、467位、468位碱基替换(C-A);保加利亚白玫瑰(444号样品)、苦水玫瑰(777号样品)、丰花一号(888号样品)、紫枝玫瑰(999号样品)在468位、469位、470位碱基缺失;北京妙峰山单瓣白玫瑰(222号样品)在466位、469位、470位碱基替换(A-C);苦水玫瑰(777号样品)在475位、561位碱基缺失。序列对比见图2。
结果表明不同品种玫瑰花的ITS序列有一定的差异,所以玫瑰花的ITS序列(ITS及5.8S rDNA)分析资料可为玫瑰花的品种鉴别及分类提供新的手段。
3 小结
该实验通过对玫瑰花常见种类的叶片进行核糖体rDNA ITS序列分析,证明该技术可以作为一个鉴别玫瑰花不同品种和种质资源优化的分子生物学依据。通过序列分析,可以考虑将丰花一号5.8S区第286位和409位碱基作为鉴别丰花一号的碱基鉴别位点;苦水玫瑰作为一个中国传统玫瑰的变种在ITS1区有4个变异位点,同时在ITS2区有两个位点的碱基缺失;7个实验样品在ITS2区共5个位点有碱基缺失和替换,主要集中在466~470位。丰花一号、苦水玫瑰作为玫瑰花在市场上种植面积较大、推广较为成功的品种,通过该技术建立的分子标记,更有助于两品种的推广。
由于时间和条件限制,并没有采集到所有常见玫瑰花品种,如保加利亚红玫瑰、大马士革玫瑰、格拉斯玫瑰,因此有待于以后的样品采集和测定工作。本实验对ITS测序技术在玫瑰花药材鉴定方面进行了初步探究,在以后的研究中可以结合形态学特性、主要有效化学成分、精油含量等指标全面考察,为今后进一步研究玫瑰花鉴定、新品种选育、品种优化奠定理论基础。
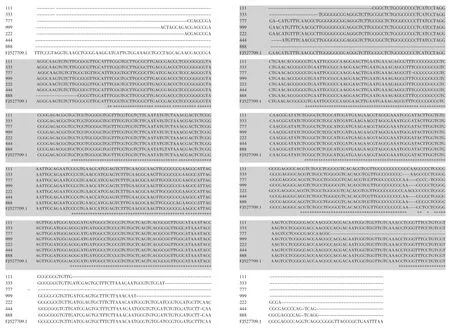
图2 7种玫瑰花ITS序列比较
[1]中华人民共和国药典委员会.中国药典(一部)[S].北京:化学工业出版社,2010:139
[2]蔡芳.玫瑰种质资源与药材鉴别研究[D]北京:北京协和医科大学,2008.
[3]顾一珠,马君一,李明,等玫瑰花聚丙烯酰胺凝胶电泳鉴别研究[J].药物分析杂志,2009,29(5):819.
[4] Baldwin BG,Sanderson MJ,Porter JM.The ITS region of nuclear ribosomal DNA,a valuable source of evidence on angiosperm phylogeny[J].Annals of the Missouri Botanical Garden,1995,(82):247-277.
[5] Bellarosa R,Simeone MC,Papini A,et al.Utitity of ITS sequence data for phylogenetic reconstruction of Itatlian Quenrces spp[J].Molecular Phytogenetics and Evolution,2005,(34):355-370.
[6]Kress WJ,Liu AZ,New man M,et al.The molecular phylogeny of Alpinia(Zingiberaceac).A complex and polyphyletic genus of gingers[J].American Journal of Botany,2005,9(2):167-178.
[7]田晓轩,刘杰,肖咪,等.基于DNA条形码区域的中药地鳖隐存物种多样性发展研究[J].天津中医药大学学报,2014,33(2):90-93.
[8]金建峰,朱思眉,蒋明,等.石斛属植物rDNA ITS序列的克隆与分析[J].浙江农业学报,2014,6(3):685.
[9]蒋明,李温平,周晶,等.10种杓兰属植物rDNA ITS序列的克隆与分析[J].浙江大学学报,2012,39(6):689-695.
[10]赵欢,吴卫,郑有良,等.核糖体DNA ITS序列分析在药用植物研究中的应用[J].时珍国医国药,2009,20(4):959
[11]顾慧芬,庄意丽,马子建,等.基于ITS序列分析铁皮石斛与近缘类群的亲缘关系[J].中成药,2010,32(4):628-632.
[12]朱颖,董玉芝,昝少平,等.基于ITS序列探讨忍冬属的系统发育关系[J].西北植物学报,2010,30(2):250-254.
[13]李艳萍,魏继承,何淼,等.基于rDNA ITS序列东北地区丁香属植物的系统发育关系[J].东北林业大学学报,2010,38(8):45-47.
[14]唐铖,张铁军,刘昌孝.基于PCR技术的中药指纹图谱研究[J]天津中医药大学学报,2007,26(3):162-165.
[15]商洪才,张伯礼,高秀梅.中医药现代化与分子生物学[J].天津中医药,2001,18(5):41-43.
[16]郑守曾,王睿林.中医药现代化研究中分子生物学技术的应用(一)[J].天津中医药,2005,22(3):186-189.
[17]郑守曾,王睿林.中医药现代化研究中分子生物学技术的应用(二)[J].天津中医药,2005,22(4):272-275.
[18]陈诗晴,蒲沁琳,陈小军,等.基于ITS序列分析技术鉴定川牛膝与常见伪品麻牛膝[J].中药与临床,2014,38(9):4-6.
[19]樊杰,白妍,束明月.远志及其近缘种的ITS序列分析及鉴定[J].山西中医学院学报,2014,15(4):31-32.
[20]朱烨,张春,庄元春.基于ITS序列分析对紫花地丁的分子鉴别[J].中华中医药,2014,28(4):918-920.
[21]曾明,马雅军,郑水庆,等.中药葛根及其近缘种的rDNA-ITS序列分析[J].中国药学杂志,2003,38(3):173.
The pre-explore of rDNA ITS sequence of Flos Rosae Rugosae in Shandong province
LI Hong-qin1,MA Chang-hao2,PENG Yan-li3
(1.Heze University,Heze 274015,China;2.Heze Institute of Food and Drug Control,Heze 274000,China;3.Shandong University of Traditional Chinese Medicine,Jinan 250355,China)
[Objective]To provide scientific evidence for molecular identification by analyzing ITS sequences in different germplasms from Flos Rosae Rugosae.[Methods]The ITS(Internal Transcribed Spacer)regions were cloned by PCR amplification and sequenced bi-directionally.The ITS sequences were aligned by CLUSTALX(2.0)and genetic distances were calculated.[Results]The genetic distances among different germplasms ranged form 0.000 to 0.011.There were a lot of variable sites in both ITS1 and ITS1 sequences from non-coding regions,as well as 5.8S from the conservative region.[Conclusion]The phylogentic analyses based on ITS sequences can provide reliable molecular evidences for identifying different genotypic germplasms of Flos Rosae Rugosae.
Flos Rosae Rugosae;rDNA;ITS sequence
R284
A
1673-9043(2015)02-0104-04
10.11656/j.issn.1673-9043.2015.02.10
山东省自然科学基金资助项目(ZR2009CM106)。
李洪芹(1987-),女,硕士研究生,助教,研究方向为生药、中药质量控制和品质评价。
彭艳丽,E-mail:py1567@163.com。
(2014-10-20)
