图顶点着色问题的质粒DNA计算
2015-08-19马莹殷志祥
马莹++殷志祥

摘 要:图的着色问题是著名的NP问题,有着重要的实际意义。比如通讯系统的频道分配、考试排考场问题等方面有直接应用。图的着色问题采用DNA计算方法很多,有表面DNA计算,粘贴DNA计算。本文提出质粒DNA计算,首先把顶点着色问题转化为求最大独立集问题,然后给出了图顶点着色问题的质粒DNA分子生物实验,利用限制性内切酶的特性切割有边相连的顶点,得到最大独立集,在试验中特别引入了一个备用试管,最后给出一个具体的实例。实例给出具体的着色方案,证明了该质粒DNA算法有效并且是可行的。
关键词:DNA计算;顶点着色;最大独立集;质粒
中图分类号:TP301 文献标志码:A 文章编号:1672-1098(2015)02-0064-04
DNA Computing Model for the Graph Vertex Coloring Problem by Plasmids
MA Ying, YIN Zhi-xiang
(School of Science,Anhui University of Science and Technology,Huainan Anhui 232001, China)
Abstract:Graph coloring is a famous NP-problem, which has important practical significance, which applies in channel allocation of communication system, examination room arrangement and so on. There are a lot of DNA computing methods for graph coloring problem, such as surface DNA computing, pasting DNA computing. In the paper the plasmid DNA computing for the graph vertex coloring problem was presented. At first, the graph vertex coloring problem was transformed into a vertex of maximum independent set problem. Then the plasmid DNA molecular biological experiment of graph vertex coloring problem was conducted. The vertexes connected with edges were cut by using the feature of restriction enzymes, and the maximum independent set was obtained. In the experiment a spare tube was introduced. At last, the method was illustrated by an example. In the example the final coloring schemes were given. It was verified that the plasmid DNA algorithm is effective and feasible.
Key words:DNA computing; vertex coloring problem; maximum independent set; plasmid
1994年,文献[1]首次用DNA计算解决有向图的哈密顿问题;1995年文献[2]在Adleman思想的启发下,解决了可满足性问题;1997年,文献[3]利用DNA计算解决了另一个NP完全问题,图的最大团问题;2000年,文献[4]提出了用质粒DNA分子来解决可满足性问题;2004年,文献[5]提出了基于质粒DNA匹配问题的分子算法。
图顶点着色问题是著名的NP问题。2003年,文献[6]讨论了图的3-顶点着色的DNA算法;2005年,文献[7]提出了先把着色问题分解成顶点独立集问题和顶点划分问题并给出这两个问题的DNA 粘贴算法,并调用这两个算法解决了图顶点着色问题;2006年,文献[8]提出将图的顶点着色问题转化为SAT-问题来解,并且利用粘贴DNA计算来解决;2009年,文献[9]建立了一种基于酶切技术和PCR技术的图顶点着色DNA计算模型,给出了实现该模型的双编码的编码方案。
本文提出基于质粒的DNA计算求解图的顶点着色问题,从图顶点着色问题的本质出发,把图的顶点着色问题转化成图的顶点划分问题。
1 质粒
质粒是染色体外小型的共价、闭合、环状的双链DNA分子,是含有复制功能的遗传结构的DNA分子。目前DNA计算中采用的质粒是闭环状的双链DNA分子,常用人工构建的质粒作为载体。常用的人工质粒运载体有pBR322、pSC101,必须包含三个特征:复制子、选择性标志和克隆位点。克隆位点是限制性内切酶切割位点,外源性DNA可由此插入质粒内,而且并不影响质粒的复制能力。用限制性核酸内切酶和DNA链接酶可以对质粒进行剪切和重组。每位与唯一的限制性酶相对应,每位都以它对应的限制性酶为识别位点。
11 质粒编码
给一个质粒 DNA 环状分子进行编码,设P是一个质粒载体,k是正整数,s1,s2,…,sk是P的k个相互不重叠的子段。对于每个i,核苷酸序列si不能出现在质粒P的其余位置上,并称si是质粒P的“位置”。通过切割和粘贴,质粒在“位置”处不断地修改,相应的核苷酸序列si要么在质粒上,要么不在,分别用1和0表示。质粒DNA的位为1表示这个位对应的顶点在质粒DNA对应的顶点集中,质粒DNA的位为0表示这个位对应的顶点不在对应的顶点集中。 例如: 质粒111111表示顶点集{v1,v2,v3,v4,v5,v6}, 质粒 100001 表示顶点集{v1,v6}。endprint
12 限制性内切酶
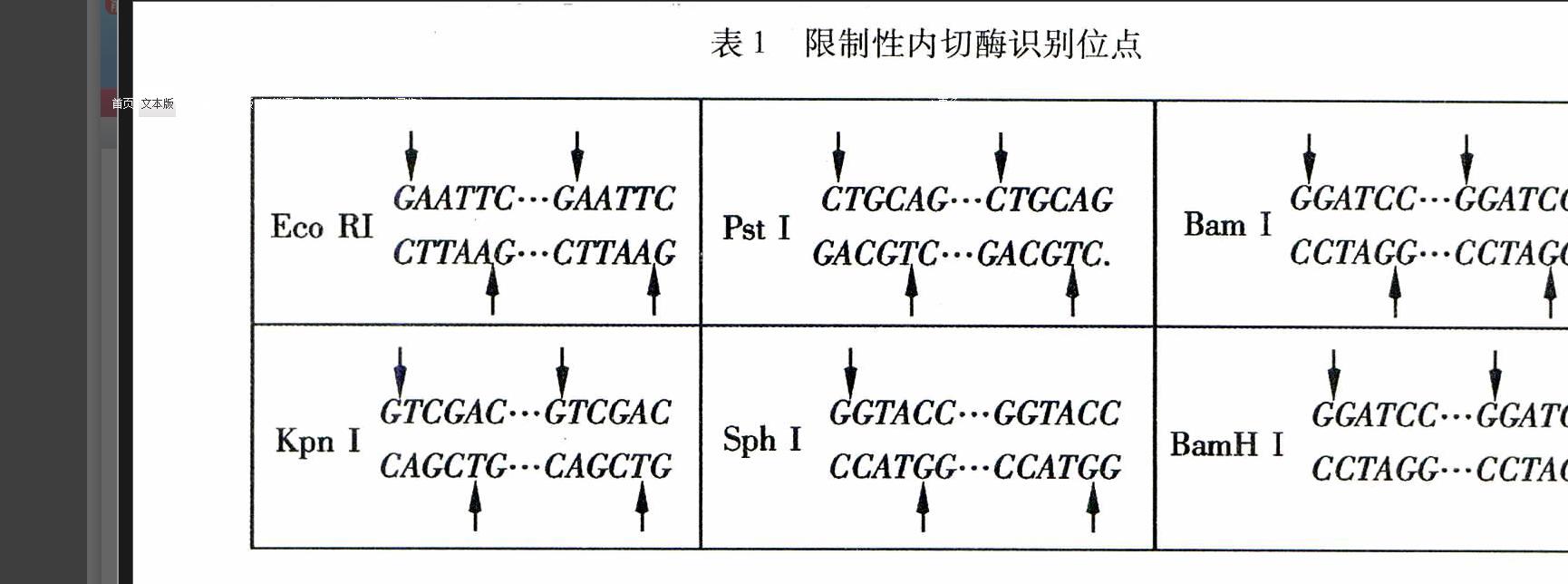
生物体内能识别并切割特异的双链DNA序列的一种内切核酸酶。限制酶的名字根据最初分离出限制酶的生物体所命名,第一个字母取自属名的第一个字母,随后的两个字母取自种名的前两个字母,第四个字母表示菌株(品系)。例如Bam H,表示从Bacillus amylolique faciens H中分离出来的限制内切酶。通常,限制性内切酶仅剪切双链DNA分子,而且只在一些特定的位置上剪切(见表1)。表1列出常用的限制性内切酶的识别位点,其中箭头表示切割位点。
表1 限制性内切酶识别位点
2 图顶点着色问题的算法设计
设G是一个图,分别用V,E,ψv(G),Δ(v),p和q表示图G的顶点集,边集,G的点色数,顶点v的度,顶点数和边数。
定义1 设G为任意图且C代表颜色集合,如果存在映射σ∶ V(G)→C使得对任意uv∈E(G),σ(u)≠σ(v),则称σ为G的顶点着色法,即对G的每个顶点用C中的某种颜色着色使得每个顶点只染一种颜色,且相邻两个顶点不能染同种颜色。
定义2 对图G的顶点着色需要的最少颜色数称为G的点色数,简称为G的色数,并用ψv(G)来表示。
定义3 对图G=(V,E)的结点子集SV,如果
1) S中的任意两个结点均不相邻,则称S为G的一个独立集。
2) 若S是G的独立集,且不存在G的另一独立集S′满足|S′|>|S|,则称S为G的最大独立集。
定义4 对于无向图G(V,E),将其顶点集V划分为互不相交的多个非空子集V1,V2,…,Vm,使得V1∩V2∩…∩Vm=V,且Vi∩Vj=,其中i,j=1,2,…,m,i≠j,则V1,V2,…,Vm称为G=(V,E)的顶点一个划分。
定理1 对任意图G,有ψv(G)≤Δ+1。
证:对图的结点数n作归纳证明,当n=1时,即图只有一个结点,ψv(G)=1,定理显然成立。设结点数为n-1时定理成立,现增加一个结点v0,则与v0邻接的结点最多有Δ个,即使这些结点都着不同颜色,也不会超过Δ种颜色,则给v0着不同颜色,色数不会超过Δ+1,定理亦成立。
由于独立集内的所有结点可着同一颜色,如果将图的顶点进行划分,使每一结点子集都是独立集,即最小划分数即是图的点色数。
21 图顶点着色的算法
图顶点着色算法:
先求图G的一个最大独立集V1,然后求G-V1的最大独立集V2,然后再求G-(V1∪V2)的最大独立集V3,如此继续直到最后一个最大独立集Vk,则所需色数ψv(G)=k,即求G的独立数I(G)。
具体算法步骤如下:
步骤1:从顶点V中选取顶点,不妨设为v1,记k=1,Vk={v1}。删除与顶点v1相邻的顶点,剩余的顶点假设记为vi,vj加入Vk={v1,vi,vj}集合中,Vk即为所有顶点中的最大独立集。n为Vk中元素的个数。
步骤2:如果n=|V(G)|,则k为顶点着色数;否则执行下一步。
步骤3:把k换成k+1,继续找剩下顶点中最大独立集Vk,按顺序找出顶点记为vm(vm为不在Vk中的顶点),记Vk={vm},删除与vm相邻的顶点,剩下的顶点加入到Vk,n为V1,V2,…Vk中所有元素的个数,转步骤2。
22 图顶点着色问题的DNA计算模型系统
1) 顶点编码。每个顶点用寡聚核苷酸片段进行编码,每段的两端都有特殊酶切位点的序列。例如,顶点v1 用A,T,C,G随意编码,长度为50 bp,其两端含有限制酶Eco RI的识别序列为5′-GAATTC,其分割点在G与A之间,这样通过Eco RI作用就可以将v1从链上切除。Station2表示顶点v2所在的位置,其两端含有Pst I的识别序列式5′-CTGCAG,类似通过Pst I作用可将v2从链上切除。顶点编码成质粒形式如图1所示。
图1 顶点编码
2) 图顶点着色问题的DNA生物算法。对应的质粒DNA算法具体步骤如下:
步骤1:编码。给顶点进行DNA编码,每个顶点用A,T,G,C进行编码,长度可以在40 kb至50 kb之间,在每个顶点的两端连接限制性内切酶。将编码好的DNA片段放入试管中。将编码好的DNA片段链插入到已开口质粒中,这样可以形成环状的双链DNA质粒。
步骤2:扩增。将细菌质粒转化到大肠杆菌中进行扩增,可以得到数量足够多的新质粒,用试管T0表示。
步骤3:切割。检查试管T0中任意两条边之间是否有顶点连接。如果都没有顶点相连,转入步骤4,否则假设有边ei和边ej相连对应的顶点为vm,将步骤2所产生的新的质粒的试管T0分成相等的三个试管,一个试管单独放置(步骤4使用),然后检查所有和vm相连的边,另外两个试管中分别加入切割有边相连的两个顶点所对应内切酶,然后把切割下小的DNA片段和质粒分离,最后使质粒重新环化,合成一个试管。返回步骤2。
步骤4:找到最大独立集。测序而且记录DNA分子片段,找到该分子片段所对应的顶点,令其记为Vk(k=1,2…)。如果V1,V2,…,Vk中总的顶点数恰好等于图G的顶点数,则转步骤5;否则,再用试管T0(步骤2中的初始试管)切割V1,V2,…,Vk顶点,把切割下小来的DNA分子片段和质粒分离,使质粒重新环化,合成一个试管,转步骤2。
步骤5:k即为着色数,则V1,V2,…,Vk中的顶点集为同一种颜色。
3 一个实例
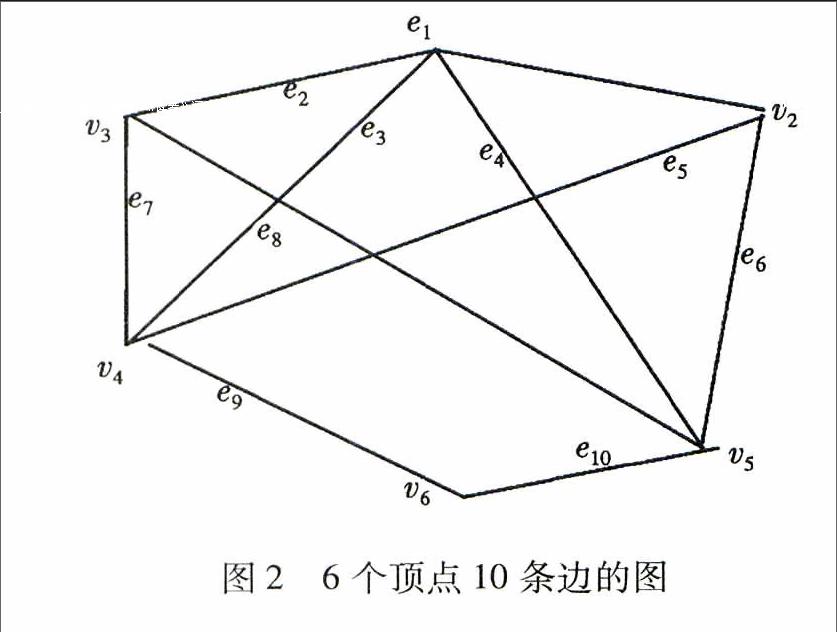
下面对图1所示顶点着色问题来详细讨论上述算法生物实现步骤。
步骤1:编码输入。对图1中的每个顶点进行编码。
图2 6个顶点10条边的图endprint
步骤2:扩增。将合成的DNA分子片段链插入到已经开口的质粒中,这样形成闭环状的质粒,再转入大肠杆菌进行扩增,容易得到数量足够多的实验所需要的新质粒,仍用用试管T0表示。
步骤3:剪切。将试管T0分成三个相同的试管T′ 0、T1和T2。T′ 0为备用试管,在步骤4中使用。此时试管中质粒位为111111,即点集{v1,v2,v3,v4,v5,v6}。首先,对顶点v1所有连接的点全部切割掉,则剩下的点即与v1不连接。对边e1顶点v1与其相邻的顶点v2,在试管T1中用对应的酶Eco RI切掉v1所表示的粘性末端DNA分子片段,并且把切割下来的DNA分子片段和质粒分离,使T1中的质粒重新环化,这时T1中质粒表示位为011111,即点集{v2,v3,v4,v5,v6}。在试管T2中用对应的酶Pst I切掉v1所表示的粘性末端DNA分子片段,把切割下来的DNA分子片段和质粒分离,并且使T1中的颗粒重新环化,这时T2中质粒表示位为101111,即点集{v1,v3,v4,v5,v6}。把试管T1和T2合成一新的试管,仍记为T0。若图中无边则表示图可划分为{v1,v3,v4,v5,v6}和{v2},或者{v2,v3,v4,v5,v6}和{v1},图为2-可着色。
将试管T0分成两个相同的试管T1和T2。对于边e2对应顶点v1和v3,在试管T1中用限制性酶Eco RI切割掉v1顶点所表示的粘性末端DNA分子片段,把切割下来的DNA分子片段和质粒分离,并且使T1中的质粒重新环化,这时T1中含质粒为011111和001111,在T2试管中加入BamH I切割掉e2对应顶点v3,并使T2中的颗粒重新环化,此时T2中质粒表示011111和100111。把试管T1和T2合成一新的试管,仍记为T0。T0中含有三种新的质粒对应的为表示为011111和001111以及100111。
将试管T0分成两个相同的试管T1和T2。对于边e3对应顶点v1和v4,在试管T1中用相应的限制性酶Eco RI切割掉v1所代表的粘性末端DNA片段,把切下来的DNA片段和质粒分离,并使T1中的颗粒重新环化,此时T1中含质粒为011111, 001111和000111,在T2试管中加入Sal I切割掉e3对应顶点v4,并使T2中的颗粒重新环化,此时T2中质粒表示011111,001111和100011。把试管T1和T2合成一新的试管,仍记为T0。T0中含有四种新的质粒对应的为表示为011111,001111和000111以及1000111。
再切割和顶点v2相连的顶点(v1除外),依次切割和v3相连的顶点(v1和v2除外),经过反复切割,最终可得试管中的质粒表示为000001,000010,001001,000110,10000,011001,即表示顶点集{v6},{v5},{v3,v6},{v4,v5},{v1,v6},{v2,v3,v6},即最大独立集为{v2,v3,v6}。
步骤4:测序。通过凝胶电泳实验找出链长最长的质粒,用分子克隆技术确定长度最大的质粒,它所代表的编码就是最大顶点独立集{v2,v3,v6}。
步骤5:从剩余顶点中求最大顶点独立集。在步骤2中备用试管T′0中切割v2,v3,v6,则试管T′0中仍含有顶点v1,v4,v5,转入步骤2,寻找T′0的最大独立集,可求得{v4,v5}为最大独立集。
从剩余顶点中求最大顶点独立集。在步骤2中备用试管T′0中切割v4,v5,则试管T′0中只含有顶点v1,显然,图可划分为{v2,v3,v6},{v4,v5},{v1},即图可3-着色。endprint
