一种从富含次生代谢产物茶树嫩叶中高效提取基因组DNA的方法
2015-08-18唐祥凯谭和平沈兴中李怀平
唐祥凯,谭和平,沈兴中,李怀平,许 洋,管 驰
一种从富含次生代谢产物茶树嫩叶中高效提取基因组DNA的方法
唐祥凯1,2,谭和平1,2,沈兴中2,李怀平1,2,许洋1,2,管驰1,2
(1.茶叶标准与检测技术四川省重点实验室,四川成都610021;2.中国测试技术研究院,四川成都610021)
建立一种从富含次生代谢产物的茶树嫩叶中高效提取基因组DNA的方法。提出使用冷冻研磨机来破碎细胞,在确保低温环境保护下,快速、均匀地破碎茶树细胞,解决细胞破碎后易氧化、褐变的关键难点;同时在提取液中加入β-巯基乙醇等抗氧化剂进一步防止多酚类化合物的氧化;经过多次纯化有效去除多酚、多糖、蛋白质等杂质;所提取的基因组DNA经验证完整性好、纯度高,完全能进行酶切和聚合酶链式反应(PCR)。此外,进行系统性的方法学考察,结果表明该提取方法稳定、可靠、重现性好。该研究为茶树基因组DNA提取标准方法的建立奠定基础。
DNA提取;茶树;方法论;稳定性分析
doi:10.11857/j.issn.1674-5124.2015.09.011
0 引言
基因组DNA是分子生物学研究的基础,如何高效提取完整、纯净的DNA是生物技术工作者一直关注的问题[1]。茶树富含多酚、多糖、单宁等次生代谢产物[2-5],茶叶细胞破碎后,多酚类化合物极易发生氧化、褐变,并与基因组DNA粘附在一起[6],使得从茶树中提取完整性好、纯度高的基因组DNA变得异常困难,从而也极大制约了遗传多样性检测、基因映射、图位克隆、基因型鉴定等后续研究。
目前常规的基因组DNA提取方法,植物细胞的破碎一般采用手工研磨,耗时长、效率低,温度和细胞破碎程度难控制;在研磨过程中极易发生多酚的氧化,提取的基因组DNA不完整,质量稳定性、重复性差。为了解决这些难题,本研究对现有的植物基因组提取方法进行了改良和优化,提出一种从富含次生代谢产物的茶树嫩叶中高效提取基因组DNA的方法。
1 材料和方法
1.1植物材料
本研究选取国家级良种福鼎大白茶(Camellia sinensis cv.Fuding dabaicha)的一芽一叶为材料,材料均来自茶叶标准与检测技术四川省重点实验室盆栽场。
1.2仪器与试剂
6770冷冻研磨机(美国SPEX CertiPrep公司);UV-1700分光光度计(日本SHIMADZU公司);水净化系统(美国Millipore公司);Veriti 96孔热循环梯度PCR仪(美国Applied Biosystems公司);恒温系统(美国JULABO公司)。
DNA提取液I:100mmol/L Tris-Cl,20mmol/L EDTA,500mmol/L NaCl,1.5%SDS;DNA提取液II:18.6g葡萄糖,6.9g Na2S2O5,6.0g PVP,240μLβ-巯基乙醇,加水至300m L;乙酸钠溶液:3mol/L,用乙酸调pH至5.2;氯仿∶异戊醇∶乙醇溶液(ν∶ν∶ν,80∶4∶16);TE缓冲液:10mmol/L Tris-Cl,1mmol/L EDTA,使用HCl或NaOH调节pH至8.0;异丙醇;RNase A酶;无水乙醇;70%乙醇;EcoR I内切酶;Pst I内切酶;Taq DNA聚合酶;无菌双蒸水。
1.3提取方法
1)取植物新鲜嫩叶,用无菌超纯水冲洗1min,70%乙醇消毒2min,再用无菌超纯水冲洗2min,最后用无菌吸水纸吸干水分,称取约0.5 g备用(精确至0.1mg)。
2)将干净样品放于灭菌的样品研磨瓶中,按表1程序将样品研磨成粉状,迅速用无菌玻棒将磨碎样品转移至预冷的含有5mL DNA提取液II 的15mL离心管中,加入0.09 g Na2S2O5,0.2 g PVP,500μLβ-巯基乙醇,颠倒混匀,4℃5 000 r/min离心5min。

表1 研磨机研磨条件
3)去上清液,加入5mL预热至65℃的DNA提取液I,温和颠倒混匀,65℃温浴1 h,温浴期间每10min摇动混匀一次。
4)将离心管放置于冰上冷却,加入1m L预冷的NaAc溶液,混匀后加入5mL氯仿∶异戊醇∶乙醇溶液,温和颠倒混匀(需带手套,防止损伤皮肤),冰上静置10min,使水相和有机相分层(必要时可重新混匀),4℃10000 r/min离心10min。
5)仔细移取上清液至一无菌15mL离心管中,如果在水相和有机相交界处有白色沉淀物,则重新抽提有机相,合并水相。
6)重复上述抽提步骤,向抽提得到的上清液中加入等体积异丙醇,温和倒转数次使之混匀,-20℃放置20min,出现絮状DNA沉淀。
7)将絮状DNA沉淀转入含600μL TE的无菌离心管中(如DNA不形成絮状沉淀,则可在4℃10000 r/min离心5min,再将沉淀移入TE管中。这样收集的沉淀,往往难溶解于TE,可在60℃水浴放置15min以上,以帮助溶解)。
8)加入RNaseA溶液至RNase A终质量浓度为10μg/mL,37℃温浴30min;加入0.1g PVP,加入等体积氯仿∶异戊醇∶乙醇溶液,温和颠倒混匀,并以10000 r/min离心10min。
9)取上清液于新的1.5mL灭菌离心管内,重复抽提一次。
10)向抽提得到的上清液中加入1/10体积的NaAc溶液,加入2倍体积预冷的无水乙醇,混匀,-20℃放置20min左右。
11)4℃10 000 r/min离心10min,弃上清液,加入1 mL 70%乙醇,温和颠倒离心管数次,洗涤沉淀。
12)重复11)步骤2次,向沉淀加入500μL预冷的无水乙醇,4℃10000 r/min离心5~8s。
13)弃去乙醇溶液,将离心管开盖放置于室温下,待乙醇挥发干后将DNA重新溶解于200μL TE缓冲液中。
1.4纯度测试方法
1.4.1紫外光谱法
通过测定260nm/280nm吸光度比值来判断所提取基因组DNA是否含有蛋白质污染物;260 nm/ 230nm的吸光度比值来判断基因组DNA是否含有盐、碳水化合物等杂质;260 nm/270 nm的吸光度比值来判断基因组DNA是否含有多酚类杂质[7];DNA的浓度可以通过测定260nm处的吸光度值来换算,但是测定前,应将DNA溶液的浓度稀释到260 nm处吸光度值介于0.1~0.5之间。

表2 基因组DNA样品纯度
1.4.2琼脂糖凝胶电泳
通过琼脂糖凝胶电泳结果来判断所提取的基因组DNA的完整性和纯度,取3μL DNA溶液在0.8%琼脂糖凝胶上,以4V/cm的电压电泳50min。
1.4.3限制性内切核酸酶
通过限制性内切酶反应结果来判断基因组DNA中是否含有限制性内切酶的抑制剂,将500 ng基因组DNA和EcoR I酶混匀,在37℃水浴1h,将消化产物在0.8%琼脂糖凝胶上进行电泳。
1.4.4PCR
在反应体系为50μL的离心管中加入300ng基因组DNA,5μL 10×PCR缓冲液,3μL 25mmol/L的氯化镁,8μL 2.5 mmol/L的dNTP,2μL 10μmol/L的上引物,2μL 10μmol/L的下引物和1U 5 U/μL的Taq DNA聚合酶,无菌双蒸水补齐至50μL,引物为5′-ATAAGCCAACTACAAGTCCAAGC-3′和5′-CACTATTCACCCAGTATTCCACC-3′。扩增程序为94℃保持5min,1个循环;94℃保持30 s,53℃保持30 s,72℃保持30s,30个循环;最后72℃保持7min。扩增结束后用凝胶纯化试剂盒(天根,中国)纯化PCR产物,并通过Pst I裂解,将PCR产物和酶切产物在1.5%琼脂糖凝胶上进行电泳分析,PCR产物的大小应为364bp,产物DNA经酶切后被分解为两个片段,大小分别为76bp和288bp。
1.5方法学考察
稳定性是评价提取方法的一个主要因素,进行6次重复性试验,提取的基因组DNA的纯度通过紫外分光光度法和琼脂糖凝胶电泳进行检测。
2 结果与分析
2.1紫外光谱法
如表2所示,260 nm/280 nm吸光度比值介于1.81~1.83,这表明提取的基因组DNA中基本不含蛋白质等杂质,260 nm/230 nm吸光度比值>2.00,这表明基因组DNA中基本不含盐、碳水化合物等杂质,260nm/270nm吸光度比值为1.20,表明多酚类杂质也被去除。
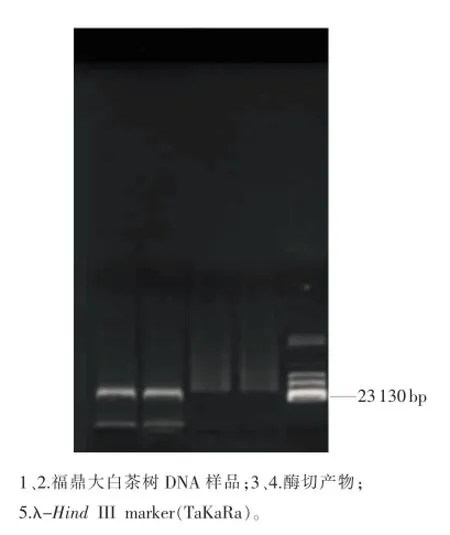
图1 基因组DNA及酶切产物电泳图
2.2琼脂糖凝胶电泳
如图1所示,DNA条带集中在泳道中,在紫外灯照射下明亮且无拖尾现象,分子量约为23 kbp,能被EcoR I消化(图1中泳道3和4),表明提取的基因组DNA完整,纯度高,不含抑制限制性内切酶的杂质。
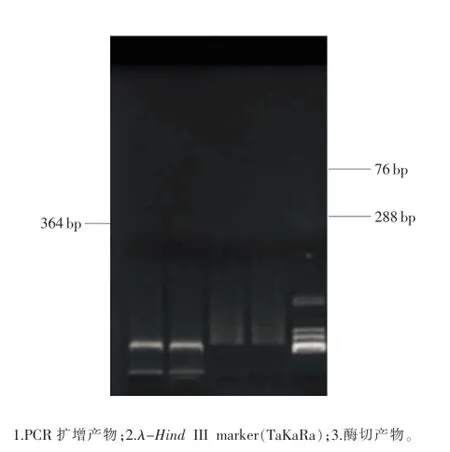
图2 PCR产物、酶切产物电泳图
2.3PCR
如图2所示,以提取的基因组DNA样品为模板,通过PCR可成功扩增出364 bp的预期条带,而PCR扩增片段经Pst I酶切后,可被成功酶切为76 bp和288bp的两个片段,这表明所提取的基因组DNA样品不含抑制PCR扩增反应的杂质,可作为PCR反应模板。
2.4重复性考察
如表3所示,经过6次重复性试验,基因组DNA样品的260nm/280 nm吸光度比值介于1.79~1.83,260nm/230nm吸光度比值均大于2.00,260nm/270nm吸光度比值介于1.20~1.22。如图3所示,6个DNA样本条带集中在泳道中,在紫外灯照射下明亮且无拖尾现象,所提取基因组DNA不含蛋白质、盐、碳水化合物、多酚等杂质,且纯度基本一致,表明该基因组DNA提取方法稳定、可靠、重现性高。

表3 6个基因组DNA样品的纯度分析

图3 6个基因组DNA样品凝胶电泳图
3 结束语
本研究建立了一种从富含次生代谢产物的茶树嫩叶中高效提取基因组DNA的方法。在该方法中首次提出使用冷冻研磨机来破碎细胞,通常磨碎一个样品只需1~2min;细胞的破碎时刻处于液氮环境保护下,同时在提取液中加入β-巯基乙醇等抗氧化剂[8-10],可有效避免多酚类物质的氧化;细胞破碎程度可根据需求进行控制,使得每次提取的DNA完整性、质量稳定性和重复性都能得到有效保证。经过多次纯化去除多酚、多糖、蛋白质等杂质,所提取的基因组DNA经验证完整性好、纯度高,完全能进行酶切和聚合酶链式反应(PCR);此外,系统性的方法学考察也表明该提取方法稳定、可靠、重现性高;本研究为茶树基因组DNA提取标准方法的建立奠定了基础。
研究还表明,适合茶树基因组DNA提取的最佳材料为茶树嫩叶,在提取过程中加入β-巯基乙醇、PVP、焦亚硫酸钠等抗氧化剂对于防止多酚类物质的氧化效果明显,但β-巯基乙醇有刺激性气味且有毒,操作过程中需谨慎;而高浓度的盐会严重抑制PCR反应,所以基因组DNA必须用70%乙醇多次洗涤以除去这些杂质。
[1]Dehestani A,Kazem i S K,Tabar.A rapid efficient method for DNA isolation from plants with high levels of secondarymetabolites[J].Asian Journal of Plant Sciences,2007,6(6):977-981.
[2]Mondal T K,Singh H P,Ahuija P S.Isolation of genomic DNA from tea and other phenol rich p lants[J]. Journal of Plantation Crops,2000(28):30-34.
[3]Horne E C,Kumpatla SP,Patterson K A,etal.Improved high-throughput sunflower and cotton genomic DNA extraction and PCR fidelity[J].Plant Mol Biol Rep,2004 (22):83-83.
[4]Khan I A,Awan F S,Ahmad,et al.A modified mini-prep method for economical and rapid extraction of genom ic DNA in plants[J].Plant Mol Biol Rep,2004 (22):89-89.
[5]陈亮,陈大明,高其康,等.茶树基因组DNA提纯与鉴定[J].茶叶科学,1997,17(2):177-181.
[6]Zhang L L,Wang B,Pan L.Recycling Isolation of Plant DNA,A Novel Method[J].Journal of Genetics and Genomics,2013(1):45-54.
[7]谭和平,沈兴中,唐祥凯,等.茶树基因组DNA测试方法概述[J].中国测试,2010,1(1):49-52.
[8]乔玉山,章镇,沈志军.中国李基因组DNA提取方法的优化[J].上海交通大学学报,2004,22(2):138-142.
[9]黄瑞安,黄意欢,罗君武,等.茶树基因组DNA的高效提取方法[J].湖南农业大学学报,2003,29(5):402-407.
[10]易庆平,罗正荣,张青林.植物总基因组DNA提取纯化方法综述[J].安徽农业科学,2007,35(25):7789-7791.
Efficient method for isolation of genom ic DNA from tender tea leaves rich in secondary metabolites
TANG Xiangkai1,2,TAN Heping1,2,SHEN Xingzhong2,LI Huaiping1,2,XU Yang1,2,GUAN Chi1,2
(1.Standard and Testing Technology of Tea Key Laboratory of Sichuan Province,Chengdu 610021,China;2.National Institute of Measurement and Testing Technology,Chengdu 610021,China)
An efficient method for isolation of genomic DNA from tender tea leaves rich in secondary metabolites is established.First,the cells of tea leaves were grinded quickly and uniform ly with a 6770 Freezer/Mill at low temperature.The difficulty of how to avoid the oxidation and browning of polyphenols has hence solved.Additionally,β-mercaptoethanol and other antioxidants were added into an extraction buffer to prevent the polyphenols from oxidizing.After several purification steps,impurities,such as polyphenols,polysaccharides,and proteins,were removed.The genomic DNA extracted from tea leaves is intact,pure and suitable for restriction digestion and polymerase chain reaction.Moreover,the systematic methodological study has indicated that the method is stable,reliable,and reproducible.The study has laid a foundation for the establishment of a standard method to extract genomic DNA from young leaves of tea.
DNA extraction;tea;methodology;stability analysis
A
1674-5124(2015)09-0047-04
2015-03-09;
2015-04-17
国家科技支撑计划项目(2008BAK41B01-7);四川省科技支撑计划项目(2009NZ0015)
唐祥凯(1986-),男,四川成都市人,硕士,主要从事茶树生物技术研究。
谭和平(1957-),男,重庆市人,研究员,享受国务院政府津贴专家,从事生物化学茶产业链研究。
