基于DNA链置换的分子逻辑门计算模型
2015-07-21张文逸殷志祥
张文逸++殷志祥

摘要:DNA计算是近年来的研究热点,分子逻辑门是DNA计算机体系结构和运算实现的重要基础。将DNA自组装与链置换技术和荧光标记相结合,在现有的链置换逻辑计算模型的基础上,构造了非门,与门,与非门,或门和或非门。可在室温下进行,减少了因复杂的生物操作步骤带来的误差。使用荧光检测来判断逻辑结果,操作简单,容易检测,且灵敏度高。
关键词:DNA计算;自组装;链置换;荧光标记;逻辑门
中图分类号:TP301.6 文献标志码:A
文章编号:1672-1098(2015)01-0007-04
自从1946年科学家发明了第一台电子计算机ENIAC,计算机迅猛发展并时刻影响着人类的生活。但随着科技的进步,传统计算机已无法满足呈指数增长的大规模运算需求,加之“Moore定律”导致的芯片研究成本和半导体物理极限,集成电路的时代不可能永远下去。美国物理学家Feynman于1959年首次提出了分子计算机的思想[1]。20世纪90年代,美国加州大学的Leonard Adleman于1994年第一次在试管中通过利用寡核苷酸链进行实验,解决了经典的有向Hamilton路问题[2],首次从实验上证明了分子计算机的可行性。
分子逻辑门是实现分子计算机的基础。文献[3]首次提出布尔逻辑分子计算机模型,这是使用DNA分子模拟布尔电路终将实现以DNA为核心的分子计算机。文献[4]构建了DNA核酸分子逻辑非门、与门和异或门,文献[5]利用DNA核酶构建了多种DNA逻辑运算模型,文献[6]构建了半加器的复杂逻辑门,文献[7]首次构建了逻辑与门、与非门和禁门,文献[8]构建了一套完整的无酶逻辑电路,文献[9]构建了环状DNA逻辑门,文献[10]使用多种DNA酶构建逻辑运算模型。
近年来,DNA计算发展迅速[11],融合了荧光标记、链置换、自组装和纳米颗粒[12]等多种分子操作技术。其中,结合荧光标记的DNA链置换技术更是因在Science和Nature等杂志[13-15]上发表的工作而具有广泛的应用前景。本文在文献[16]的基础上,结合荧光标记技术[17-18],改进并构造了基本的逻辑门单元,以寡核苷酸链作为输入信号,荧光检测作为输出信号,能在室温下自发进行,对结果有更好的可读性。
1DNA自组装和链置换
11DNA自组装
DNA自组装是指一些带有输入信息的DNA分子根据碱基互补配对原则,在一定的温度、浓度、酸碱度以及特定酶的作用下,自组装生成带有输出信息的新的DNA分子的过程。自组装计算模型是通过DNA分子间的相互作用形成特定的构型来完成计算过程,是由Winfree等首次提出来的一种DNA计算模型[19-21]。文献[22]首先使用复杂DNA自组装结构实现了简单的逻辑运算。文献[23]将自组装DNA计算的基本思想用于求解布尔逻辑表达式并将其实现逻辑电路。该模型目前在生物计算中得到了良好的应用。
12链置换技术
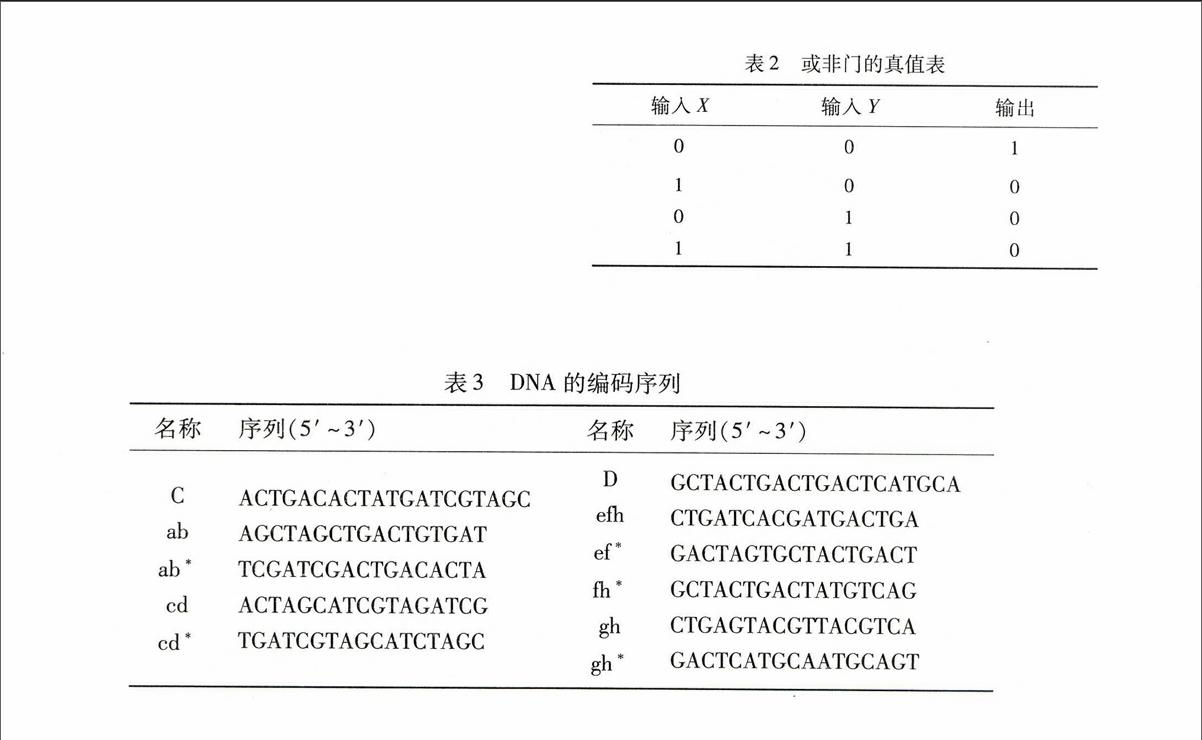
链置换技术是利用分子杂交系统趋向稳定能级的特点,通过加入不同长度和序列的输入链来诱导控制链置换反应,从而释放另一条DNA链的过程。DNA链置换的基本原理如图1所示。链A/B是由两条部分互补的DNA链结合成的部分双链,单链部分在反应中被称作立足点,发生链置换反应的可能性随立足点的增长而增大,链A*是序列与A完全互补的单链。单链A*先特异性识别结合立足点,为达到最稳态分支开始迁移,A*与A的碱基对逐渐取代B与A的碱基对,直至完全置换释放出B链。该反应过程具有自引发性,灵敏性和准确性等特点,近年来发展迅速,已成为分子计算领域的研究热点。
图1链置换反应的基本过程
2链置换逻辑门的计算模型
21非门
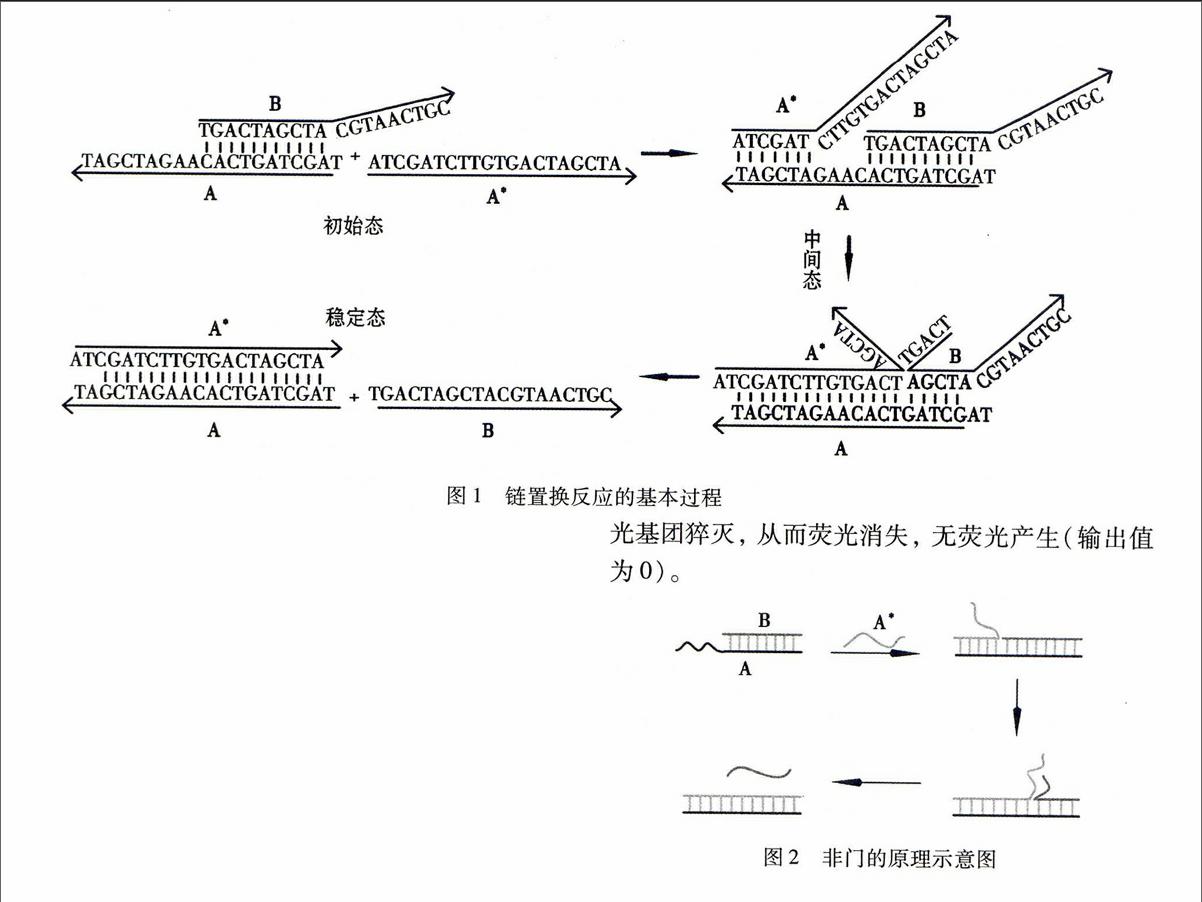
在反应底物自组装结构的部分双链中长链A的5′端标记荧光基团FAM,与A链完全互补的单链DNA链A*的5′端标记荧光猝灭基团DABCYL, 当无DNA链输入(输入值为0)时, 有荧光产生(输出值为1);当加入互补链A*(输入值为1)时, 因A链与A*链完全互补结合, 荧光猝灭基团将荧光基团猝灭, 从而荧光消失, 无荧光产生(输出值为0)。
图2非门的原理示意图
22与门
反应底物自组装结构由主链C和单侧信号识别链ab、cd杂交而成,主链C长20 bp,分为左右各长10 bp的碱基片断,信号识别链由长10 bp的结构区域b、c和长约6~7 bp的特异性识别位点a、b组成,输入链ab*和cd*为分别与ab和cd完全互补的信号链。对所需要的DNA序列进行编码后,在主链C的5′和3′端分别标记荧光基团FAM和ROX,在b的5′端和c的3′端分别标记荧光猝灭基团DABCYL。定义同时有两种不同的荧光产生时为输出1,否则为输出0。
当无信号输入(0,0)时,自组装结构无变化,无荧光产生(输出值为0);当输入信号1(1,0)时,ab*将ab完全置换,产生FAM荧光,但无ROX荧光(输出值为0);当输入信号2(0,1)时,cd*将cd完全置换,产生ROX荧光,但无FAM荧光(输出值为0);当同时输入信号1和2(1,1)时,ab*将ab完全置换,产生FAM荧光,cd*将cd完全置换,产生ROX荧光(输出值为1)。
23与非门
自组装结构同与门相同(见图3),对所需要的DNA序列进行编码后,在主链C的5′和3′端分别标记荧光基团FAM和ROX,在输入链ab*中b*的5′端和cd*中c*的3′端分别标记荧光猝灭基团DABCYL。有荧光产生时为输出1,无荧光则为0(见表1)。
图3与门的原理示意图
当无信号输入(0,0)时,自组装结构无变化,有荧光FAM和ROX产生(输出值为1);当输入信号1(1,0)时,ab*将ab完全置换,荧光猝灭基团将荧光FAM基团猝灭,FAM荧光消失,但有ROX荧光(输出值为1);当输入信号2(0,1)时,cd*将cd完全置换,荧光猝灭基团将荧光ROX基团猝灭,ROX荧光消失,但有FAM荧光(输出值为1);当同时输入信号1和2(1,1)时,ab*将ab完全置换, FAM荧光消失,cd*将cd完全置换,ROX荧光消失,无荧光(输出值为0)。endprint
表1与非门的真值表
输入X输入Y输出
001
101011110
24或门
反应底物自组装结构由主链D和多信号识别区域(e,f,g,h)组成。单种输入为混合输入信号,信号3为输入链(ef*,gh*),信号4为输入链(fh*,gh*)。对所需要的DNA序列进行编码后,在主链B的中间部位和3′端分别标记荧光基团FAM和ROX,在efh中f和gh中g的3′端分别标记荧光猝灭基团DABCYL。定义同时有两种不同的荧光产生时为输出1,无荧光为输出0。
当无信号输入(0,0)时,自组装结构无变化,无荧光产生(输出值为0);当输入信号3(1,0)时,ef*将efh置换成部分双链,产生FAM荧光,gh*将gh完全置换,产生ROX荧光(输出值为1);当输入信号4(0,1)时,fh*将efh置换成部分双链,产生FAM荧光,gh*将gh完全置换,产生ROX荧光(输出值为1);当同时输入信号3和4(1,1)时,ef*和fh*将efh置换成部分双链,产生FAM荧光,gh*将gh完全置换,产生ROX荧光(输出值为1)。
25或非门
自组装结构同或门相同(见图4),对所需要的DNA序列进行编码后,在主链D的中间部位和3′端分别标记荧光基团FAM和ROX,在ef*的5′端、fh*中f和gh*中g的3′端分别标记荧光猝灭基团DABCYL。定义同时有两种不同的荧光产生时为输出1,否则为输出0(见表2)。
图4或门的原理示意图
当无信号输入(0,0)时,自组装结构无变化,有荧光FAM和ROX(输出值为1);当输入信号3(1,0)时,ef*将efh置换成部分双链,荧光猝灭基团将荧光FAM基团猝灭,FAM荧光消失,gh*将gh完全置换,荧光猝灭基团将荧光ROX基团猝灭,ROX荧光消失(输出值为0);当输入信号4(0,1)时,fh*将efh置换成部分双链,荧光猝灭基团将荧光FAM基团猝灭,FAM荧光消失,gh*将gh完全置换,荧光猝灭基团将荧光ROX基团猝灭,ROX荧光消失(输出值为0);当同时输入信号3和4(1,1)时,ef*和fh*将efh置换成部分双链,荧光猝灭基团将荧光FAM基团猝灭,FAM荧光消失,gh*将gh完全置换,荧光猝灭基团将荧光ROX基团猝灭,ROX荧光消失(输出值为0)(见表3)。
表2或非门的真值表
输入X输入Y输出
001
101011000
表3DNA的编码序列
名称序列(5′~3′)名称序列(5′~3′)
Cabab*cdcd*ACTGACACTATGATCGTAGC
AGCTAGCTGACTGTGATTCGATCGACTGACACTA
ACTAGCATCGTAGATCGTGATCGTAGCATCTAGCDefhef*fh*ghgh*
GCTACTGACTGACTCATGCACTGATCACGATGACTGA
GACTAGTGCTACTGACTGCTACTGACTATGTCAG
CTGAGTACGTTACGTCAGACTCATGCAATGCAGT
3结论
本文以现有的链置换逻辑计算模型为基础,结合荧光标记技术,用荧光检测作为输出信号,并对输入信号进行相应的标记,实现了非门,与门,与非门,或门和或非门的逻辑门操作。该逻辑门可在室温下自发反应,减小了实验误差。在读取逻辑结果时,通过荧光的出现情况来判断逻辑真值,操作简单,容易检测,且灵敏度高。但是对于不同的逻辑门,需要对自组装结构进行不同的设计,且在实验过程中,复杂体系的DNA序列设计难度增加,可能会出现碱基错配的现象,需要进一步进行实验验证和改进。
参考文献:
[1]FEYNMAN R P. Miniaturization[J]. Reinhold, 1961: 282-296.
[2]ADLEMAN L M. Molecular computation of solutions to combinatorial problems[J].Science,1994,266(5 187):1 021-1 024.
[3]OGIHARA M,RAY A.Simulating Boolean circuits on a DNA computer[J].Algorithmica 1999,25(2-3):239-250.
[4]STOJANOVIC MN,MITCHELL TE,STEFANOVIC D.Deoxyribozyme based logic gates[J]. Am Chem Soc,2002, 124(14):3 555-3 561.
[5]MILLIAN N S,DARKO S.A deoxyribozyme-based molecular automaton[J]. Nat Biotechnol, 2003,21:1 069-1 074.
[6]STOJANOVIC MN,STEFANOVIC D. Deoxyribozyme based half-adder[J]. Am Chem Soc,2003,125(22):6 673-6 676.
[7]SAGHATELIAN A,VOLCKER NH,GUCKIAN KM,et al. DNA-based photonic logic gates: AND,NAND,and INHIBIT[J].Am Chem Soc,2003,125(2):346-347.
[8]SEELIG G,SOLOVEICHIK D,ZHANG DY,et al. Enzyme-free nucleic acid logic circuits[J].Science,2006,314:1 585-1 588.
[9]ZHANG C,YANG J,XU J.Circular DNA logic gates with strand displacement[J].Langmuir,2010, 26(3):1 416-1 419.
[10]ELBAZ J,LIOUBASHEVSKI O,WANG F,et al. DNA computing circuits using libraries of DNAzyme subunits[J]. Nat Biotechnol, 2010, 5: 417-422.
[11]万菲,董晨,杨静,等. DNA 计算技术的发展与应用[J].中国科学院院刊,2014,29(1):94-105.endprint
