靶向膜型1基质金属蛋白酶反义肽的虚拟筛选与分子模拟
2015-07-19曾丽谭博文杨亚蓝邱槿怡熊莉丽茆灿泉
曾丽,谭博文,杨亚蓝,邱槿怡,熊莉丽,茆灿泉
靶向膜型1基质金属蛋白酶反义肽的虚拟筛选与分子模拟
曾丽,谭博文,杨亚蓝,邱槿怡,熊莉丽,茆灿泉
西南交通大学生命科学与工程学院分子进化与应用生物学实验室,四川成都 610031

曾丽, 谭博文, 杨亚蓝, 等. 靶向膜型1基质金属蛋白酶反义肽的虚拟筛选与分子模拟. 生物工程学报, 2015, 31(2): 269–280.Zeng L, Tan BW, Yang YL, et al. Virtual screening and molecular simulations of antisense peptides targeting MT1-MMP. Chin J Biotech, 2015, 31(2): 269–280.
膜型1基质金属蛋白酶 (Membrane type-1 matrix metalloproteinase, MT1-MMP, MMP14) 在肿瘤的发生发展及转移中起着重要作用,是肿瘤潜在理想的药物靶标。为了获得MT1-MMP结合肽,我们首先采用生物信息学方法分析MMPs序列,获得MT1-MMP差异大且特异的序列。以此为正义肽靶标,设计反义肽库,然后通过分子对接、分子动力学模拟以及体外细胞实验等多种方法,进行靶向MT1-MMP反义肽的筛选与活性研究。多序列比对确定了位于MT1-MMP环区的特异序列AYIREGHE (简称MT1-loop),并构建1 536条反义肽。经两轮虚拟筛选,选取打分位于前五的反义肽用于后续研究。该五条反义肽与MT1-MMP存在较强的相互作用且能很好地对接于正义肽区域。进一步分析其与MMPs其他家族成员 (MMP1-3, MMP7-13, MMP14 HPX, MMP16) 的亲和力,发现反义肽FVTFPYIR对MT1-MMP具有更强的特异性。分子动力学模拟表明,反义肽FVTFPYIR可能是通过影响受体MT1-MMP的构象稳定性,进而影响其功能活性。体外细胞实验初步确定反义肽FVTFPYIR可选择性地抑制表达MT1-MMP的人成骨肉瘤细胞MG63和乳腺癌MDA-MB-231细胞的增殖。本研究为抗肿瘤反义肽先导药物的研发提供了一种新的思路与途径。
膜型1基质金属蛋白酶 (MT1-MMP,MMP14),反义肽,分子对接,分子动力学模拟
基质金属蛋白酶 (Matrix metalloproteinases, MMPs) 属于锌离子依赖性內肽酶家族成员,参与体内多种生理和病理过程。研究显示,MMPs的过量表达在肿瘤的浸润、转移和新生血管形成中起着重要的作用,与癌细胞的转化、生长和信号传递等密切相关。作为MMPs家族的重要成员之一,MT1-MMP能够直接或间接降解细胞外基质中的多种成分如纤连蛋白、层粘蛋白、Ⅰ型和Ⅱ型胶原[1],并调控许多细胞粘附和信号受体功能。它在多种肿瘤细胞中高度表达,促进肿瘤细胞在体内外迁移、浸润和转移,同时MT1-MMP的表达对肿瘤细胞在3D胶原基质中生长至关重要的。MT1-MMP具有MMPs成员的典型结构:前肽结构区、催化结构域、铰链结构域和血红素样结构域 (HPX)[2-3],同时具有跨膜区、胞质尾端以及furin蛋白识别等位点。其中催化结构域是MT1-MMP致癌特性最重要的区域[4],被认为是恶性肿瘤适宜的药物靶点而成为国内外研究的热点[5-6]。随着HPX[4,7-10]、跨膜胞质区[11]等其他区域作用功能的逐渐阐明,MT1-MMP在整个肿瘤发生、发展中的重要作用被更加确定。
反义肽即DNA反义链编码的多肽,如同DNA正义链和反义链那样,其与正义肽也存在相互作用。目前有Mekler-Idlis (M-I) 配对理论、AHBs (反义同源盒) 理论和分子识别理论对其作用原理和机制进行了较为详细的描述[12],其相互作用主要是疏水性、静电引力、氢键。反义肽和正义肽之间特殊的作用模式,已被用于多个领域,如Jackson等用血管紧张素Ⅱ的反义肽证实了反义肽的药物功能。作为一种新型多肽先导药物来源,反义肽在生物医药等领域具有重要的研发前景。
国内外有大量的MMPs抑制剂 (Matrix metalloproteinase inhibitors, MMPIs) 研究报道,但大部分的MMPIs被扼杀在Ⅱ、Ⅲ临床阶段[13],其主要原因是MMPs家族序列同源性高、功能区域三级结构相似,所研发的MMPIs广谱作用而缺乏选择性,以致特异性低和毒副作用大。筛选和获得高选择性的MMPIs已成为该领域研发的关键。本研究以MT1-MMP膜表面蛋白为对象,采用生物信息学与基于反义肽理论的虚拟筛选技术,筛选与发现分子靶向MT1-MMP特异区域的结合肽,期望为靶向MT1-MMP抗肿瘤反义肽先导药物的研发提供一种新的思路与途径。
1 材料与方法
1.1 材料及数据来源
MMPs蛋白序列下载自NCBI蛋白数据库 (http://www.ncbi.nlm.nih.gov/protein/),MMPs三维结构下载自PDB数据库 (http://www.rcsb.org/ pdb/home/home.do),蛋白序列及三维结构信息见表1;人成骨肉瘤MG63、乳腺癌MCF7为本实验室保存,人乳腺癌MDA-MB-231和MDA-MB-453细胞为成都医学院公共卫生系余小平教授惠赠;FITC标记和不标记的多肽由上海强耀生物科技有限公司合成 (纯度99%);细胞培养基DMEM粉末和胎牛血清购自Gibco公司;MTT购自Amresco公司;其他试剂均购自成都试剂公司,化学试剂为分析纯。
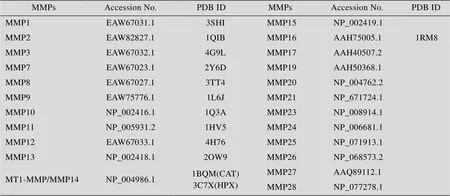
表1 MMPs三维结构信息
1.2 序列分析与反义肽设计
在NCBI数据库中下载所有人类MMPs (MMP1-3、MMP7-17、MMP19-21、MMP23-28) 等23种已知的蛋白质序列,利用Clustal X 1.81软件进行序列比对及多重序列比对,获得与MMPs其他家族成员差异性大、MT1-MMP特异的多肽序列。以该特异序列为正义肽,根据Meker-Idlis (M-I) 理论,设计相应的反义肽,运用Discovery Studio (DS) 2.0软件中的Protein Modeling模块构建反义肽的三维结构,采用CHARM力场进行结构优化。
1.3 虚拟筛选
为获得MT1-MMP特异性结合的反义肽,利用DS2.0中的Libdock模块和Molegro Virtual Docker (MVD) 分子对接软件进行两轮筛选。 Libdock初筛以MT1-MMP催化域为受体,以构建的反义肽库为配体,正义肽为对接中心,半径16.5 Å,每次保留10个构象,配体构象产生方法为best,Hotspots为150,其他参数默认,对接结果以Libdockscore排序。第二轮采用MVD重筛,使用半柔性对接,对接算法采用MolDock Optimizer,每个配体运行10次,每次最大迭代5 000步,对接结果以Rerankscore排序。
1.4 反义肽与其他MMPs成员分子对接
为了确定虚拟筛选获得的反义肽对MT1-MMP的特异性,以经上述两轮筛选获得的反义肽为配体,利用MVD软件对PDB数据库中包括MT1-MMP的HPX结构域在内的已解析三维结构的MMPs成员进行分子对接研究,对接方法参照1.3。
1.5 空载体与复合物的分子动力学模拟
分子动力学模拟和结果处理分别采用NAMD和VMD1.9.1软件。蛋白质-反义肽溶质化采用边长为70 nm的立方体,加入0.15 mmol/L的NaCl。先限制蛋白及反义肽,进行能量最小化 (10 000步,步长2 fs) 和能量平衡 (200 000步,步长2 fs),然后允许蛋白、反义肽和水分子同时运动,再进行能量最小化 (10 000步,步长2 fs) 和能量平衡 (2 500 000步,步长2 fs)。分子动力学模拟采用CHARM力场,使用恒温310 K和常压1×105Pa。
1.6 体外肿瘤细胞活力测定
MG63、MDA-MB-231、MCF7、MDA-MB-453细胞复苏,常规培养,以5×103个细胞/孔的浓度铺于96孔板中培养过夜,加入不同浓度的合成肽 (0.1、1、10、100 µg/mL,=6) 继续培养,24 h后多肽换液一次,48 h后进行细胞形态学观察与MTT测定,方法参照文献[14]进行。
1.7 合成肽体外细胞亲和力测定
选用MDA-MB-231和MCF7细胞,以5×103个细胞/孔的浓度铺于96孔板中培养过夜,加入不同浓度的FITC标记的多肽 (0.1、1、10、50、100 µg/mL,=5) 继续培养,24 h后荧光观察后换多肽液一次,48 h再进行观察。
2 结果与分析
2.1 MT1-MMP特异的多重序列分析及反义肽设计
对23种已知的人MMPs家族成员进行多重序列比对。根据比对结果,以MT1-MMP为研究对象,选取与其他MMPs家族成员序列差异性相对较大的AYIREGHE (简称MT1-loop) 作为MT1-MMP的特异序列(图1),该序列位于MT1-MMP的催化结构域。以此为正义肽,运用Meker-Idlis (M-I) 理论,设计反义肽,共获得1 536条反义肽(表2)。

图1 ClustalX多序列比对人MMPs序列
2.2 虚拟筛选及反义肽与MMPs的分子对接
Libdock对接打分具有速度快和能够并行运算等特点,广泛用于大规模的分子对接模拟与快速筛选;MVD则可根据配体准确预测大分子蛋白的活性位点,是一款精确的半柔性分子对接软件,适合于二次筛选或精筛。经过Libdock第一轮筛选,共获得96条LibDockScore高于200的反义肽,再将其进行MVD第二轮筛选,Rerankscore分值前5位的反义肽分别为FVTFPYIR、LVSFPYVR、FVTFSYVG、FMAFSYVS和FVTLPYVR,后者对接结果和参数见图2和表3。

表2 基于Meker-Idlis (M-I) 理论设计反义肽
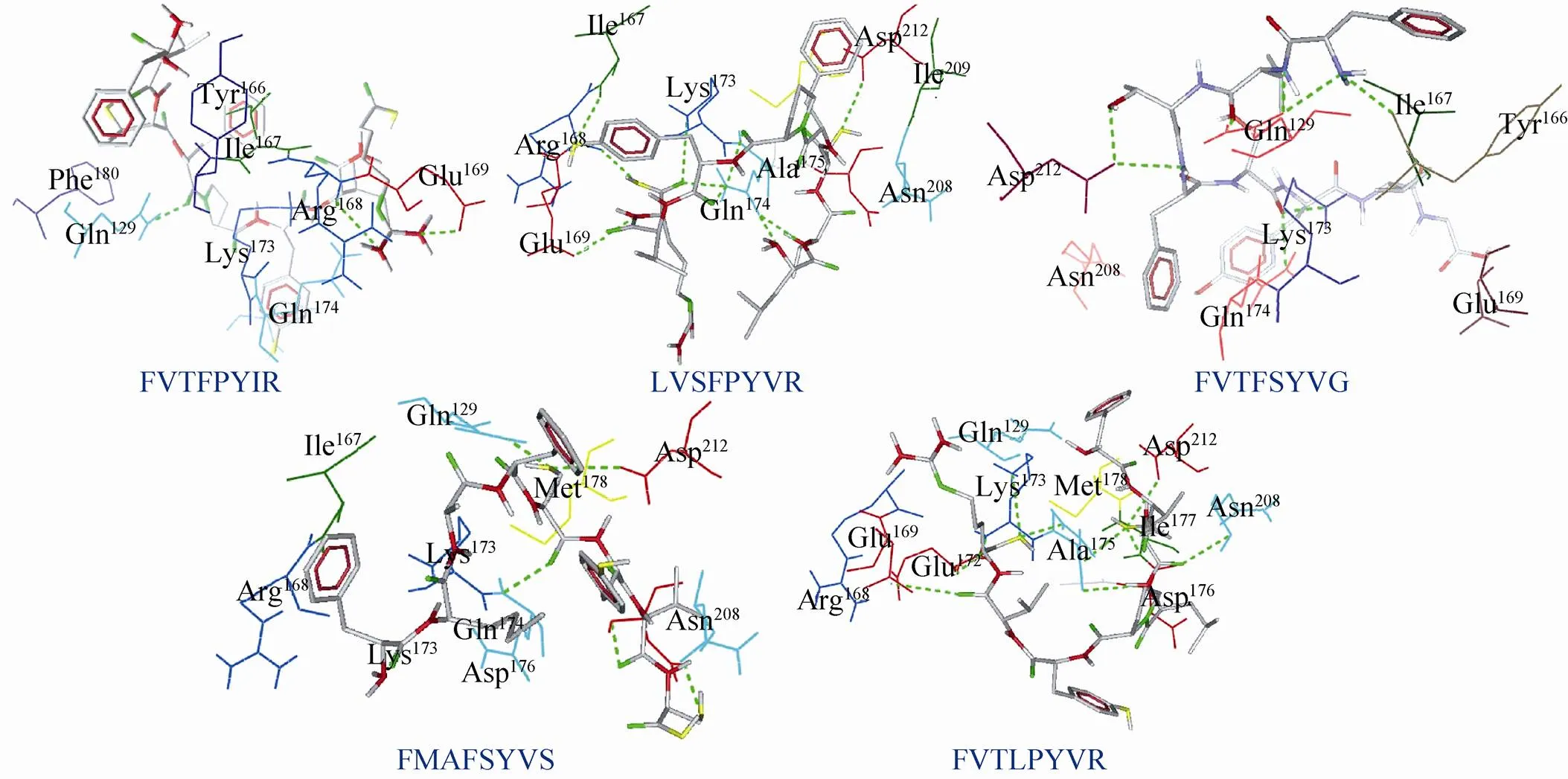
图2 反义肽与MT1-MMP分子对接图
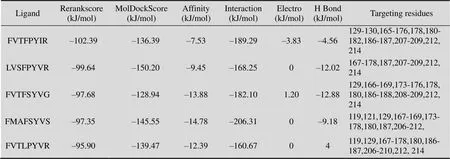
表3 反义肽与MT1-MMP催化域的对接参数
以反义肽FVTFPYIR与MT1-MMP催化域对接结果为例,图3展示两者间的相互作用方式。首先,反义肽的Phe4与MT1-MMP的Gln129、Arg8与MT1-MMP的Arg168及Glu169形成3个稳定的氢键 (图3A);另一方面,反义肽位于Ala165、Ile167、Gly170和Phe180等氨基酸形成的疏水腔中,两者之间存在较强的疏水作用 (图3B);同时,该反义肽与受体间存在着较强的静电作用力 (表3,图3C)。综合分析,FVTFPYIR与MT1-MMP结合的主要驱动力是疏水作用力、氢键以及静电作用力。
与其他MMPs家族成员的分子对接结果表明,相对于其他4条反义肽,多肽FVTFPYIR特异性更好。除MMP13外,FVTFPYIR与其余的MMPs家族成员的对接能量均高于MT1-MMP (表4)。
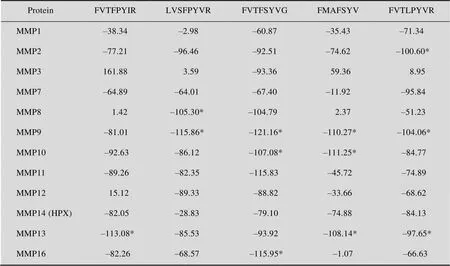
表4 反义肽与MMPs家族成员分子对接的Rerankscores值(kJ/mol)
* indicates the rerankscore was better than those docking to the catalytic of MT1-MMP.
2.3 反义肽与MT1-MMP的分子动力学模拟
为深入了解所确定的反义肽与MT1-MMP的作用模式,对每个复合物体系进行了5 ns的常规分子动力学模拟,用均方根偏差 (Root mean square deviation,RMSD) 评价体系的稳定性。图4显示动力学模拟过程中MT1-MMP和反义肽主链骨架的RMSD的变化情况,起初 0.5 ns,蛋白内部结构优化主链的RMSD上升较快。首先模拟MT1-MMP在水溶质中的稳定性,结果显示在1.7 ns后就达到平衡状态,RMSD约 0.35 nm (图4A)。而在MT1-MMP-FVTFPYIR复合物中,MT1-MMP的RMSD呈上升趋势,配体约在3.5 ns达到平衡状态 (图4B),这说明配体能够引起MT1-MMP的不稳定,推测MT1-MMP的功能活性受到影响。在其他模拟过程中发现,反义肽LVSFPYVR、FVTFSYVG能稳定MT1-MMP其RMSD均在0.25 nm左右,而反义肽自身的RMSD波动较大 (图4C–E);FVTLPYVR、FMAFSYVS体系在2 ns后均达到稳定 (图4F)。
2.4 反义肽对肿瘤细胞活力的抑制及亲和力
为了确认虚拟筛选获得的反义肽是否对肿瘤细胞真实有效,综合分子对接和分子动力模拟结果,选取反义肽FVTFPYIR作为体外细胞实验的候选肽。实验结果表明,用该反义肽作用肿瘤细胞 48 h后,加药组MG63细胞密度明显低于对照组(图5),MDA-MB-231也有着相似的变化,而MDA-MB-453和MCF7差异不明显,且MDA-MB-453实验组较对照组有相逆的趋势 (后三者图未显示);MTT测定显示,MG63细胞增殖受到明显抑制,随着多肽浓度的增大,抑制作用也增强,呈剂量依赖关系 (图6A),同样反义肽也能抑制MDA-MB-231细胞的增殖 (图6B);与之不同的是,反义肽对MCF7细胞活力几乎无剂量依赖抑制作用,对MDA-MB-453细胞在低剂量下促进其生长,高剂量下抑制其生长 (结果未显示)。
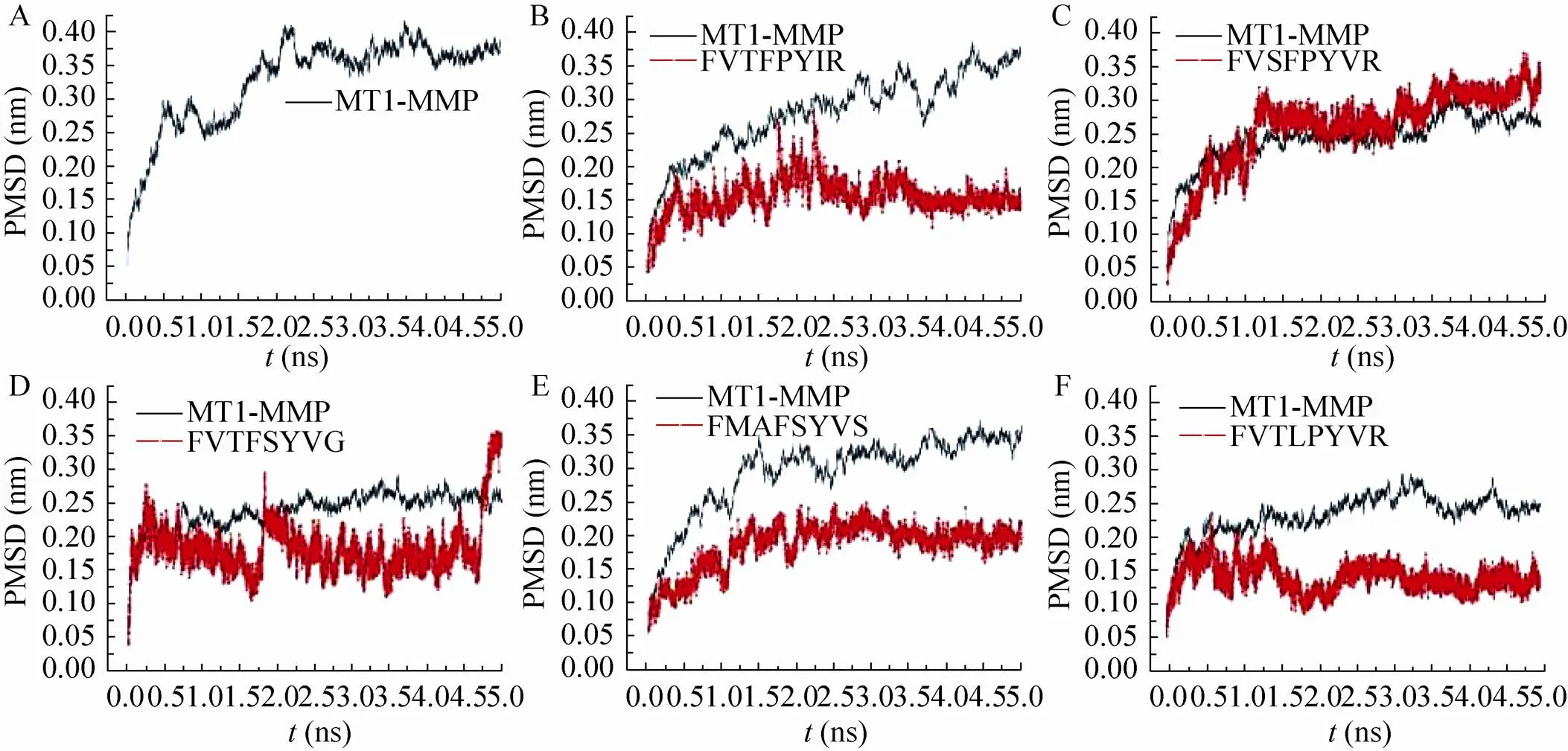
图4 MD模拟过程中MT1-MMP及反义肽主链骨架的RMSD值

图5 倒置显微镜下MG63细胞形态的变化(100×)
MDA-MB-231和MCF7细胞亲和力测定结果显示,培养24 h后,100 µg/mL的反义肽共培养的MDA-MB-231细胞检测到荧光信号;而培养48 h后,100 µg/mL和50 µg/mL的反义肽共培养的MDA-MB-231细胞均检测到荧光信号,浓度越大,荧光信号越强 (图7)。但无论培养24 h还是48 h,MCF7细胞均检测不到荧光。

图6 FVTFPYIR对肿瘤细胞活力的影响
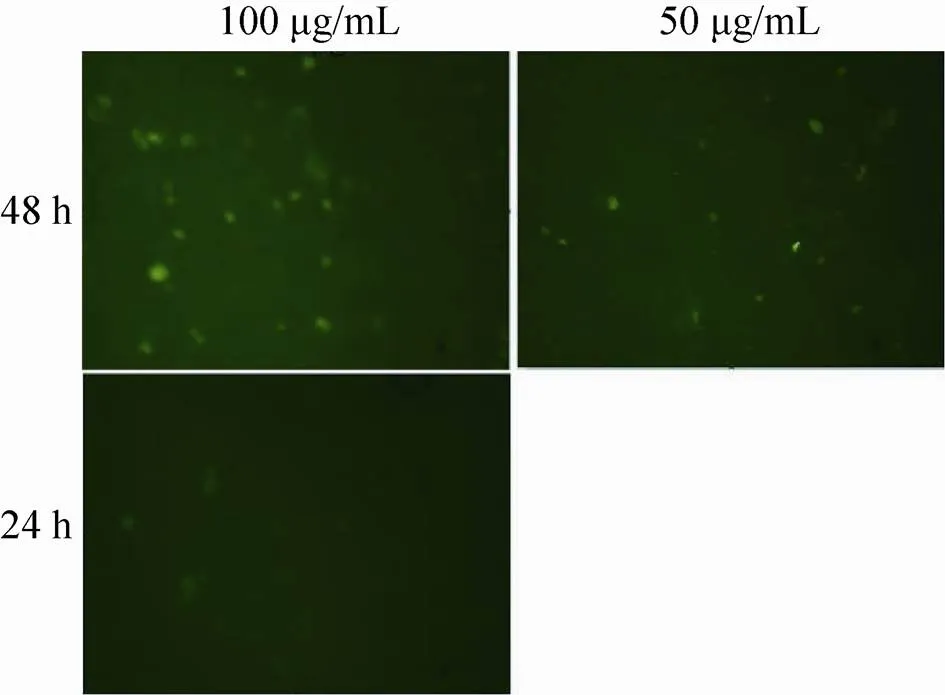
图7 FVTFPYIR与MDA-MB-231细胞亲和力检测结果(200×)
3 讨论
MMPs家族成员具有高度的同源性,加之MMPs各成员对机体作用不一,导致靶向MMPs抗肿瘤抑制剂研发十分困难。因此,寻找合适的MMPs家族成员以及序列及功能差异性大的区域作为其药物筛选的靶标是解决此问题的关键。MT1-MMP作为MMPs家族的重要成员,参与proMMP2的激活,直接或间接降解细胞外基质中的多种组分。一方面它是细胞表面膜蛋白,可以克服靶向胞内蛋白药物需要进入细胞的难点,同时其被认为是MMPs 家族中与肿瘤关系最密切的酶[15],因此,MT1-MMP是MMPs家族中最理想的抗肿瘤药物筛选靶蛋白之一[4]。不仅如此,在MT1-MMP的催化域上存在一段独特的序列MT-loop (aa.163–170),有研究表明,MT-loop对proMMP2有效激活是不可缺少的。综合考虑以上因素,我们选取MT1-MMP的特有序列MT1-loop为靶标进行反义肽库的虚拟筛选与研究。与国内房学讯实验室[6]开展的靶向MT1-MMP loop区 (aa.160–174)的噬菌体随机肽库筛选不同的是,本研究在多重序列比对分析的基础上,确定和选取的MT1-loop区序列更短既利于正义肽的选择和反义肽库的设计,差异性又更强。以MT1-loop作为研究靶标,有望获得MT1-MMP特异性的抑制剂和先导药物,同时因为MT1-loop位于MT1-MMP三维结构的表面,更利于药物作用的直接发挥。
多肽药物具有与靶蛋白结构相容性高、能有效破坏蛋白-蛋白作用界面以及分子量小等优点,加上近年在药物修饰、给药途径以及膜渗透等领域的突破,使得多肽药物又引起了极大的关注[16],2012年FDA批准了6个多肽药物上市便是很好的说明。自2005年始,本实验室致力于靶向MT1-MMP的多肽抑制剂筛选与研究并取得了一定的成效[10,13-14,17]。本研究运用反义肽原理设计靶向MT1-loop的反义肽库,依次采用Libdock和MVD分子对接软件进行虚拟筛选。综合两种软件的对接结果,获得高效可靠的目标反义多肽,最终选取了综合排名前五位的反义肽进一步的研究分析。首先从理论上考察其专一性,即采用分子模拟与对接分析反义肽与已解析三维结构的MMPs的作用特性,由于在PDB数据库中已解析的大多数MMPs三维结构并非全长,又基于正义肽位于MT1-MMP的催化域中,因此在选择MMPs三维结构时也主要将相应的催化域作为研究对象,其中MMP20三维结构太大,没有对其进行研究。结果显示FVTFPYIR特异性最好。其次通过动力学模拟研究复合物体系的稳定性,为此进行5 ns的常规动力学模拟。在分子动力学模拟过程中,小分子配体在模拟起初阶段RMSD上升较快,表明小分子比较灵活,在结合位点附近略显波动,并能周期性形成氢键进而使结合更加稳定[18];而后很快在0.15 nm左右波动 (除LVSFPYVR外),受体蛋白同样在起始阶段RMSD上升较快,这主要是蛋白内部结构优化造成[19],在0.25 nm和0.35 nm后我们也发现有两条多肽的RMSD值发生突变 (图4C,D),极有可能碳骨架发生断裂,不宜作为候选药物。令人欣喜的是,反义肽FVTFPYIR与受体结合后,能在影响受体构象稳定的同时而保持自身的稳定。
早有研究表明VEGF-C反义肽可抑制骨肉瘤细胞的增殖、侵袭转移能力以及抑制肿瘤组织VECFR-3阳性血管的形成,发挥抗骨肉瘤作用[20]。因此在以上工作的基础上,我们也初步探究了本研究筛选获得的反义肽对肿瘤细胞增殖的影响。依据反义肽的特异性和动力学模拟结果,我们合成了评价最优的反义肽FVTFPYIR,同时选取MT1-MMP表达的MG63、MDA-MB-231细胞和不表达的乳腺癌细胞MDA-MB-453、MCF7作为实验对象,结果表明该反义肽对MG6和MDA-MB-231细胞有很好的抑制作用,而对MDA-MB-453细胞几乎是促进生长,对MCF7细胞活力几乎无剂量依赖抑制作用。有研究报道,人工合成的十九肽[21]或者通过噬菌体文库筛选得到的短肽[22]能够促进轴突的生长,从另一角度佐证了我们合成的多肽在较低浓度下促进乳腺癌MDA-MB-453细胞生长的可能性。结合4种细胞的实验结果,我们推测这很可能与细胞是否表达MT1-MMP有关,该结论也与动力学模拟结果相得益彰。进一步的细胞亲和力测定结果也证明了反义肽对MT1-MMP表达的MDA-MB-231细胞的靶向结合能力。当然,更可靠的结论将有待于在MT1-MMP蛋白表达、酶蛋白活性以及信号转导等水平的检测与分析来进一步确认。
综上所述,本研究通过生物信息与计算机辅助药物设计的方法筛选与获得了5条能与MT1-MMP有效对接的反义肽,体外实验初步确认反义肽FVTFPYIR对MG63和MDA-MB-231细胞增殖有一定的抑制作用以及对后者的亲和力。进一步的多肽体外作用研究尚在进行中。本研究也为靶向MT1-MMP抗肿瘤反义肽先导药物的研发提供了一种新的思路与途径。
REFERENCES
[1] Seiki M. Role of MT1-MMP in tumor-stromal interaction//New Trends in the Molecular and Biological Basis for Clinical Oncology. New York: Springer, 2009: 86–91.
[2] Albert GR, Dmitri VR, Peter CB, et al. The transmembrane domain is essential for the microtubular trafficking of membrane type-1matrix metalloproteinase. J Cell Sci, 2005, 118(21): 4975–4984.
[3] Barbolina MV, Stack MS. Membrane type-l matrix metalloproteinase: substrate diversity in pericellular proteolysis. Semin Cell Dev Biol, 2008, 19(1): 24–33.
[4] Albert GR, Vladislav SG, Sergey AS, et al. Novel MT1-MMP small-molecule inhibitors based on insights into hemopexin domain function in tumor growth. Cancer Res, 2012, 72(9): 2339–2349.
[5] Cao J, Pallavi K, Maria P, et al. Distinct roles for the catalytic and hemopexin domains of membrane type-1 matrix metalloproteinase in substrate degradation and cell migration. J Biol Chem, 2009, 279(14): 14129–14139.
[6] Zhu L, Wang HL, Wang Y, et al. High-affinity peptide against MT1-MMP fortumor imaging. J Controlled Release, 2011, 150(3): 248–255.
[7] Basu B, de Sampaio PC, Mohammed P, et al. Inhibition of MT1-MMP activity using functional antibody fragments selected against its hemopexin domain. Int J Biochem Cell Biol, 2012, 44(2): 393–403.
[8] Kevin Z, Antoine D, Jian L, et al. Inhibition of matrix metalloproteinase 14 (MMP-14)-mediated cancer cell migration. J Biol Chem, 2011, 286(38): 33167–33177.
[9] Wang P, Nie J, Pei DQ. The hemopexin domain of membrane-type matrix metalloproteinase-1 (MT1-MMP) is not required for its activation of proMMP2 on cell surface but is essential for MT1-MMP-mediated invasion in three-dimensional type I collagen. J Biol Chem, 2004, 279(49): 51148–51155.
[10] Huang TT, Cui J, Liang ZJ, et al. Virtual screening of lead chemicals based on HPX domain of MT1-MMP. The 5th International Conference on Bioinformatics and Biomedical Engineering. USA: IEEE Computer Society, 2011: 1–4.
[11] Hidetoshi M, Alvin TL, Jamie LI, et al. Transmembrane/cytoplasmic, rather than catalytic, domains of MMP14 signal to MAPK activation and mammary branching morphogenesis via binding to integrin β1. Development, 2013, 140(2): 343–352.
[12] Wang LH, Bao CY, Shi W, et al. Protein codes: the interaction between sense peptides and antisense peptides. Chem Life, 2003, 23(1): 31–34 (in Chinese).王梁华, 包晨颖, 史薇, 等. 蛋白质密码—有义多肽与反义多肽间的相互作用. 生命的化学, 2003, 23(1): 31–34.
[13] Liang ZJ, Huang JS, Cui J, et al.Selection and molecular simulation of binding peptides dual-targeting MMP14 and metal ions. Chin J Biochem Mol Biol, 2011, 27(4): 341–349 (in Chinese).梁志娟, 黄敬双, 崔健, 等. 双靶向MMP-14和金属离子的结合肽筛选与分子模拟. 中国生物化学与分子生物学报, 2011, 27(4): 341–349.
[14] Liang ZJ, Huang JS, Huang TT, et al. Selection and finding of lead peptides dual-targeting MMP14 and metal ions by subtractive cell surface panning and molecular docking, Int J Pept Res Ther, 2012, 18(1): 31–40.
[15] Sounni NE, Noel A. Membrane type-matrix metalloproteinases and tumor progression. Biochimie, 2005, 87(34): 329–342.
[16] Peter V, Almer MS, Erik V, et al. Computational design of peptide ligands. Trends Biotechnol, 2011, 29(5): 231–239.
[17] Zeng L, Tan BW, Mao CQ. Design and molecular docking of antisense peptides targeting MT1-MMP. The 9th National Conference of the Chinese Society of Genetics and the Abstracts of Academic Symposium (http://cpfd.cnki.com.cn/ Article/CPFDTOTAL-ZGYL201309001150.htm), 2013: 180 (in Chinese).曾丽, 谭博文, 茆灿泉. 肿瘤靶向MT1-MMP反义肽的筛选与分析. 中国遗传学会第九次全国会员代表大会暨学术研讨会论文摘要汇编(http://cpfd.cnki.com.cn/Article/CPFDTOTAL-ZGYL201309001150.htm), 2013: 180.
[18] Li B, Zhou R, He G, et al. Molecular docking, QSAR and molecular dynamics simulation on spiro-oxindoles as MDM2 inhibitors. Acta Chim Sin, 2013, 71(10): 1396–1403 (in Chinese).李博, 周锐, 何谷, 等. 螺环吲哚类MDM2抑制剂的分子对接、定量构效关系和分子动力模拟. 化学学报, 2013, 71(10): 1396–1403.
[19] Lü J, Jiang YJ, Yu QS, et al. Molecular docking and molecular dynamics simulations of inhibitors binding to jack bean urease. Acta Chim Sin, 2013, 69(20): 2427–2433 (in Chinese).吕婧, 蒋勇军, 俞庆森, 等. 洋刀豆脲酶与抑制剂相互作用的分子对接和分子动力学研究. 化学学报, 2013, 69(20): 2427–2433.
[20] Luo SM, Xia YX. Studying on the cellular biologic behavior effects of human antisense-VEGF gene C antisense polypeptide to human osteosarcoma. J Clin Exp Med, 2008, 7(2): 17–19 (in Chinese).罗胜明, 夏永新. 血管内皮生长因子-C反义肽对人成骨肉瘤细胞生物学行为影响的研究. 临床和实验医学杂志, 2008, 7(2): 17–19.
[21] Ken-ichiro T, Gregory CS,Benjamin W, et al. A synthetic peptide containing the IKVAV sequence from a chain of laminin mediates cell attachment, migration, and neurite outgrowth. J Biol Chem, 1989, 264(27): 6174–16182.
[22] Deng QY, Cai WQ, Li SR, et al. Small nogo-66-binding peptide promotes neurite outgrowth through RhoA inhibition after spinal cord injury. Brain Res Bull, 2013, 99: 140–144.
(本文责编陈宏宇)
Virtual screening and molecular simulations of antisense peptides targeting MT1-MMP
Li Zeng, Bowen Tan, Yalan Yang, Jinyi Qiu, Lili Xiong, and Canquan Mao
Laboratory of Molecular Evolution and Applied Biology, School of Life Science and Engineering, Southwest Jiaotong University, Chengdu 610031, Sichuan, China
Membrane type-1 matrix metalloproteinase (MT1-MMP or MMP14) plays the pivotal role in tumor development and metastasis, so it is a promising drug target in malignancy. To acquire MT1-MMP specific binding peptides, we first analyzed MMPs sequences to find the divergent and specific sequence of MT1-MMP by bioinformatics approach, then set the specific sequence as the sense peptide target and designed antisense peptide library. Finally, by means of molecular docking, molecular dynamics simulation andcell assays, we screened the antisense peptide library against MT1-MMP and further studied the obtained specific peptides. Here, we identified the divergent and specific sequence of AYIREGHE (Named MT1-loop) located in MT1-MMP loop by multiple sequence alignment and established the antisense peptides library with capacity of 1 536 sequences. After two rounds of virtual screening, we obtained five antisense peptides with Rerankscores in the top for further screening. They all interacted with MT1-MMP, and docked well at the active site composed of MT1-loop sequence. Analysis of the affinities of these five antisense peptides to other MMPs (MMP1-3, MMP7-13, MMP14 HPX, MMP16) revealed that the peptide FVTFPYIR was more specific to MT1-MMP. Molecular dynamics simulation showed that the peptide FVTFPYIR might affect the stability of MT1-MMP and thus have effects on its activities. Meanwhile, the peptide FVTFPYIR could specifically inhibit the growth of MG63 and MDA-MB-231 tumor cells both of which expressed MT1-MMP. The work provides a new insight and way for the development of antitumor lead peptides targeting MT1-MMP.
membrane type-1 matrix metalloproteinase (MT1-MMP, MMP14), antisense peptide, molecular docking, molecular dynamics simulation
April 2, 2014; Accepted: May 7, 2014
Canquan Mao. Tel: +86-28-87634296; E-mail: maocq@swjtu.edu.cn
Supported by: the Fundamental Research Funds for the Central Universities of China (No. SWJTU09ZT28), National High Technology Research and Development Program of China (863 Program) (No. 2006AA06Z353).
中央高校基本科研业务费专题研究项目 (No. SWJTU09ZT28),国家高技术研究发展计划 (863计划) (No. 2006AA06Z353) 资助。
网络出版时间:2014-05-28
http://www.cnki.net/kcms/doi/10.13345/j.cjb.140199.html
