半红树植物水黄皮分子生物学研究进展
2015-07-19黄健子张万科黄荣峰郑易之
黄健子,张万科,黄荣峰,郑易之
半红树植物水黄皮分子生物学研究进展
黄健子1,张万科2,黄荣峰3,郑易之1
1 深圳大学生命科学学院,广东深圳 518060 2 中国科学院遗传与发育生物学研究所,北京 100101 3 中国农业科学院生物技术研究所,北京 100081

黄健子, 张万科, 黄荣峰, 等. 半红树植物水黄皮分子生物学研究进展. 生物工程学报, 2015, 31(4): 461–468.Huang JZ, Zhang WK, Huang RF, et al. Progress in molecular biology of a semi-mangrove, Millettia pinnata. Chin J Biotech, 2015, 31(4): 461–468.
水黄皮L. 是豆科的一种具有巨大发展潜力的生物柴油能源树种,同时也是一种典型的半红树植物。文中综述了近年来国内外关于水黄皮分子生物学方面的研究进展,重点包括基于分子标记的水黄皮遗传多样性和系统发育研究,基于高通量测序技术的水黄皮基因组和转录组研究,以及水黄皮基因和基因组片段的分离及相关分析;最后还结合当前研究的现状,展望了水黄皮未来研究工作的发展前景。
水黄皮,分子标记,基因组,转录组,油脂代谢,盐胁迫
水黄皮L.为多年生中型木本植物,隶属于豆科Fabaceae蝶形花亚科水黄皮属,在印度被称为卡兰贾树Karanj,在我国民间又名水流豆或九重吹。该物种原产于印度次大陆及东南亚,后被引种到中国、澳大利亚、新西兰和美国等国家的湿热气候地区。在我国,水黄皮主要分布于广东、广西、海南和台湾四省,是一种典型的半红树植物,通常生长在海岸或沿河的滩涂地区,亦可作为庭园树或行道树栽培于内陆地区,具有耐热、耐旱、耐盐碱、抗风、抗污染和易移植等特性。水黄皮树高8−10米,主根可延伸至地下10米;叶互生,为一回羽状复叶;花呈白色或淡紫色,主要通过昆虫进行异花授粉;果实初为绿色,成熟后变为褐色,内含1−2粒种子。
近年来,水黄皮作为具有巨大发展潜力的非粮(Non-edible) 油料植物之一,吸引着越来越多的研究者关注[1-2]。从水黄皮种子中提取油脂用于制备生物柴油的报道最早见于印度[3],其后有关该物种制备生物柴油的可行性得到多项研究证明[4-6]。水黄皮作为生物质能源树种的优势主要体现在以下几个方面:其一,种子具有较高含油量,且其结实率高,单位面积的年产油量远高于玉米、大豆、芥花和麻疯树等物 种[7-8];其二,种子油的脂肪酸组成中,油酸比率较高,由其所制备的生物柴油具有较低的浊点和流点,有利于提升发动机性能[9-11];其三,根部的根瘤菌可进行生物固氮,在减少氮肥施用的同时亦可降低其所制备生物柴油在燃烧过程中含氮温室气体的排放量[2,8,12];其四,植株具有耐盐和耐旱的特点[8,13],可种植于土壤贫瘠地区而“不与粮争地”,还可作为防风林抵御风暴及保护海岸生态系统。此外,水黄皮植株各部分均具有不同的经济价值和药用价值,这将为其生物柴油产业化发展增添不少附加值[14]。
利用各种分子生物学手段对水黄皮开展种质资源调查、功能基因发掘及品种遗传改良等研究工作,不仅可以为水黄皮资源保护策略的制定提供有力依据,也将为其大规模推广种植及产业化制备奠定坚实基础。基于此,本文综述了近年来国内外有关水黄皮遗传多样性分析、基因组学和转录组学研究以及功能基因分析等方面的研究进展,并结合当前研究的不足探讨了该物种未来分子生物学研究的方向。
1 分子标记和遗传多样性分析
现有的水黄皮种质资源研究工作主要基于表型(性状) 标记和分子标记两个方面。与前者相比,分子标记具有信息量大、重复性好、不受环境和取材部位影响等优点,可应用于遗传多样性分析、遗传图谱构建、标记辅助选择和资源评价等方面。此外,分子标记在进行水黄皮良种选育时的优势还在于其可对幼苗期植株进行筛选,而不必等到植株挂果期(一般需要5年)。迄今已在水黄皮中应用的分子标记技术包括:随机扩增多态性DNA (Random amplified polymorphic DNA,RAPD) 标记、简单重复序列区间(Inter simple sequence repeat,ISSR) 标记和扩增片段多态性(Amplified fragment length polymorphism,AFLP) 标记。
最早运用于水黄皮遗传多样性分析的是ISSR标记。Sahoo等[15]采用10组ISSR引物对来自印度奥里萨邦7个水黄皮种群的226个样品进行遗传多样性分析,结果显示所选样品具有中等的遗传变异水平,种群间遗传分化与地理距离之间存在正相关。Sujatha等[16]同样采用ISSR标记对来自印度两个水黄皮种群的10个品种进行分析却揭示较高的遗传变异水平,然而该研究并未在具有高含油量的品种中检测到特征条带。Jiang等[17]基于高通量测序所得基因组序列结果设计出10组PISSR引物,对来自澳大利亚和马来西亚的29个品种进行研究,也发现品种间存在较高的遗传变异,而遗传相似度与地理位置间并无显著相关性。
在水黄皮遗传多样性研究中运用较多的另一种分子标记是AFLP技术。Thudi等[18]采用5组AFLP引物对印度6个邦48个种子含油量差别较大的水黄皮样品进行研究,获得了较多的多态性位点,并发现其中卡纳塔卡邦样品的遗传多样性明显高于另外几个邦。Sharma等[19]运用AFLP和三酶切TE-AFLP标记分析20个印度水黄皮品种的遗传多样性,发现两种标记都可以有效用于水黄皮种质评价研究,且后者比前者更适于样品数量较大的研究。Pavithra等[20]近期采用11组AFLP引物对印度33个水黄皮品种的研究也检测到较高的遗传变异水平。Kesari等[21-22]综合运用RAPD、ISSR和AFLP标记对印度 10个水黄皮品种的遗传多样性分析表明3种分子标记均可有效区分不同品种,且AFLP标记在重复性、稳定性和信息量方面要优于另外两种标记。
总的来说,尽管采用的引物组合不多,上述分子标记研究还是揭示水黄皮种内存在较高的遗传变异,这与其异花授粉的繁殖特点是相符的。然而,这些研究所选取的水黄皮样品来源较为集中,取样地点多为印度境内,只有少量样品取自澳大利亚和马来西亚,而对我国水黄皮品种的研究工作尚未见报道(表1)。此外,这些研究所采用的分子标记基本上都是显性标记而非共显性标记,不能有效鉴别杂合子和纯合子,在对水黄皮这种异花授粉的物种进行遗传分析时会丢失一些信息。将分子标记运用于我国野生水黄皮种群遗传多样性研究,不仅有利于保护我国水黄皮种质和基因资源,也有助于筛选合适品种在我国沿海地区进行推广 种植。
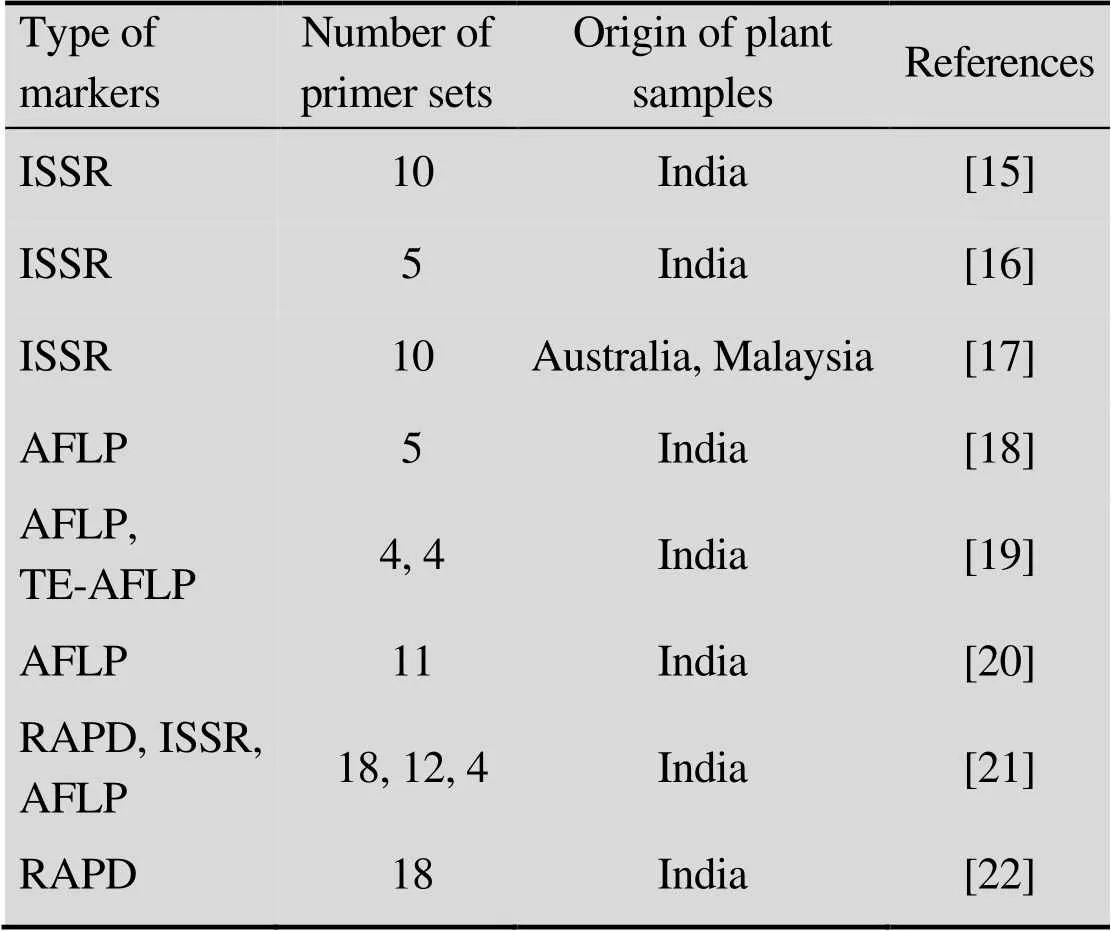
表1 水黄皮中开展的分子标记研究
2 基因组学和转录组学研究
2.1 基因组学
水黄皮是具有22对染色体的二倍体木本植物[23],其核基因组大小据估计约为1 300 Mb[24]。目前,来自澳大利亚昆士兰大学的Gresshoff研究团队正运用第二代高通量测序技术测定水黄皮核基因组序列,并已在NCBI网站上发布两组单端读长分别为36 bp和75 bp的短序列数据集,两者各包含了450 Mb和2.2 Gb数据。同时,这些基因组序列数据也被收录到TAGdb网站(http://flora.acpfg.com.au/tagdb/),该网站主要利用一些非模式植物全基因组测序时生成的短序列进行基因和启动子的发掘[25]。现有的水黄皮核基因组测序结果已经被成功应用于发掘油脂代谢相关基因及设计ISSR标记引物[8,17]。
水黄皮通过叶绿体捕获的太阳光能,经过线粒体、质体和内质网等细胞器的代谢转化后,以油体的形式将能量贮存在种子里。叶绿体和线粒体这两个直接参与光合作用和能量代谢的细胞器代表着水黄皮所产出原油中生物质能量的源头。Gresshoff研究团队同样采用Illumina测序平台测定并注释了水黄皮叶绿体基因组(cpDNA) 和线粒体基因组(mtDNA)[26]。其中,水黄皮cpDNA全长152 968 bp,含有8个rRNA基因、37个tRNA基因和83个蛋白质编码基因,GC含量为34.8%。水黄皮cpDNA除了具有在豆科植物cpDNA中普遍存在的50 kb倒置区外,还包含一个该物种特有的6.5 kb小倒置区。水黄皮mtDNA全长425 718 bp,具有两组正向重复单元和两组反向重复单元,包含3个rRNA基因、24个tRNA基因、37个蛋白质编码基因及 7个假基因,GC含量为45.0%。总体而言,水黄皮cpDNA具有比其mtDNA更高的基因密度。此外,该研究还将水黄皮cpDNA和mtDNA中的120个蛋白质编码基因与百脉根及绿豆中的同源基因进行比较分析,发现后两个物种间的序列分歧度较大,而水黄皮跟这两个物种间的序列分歧度较小。
2.2 转录组学
作为一种半红树植物,水黄皮对土壤盐度有一个较宽的适应范围[27],而其并不像其他红树植物那样具有根部拒盐和叶片泌盐等形态生理特征。因此,该物种对逆境的适应性更有可能归因于其基因表达调控[28]。研究水黄皮的耐盐分子机理,有助于改善其在逆境条件下的生长状态,进而对其在生物柴油方面的开发应用提供支持。通过提取海水和淡水处理下若干时间点的水黄皮根部和叶片RNA,我们构建了4个文库并采用Illumina平台进行转录组测序,将初始测序结果进行组装和拼接后,共得到108 598条平均长度为606 bp的独立基因序列(Unisequences)[29]。通过比较这些独立基因序列在海水和淡水处理下的表达量,我们从中筛选出23 815条在根部或叶片具有显著表达量变化的序列,作为进一步功能分析的候选基因序列。同时,对这些候选基因的GO和KEGG pathway富集分析显示水黄皮的根和叶器官在响应盐胁迫时存在一些明显的时空表达差异,这一结果为全面探讨不同类型植物响应盐胁迫的分子机制提供了良好的素材。基于上述候选基因的功能注释和表达量变化情况,我们初步挑选了若干基因,在获得其全长序列后构建载体转化拟南芥,已获得部分耐盐能力显著提升的转基因株系,对这些基因作用机制的深入研究仍在进行中。
近期,笔者所在研究团队又基于水黄皮开花后15周至40周时期内的种子样品,同样采用Illumina平台进行转录组测序。该采样时期涵盖了水黄皮种子发育的早、中、晚3个阶段[10]。所得到的种子转录组包含了53 586条平均长度为787 bp的独立基因序列。通过对这些独立基因序列的功能注释,我们发现其中有2 093条序列直接参与各种油脂代谢途径(结果未发表)。上述水黄皮3个器官转录组的构建及相关基因的功能分析,不仅将为该物种的育种工作提供丰富而有价值的基因资源,还可以为其遗传多样性分析提供大量的候选分子标记。
3 基因片段及相关分析
目前,已经报道的水黄皮基因数目不多,且大多数基因片段用于系统发育分析,只有少数基因用于功能研究。Lavin等[30]利用光敏色素A基因() 序列分析了豆科内几个属代表物种的系统发育关系,并结合这些物种的花序特点证明了Millettieae族的单系起源。Hu等[31]则利用核糖体内转录间隔区(ITS) 序列和5.8S rRNA基因序列再次对Millettieae族内包括水黄皮在内的多个物种进行系统发育关系分析。Shi等[32]基于核基因组的18S rRNA基因序列,线粒体基因组的成熟酶R基因() 序列和叶绿体基因组的核酮糖1,5-二磷酸羧化/氧化酶大亚基基因() 序列,采用最大似然(Maximum likelihood) 法对来自17个科的26个真红树和半红树植物代表物种进行系统发育重建,其结果验证了胎萌和泌盐这两个适应性性状在红树植物中是多次起源的。近期,Sirichamorn等[33]对Millettieae族内50个物种进行系统发育关系分析时,扩增到了水黄皮的3个叶绿体基因序列(,基因间隔区和基因间隔区) 及1个核基因序列(ITS/5.8S)。Arpiwi等[13]采用ITS序列对来自澳大利亚、印度和印度尼西亚的49个水黄皮样品进行了谱系分析,结果表明来自印度尼西亚的样品与其他两国样品的亲缘关系较远。
在基因功能研究方面,笔者所在研究团队基于水黄皮转录组测序结果,选择了一个在海水和淡水处理下有显著差异表达的查尔酮异构酶基因(),借助RACE技术获取其666 bp全长cDNA,Blast结果显示其氨基酸序列和核苷酸序列均与豆科其他物种的基因具有高同源性,且其编码蛋白的底物结合位点和反应活性位点也与其他在豆科中发现的查尔酮异构酶相似[34]。通过构建载体将该基因转化盐敏感型酵母突变体后,得到耐盐能力显著提高的酵母菌株,暗示水黄皮查尔酮异构酶可能直接参与其盐胁迫响应过程。水黄皮中的另一个功能基因——硬脂酰载体蛋白去饱和酶基因() 则由印度的Rangan研究团队分离鉴定[35],该基因含有一个1 182 bp的开放阅读框,编码一个与其他植物SAD酶高度相似的蛋白。Southern印迹分析显示在水黄皮中为多拷贝基因,这组基因与水黄皮种子生长发育调控密切相关。除了上述两个已报道的功能基因外,研究者还从水黄皮中分离到若干与油脂合成相关基因(如:乙酰辅酶A羧化酶基因和酮脂酰载体蛋白合成酶基因等) 及与结瘤固氮相关基因(如:黄嘌呤脱氢酶基因和结瘤素基因等),对这些基因的功能研究仍在进行中[8]。
4 小结与展望
尽管水黄皮具有许多优良生物学特性,目前尚未见其商业化种植的报道。该物种主要借助昆虫进行异交繁殖,不仅在不同个体间存在较大的表型和基因型差异,且同一个体的若干后代株系间也存在一定的表型性状差异[17],即其在自然条件下繁殖后代的性状表现较不稳定。因此,对该树种进行良种选育结合后续营养(无性) 繁殖是其大规模推广种植的必要基础。在进行良种选育时,相较于传统筛选手段,借助转基因或RNA干扰等技术进行遗传改良被认为是更适合于能源植物的育种途径[36],而这些技术的实施仍然有赖于对水黄皮分子遗传背景的阐明。然而,对于这一颇具发展潜力的生物质能源树种,目前只有印度、澳大利亚和我国的少数几个研究团队开展了分子生物学研究工作;在水黄皮中开发的分子标记及分离的基因片段均数目有限,对于基因功能研究的报道更是寥寥无几;同时,水黄皮叶绿体基因组、线粒体基因组及转录组测序工作虽已完成,但对于序列数据的解析还不够深入。未来水黄皮分子生物学相关研究将主要围绕以下几个方面开展:其一是应用更多分子标记进行种质遗传多样性分析和谱系分析,找到与感兴趣表型性状相关联的标记位点,辅助相关基因定位;其二是从基因组和转录组数据中发掘更多与种子生长发育、油脂代谢及植株抗性相关的基因,获取基因全长序列,通过异源转基因植物对其功能进行详细的分析和鉴定;其三是充分利用豆科植物的特点,借助发根农杆菌介导的基因转化体系,尝试获得具有推广前景的转基因水黄皮,为后续制备生物柴油的产业化发展提供高产量高品质的油料来源。
REFERENCES
[1] Scott PT, Pregelj L, Chen N, et al.: an untapped resource for the biofuels industry of the future. Bioenergy Res, 2008, 1(1): 2−11.
[2] Murphy HT, O’Connell DA, Seaton G, et al. A common view of opportunities, challenges, and research actions forin Australia. Bioenergy Res, 2012, 5(3): 778−800.
[3] De BK, Bhattacharyya DK. Biodiesel from minor vegetable oils like karanja oil and nahor oil. Lipid Fett, 1999, 101(10): 404−406.
[4] Karmee SK, Chadha A. Preparation of biodiesel from crude oil of. Bioresour Technol, 2005, 96(13): 1425−1429.
[5] Naik M, Meher LC, Naik SN, et al. Production of biodiesel from high free fatty acid Karanja () oil. Biomass Bioenerg, 2008, 32(4): 354−357.
[6] Sharma YC, Singh B, Korstad J. High yield and conversion of biodiesel from a nonedible feedstock (). J Agr Food Chem, 2010, 58(1): 242−247.
[7] Biswas B, Scott PT, Gresshoff PM. Tree legumes as feedstock for sustainable biofuel production: opportunities and challenges. J Plant Physiol, 2011, 168(16): 1877−1884.
[8] Biswas B, Kazakoff S, Jiang Q, et al. Genetic and genomic analysis of the tree legumeas a feedstock for biofuels. Plant Genome, 2013, 6(3): doi: 10.3835/plantgenome2013.05.0015.
[9] Mukta N, Murthy IYLN, Sripal P. Variability assessment in(L.) Pierre germplasm for biodiesel traits. Ind Crops Prod, 2009, 29(3): 536−540.
[10] Pavithra HR, Gowda B, Kumar KR, et al. Oil, fatty acid profile and Karanjin content in developing(L.) Pierre seeds. J Am Oil Chem Soc, 2012, 89(12): 2237−2244.
[11] Bala M, Nag NT, Kumar S, et al. Proximate composition and fatty acid profile of, a potential biodiesel crop. J Am Oil Chem Soc, 2011, 88(4): 559−562.
[12] Hill J, Nelson E, Tilman D, et al. Environmental, economic and energetic costs and benefits of biodiesel and ethanol biofuels. Proc Natl Acad Sci USA, 2006, 103(30): 11206−11210.
[13] Arpiwi NL, Yan GJ, Barbour EL, et al. Genetic diversity, seed traits and salinity tolerance of(L.) Panigrahi, a biodiesel tree. Genet Resour Crop Evol, 2013, 60(2): 677−692.
[14] Sangwan S, Rao DV, Sharma RA. A review on: a great versatile leguminous plant. Nat Sci, 2010, 8(11): 130−139.
[15] Sahoo DP, Aparajita S, Rout GR. Inter and intra-population variability of: a bioenergy legume tree. Plant Syst Evol, 2010, 285(1): 121−125.
[16] Sujatha K, Rajwade AV, Gupta VS, et al. Assessment of-a biodiesel producing tree species using ISSR markers. Curr Sci, 2010, 99(10): 1327−1329.
[17] Jiang Q, Yen SH, Stiller J, et al. Genetic, biochemical, and morphological diversity of the legume biofuel tree. J Plant Genome Sci, 2012, 1(3): 54−67.
[18] Thudi M, Manthena R, Wani SP, et al. Analysis of genetic diversity in Pongamia [(L.) Pierrre] using AFLP markers. J Plant Biochem Biotechnol, 2010, 19(2): 209−216.
[19] Sharma SS, Negi MS, Sinha P, et al. Assessment of genetic diversity of biodiesel speciesaccessions using AFLP and three endonuclease-AFLP. Plant Mol Biol Rep, 2011, 29(1): 12−18.
[20] Pavithra HR, Shivanna MB, Chandrika K, et al. Genetic analysis ofpopulations using AFLP markers. Tree Genet Genomes, 2014, 10(1): 173−188.
[21] Kesari V, Sathyanarayana VM, Parida A, et al. Molecular marker-based characterization in candidate plus trees of, a potential biodiesel legume. AoB Plants, 2010, 2010: plq017, doi: 10.1093/aobpla/plq017.
[22] Kesari V, Rangan L. Genetic diversity analysis by RAPD markers in candidate plus trees of, a promising source of bioenergy. Biomass Bioenerg, 2011, 35(7): 3123−3128.
[23] Sarbhoy RK. Cytogenetical studies in(L.) Pierre. Cytologia, 1977, 42(3): 415−423.
[24] Choudhury RR, Basak S, Ramesh AM, et al. Nuclear DNA content ofL. and genome size stability of-regenerated plantlets. Protoplasma, 2014, 251(3): 703−709.
[25] Marshall DJ, Hayward A, Eales D, et al. Targeted identification of genomic regions using TAGdb. Plant Methods, 2010, 6(1): 19.
[26] Kazakoff SH, Imelfort M, Edwards D, et al. Capturing the biofuel wellhead and powerhouse: the chloroplast and mitochondrial genomes of the leguminous feedstock tree. PLoS ONE, 2012, 7(12): e51687.
[27] Singh K, Yadav J. Effect of soil salinity and sodicity on seedling growth and mineral compsition of. Indian Forester, 1999, 125(6): 618−622.
[28] Parida AK, Jha B. Salt tolerance mechanisms in mangrove: a review. Trees, 2010, 24(2): 199−217.
[29] Huang J, Lu X, Yan H, et al. Transcriptome characterization and sequencing-based identification of salt-responsive genes in, a semi-mangrove species. DNA Res, 2012, 19(2): 195−207.
[30] Lavin M, Eshbaugh E, Hu JM, et al. Monophyletic subgroups of the tribe Millettieae (Leguminosae) as revealed by phytochrome nucleotide sequence data. Am J Bot, 1998, 85(3): 412−433.
[31] Hu JM, Lavin M, Wojciechowski MF, et al. Phylogenetic analysis of nuclear ribosomal ITS/5.8S sequences in the tribe Millettieae (Fabaceae): the core Millettieae, and the Callerya group. Syst Bot, 2002, 27(4): 722−733.
[32] Shi SH, Huang YL, Zeng K, et al. Molecular phylogenetic analysis of mangroves: independent evolutionary origins of vivipary and salt secretion. Mol Phylogenet Evol, 2005, 34(1): 159−166.
[33] Sirichamorn Y, Adema F, Gravendeel B, et al. Phylogeny of palaeotropic-like (Fabaceae) taxa based on chloroplast and nuclear DNA sequences shows reorganization of (infra) generic classification is needed. Am J Bot, 2012, 99(11): 1793−1808.
[34] Wang H, Hu T, Huang J, et al. The expression ofchalcone isomerase insalt-sensitive mutants enhances salt-tolerance. Int J Mol Sci, 2013, 14(5): 8775−8786.
[35] Ramesh AM, Kesari V, Rangan L. Characterization of a stearoyl-acyl carrier protein desaturase gene from potential biofule plant,L. Gene, 2014, 542(2): 113−121.
[36] Gressel J. Transgenics are imperative for biofuel crops. Plant Sci, 2008, 174(3): 246−263.
(本文责编郝丽芳)
Progress in molecular biology of a semi-mangrove,
Jianzi Huang1, Wanke Zhang2, Rongfeng Huang3, and Yizhi Zheng1
1,,518060,,2,,100101,Biotechnology Research InstituteChinese Academy of Agricultural SciencesBeijingChina
L. is a leguminous tree with great potential in biodiesel applications and also a typical semi-mangrove. In this review, we presented several aspects about the recent research progress in molecular biology of. We descrived several types of molecular markers used to assess the genetic diversity and phylogeny of this species, genome and transcriptome analyses based on high-throughput sequencing platform accomplished for this species, and several gene and genomic sequences of this species isolated for further research. Finally, based on the current research progress, we proposed some orientations for future molecular biology research on.
, molecular marker, genome, transcriptome, lipid metabolism, salt stress
July 23, 2014; Accepted: October 28, 2014
Yizhi Zheng. Tel: +86-755-26558400; E-mail: yzzheng@szu.edu.cn
Supported by:National Natural Science Foundation of China (Nos. 31300275, 31370289).
国家自然科学基金(Nos. 31300275, 31370289) 资助。
网络出版时间:2014-11-17
http://www.cnki.net/kcms/doi/10.13345/j.cjb.140388.html
