陆地棉‘徐州142’纤维不同发育时期蛋白质组比较分析
2015-07-04李波,王娟,杨洋,范玲*
李 波,王 娟,杨 洋,范 玲*
(1 新疆农业科学院 核技术生物技术研究所,乌鲁木齐830091;2 新疆农业科学院 经济作物研究所,乌鲁木齐830091)
蛋白质组研究是一种分子生物学研究方法,并在棉花纤维发育机制研究方面已取得较大进展,但利用蛋白质组学技术研究棉花纤维发育的报道很少。与基因组学的遗传标记相比,由于蛋白质组学的研究对象是基因表达的产物,是介于基因型和表型之间的特性,因而蛋白质组学标记是联系基因多样性和表型多样性的纽带,具有独特的意义。
棉纤维是由胚珠表皮细胞发育而形成的单细胞纤维,起始分化后迅速伸长,一般不再分裂,最终纤维长度可达细胞纤维直径数千倍[1]。棉花纤维形成和发育过程可分为:纤维原始细胞分化和突起(起始期)、初生壁伸长、次生壁增厚和脱水成熟4个相互重叠的时期[2-3]。在每个发育时期棉纤维都具有不同的内容和特点,分别决定着纤维的不同性状,但相邻时期没有截然区分的界限,并且有所重叠[3]。Graves等[4-5]最早利用双向电泳研究了棉花胚珠或纤维的蛋白差异图谱,分别比较了-3DPA(开花后天数)、-2DPA 和2DPA 胚珠的差异,从而确定了棉纤维起始发育的开始。Turley[6]利用双向电泳技术,在离体培养时产生纤维的胚珠中发现了5个特异表达蛋白。Ferguson 等[7]比较了纤维发育伸长期棉纤维的蛋白质图谱,发现了46个差异表达的蛋白质点,并鉴定了其中2个蛋白质点。近年来,中国棉花研究者也利用双向电泳研究蛋白质与棉纤维发育的关系。刘康等[8]和徐子健等[9]对棉花纤维蛋白质的提取方法和双向电泳条件等进行了初步探索和改良。有研究者比较了5~25DPA 棉花胚珠和纤维2-DE图谱差异发现,在整个纤维发育中大约有1 600个蛋白点处于稳定表达水平[10-11]。
棉纤维蛋白质组中有相当多的蛋白质是随不同发育阶段而差异表达,这些差异表达的蛋白质与不同时期纤维的各种变化密切相关。研究这些差异表达蛋白质的功能及其与纤维发育之间的关系,有利于阐述棉纤维伸长和次生壁增厚的分子调控机制。本研究以陆地棉遗传标准系‘徐州142’为实验材料,采取5、10、15、20、25和30DPA 分别代表细胞分化期、初生壁伸长高峰期、初生壁合成向次生壁合成转变前期、中期、末期以及次生壁增厚期等6个不同时期的棉纤维,比较不同时期棉纤维蛋白质组2-DE图谱,对其可能的功能进行讨论,以了解不同发育阶段差异表达蛋白质及其功能与纤维发育之间的关系,为棉花产量和品质的改良提供理论依据。
1 材料与方法
1.1 材 料
供试材料为陆地棉‘徐州142’(Gossypium hirsutum L.cv.Xuzhou 142),种植于新疆农业科学院玛纳斯试验基地,取5、10、15、20、25和30DPA 棉铃,剥除棉籽置-70 ℃冰箱备用。
1.2 方 法
1.2.1 不同阶段棉纤维蛋白的提取 取棉花纤维,去除棉籽,于液氮中迅速研磨成细粉状,加入3倍体积的蛋白提取缓冲液[500 mmol/L Tris-HCl(pH 8.0),50 mmol/L EDTA,700 mmol/L 蔗 糖,2%PVP-40,1mmol/L PMSF],漩涡,在冰浴上静置10 min;加入等体积Tris饱和酚,溶液在室温下混匀10min;4 ℃、12 000×g离心10min,将酚相转移至新的离心管中,加入等体积蛋白提取缓冲液,漩涡,在冰浴上静置3min后离心;取酚相,在酚相中加入5倍体积的甲醇-醋酸铵,-20 ℃过夜,进行蛋白沉淀;4 ℃、12 000×g离心10min,收集沉淀,用甲醇-醋酸铵溶液洗涤3次,然后用冷丙酮洗涤1次,真空干燥蛋白,-70 ℃冰箱保存备用[12-13]。
1.2.2 蛋白质浓度测定 蛋白质浓度测定参考Bradford[14]的方法。在595nm下测定吸光度值A595,重复3次求吸光度平均值,制作标准曲线,计算样品蛋白质含量。
1.2.3 双向电泳 采用Protean IEF Cell等电聚焦系统(Bio-Rad),条件略有改变,取适量蛋白样品加裂解液至总体积为400μL,沿IPG 胶条槽缓慢均匀加入,将IPG 胶条胶面朝下覆盖在样品上,在胶面上加少许矿物油,置于Protean IEF Cell型电泳仪上,20℃恒温进行,50V 主动水化12h后,250V 30min;500V1h;1 000V30 min;5 000 V1h,10 000V5h;最后稳压在10 000V 进行等电聚焦,总电压时间积为60 000Vh时结束电泳。第一向等电聚焦结束后,将IPG 胶条放于2.5mL 胶条平衡缓冲液I中振荡平衡15min,再转入2.5mL 胶条平衡缓冲液II中振荡平衡15min。
用PROTEAN XL(Bio-Rad)垂直电泳系统进行第二向电泳。平衡完后将胶条转移到12%SDSPAGE凝胶上,用琼脂糖封胶,以120 V 电压进行电泳,待溴酚蓝移至凝胶底部时停止电泳。电泳结束后,用考马斯亮蓝染色方法[8]进行凝胶染色。

表1 实验中所用的引物序列Table1 Primer sequences applied in this study
1.2.4 RNA提取与cDNA第一链合成 采用热硼酸-蛋白酶K 法提取各时期棉纤维的总RNA。利用反转录酶反转录合成cDNA 第一链,首先将RNA与随机引物混合,在PCR 仪上72 ℃变性5min后,及时暂停冰上依次后续组分,混匀后于25 ℃复性5 min,42 ℃延伸60min,70 ℃加热15min 使反转录酶失活,cDNA 产物于-20 ℃保存。
1.2.5 半定量RT-PCR 利用半定量RT-PCR 方法,以5、10、15、20和25DPA 棉花纤维反转录产物为模板,检测GhMDH、GhSAM、GhPGK1、GhCSD和GhFB在10 和25DPA 棉纤维中的表达情况。根据Mascot搜索结果,利用DNAMAN6.0软件根据目的蛋白的核苷酸序列进行引物设计。引物序列和退火温度如表1。以GhUBI(登录号AY18997)作为内标基因,扩增引物为GhUBI-F和GhUBI-R。GhUBI与目的基因同机分管扩增。20μL 体系中含cDNA(20ng/μL)1μL,2×Mix 10μL,引物(25 μmol/L)0.5μL,ddH2O 8μL。扩增条件为:94 ℃预变性4min;94℃变性30s,60℃复性45s,72℃延伸1min,共28个循环;72 ℃延伸10min。PCR产物置1%琼脂糖中电泳,用紫外凝胶成像仪(Bio-Rad)观察结果。实验重复3次。
2 结果与分析
2.1 不同阶段棉花纤维总蛋白含量的变化
利用Bradford法,根据BSA 标准曲线,计算蛋白的百分含量。结果表明,在棉纤维发育过程中,蛋白的百分含量呈动态变化(图1)。在5DPA 棉花纤维中,总蛋白含量最高达到11.7%,其后随着发育时期的发展,总蛋白含量逐渐降低。这一结果与棉纤维发育过程中生理变化相关,在棉纤维快速延伸阶段(5~15 DPA)和次生壁增厚阶段(20~30 DPA),纤维迅速增加,蛋白含量降低。

图1 棉纤维发育过程中总蛋白百分含量的动态变化Fig.1 The dynamic variation analysis during the cotton fiber development
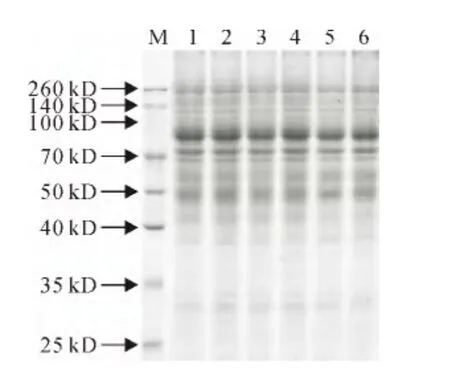
图2 不同时期棉纤维总蛋白的SDS-PAGE分析Fig.2 The dynamic variation analysis during the cotton fiber development
2.2 不同发育阶段棉纤维总蛋白SDS-PAGE分析
利用饱和酚-甲醇醋酸铵法,分别提取6个不同时期棉纤维总蛋白,并利用SDS-PAGE凝胶电泳进行检测,结果(图2)显示,这6个时期都可提取出蛋白,且在分子量50kD左右蛋白含量较多,条带较亮。
2.3 不同发育阶段棉纤维蛋白的2-DE图谱比较
6个不同时期棉纤维蛋白质组的2-DE 图谱共检测到1 000多个蛋白质点。根据质谱鉴定结果,比较了6个样品2-DE 图谱中15个差异点(图3)。在NCBI上进行数据查询,分别是F-box家族蛋白、肌动蛋白、β-微管蛋白、F1-ATP 合成酶、ATP 酶β亚基、膜联蛋白、磷酸甘油酸酯激酶I、胞质苹果酸脱氢酶、S-腺苷-L-高半胱氨酸水解酶、谷氨酰胺合成酶、Cu/Zn超氧化物歧化酶、profilin、4-香豆酸辅酶A 连接酶。选取5个在不同样品中表达量差异明显的蛋白质点进行分析。结果表明,蛋白质在不同发育时期的表达有所不同,蛋白点1只在20~30 DPA 时期表达,蛋白点14只在15~30DPA 时期表达,其它3个蛋白点在不同发育时期表达量都不同,说明不同发育时期纤维的蛋白质在不同时期表达量不同,其中一部分蛋白在特定时期不表达,这是蛋白质以适应不同纤维发育时期纤维细胞活动的需要。
2.4 部分差异蛋白在不同发育阶段的表达量分析
利用PDQuest 8.0.1 软 件 对 其 中5 个 差 异 点进行分析,结果显示,胞质苹果酸脱氢酶(GhMDH)存在于棉纤维发育的各个时期,并且随着纤维的发育表达量升高;S-腺苷-L-高半胱氨酸水解酶(Gh-SAM)同样存在于棉花纤维发育的各个阶段,其在15DPA 表达量最高;胞质磷酸甘油酸酯激酶I也存在于整个棉纤维发育阶段,在10DPA 时表达量最高(GhPGK1),之后随着时间的增加蛋白的表达量逐渐减少;Cu/Zn 超氧化物歧化酶(GhCSD)从15 DPA 开始表达,并且随着纤维的发育含量逐渐增加;F-box家族蛋白(GhFB)在棉纤维的伸长期未见表达,其于20DPA 开始表达,含量随着时间的增加而升高(图4)。
2.5 部分差异蛋白的qRT-PCR分析
利用qRT-PCR 方法,分析5 种蛋白基因在棉纤维发育不同时期的表达情况(图5)。S-腺苷-L-高半胱氨酸水解酶基因(GhSAM)在10DPA 棉花纤维中表达量相对较高;胞质磷酸甘油酸酯激酶I基因(GhPGK1)在2 0DPA棉纤维中表达量相对较高;Cu/Zn 超氧化物歧化酶基因(GhCSD)在15 DPA 棉纤维中表达量相对较高;胞质苹果酸脱氢酶基因(GhMDH)和F-box家族蛋白基因(GhFB)在25DPA 棉纤维中表达量最高。
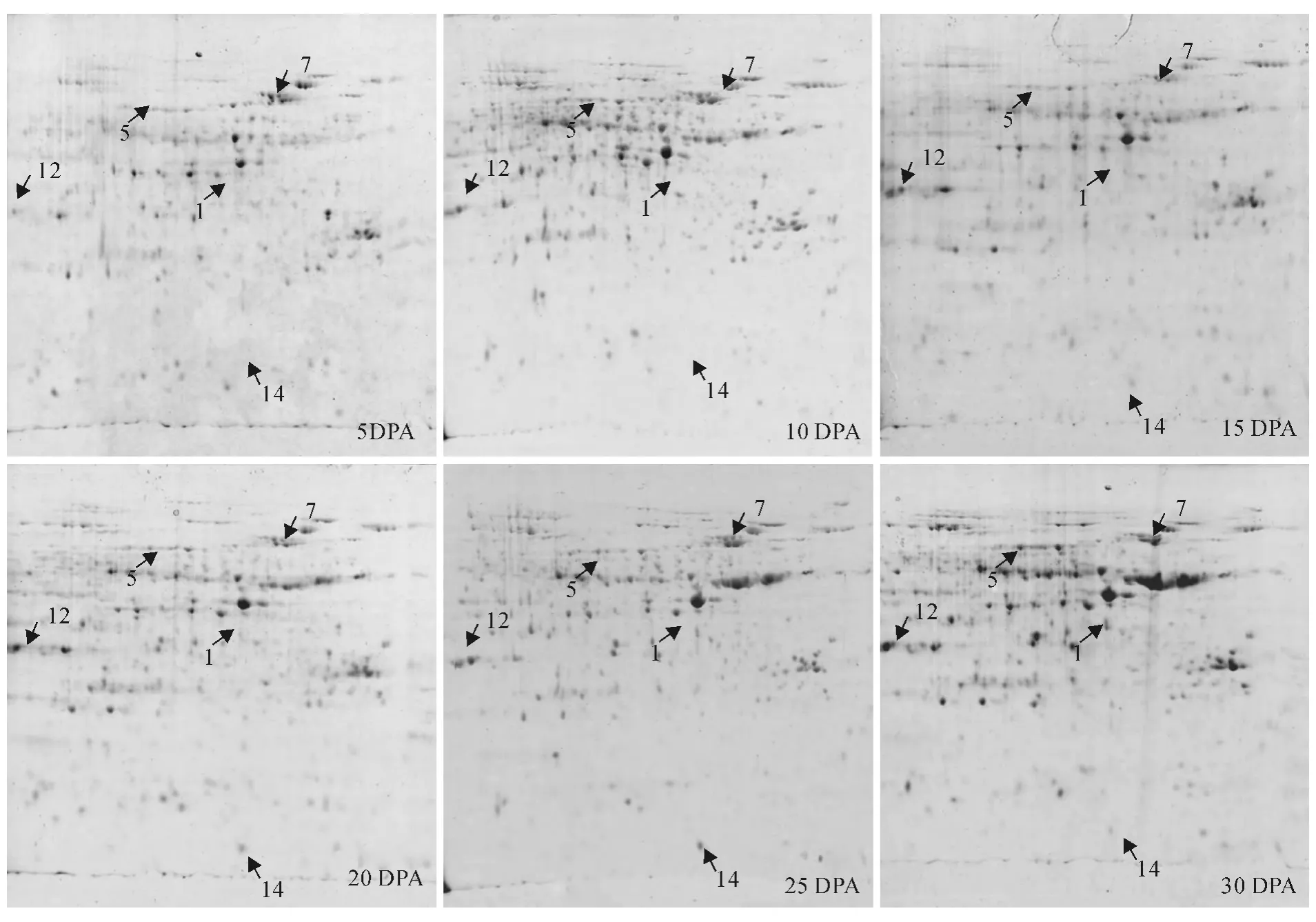
图3 不同发育时期棉花纤维蛋白的2-DE图谱分析Fig.3 The 2-DE analysis during the different cotton fiber development stages
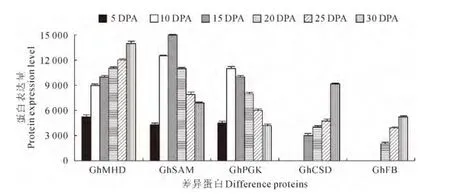
图4 部分差异蛋白在不同发育阶段的表达量Fig.4 The protein expression quantity analysis during the cotton fiber development stages

图5 棉纤维不同发育时期部分基因的qRT-PCR表达分析Fig.5 The genes expression analysis at the different cotton fiber development stages
3 讨 论
纤维生长是棉花发育周期中一个主要的过程,尤其是在次生壁增厚阶段产生94%左右的纤维素。之前研究中,利用MADLI-TOF-MS 对延伸初期(10DPA)和次生壁合成中期(25DPA)的差异蛋白进行了初步鉴定。根据鉴定结果,对6个不同发育阶段的棉纤维蛋白进行2-DE 分析,并对部分蛋白进行了蛋白水平和转录水平上的比较。
胞质苹果酸脱氢酶(MDH)是苹果酸代谢过程中的关键酶,而苹果酸在棉花纤维发育过程中起着重要的作 用,Dhindsa[15]和Basra[16]发现棉纤维延伸初期苹果酸含量显著提升,在次生壁合成初期开始减少。而Ferguson等[7]则认为,从棉花纤维延伸到次生壁合成过程中,苹果酸脱氢酶的活性逐渐增加;佘义斌等[17]研究发现,S-腺苷-L-高半胱氨酸水解酶基因随着纤维伸长的发育进程,表达量逐渐减弱;胞质磷酸甘油酸酯激酶I是在糖酵解途径中为纤维的延伸提供能量的关键酶[9];Cu/Zn超氧化物歧化酶与棉花纤维次生壁发育相关[18];F-box family蛋白参与细胞周期调控、转录调控、细胞凋亡、细胞信号转导等生命活动[19]。另外,F-box家族蛋白还通过其它作用方式参与了体内众多生化过程[20]。
本研究分别在蛋白水平和转录水平上对部分差异蛋白在棉花纤维发育过程中的作用进行了分析和讨论。结果显示,在蛋白水平上,胞质苹果酸脱氢酶、Cu/Zn超氧化物歧化酶以及F-box家族蛋白在棉花纤维次生壁增厚阶段表达量明显增加,推测其可能在棉花纤维次生壁发育阶段起重要作用。在转录水平上,存在着和蛋白水平不一致的结果,可能是由于真核生物在转录与翻译不同时也不同地,基因组与染色体结构复杂,有着更为复杂的调控机制。真核生物的基因表达具有多层次性,可发生在染色质水平、转录起始水平、转录后水平、翻译水平以及翻译后水平[21];另外,在多肽链加工折叠为蛋白质的过程中,经过不同修饰加工的蛋白质其稳定性会有差异。
棉纤维发育是一个经历时间长、形态和生理等发生剧烈变化的过程,研究不同时期棉纤维蛋白的动态变化,可以将不同发育时期纤维的形态和生理变化与相关蛋白的活动联系起来,从而了解这些变化的分子机制,最终为纤维品质和产量的遗传工程改良提供依据。由于棉纤维细胞壁随着细胞的发育而逐渐增厚,使得棉纤维细胞不易破碎,为实验带来一定的难度,且随着时间变化蛋白含量逐渐减少。
[1] LI B(李 波),NI ZH Y(倪志勇),etal.Prokaryotic expression,protein purification and western blotting identification ofGhCOMT2gene fromGossypiumhirsutumL.[J].ActaBot.Boreal.-Occident.Sin(西北植物学报),2010,30(9):1 738-1 743(in Chinese).
[2] BASRA A S,MALIK C P.Development of the cotton fiber[J].InterantionalReviewofCytology,1984,89:65-113.
[3] DELANGHE E A L.Cotton Physiology[M].Memphis:Lint Development,1986:325-349.
[4] GRAVES D A,STEWART J M.Chronology of the differentiation of cotton(GossypiumhirsutumL.)fiber cells[J].Planta,1988,175(2):254-258.
[5] GRAVES D A,STEWART J M.Analysis of the protein constituency of developing cotton fibers[J].JournalofExperimentalBotany,1988,39:59-69.
[6] TURLEY R B,FERGUSOND L.Changes of ovule proteins during fiber development in a normal and a fiberless line of cotton(Gossypium hirsutumL.)[J].JournalofPlantPhysiol.,1996,149:695-702.
[7] FERGUSON D L,TURLEY R B,TRIPLETT B A,etal.Comparison of protein profiles during cotton(GossypiumhirsutumL.)fiber cell wall development with partial sequences of two proteins[J].JournalofAgriculturalandFoodChemistry,1996,44(12):4 022-4 027.
[8] LIU K(刘 康),HU F P(胡凤萍),ZHANG T ZH(张天真).Effect of two methods of protein extraction from cotton ovule and fiber[J].CottonScience(棉花学报),2005,17(6):323-327(in Chinese).
[9] XUE Z J(徐子剑),SHU X(舒 晓),etal.Three techniques on protein extraction from cotton fibers and two-dimensionalelectrophoreses[J].ChineseJournalofBiochemistryandMolecularBiology(中国生物化学与分子生物学报),2006,22(1):77-80(in Chinese).
[10] WU Y T,LIU J Y.Molecular cloning and characterization of a cotton glucuronosyltranferase gene[J].JournalofPlantPhysiology,2005,162(5):573-582.
[11] YANG Y W,BIAN S M,YAO Y,etal.Comparative proteomic analysis provides new insights into the fiber elongating process in cotton[J].JournalofProteomeResearch,2008,7(11):4 623-4 637.
[12] WU X L,XIONG E H,WANG W,etal.Universal sample preparation method integrating trichloroacetic acid/acetone precipitation with phenol extraction for crop proteomic analysis[J].s.Nat.Protoc,2014,9:362-374.
[13] ZHENG R,YUE S J,HAN L,etal.Applied and establishment of two-dimensional polyacrylamide gel electrophoresis system for antherproteome ofLyceumbarbarum[J].ActaBot.Boreal.-Occident.Sin(西北植物学报),2011,31(12):2 443-2 448(in Chinese).
[14] BRADFORD M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of proteindye binding[J].AnalyticalBiochemistry,1976,72(1/2):248-254.
[15] DHINDSA R S,BEASLEY C A,TING I P.Osmoregulation in cotton fiber:Accumulation of potassium and malate during growth[J].PlantPhysiol,1975,56:394-398.
[16] BASRA A S,MALIK C P.Dark metabolism of CO2during fibre elongation of two cottons differing in fibre lengths[J].JournalofExperimentalBotany,1983,34:1-9.
[17] SHE Y B(佘义斌),ZHU Y CH(朱一超),ZHANG T ZH(张天真).Cloning,expression,and mapping of S-adenosyl-L-homocysteine hydrolase(GhSAHH)cDNA in cotton[J].ActaAgronomicaSinica(作物学报),2008,4(6):958-966(in Chinese).
[18] KIM H J,TRIPLETT B A.Involvement of extracellular Cu/Zn superoxide dismutase in cotton fiber primary and secondary cell wall biosynthesis[J].PlantSignalingandBehavior,2008,3(12):1 119-1 121.
[19] HERBST A,HEMANN M T,TWORKOWSKI K A.A conserved element in Myc that negatively regulates its proapoptotic activity[J].EMBOReports,2005,6:177-183.
[20] MATSUZAWA S I,REED J C.SIAH-1,SIP and Ebi collaborate in a novel pathway for B-Catenin degradation linked to p53response[J].MolecularCell,2001,7:915-926.
[21] DIRK B,NORBERT L,WOLFGANG B.Induction of heat shock proteins in response to primary alcohols inAcinetobaetercalcoaceticus[J].Electrophoresis,1999,20(4-5):781-789.
