基于氢键诱导的纳米金比色传感器实时检测脂肪酶活性
2015-06-08刘佳等
刘佳等
关键词 脂肪酶; 酶活性; 纳米金; 氢键; 比色传感
1 引 言
脂肪酶(3.1.1.3)被认为是继蛋白酶、糖化酶之后的又一重要工业用酶。工业用脂肪酶大多来源于微生物,筛选活力高、产量高及低成本的脂肪酶菌种是其关键技术环节,在脂肪酶的改造、分离、纯化以及重组蛋白的筛选等领域,脂肪酶活力的测定都是不可缺少的关键技术,因此, 建立高通量的酶活性检测方法对于促进酶学研究和酶工程技术的发展都具有重要意义[1~4]。
目前,脂肪酶活性检测普遍采用酸碱滴定法[5]、浊度法[6]及光度法[7],但这些方法多存在灵敏度不高、误差大、且费时费力、不适用于高通量检测等不足。荧光法[7]虽然灵敏度较高,但大部分可被脂肪酶水解的荧光底物均受限于反应介质的酸度,另外荧光猝灭等因素还可能影响实验结果的精确性。对于动辄数以千计的定向进化库而言,要进行高通量酶检测,这些方法存在较大局限性。因此迫切需要建立高灵敏度、高选择性,更加简单、快速、准确的脂肪酶检测方法。
纳米金因其独特的表面等离子体共振(SPR)效应成为构建生物传感器的优良材料,已被用于多种酶分析研究中[8~11],尤其是基于纳米金的比色检测方法由于其简单、快速已成为高通量检测方法之一[12]。
本实验以巯基乙酸甲酯修饰的纳米金(MTAuNPs)为比色探针,构建检测脂肪酶活性的生物传感器。在弱酸性磷酸缓冲溶液中,脂肪酶水解切割MTAuNPs 上的MT,生成羧酸根。通过调节溶液pH值至酸性,带有羧酸根残基的纳米金之间能够形成较强氢键,诱导AuNPs聚集变色,基于此建立了氢键诱导纳米金比色传感方法检测脂肪酶的活性(如图1所示)。与传统方法相比,本方法具有快速、简单、价廉、灵敏度高等优势,可用于高通量、实时检测脂肪酶活性。
2 实验部分
2.1 仪器与试剂
SpectraMax M3微孔板检测系统 (美国分子仪器公司); JEM200 CX透射电子显微镜 (日本电子株式会社); B260恒温水浴锅 (上海亚荣生化仪器厂); 电子天平(德国赛多利斯公司); PHS3C pH计(上海雷磁公司); 氯金酸、NaH2PO4、Na2HPO4、柠檬酸钠、柠檬酸、NaOH均为分析纯(国药集团化学试剂有限公司); 巯基乙酸甲酯(MT, 95%,百灵威科技有限公司); 诺维信435脂肪酶(Novozymes 435,诺维信投资有限公司); 根霉脂肪酶(RNL, 1.5 U/mg)、猪胰脂肪酶(PPL)、洋葱假单胞脂肪酶(CRL)、假单胞菌脂肪酶(PSL)、食品脂肪酶(FNL, SigmaAldrich (上海)贸易有限公司)。
2.2 纳米金的合成
采用柠檬酸钠还原法合成纳米金[12]: 300 μL HAuCl4 (0.1 mol/L) 溶液加入含有100 mL超纯水的三口烧瓶中,剧烈搅拌且加热至沸腾; 2 min后加入4 mL现配的柠檬酸钠溶液(38 mmol/L),溶液颜色由黄色逐渐变为透明酒红色,继续反应10 min后停止加热,自然冷却到室温。制得的AuNPs溶液在4 ℃保存备用。
2.3 巯基乙酸甲酯(MT)修饰纳米金
移取适量AuNPs溶液(2.3 nmol/L)和MT溶液(1.0 μmol/L)混合,使其最终摩尔比为1∶50。在室温下,轻微振荡2 h, 使MT在溶液中分散均匀,再静置 10 h,即得MT修饰的纳米金(MTAuNPs)。
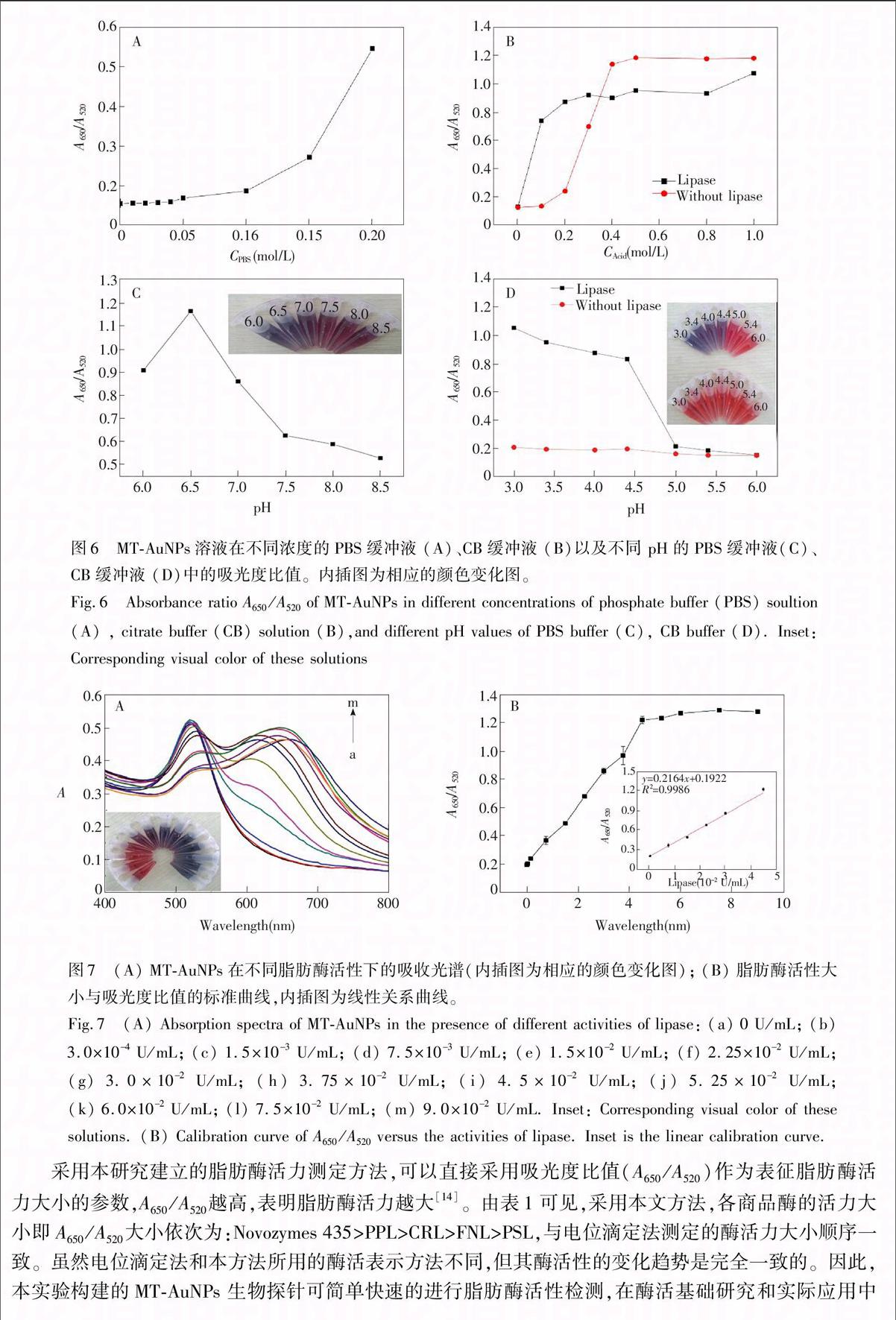
采用本研究建立的脂肪酶活力测定方法,可以直接采用吸光度比值(A650/A520)作为表征脂肪酶活力大小的参数,A650/A520越高,表明脂肪酶活力越大[14]。由表1可见,采用本文方法,各商品酶的活力大小即A650/A520大小依次为:Novozymes 435>PPL>CRL>FNL>PSL,与电位滴定法测定的酶活力大小顺序一致。虽然电位滴定法和本方法所用的酶活表示方法不同,但其酶活性的变化趋势是完全一致的。因此,本实验构建的MTAuNPs 生物探针可简单快速的进行脂肪酶活性检测,在酶活基础研究和实际应用中具有潜在的应用价值。
References
1 Zhu Y T, Jia Y W, Liu Y M, Liang J, Ding L S, Liao X. J. Agric. Food Chem., 2014, 62(44): 10679-10686
2 Nielsen A V F, Andric P, Nielsen P M, Pedersen L H. Langmuir., 2014, 30(19): 5429-5434
3 Saha K, Agasti S S, Kim C, Li X, Rotello V M. Chem. Rev., 2012, 112(5): 2739-2779
4 Yang G, Wu J P , Xu G, Yang L R. Colloids. Surf. B., 2010, 78(2): 351-356
5 Ferrato F, Carriere F, Sarda L, Verger R. Method. Enzymol., 1997, 286: 327-347
6 Francisco J P, Manue F, Oscar M N. Biotechnol. Tech., 1998, 12(3): 183-186
7 Jette J F, Ziomek E. Anal. Biochem., 1994, 219: 256-260
8 Hutter E, Maysinger D. Trends. Pharmaco. Sci., 2013, 34(9): 497-507
9 Kim G B, Kim K H, Park Y H, Ko S, Kim Y. Biosens. Bioelectron., 2013, 41(1): 833-839
10 Zhang L, Zhao J, Jiang J, Yu R. Chem. Commun., 2012, 48(89): 10996
11 Bonomi R, Cazzolaro A, Sansone A, Scrimin P, Prins L J. Angew. Chem. Int. Edit., 2011, 50(10): 2307-2312
12 Grabar K C, Freeman R G, Hommer M B, Natan M J. Anal. Chem., 1995, 67(4): 735-743
13 ZHAO Wei, HUANG ZhuoNan, LI Na. Chinese J. Anal. Chem., 2011, 39(3): 351-355
赵 炜, 黄卓楠, 李 娜. 分析化学, 2011, 39(3): 351-355
14 Zhang W, Tang Y, Liu J, Jiang L,Huang W, Huo F W, Tian D B. J. Agric. Food Chem., 2015, 63(1): 39-42