猪瘟病毒广西流行毒株E0基因的克隆与分析
2015-05-31曾咏芳曹佳媛杨可妍覃雨阳
曾咏芳,曹佳媛,杨可妍,吴 军,覃雨阳,熊 毅
(1.广西大学动物科学技术学院,广西南宁 530005;2.广西区动物疫病预防与控制中心,广西南宁 530001)
猪瘟是由猪瘟病毒 (Classical swine fever virus,CSFV)引起的一种对养猪业危害严重,造成了严重的损失,是世界粮农组织和各国政府严密监控和检疫的主要传染病之一[1]。猪瘟病毒是黄病毒科瘟病毒属成员,具有囊膜的单股正链RNA病毒,基因组长度约12.5kb,含单一的开放阅读框架,编码4种结构蛋白,8种非结构蛋白,其中E0和E2基因编码主要的囊膜糖蛋白,诱导机体产生中和抗体。E0蛋白是一种结构蛋白,也是一种多功能性蛋白,具有神经毒性及转导特性参与病毒粒子黏附和侵入宿主细胞的过程,并且能够通过介导宿主淋巴细胞的凋亡而使宿主产生免疫抑制导致持续感染[2]。在病毒粒子中,E0糖蛋白以分子质量为100ku的同源二聚体的形式存在,但E0并不像E2那样具有跨膜区,而是以一种未知机制连接在病毒粒子的表面上,对CSFV在细胞中的复制、表达和调控过程起重要作用[3]。E0蛋白具有RNase活性,其C端区域可控制CSFV感染过程中的病毒对真核细胞膜的迁移并具有抑制双链RNA诱导细胞反应。王琴等[4]通过对10个流行株与SM株E0基因的比较分析,发现我国CSFV具有复杂性、多样性和相对的遗传稳定性;王家富等[5]利用RT-PCR的方法,扩增并克隆了HCLV和石门强毒株完整的E0糖蛋白基因并进行了序列测定。E0是CSFV的一个重要蛋白,在长期的进化中仍保持RNase活性,其RNase具有多种功能,与许多生物过程有关。E0可抑制多种动物淋巴细胞的蛋白合成及引起淋巴细胞凋亡,CSFV感染的猪存在白细胞减少和免疫抑制,这些结果提示E0在CSFV的致病过程中起着重要作用。因此,对E0基因的研究有十分积极的意义,通过对6株广西猪瘟病毒流行株与4个标准株E0基因的序列及推导蛋白的分析,可以更切合实际的了解广西猪瘟流行毒株与猪瘟4个标准株之间的差异,为其变异规律提供理论依据,从而为制定更有效防控CSFV的措施奠定了基础。
1 材料与方法
1.1 材料
1.1.1 主要仪器与试剂 总RNA抽提试剂盒、DNA Marker DL 2 000、胶回收试剂盒、DH5α大肠埃希菌感受态细胞、质粒抽提取试剂盒等为天根生化有限公司产品;Ex Taq DNA聚合酶、M-MLV反转录酶、HIR 抑制酶、dNTPs、Mgcl2、PMD18-T 质粒载体、限制性内切酶等为宝生物工程(大连)有限公司产品;PCR仪、超速离心机、移液器等。
1.1.2 病毒 猪瘟病毒野毒株阳性病料由广西动物疫病预防控制中心家畜诊断科鉴定并保存,扩增的6株E0基因分别编号为GX1~GX6,病料来自广西陆川县。
1.2 方法
1.2.1 引物的合成与设计 根据GenBank中已发表的CSFV全基因组序列,通过Oligo软件设计了扩增E0基因的引物对E0-1/E0-2,预期扩增长度为887bp,引物由宝生物工程(大连)有限公司合成。将其稀释成25pmol/μL,置-20℃分装,备用。
1.2.2 病毒RNA的提取 按天根生化有限公司总RNA提取试剂盒方法提取RNA,最后溶于30μL ddH2O。
1.2.3 反转录合成病毒cDNA 将获得的病毒RNA根据M-MLV反转录酶产品说明书进行操作。反应体系为5×M-MLV RTase Reaction buffer 5μL,dNTPs(10mmol/L)2μL,下游引物 E0-2(25pmol/μL)2μL,RNasin inhibitor(40U/μL)0.5μL,M-MLV RTase(200U/μL)0.5μL,RNA模板15μL。PCR反应条件:42℃,1h;94℃,5min,反应结束后获得猪瘟病毒cDNA。
1.2.4 目的基因的PCR扩增 用E0引物对目的基因进行PCR扩增。PCR反应体系为:ddH2O 14.5μL,10×ExTaq bufffer 2.5 μL,dNTPs(2.5mmol/L)2μL,上、下游引物各0.5μL,ExTaq酶0.1μL,cDNA 模板5.0μL,共25μL 体系。PCR反应程序为:94℃5min;94℃45s,56℃45s,72℃1min 30s,进行35个循环;最后72℃10min。反应结束后,取PCR产物于10g/L琼脂糖凝胶上电泳,125V电泳35min,置凝胶成像系统中观察结果。
1.2.5 目的基因的胶回收、连接、酶切鉴定及克隆测序 胶回收按照天根生化有限公司胶回收试剂盒进行。按DNA连接试剂盒将回收纯化后的目的基因与pMD18-T载体连接,连接后4℃过夜。将连接产物转化至感受态细胞DH5ɑ中,涂布于添加有Amp(100μg/mL)的LA培养基上,37℃培养12h~16h,挑取上述生长良好的白色菌落,接至含有Amp(100μg/mL)的LB液体培养基,37℃摇床培养16h。取3mL培养物提取重组质粒,进行双酶切及质粒PCR鉴定,筛选出两者均为阳性的菌株送宝生物工程(大连)有限公司进行测序。
2 结果
2.1 E0基因扩增结果
PCR扩增产物经10g/L琼脂糖凝胶电泳分析表明,利用自设E0基因的特异性引物扩增出长度为887bp左右的条带,与预期结果相符(图1)。阳性对照为猪瘟兔化弱毒苗(HCLV)。

图1 CSFV E0基因RT-PCR产物电泳图Fig.1 Electrophoresis of RT-PCR products of CSFV E0gene
2.2 目的基因的重组质粒鉴定结果
6株E0基因的2个片段重组质粒用(SalⅠ和EcoRⅠ双酶切),可见887bp和2 650bp左右的2个片段,分别为目的基因和pMD18-T载体,与预期结果相符(图2)。
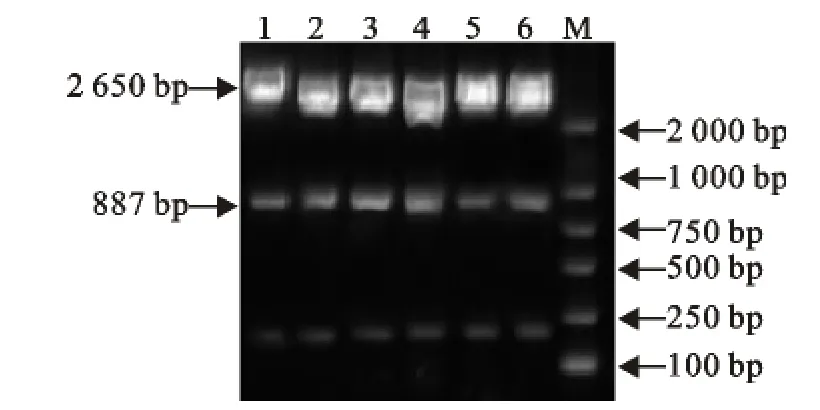
图2 CSFV E0基因酶切产物电泳图Fig.2 Electrophoresis of enzyme digestion products of CSFV E0gene
2.3 核苷酸和氨基酸的同源性分析
本试验分离得到的6株广西毒株与HCLV(登录号:AF091507.1)、Shimen(登录号:AF092448.2)、Paderborn(登录号:GQ902941.1)、GXWZ02(登录号:AY367767)核苷酸和氨基酸的同源性比对结果如表1所示。序列分析可知,广西流行株与HCLV株、Shimen株、Paderborn株、GXWZ02株之间核苷酸的同源性分别为80.8%~81.5%、82.8%~83.3%、93.1%~94.2%、93.7%~94.2%,推导的氨基酸同源性分别为88.3%~89.7%、89.7%~91.1%、96.2%~97.7%、97.7%~99.1%,表明研究的6株广西猪瘟流行株的E0基因发生了较大变异。
2.4 系统进化树分析
使用软件MEGA4.1对6株毒株的E0基因进行序列比对,并从GenBank上下载国内外发表的毒株,构建遗传进化树(图3)。

表1 6株广西猪瘟病毒流行毒株与HCLV株、Shimen株、Paderborn株、GXWZ02株E0基因核苷酸和氨基酸同源性比较Table 1 The nucleotide and amino acid homologous comparisons of E0gene among 6Guangxi CSWFV strain and HCLV strain,Shimen strain,Paderborn strain,GXWZ02strain %
图3 CSFV E0基因的遗传进化树Fig.3 Phylogenetic tree based on nucleotide sequences of CSFV E0
如图3所示,广西流行毒株均分布于GenogroupⅡ,在遗传关系上与 FJFZ、Paderborn、CSF0283、GXWZ02、GXBH、HeNXH等毒株亲缘性较近,而与GenogroupⅠ中的毒株Shimen、HCLV的关系相距较远。
2.5 E0蛋白Rnase活性区域氨基酸序列分析
E0蛋白中存在2个具有Rnase活性的氨基酸基序SLHGIWPE和EWNKHWC,在广西6株流行毒株的氨基酸序列中分别位于9-16位和56-63位(图4)。由图4可知,这两个活性区域内除了HCLV在16位上由E→G,其他氨基酸均没有任何变异。
2.6 糖基化位点分析
广西流行野毒株的E0基因上共有6个糖基化位点,分别在7、46、76、81、124、139位上,与Paderborn株、GXWZ02株相同,而HCLV株的E0基因上缺少了81位糖基化位点,标准强毒株Shimen缺少了7和81位糖基化位点(表2)。

图4 广西CSFV流行毒株与参考毒株E0 2个Rnase活性区域氨基酸基序的比较Fig.4 Comparison of deduced amino acid sequences of two domains of Rnase activity among reference strains and Guangxi strain

表2 糖基化位点的比较Table 2 Comparison of glycosylation sites
3 讨论
随着科学的进步和科技的发展,猪瘟病毒在国内的传播和扩散已经找到了较为系统的防控方法,但是,某些地区的猪瘟病毒还是存在着以隐性感染的形式传播扩散。本文研究的6株阳性毒株发病急,病程短,病死率高达50%,25d~30d阶段都已注射过猪瘟疫苗。序列分析结果表明,广西流行株与 HCLV、Shimen、Paderborn、GXWZ02株之间核苷酸的同源性分别为80.8%~81.5%、82.8%~83.3%、93.1%~94.2%、93.7%~94.2%,推导的氨基酸同源性分别为88.3%~89.7%、89.7%~91.1%、96.2%~97.7%、97.7%~99.1%,广西流行株与HCLV株、Shimen株相比同源性较低,表明广西近期流行的CSFV与两个标准对照株之间存在较大的差异;相对而言,与Paderborn株、GXWZ02株亲缘关系比较近。遗传进化树结果表明,广西流行毒株与疫苗株HCLV、Shimen株关系较远,而与Paderborn株、GXWZ02株较为相近。说明广西CSFV野毒株正向着远离疫苗株的方向发展,这可能也是注射了猪瘟疫苗以后未能得到免疫保护,导致不完全免疫甚至免疫失败的原因。
目前,关于CSFV的分子流行病学调查主要是通过对其全基因或某个特定的区域(如E0基因)进行序列测定,分析其核苷酸及推导的氨基酸序列的变异,然后构建遗传进化树,追溯病毒的起源,这对CSFV的遗传变异的研究及对其的防治有着重要意义。CSFV的E0蛋白是一种保护性抗原,能够刺激机体产生中和性抗体[6],从而获得对CSFV的免疫力。其RNase活性可能对病毒在宿主体内的持续感染有直接关系[7],而且在病毒复制过程中有可能发挥了重要作用。E0蛋白中存在2个具有RNase活性的氨基酸基序SLHGIWPE和EWNKHWC,在广西6株流行毒株的氨基酸序列中分别位于第9位-16位和第56位-63位,这两个活性区域内除了HCLV在第16位上由E→G,其他氨基酸均没有任何变异,HCLV在第16位上的氨基酸变异有可能是导致广西流行毒株未被兔化弱毒苗免疫保护的原因,具体原因还需进一步的研究。广西流行株GX1-GX6的E0基因上共有6个糖基化位点,分别在第7、46、76、81、124、139 位 上,与 Paderborn 株、GXWZ02株相同,而HCLV株的E0基因上缺少了第81位糖基化位点,标准强毒株Shimen株缺少了第7和81位糖基化位点,这些变异有可能导致了抗原性差异,从而使广西流行毒株逃脱兔化弱毒株的免疫保护,导致不完全免疫或活免疫失败。猪瘟病毒仅一个血清型,我国传统疫苗株被证明是十分可靠且有效的疫苗,但是病毒的变异与疫苗保护的背离最终是否会导致疫苗不能保护未来的病毒变异株是个非常值得探讨的问题。
通过对广西猪瘟病毒流行株与4个标准株E0基因的序列及推导蛋白的分析可知,广西CSFV流行株E0基因的抗原性发生了改变,CSFV野毒株正向着远离疫苗株的方向发展,这为今后研究猪瘟病毒与疫苗株的差异和变异规律提供理论依据,也为进一步探讨E0蛋白抗原结构提供了依据。
[1]毋 婷 .目前猪瘟免疫失败的主要原因及综合防治措施[J].新疆畜牧业,2011,21(1):21-22.
[2]谭颖翌.广西部分规模猪场种猪群猪瘟感染情况调查及免疫效果评估[D].广西南宁:广西大学,2012.
[3]Hulst M M,Panote F E,Hoekman A,et al.Inactivation of the RNase activity of glycoprotein Erns of classical swine fever virus results in a cytopathogenic virus[J].J Virol,1998,72:151-157.
[4]王 琴,宁宜宝,王在时,等.猪瘟病毒流行株与疫苗株Erns基因的序列分析[J].中国农业科学,2004,37(3):446-452.
[5]王家富,张楚瑜,王 宁,等.猪瘟病毒石门株与兔化弱毒株E0糖蛋白基因的克隆及序列分析[J].微生物学报,2000,40(1):32-37.
[6]张富强,李志华,张念祖.猪瘟病毒囊膜结构(糖)蛋白Erns和E2的生物学特性研究[J].中国预防兽医学报,2005,27(6):465-468.
[7]Luo Xuelian,Pan Ruangang,Wan Chao,et al.Glycosylation of classical swine fever virus Erns is essential for binding doublestranded RNA and preventing interferon-beta induction[J].Virus Res,2009,146(10):135-139.
[8]尹 珊.猪瘟病毒Erns、NS4A、NS4B、NS5A蛋白的原核表达及多克隆抗体的制备[D].广西南宁:广西大学,2013.
[9]彭志成.广东省部分地区猪瘟分子流行病学调查[D].山东泰安:山东农业大学,2013.
[10]陈 锴.猪瘟慢性感染对猪免疫功能影响的细胞与分子机制研究[D].四川雅安:四川农业大学,2012.
[11]刘 坤,兰邹然,姜 平,等.猪瘟病毒分子生物学及检测技术研究进展[J].动物医学进展,2012,33(10):99-104.
[12]王晓燕,张 月,孙 娟,等.猪瘟病毒Erns基因的克隆与序列分析[J].动物医学进展,2011,32(1):15-18.
[13]Sun Y,Yang Y,Zheng H,et al.Co-expression of Erns and E2genes of classical swine fever virus by replication-defective recombinant adenovirus completely protects pigs against virulent challenge with classical swine fever virus[J].Res Vet Sci,2013,94(2):354-360.
[14]Li X,Wang L,Shi X,et al.Development of an immunochromatographic strip for rapid detection of antibodies against classical swine fever virus[J].J Virol Meth,2012,180(1-2):32-7.
[15]Ahuja A,Sen A,Yogisharadhya R,et al.Prokaryotic expression and purification of highly soluble partial glycoprotein Erns of Indian strain ofclassical Swine Fever virus[J].Indian J Virol,2012,23(3):397-401.
[16]张民秀.广西CSFV地方株的分离、全基因序列测定分析和致病性研究[D].广西南宁:广西大学,2012.
[17]Hulst M,Loeffen W,Weesendorp E.Pathway analysis in blood cells of pigs infected with classical swine fever virus:comparison of pigs that develop a chronic form of infection or recover[J].Arch Virol,2013,158(2):325-339.
