卡特兰 ChCHS1和ChDFR1基因的克隆及表达分析
2015-05-30王紫珊周琳王雁
王紫珊 周琳 王雁


摘 要 以卡特兰紫色花品种‘粉女郎(Cattleya hybrid‘Pink Lady)为材料,采用RT-PCR和RACE方法从花瓣中分离得到了一个查尔酮合酶(chalcone synthase, CHS)和一个二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase, DFR)同源基因的cDNA全长,分别命名为ChCHS1和ChDFR1,GenBank登录号分别为KP171693和KP171694。序列分析结果发现:ChCHS1的cDNA全长1 508 bp ,编码394个氨基酸;ChDFR1基因的cDNA全长1 250 bp,编码350个氨基酸。同源性检索和生物信息学分析表明,这2个基因编码的蛋白都具有各自的功能位点和保守性特征多肽序列。氨基酸序列比对与系统进化树分析显示,ChCHS1、ChDFR1基因在兰科中与蕙兰、石斛兰关系最近,与百合科关系较近。相对实时荧光定量PCR结果表明,ChCHS1 和ChDFR1都在蕾期表达量最低,伴随花朵的开放,表达量逐渐上升,最终分别在花朵盛开期和接近开放时达到最高。
关键词 卡特兰;查尔酮合酶基因;二氢黄酮醇4-还原酶基因;克隆;表达分析
中图分类号 S682.3 文献标识码 A
Abstract The full-length cDNA sequences of chalcone synthase(CHS)and dihydroflavonol 4-reductase(DFR)genes were cloned from Cattleya hybrida‘Pink ladyby using RT-PCR and RACE, named ChCHS1 and ChDFR1. The GenBank accession was No. KP171693 and No. KP171694 recpectively. The bioinformatics analysis indicated that ChCHS1 is 1 508 bp in full length, encoding a polypeptide of 394 amino acids while ChDFR1 is 1 250 bp in full length, encoding a 350 predicted amino acids residues. The sequence and homology of GenBank revealed that CHS and DFR had the conserved active sites and the family signture. Sequence and phylogenetic analysis indicated a high degree homeotic between Cattleya and Cymbidium or Dendrobium, and in different family CHS and DFR shared more than 70% homology with Liliaceae. Relative real-time PCR analysis showed that the highest expression of ChCHS1 was when flower was in bloom. When the flower was nearly open, ChDFR1 had a highest abundant transcript. In bud stage, ChCHS1 and ChDFR1 had the low level expression.
Key words Cattleya hybrida; Chalcone synthase gene; Dihydroflavonol 4-reductase gene; Cloning; Expression analysis
doi 10.3969/j.issn.1000-2561.2015.07.016
花色是观花植物最重要的观赏特征之一,直接影响花卉的观赏价值和经济价值,新奇花色的培育和创造一直是观花植物育种的重要目标和研究热点[1]。类黄酮是花色形成中最主要的色素种类,也是植物中研究最透彻的第二大次生代谢产物。通过对拟南芥、玉米和矮牵牛等模式植物类黄酮化合物合成突变体进行筛选,很多相关功能基因及调节基因得以分离[2-3],类黄酮生物合成途径也已非常清楚。在此基础上,对多种植物花色分子调控机理的研究正逐步深入[4-7]。
在类黄酮色素的合成途径中,查尔酮合成酶 (chalcone synthase, CHS)是一个重要的关键酶,其催化1分子的香豆酰CoA(coumary CoA)与3分子的丙二酰CoA(malonyl CoA)缩合形成4,5,7-三羟基黄烷酮(narigeninchalcone),再经过异构后在不同酶作用下形成花色素苷、黄酮及异黄酮类等黄酮物质。自Reimold等[8]在1983年分离得到了第一个欧芹CHS基因以来,矮牵牛[9]、金鱼草[10]、兰花[11]、金花茶[12]和牡丹[13]等多种观赏植物的CHS基因也相继克隆。二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase, DFR)是花色素苷合成途径下游中的第一个关键酶,可以催化花色素苷必要的前体物质二氢山奈酚(DHK)、二氢栎皮酮(DHQ)、二氢杨梅黄酮(DHM)最终形成矢车菊素、天竺葵素、飞燕草素[2]。目前,利用DFR基因开展花色基因工程育种的报道有很多。Meyer等[14]将玉米中分离得到的DFR基因转入矮牵牛中,最终培育出橘红色花的矮牵牛植株。Tanaka等[15]将玫瑰中分离的DFR基因转入粉红色的矮牵牛中得到了开橙红色的花。Holton[16]将正义DFR基因与正义矮牵牛F3′5′H基因导入白色的康乃馨,最终得到开紫色花的转基因植株。
卡特兰(Cattleya hybrida)因其花大形美、色彩丰富艳丽被世界公认为“洋兰之王”,经长期传统育种,已形成紫红色系、橙红色系及砖红色系、浅色及粉色系、白色系、白花红(紫)唇及白底五剑花、蓝色系、黄色系、绿色系等八大色系[17]。目前通过杂交育种手段很难定向培育出新型花色,利用基因工程技术增强或抑制基因的表达从而改变花朵颜色已被广泛利用到花卉基因工程育种中,但卡特兰花色分子研究机理的缺乏影响了其花色新品种的进程。本研究选择紫色卡特兰品种‘粉女郎为试材,利用RT-PCR和RACE技术分离克隆了ChCHS1和ChDFR1基因的cDNA全长,并对这2个基因在花朵开放进程中的表达变化进行分析,旨在为进一步研究卡特兰CHS及DFR基因功能,以及对未来通过转基因技术培育新型花色的卡特兰品种奠定基础。
1 材料与方法
1.1 材料
卡特兰‘粉女郎(Cattleya Acker's Spotlght ‘Pink lady)种植于中国林业科学研究院国家重点实验室智能温室。选取5个不同发育阶段(图1)的花瓣,P1(花芽1.5 cm长)、P2(花芽2.5 cm长)、P3(花芽4 cm长)、P4(花芽5 cm长,即将开放)、P5(盛开期),取后立将材料用液氮速冻,于-80 ℃冰箱储存备用。
1.2 方法
1.2.1 RNA提取和cDNA第一链的合成 总RNA提取按照天根生化科技有限公司RNA prep Pure Plant Kit试剂盒说明书进行,取1 μg RNA,分别使用PrimeScriptTM II 1st Strand cDNA Synthesis Kit、SMARTTM RACE cDNA Amplification Kit反转录合成cDNA,然后进行RT-PCR、RACE-PCR。
1.2.2 CHS及DFR基因的克隆 (1)中间同源片段扩增。在NCBI上搜索兰科CHS、DFR基因,比对金钗石斛(Dendrobium nobile)、白苇兰(Bromheadia finlaysoniana)、蝴蝶兰(Phalaenopsis hybrid cultivar)、蕙兰杂交种(Cymbidium hybrid cultivar)、文心兰(Oncidium Gower Ramsey)等CHS、DFR基因的mRNA序列,根据基因保守区分别设计中间片段的一对简并引物CHS-F: 5′-AT(C/A)(T/A)C(T/C)CA(C/T)CT(A/C)ATCTTCTGCAC(C/G)AC-3′,CHS-R: 5′-AT(A/G)TT(C/G/A)CC(A/G)TACTC(C/T)GC(A/C)AGCAC-3′;DFR-F: 5′-C(T/G/C)GCAGGAACAGT(A/G/C)AA(C/T)GTGGA(A/G)GA-3′,DFR-R: 5′-GAGGAATG(G/T)CATA(T/C)GTG(G/A)(G/C)ATATCT-3′。以蕾期P3期中提取的总RNA反转录后得到的cDNA第一链为模板进行RT-PCR扩增,PCR条件为94℃预变性3 min;94℃变性30 s,57~59 ℃退火30 s,72 ℃延伸40 s,30个循环;72 ℃延伸7 min。将PCR产物以1.2%的琼脂糖凝胶中进行电泳分析,并用AxyPrep DNA凝胶回收与预期片段大小一致的条带,将目的片段与载体pMD 19-T vector(TaKaRa)进行连接,转化大肠杆菌Top 10(TIANGEN),在涂有X-gal/IPTG平板上进行蓝白斑筛选,挑取白色菌落,委托上海生物工程服务有限公司测序。
(2)CHS和DFR基因3′RACE及5′RACE扩增。依据所得到的中间序列,分别设计CHS及DFR 5′端下游引物与CHS及DFR 3′端上游引物。CHS基因3′RACE引物:5′-CGTCTCGGCTTCCCAAACCA
TCCT-3′;DFR基因3′RACE引物:5′-CCCGAAGCA
AACGGCAGATACATT-3′;CHS基因:5′RACE引物5′-GGTAGAGCATTATGCGGTTGACGGAC-3′;DFR基因5′RACE引物:5′-GCTGCTTGGTGTTCCTCCA
CATTGAC-3′。以SMARTTM RACE cDNA Amplification Kit(Clontech)反转录合成的cDNA为模板进行3′RACE扩增,将与预期片段大小一致的PCR产物回收后连接到pMD 19-T载体上,委托上海生物工程服务有限公司测序。
1.2.3 基因序列分析 将测序所得的CHS及DFR基因不同片段分别用Contig Express软件进行拼接,并将获得的cDNA全长通过ORF Finder软件进行开放阅读框分析。用ProtParam软件分析ChCHS1及ChDFR1蛋白的基本性质(分子质量、理论等电点);使用BioEdit软件对搜索得到的蛋白序列进行多重比对,最后使用MEGA6.0软件中的Neighbor-joining法进行系统进化树的构建,并用Bootstrap进行检测。
1.2.4 实时荧光定量表达分析 提取卡特兰5个发育阶段的花瓣总RNA,反转录后得到不同时期的cDNA。采用SYBR Premix Ex Taq II(TaKaRa)为荧光染料,以卡特兰中分离得到的Actin基因为内参,在Applied Biosystems 7500 Real-Time PCR System荧光定量仪上进行时期特异性表达的RT-PCR分析。反应体系参照SYBR Premix Ex TaqTM II kit (TaKaRa)说明书。PCR条件为95 ℃ 30 s;95 ℃ 5 s,60 ℃ 35 s,40个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。Actin基因的上游引物为:5′-ACTGGT
ATTGTGCTGGATTCTGG-3′;下游引物为:5′-ACG
CTCTGCGGTAGTTGTGAA-3′。CHS基因的上游引物:5′-TCGGCTTCCCAAACCATCCT-3′,下游引物为:5′-TCCAATCCTGAATGCCGAGC-3′;DFR基因的上游引物为:5′-CCGAAATGCCACCGAGTTTG
G-3′;下游引物:5′-GCTTCGGGATGCTCAAATAG
AA-3′。检测目的基因和管家基因的CT值,每个样品3次PCR重复。
1.3 数据处理
采用7500 software(ver. 2.0.1)软件进行数据整理,使用Excel 2007分析数据。
2 结果与分析
2.1 ChCHS1及ChDFR1基因cDNA全长的获得
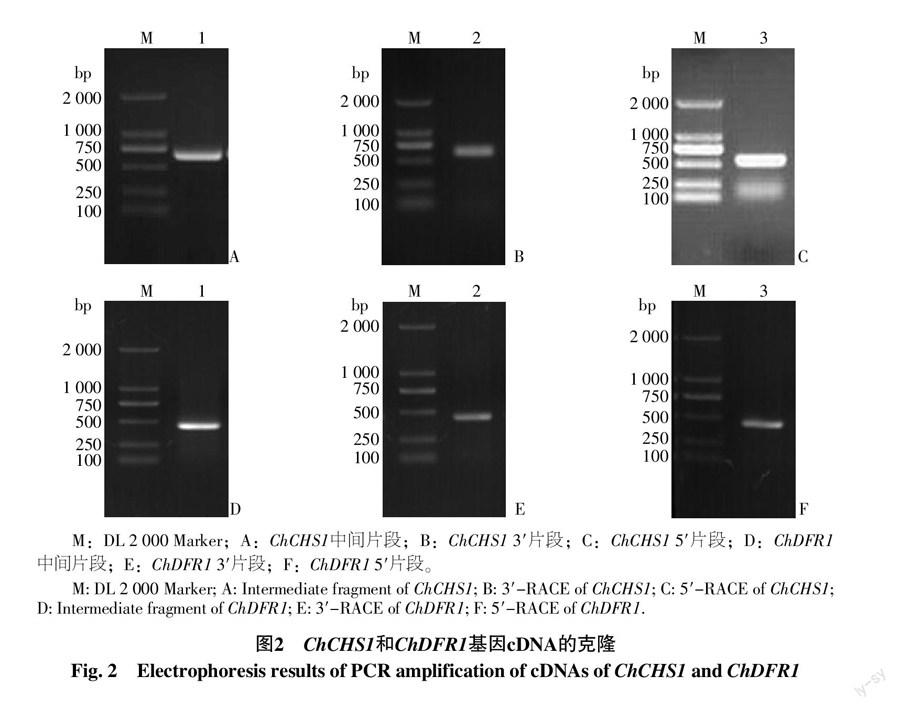
以‘Pink lady蕾期P3期 cDNA为模板,通过RT-PCR分别得到一条与预期片段大小一致的扩增片段(图2-A,D)。将测序结果用Blastn分析,2片段分别与GenBank数据库中兰科植物的CHS和DFR基因有较高的相似性。随后,采用RACE法分别获得长681 bp和 562 bp的CHS基因末端序列(图2-B,C), 以及长465 bp 和435 bp的DFR基因末端序列(图2-E,F),最后通过序列拼接得到2个基因的cDNA全长,在NCBI的ORF Finder 平台上分析发现,CHS全长1 508 bp,包含一个长度为1 185 bp的开放阅读框,该基因的cDNA序列在80 bp处有起始密码子ATG,在1 186 bp处有终止密码子TGA,在1 478 bp处有polyA附加信号,将其命名为ChCHS1,登录号为KP171693;DFR全长1 250 bp,其cDNA序列包含长度82 bp的5′非翻译区和841 bp的3′非翻译区,在1 239 bp处有polyA附加信号,将其命名为ChDFR1,登录号为KP17694。
2.2 ChCHS1及ChDFR1基因的氨基酸序列分析
ChCHS1基因编码一个包含394个氨基酸的蛋白质,用ProtParam软件预测所编码蛋白质的分子量为42.92 ku,理论等电点(pI)为5.76。
利用NCBI中的BlastP对CHS编码的蛋白保守域进行预测发现,CHS编码的蛋白含有普遍CHS蛋白共有的2个保守域:cd00831(CHS_like, Chalcone and stilbene synthases, type III plant-specific polyketide synthases, III型聚酮合酶)和PLN03172(chalcone synthase family protein, 查尔酮合成酶)。
功能位点分析发现[18-19](图3),ChCHS1含有多个在查尔酮合酶中起必要功能的活性位点,其中查尔酮合酶所特有的高度保守三联催化位点Cys(C)、His(H)和Asn(N),分别在第165、304、337处;以及7个袋状环化位点,即Thr(T)、Met(M)、2个Phe(F)、Ile(I)、Gly(G)和Pro(P),分别位于CHS蛋白序列的第133、138、216、266、255、257、376处;5个袋状香豆酰结合位点:2个Ser(S)、Glu(E)、2个Thr(T),分别位于第134和339处、第193处、第195和198处;以及辅酶A结合位点Lys(K)(第56、63处)和Arg(R)(第59处)。在CHS基因中这些位点高度保守,且各位点相对位置大体保持不变。在(http://prosite.expasy.org/)网站上分析发现,CHS还具有查尔酮合酶家族的特征多肽序列:RIMLYQQGCFAGGTVLR。
ChDFR1基因编码一个包含350个氨基酸的蛋白,预测的分子量为39.35 ku,理论等电点(pI)为5.57。在NCBI Conserved domains on检索发现,卡特兰ChDFR1编码的氨基酸序列包含PLN02650(Dihydrofl-avanol4-reductase, DFR)中多个保守域,属于SDR超家族(cl21454)。其功能位点分析发现,DFR中存在一个与NADPH结合的保守基序(V11-Y34)且含有26个氨基酸组成的底物特异结合保守基序(T135-K160);N134氨基酸是决定还原DHK活性的特异天冬酰胺位点(图4)。
通过BlastP软件与相近物种比对检索发现,卡特兰ChCHS1氨基酸序列与金钗石斛Dendrobium nobile(ABE77392)、白苇兰Bromheadia finlaysoniana(AAB62876)、蕙兰杂交种Cymbidium hybrid cultivar(AIM58717)、小兰屿蝴蝶兰Phalaenopsis equestris(AIS35912)高度同源,同源性分别达到96%、94%、94%、92%;与姜黄Curcuma longa(AEU17693)、油点草Tricyritis hirta(BAH16615)、葡萄Vitis vinifera(BAB84112)、美丽百合Lilium speciosum(BAE79201)、福斯特郁金香Tulipa fosteriana(AGJ50587)、银白杨Populus alba(ABC86919)、牡丹Paeonia suffruticosa(AIU98510)有较高的同源性,分别为85%、85%、85%、84%、84%、82%、82%。利用BioEdit软件将卡特兰ChCHS1与其他11个物种的CHS蛋白氨基酸序列进行多重比对,结果见图5。
卡特兰ChDFR1基因编码的氨基酸序列与白苇兰Bromheadia finlaysoniana(AAB62873)、细茎石斛Dendrobium moniliforme(AEB96144)、文心兰杂交种Oncidium hybrid cultivar(AAY32602)、蕙兰杂交种Cymbidium hybrid cultivar(AAC17843)、朵丽蝶兰× Doritaenopsis hybrid cultivar(AHA36975)、小兰屿蝴蝶兰Phalaenopsis equestris(AIS35914)同源性较高,分别为88%、86%、84%、84%、82%、82%;与葡萄风信子Muscari armeniacum(AIC33028)、淫羊藿Epimedium sagittatum(AFU90826)、普通小麦Triticum aestivum(BAD11018)、姜荷花Curcuma alismatifolia(ADK62520)也有较高的同源性,分别达到71%、71%、70%、68%。利用BioEdit软件将卡特兰ChDFR1与其他10个物种的DFR蛋白氨基酸序列进行多重比对(图6)。
基于不同植物的CHS基因的氨基酸序列,用Mega 6.06软件构建了ChCHS1蛋白的分子系统树,如图7所示,卡特兰ChCHS1与同科中的蕙兰杂交种、兰花、小兰屿蝴蝶兰、蝴蝶兰关系最近,分为同一分支;与石斛、细茎石斛、文心兰同属一大簇;在同科中与文心兰关系较远。相比之下,与单子叶植物中的凹唇姜、姜黄、油点草、美丽百合、福斯特郁金香亲缘关系较近,与其他双子叶植物关系较远。
为进一步了解ChDFR1基因的进化关系,选择已知植物的DFR氨基酸序列进行系统进化分析,结果表明(图8),卡特兰与石斛兰杂交种亲缘关系最近,聚为同一小分支,在同科中与朵丽蝶兰、小兰屿蝴蝶兰、白苇兰、文心兰、蕙兰亲缘关系较远;相比之下,与福斯特郁金香、美丽百合、洋葱、旱花百子莲、葡萄风信子、风信子、姜荷花关系较近,同属一大簇;与长穗薄冰草、普通小麦、拟山羊草、玉米、淫羊藿、牡丹关系较远,属于两大簇。
2.3 ChCHS1及ChDFR1基因的表达特性
对ChCHS1及ChDFR1基因在花朵开放过程中的表达模式进行分析发现,两者在花序早期发育时表达量均极低,随着花发育进程表达量呈上升趋势。ChDFR1基因在花蕾即将开放时表达量达到最大,在盛开期表达量略有下降。而ChCHS1基因的表达在花蕾发育进程中一直呈现明显上升趋势,直至盛开时达到最高。另外,对ChCHS1与ChDFR1基因表达量进行比较发现,前3期ChCHS1与ChDFR1基因表达量基本相同,而在花蕾即将开放时及盛开期ChCHS1基因的表达量明显高于ChDFR1基因(图9)。
3 讨论与结论
本研究通过RT-PCR和RACE方法从卡特兰花瓣中成功分离出CHS和DFR两个类黄酮代谢途径中的关键基因进行同源克隆。基因序列及氨基酸序列比对分析发现,这2个基因的编码区与目前已知的相应基因的氨基酸序列都有很高的同源性,在不同科植物间核苷酸水平上的同源性超过70%;在氨基酸水平上ChCHS1基因编码氨基酸的同源性超过80%,ChDFR1基因编码氨基酸同源性超过70%,这表明本次试验克隆结果可靠,ChCHS1与ChDFR1分别属于其基因家族的一员。系统进化树分析表明,这2个基因的进化具有较明显的种属特性,分别与百合科、姜科亲缘关系近;在同科之间的进化关系不太一致,在ChCHS1基因的进化进程中,文心兰与卡特兰关系较远,而文心兰ChDFR1基因与卡特兰关系较近;但卡特兰ChCHS1和ChDFR1基因都与蕙兰和石斛兰关系较近。Rasika[20]克隆了石斛兰(Dendrobium Jaquelyn Thomas)的DFR基因并构建系统进化树,发现其亲缘关系与白苇兰、蕙兰、百合关系较近,与本试验结果相似。
Mudalige-Jayawickrama等[21]用Northern blot方法对CHS和DFR基因在石斛兰花器官开放进程中不同时期的表达差异进行分析发现,这2个基因在中等蕾期时表达量最高,在开放期时几乎无法检测到其表达。Hieber等[22]发现,DFR基因的表达贯穿文心兰花朵发育整个进程,无论在蕾期还是开放期,其表达水平一直为上调。而在白苇兰中[11],DFR基因的表达仅在幼嫩叶片中,在花器官中表达量很低。Pitakdantham等[23]在2种紫色石斛兰的开花进程中发现,DFR基因花蕾接近开放时表达量高,在最小蕾期和盛开期不表达。Ying-ying等[24]从蝴蝶兰中分离了5个CHS家族基因,这5个CHS基因都在蕾期及开放期有表达,在盛开期时不表达。本试验通过相对实时荧光定量PCR的方法对CHS和DFR基因在卡特兰开花不同时期中的表达情况进行分析发现,这2个基因在每个时期都有表达,但表达水平有明显差异,ChCHS1基因随着花朵开放及着色,其表达含量逐渐升高,在盛开期时达到顶峰;ChDFR1基因在即将开放时达到最高,在盛开期时表达量略有下降。卡特兰CHS及DFR基因在花色素合成过程中表达量的变化,说明这2个基因的表达与花青素的合成有关,推测CHS及DFR可能在转录水平上对卡特兰花色的形成起到调控作用。
CHS和DFR基因分别为花色素苷生物合成途径中上游和下游的第一关键酶。目前,通过基因工程技术,利用CHS和DFR基因改良花色的研究在矮牵牛、非洲菊、月季、康乃馨、三色堇等观赏植物中均有报道,而具有丰富花色的兰科植物虽然是研究花色的理想材料[25],其分子育种却明显落后于其他观赏植物[21,26-28]。本试验成功分离了卡特兰2个花色苷合成途径中的关键基因,但如何在卡特兰中利用CHS与DFR改良其花色仍需要进一步研究。
参考文献
[1] Stephen Chandler, Yoshikazu Tanaka. Genetic Modification in Floriculture[J]. Critical Reviews in Plant Sciences, 2007, 26: 169-197.
[2] Nishihara M, Nakatsuka T. Genetic engineering of flavonoid pigments to modify flower color in floricultureal plants[J]. Biotechnol Lett, 2011, 33: 433-441.
[3] Dixon R A, Steele C L. Flavonoids and isoflavonoids-a gold mine for metabolic engineering[J]. Trends Plant Sci, 1999, 4: 394-400.
[4] Holton T A, Cornish E C. Genetics and biochemistry of anthocyanin biosynthesis[J]. Plant Cell, 1995, 7: 1 071-1 083.
[5] Mol J, Grotewold E, Koes R. How genes paint flowers and seeds[J]. Trends Plant Sci, 1998, 3: 212-217.
[6] Springob K, Nakajima J, Yamazaki M, et al. Recent advances in the biosynthesis and accumuation of anth-ocyanins[J]. Natural Products Report, 2003, 20: 288-303.
[7] Koes R, Verweij W, Quattrocchio F. Flavonoids: a colorful model for the regulation and evolution of biochemical pathways[J]. Trends Plant Sci, 2005, 10: 236-242.
[8] Reimold U, Kroeger M, Kreuzaler F, et al. Coding and 3'noncoding nucleotide sequence of chalcone synthase messenger RNA and assignment of amino acid sequence of the enzyme[J]. EMBO J, 1983, 2: 1 801-1 806.
[9] Holton T A, Brugllera F, Tanaka Y. Clong and expression of chalcone synthase from petunia hybrida[J]. Plant J, 1993, 4: 1 003-1 010.
[10] Sommer H, Saedler H. Structure of the chalcone synthase gene of Antirrhinummajus[J]. Mol Gen Genet, 1986, 202: 429-434.
[11] Liew C F, Goh C J, Loh C S, et al. Cloning and characterization of full-length cDNA clones encoding chalcone synthase from the orchid Bromheadia finlaysoniana[J]. Plant Physiol Biochem, 1998, 9: 647-655.
[12] 周兴文, 李纪元, 范正琪. 金花茶查尔酮合成酶基因全长克隆与序列分析[J]. 生物技术通报, 2011, 6: 58-64.
[13] 周 琳, 王 雁, 彭镇华. 牡丹查尔酮合酶基因Ps-CHS1的克隆及其组织特异性表达[J]. 园艺学报, 2010, 37(8): 1 295-1 302.
[14] Meyer P, Heidmann I, Forkmann G. A new petunia flower colour generated by transformation of mutant with a maize gene[J]. Nature, 1987, 330: 677-688.
[15] Tanaka Y, Fukul Y, Fukuchi-Mizutani M, et al. Moclecular cloning and characterization of Rosa hybrida dihydroflavonol 4-reductase gene[J]. Plant Cell Physiol, 1995, 36(6): 1 023-1 031.
[16] Holton T: Transgenic plants exhibiting altered flower colour and methods for producing the same[P]. PCT-International Patent Application NO. WO, 1996, 96/36716.
[17] 王 雁, 陈振皇, 郑宝强, 等. 卡特兰[M]. 北京: 中国林业出版社, 2012: 160-286.
[18] Ferrer J L, Jez J M, Bowman M E, et al. Structure of chalcone synthase and the molecular basis of plant polyketide biosynthesis[J]. Nat Struct Biol, 1999, 6(8): 775-784.
[19] Michacl B, Austin Joseph P. The chalcone synthase superfamily of type III polyketide synthases[J]. Nat Struct Biol, 2003, 20(1): 79-110.
[20] Mudalige R G. Dendrobium flower color: histology and genetic manipulation[D]. University of Hawaii, 2003.
[21] Mudalige R G, Kuehnle A R. Orchid biotechnology in production and improvement[J]. HortScience, 2004, 39: 11-17.
[22] Hieber A D, Mudalige-Jayawickrama R G, Kuehnle A R. Color genes in the orchid Oncidium Grower Ramsey: identification, expression, and potential genetic instability in an interspecific cross[J]. Planta, 2006, 223: 521-531.
[23] Pitakdantham W, Sutabutra T, Chiemsombat P. Isolation and characterization of dihydroflavonol 4-reductase gene in dendrobium flowers[J]. Journal of Plant Sciences, 2011, 6(2): 88-94.
[24] Han Y Y, Ming F, Wang J W, et al. Molecular evolution and functional specialization of chalcone synthase superfamily from Phalaenopsis Orchid[J]. Genetica, 2006, 128: 429-438.
[25] Hao Yu, Chong Jin Goh. Molecular genetics of reproductive biology in orchids[J]. Plant Physiology, 2001, 127(4): 1 390-1 393.
[26] Kuehnle A R, Lewis D H, Markham K R, et al. Floral flavonoids and pH in Dendrobium orchid species and hybrids[J]. Euphytica, 1997, 95: 187-194.
[27] Holton T A, Brugllera F, Tanaka Y. Cloning and expression of chlacone sythase from petunia hybrida[J]. Plant J, 1993, 4:1 003-1 010.
[28] Wen-Huei Chen, Chi-Yin Hsu, Hao-Yun Cheng, et al. Downregulation of putative UDP-glucose: flavonoid 3-O-glucosyltransferase gene alters flower coloring in Phalaenopsis[J]. Plant Cell Rep, 2011, 30: 1 007-1 017.
