应用Label-free技术研究人牙囊细胞和牙周膜成纤维细胞的差异蛋白质
2015-04-20弓青霞王丽颖金作林
弓青霞,宋 扬,刘 佳,王丽颖,金作林
(陕西西安:1.军事口腔医学国家重点实验室,陕西省口腔医学重点实验室,第四军医大学口腔医院正畸科,710032;2.解放军第323医院口腔科,710001)
应用Label-free技术研究人牙囊细胞和牙周膜成纤维细胞的差异蛋白质
弓青霞1,宋 扬2,刘 佳1,王丽颖1,金作林1
(陕西西安:1.军事口腔医学国家重点实验室,陕西省口腔医学重点实验室,第四军医大学口腔医院正畸科,710032;2.解放军第323医院口腔科,710001)
目的:应用Label-free蛋白定量技术研究牙囊细胞(hDFCs)和牙周膜成纤维细胞(hPDLFs)差异表达蛋白质,为发现hDFCs向hPDLFs转化过程中的重要蛋白质提供参考。方法:应用Label-free蛋白定量技术,结合ultramate 3000 nano-UPLC软件,对hDFCs和hPDLFs总蛋白提取液进行蛋白定性定量检测。然后分别对蛋白质的鉴定结果行重复性、关联性、蛋白相对含量和差异表达蛋白等进行分析;对总体蛋白质和差异表达蛋白质行生物信息学GO或KEGG分析。结果:hDFCs和hPDLFs蛋白相对含量及总体蛋白GO分析结果相似。定量分析显示,当hDFCs分化形成hPDLFs后,有22个差异表达蛋白质,其中下调蛋白15个(P<0.05,FC>2),上调蛋白7个(P<0.05,FC>2)。结论:hDFCs和hPDLFs蛋白质表达谱相似;22个差异表达蛋白质为研究hDFCs向hPDLFs的转化机制提供了方向。
牙囊细胞(hDFCs);牙周膜成纤维细胞(hPDLFs);Label-free定量蛋白组学
[Chinese Journal of Conservative Dentistry,2015,25(5):270]
从发育的角度讲,钟状期牙胚的牙乳头细胞迁移至牙囊,并成为最早的牙囊细胞(human dental follicle cells,hDFCs)[1],而后者将成为形成牙周组织的始祖细胞。牙周膜成纤维细胞(human periodontal ligament fibroblasts,hPDLFs)作为hDFCs终末分化细胞之一,其转化过程中涉及复杂的基因、蛋白质表达变化及相应细胞的代谢改变。例如:BMP3是成骨分化和骨形成的负调节蛋白,在hDFCs向牙周组织发育后期的细胞中表达上调,通过拮抗BMP2/BMP7的成骨分化和骨形成作用,以维持牙周膜内非矿化状态[2],这与Lee等[3]报道的牙周膜中BMP3的表达水平高于牙囊相一致;牙周膜形成初期,牙囊中的间充质细胞获得极性,增殖分泌功能增加,形成最早的胶原纤维和糖蛋白基质[4],其他纤维成分如耐酸纤维的早期形成也与hDFCs相关[5],而牙根发育完成后,hPDLFs则成为继hDFCs后实现胶原降解、合成和牙周膜更新改建的主要动力细胞[6-8]。以上研究虽使我们对hDFCs-hPDLFs转化过程中的单个基因、蛋白质、细胞因子的表达差异或细胞生命活动有了一定的认识,但关于总体蛋白水平差异的报道极少。
蛋白质组学技术的快速发展为我们对蛋白质进行大规模的定性、定量研究提供了方法。其中蛋白定量分析可用以筛选和寻找任何因素引起的不同样本间蛋白质的差异表达,并能对某些关键蛋白质进行定性、定量分析,从而可用于分析干细胞不同分化阶段的蛋白质组差别。鉴于hDFCs和hPDLFs在生长发育上有一定的延续性,细胞分化处于不同阶段,本实验拟应用Label-free蛋白定量技术研究hDFCs和hPDLFs在分化前后的重要差异蛋白质,以期为研究hDFCs向hPDLFs的转化机制提供线索。
1 材料和方法
1.1 主要试剂和仪器
α-MEM培养基、胎牛血清(Gibco,美国);I型胶原酶、2.5 g/L胰蛋白酶(Sigma,美国);细胞总蛋白裂解液、BCA试剂盒(武汉博士德);羊抗兔IgG/FITC、兔抗人波形蛋白、兔抗人角蛋白(北京博奥森);SDS-PAGE凝胶配制试剂盒(上海碧云天);考马斯亮蓝R250(西安国安生物科技);CO2孵箱(Forma,美国);LTQ orbitrapVelos液质联用仪(Thermo,美国)戴安ultramate 3000 nano-UPLC分析软件(Xcalibur,2.2 SP1);垂直板电泳槽(Bio-rad,美国);荧光显微镜(奥林巴斯,日本);凝胶成像仪(Bio-Best,美国)。
1.2 方法
1.2.1 hDFCs、hPDLFs的分离培养
选取12~16岁志愿者因正畸减数而新鲜拔除的前磨牙10个(均无牙体、牙周疾病),立即用PBS冲洗并刮除其冠1/3和根尖1/3的牙龈或牙周膜后,分别置于I型胶原酶中37℃条件下全牙消化3 h。同时选取来自不同患者(均知情同意)、X线显示第三磨牙阻生且为牙胚状态的牙囊10个,将牙囊组织从牙冠表面小心分离后,立即用PBS冲洗,并将其修剪成1 mm×1 mm×1 mm的组织块,然后置于I型胶原酶中37℃消化30min。牙囊和离体牙均按照收集顺序随机分配到hPDLFs1、hPDLFs2组或hDFCs1、hDFCs2组,以便于质谱分析。
上述消化结束后,分别收集两种组织的消化液,并离心弃上清;将各组织的沉淀部分滴加适量α-MEM完全培养液后,分别接种于24孔板,置于37℃、50mL/L CO2、饱和湿度条件下进行培养。待细胞从组织块边缘爬出(7~8 d),并爬满孔底80%时,胰酶消化传代,取2~3代细胞用于后续实验。
1.2.2 hDFCs、hPDLFs的免疫荧光鉴定
取3代hDFCs、hPDLFs分别制备成单层细胞爬片后,40 g/L多聚甲醛固定15 min;PBS冲洗,PBS-Triton透膜20 min;复合消化酶消化3min,5 g/mL BSA室温封闭30 min。然后分别在hDFCs、hPDLFs爬片上各滴加兔波形蛋白抗体(1∶100)、兔角蛋白抗体(1∶100)4℃过夜。18 h后,避光条件下滴加羊抗兔IgG/FITC(1∶200),37℃孵育20min;PBS冲洗,滴加DAPI(1∶200)室温孵育7min;封片,荧光显微镜观察。
1.2.3 hDFCs、hPDLFs总蛋白的SDS-PAGE电泳
提取hDFCs、hPDLFs总蛋白质,分别测定其蛋白浓度;将6×Loadingbuffer与各蛋白质提取液按1∶5混合并沸水浴5 min后置于冰上备用;配置120 g/L的SDS-PAGE胶体,连接垂直板电泳槽;两种细胞蛋白分别取20、35、50 g,加入梳孔,并同时上样5μL蛋白Marker(10~170KD)进行蛋白质凝胶电泳(压缩胶60 V、30 min,分离胶100 V、150min)。电泳结束后,考马斯亮蓝染色过夜,洗脱液漂洗胶体后凝胶成像仪照相。
1.2.4 hDFCs、hPDLFs总蛋白质色谱质谱检测及定性定量分析
分别取hDFCs1、hDFCs2、hPDLFs1、hPDLFs2 4种蛋白样品,经预处理后用LTQ orbitrap Velos液质联用仪及戴安ultramate 3000 nano-UPLC软件进行分析。上机量分别为10μL,首先进入保护柱(C18 PepMap100,300μm×1mm,5μm,100Å),随后进入分析柱(Acclaim PepMap C18,15 cm×75μm,2μm,100Å,Dionex),并以流速0.2μL/min进行分析;流动相B梯度为5%~45%,相应的流动相A梯度是95%~55%(流动相B:乙腈、1.23 g/L甲酸;流动相A:超纯水、1.23 g/L甲酸),检测时间为125 min。质谱参数:阳离子模式,CID碰撞模式,120 000分辨率,质量范围350~1 800,NCE为35%,top16离子强度用于串联质谱(tandem mass spectrometry,MS/MS)分析。
Orbitrap原始质谱数据使用maxquant(1.5.0.12)和Andromeda软件,并参考IPI人蛋白质数据V3.87对蛋白质进行定性。定性检索方法为:酶切类型为胰蛋白酶全酶切,母离子质量误差为±10 ppm,固定修饰为Carbamidomethylation(半胱氨酸),可变修饰为G1cNAc(天冬酰胺),Carbamidomethylation(赖氨酸,肽段N端)和Oxidation(蛋氨酸),IPI一个蛋白匹配的多肽最少长7个氨基酸,肽段和蛋白FDR<0.01,最少匹配unique peptide数目为1。同时以LFQ和iBAQ两参数进行蛋白定量,其中LFQ的对数(log2)用于后续蛋白定量分析。
1.2.5 hDFCs和hPDLFs蛋白组分析方案
首先用SPSS 16.0统计软件计算相同来源样本的生物学重复性及定量数据的关联性。
然后采用DAVID(http://david.abcc.ncifcrf.gov/)和WEGO(http://wego.genomics.org.cn/cgib-in/wegoindex.pl)对hDFCs和hPDLFs的总体蛋白质进行GO分类分析。
用SPSS 16.0统计软件计算hDFCs和hPDLFs中相同蛋白质LFQ值对数(log2)的比值,用以考察蛋白比例的分布;同时对相同蛋白LFQ值的对数(log2)进行t检验,分析蛋白表达量的差异,并以P<0.05和倍性变化FC>2作为最终差异蛋白。
采用DAVID和WEGO对所有蛋白和差异蛋白进行GO分类分析,研究差异蛋白和所有蛋白GO term的异同。采用DAVID对所有差异蛋白进行KEGG Path-way分析。
2 结果
2.1 hDFCs和hPDLFs的鉴定结果
免疫荧光检测显示,间充质来源的hDFCs和hPDLFs均阳性表达波形丝蛋白,阴性表达角蛋白(图1)。

图1 hDFCs和hPDLFs细胞角蛋白和波形丝蛋白的表达(×10)
2.2 SDS-PAGE电泳结果
hDFCs和hPDLFs的细胞总蛋白质分布均匀,均无极高丰度的蛋白质,符合色谱质谱检测对蛋白丰度的要求;等上样量时在10~170 KD二者具有相似的蛋白条带(图2)。
2.3 hDFCs和hPDLFs的蛋白质质谱鉴定
在样本hPDLFs1的二级质谱(MS/MS)中鉴定出2 305个肽段,其中可识别的唯一肽段有1 613个,这些唯一肽段在IPI人蛋白质数据库中可以匹配到565种蛋白质上,其他样本质谱结果见表1。4样本共鉴定出907个蛋白组。

表1 hDFCs1、hDFCs2、hPDLFs1、hPDLFs2的质谱鉴定

图2 hDFCs和hPDLFs的SDS-PAGE电泳
2.4 hDFCs1和hDFCs2、hPDLFs1和hPDLFs2的生物学重复性及数据关联性分析
hDFCs1和hDFCs2共有68.1%和73.3%的蛋白数量一致,样本间蛋白定量的关联性为r=0.73;hPDLFs1和hPDLFs2共有84.3%和65.3%蛋白数量一致,关联性r=0.87(图3)。提示,样本的生物学重复检测良好,同种样本间一致程度较高。

图3 生物学重复性(左)和数据关联性(右)
2.5 hDFCs和hPDLFs中相同蛋白比例的分布情况
SDS-PAGE显示,两细胞间相同分子量区域内蛋白质的丰度极其相似,故对两细胞同一种蛋白质的定量数据(LFQ值)的比值进行柱状图分析;从图中可以看出,其蛋白的比例呈正态分布,大多数蛋白的比例均位于1[log2(hPDLCs/hDFCs)=0]附近(图4)。

图4 相同蛋白比值分布
2.6 hDFCs和hPDLFs总体蛋白的GO分析
GO分析显示,两种细胞的总蛋白质在大多数GO分类项目(细胞成分、分子功能、生物过程及其子分类)下非常相似,只有在生物过程下的rhythmic process(节律性过程)中hDFCs较hPDLFs有更多相关蛋白表达(图5)。
2.7 差异蛋白分析
对hDFCs和hPDLFs中相同蛋白质进行定量分析后显示,22个蛋白质表达量的差异有统计学意义(P<0.05,FC>2),其中下调蛋白质15个(AKR1A1、ALCAM、CDC42、CTGF、CYR61、DDX1、DDX17;DDX5、DDX39A、F2、FKBP4、FST、GDI1;GDI2、MAT2A、RPL18A、TOMM6),上调蛋白质7个(DRAP1、ECI1、EIF3G、HSP90B1、NIT1、PPP1CB、STX7)(图6)。

图5 hDFCs和hPDLFs总体蛋白GO分析
2.8 差异蛋白和总体蛋白的GO分析
使用DAVID分析软件将所有蛋白质和差异蛋白质向GO数据库的各节点映射后,可见CTGF,CYR61(分化后均下调)在分子功能上富集于胰岛素样生长因子结合蛋白(图7)。
2.9 差异蛋白质KEGG分析
22个差异蛋白质中只有3个对比到KEGG Pathway,分别为CDC42、F2、PPP1CB,参与细胞骨架调节相关通路(图8)。
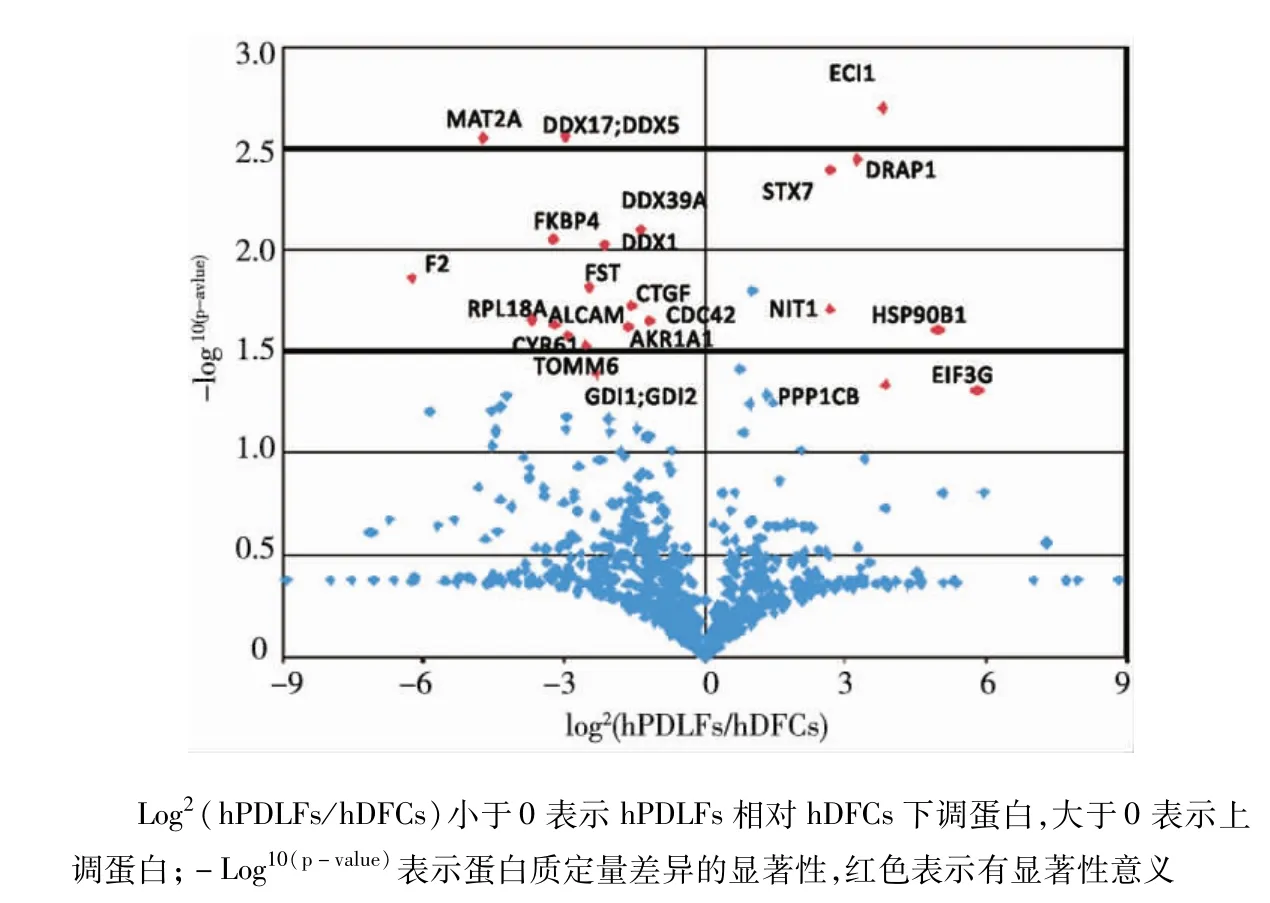
图6 差异蛋白质表达谱

图7 差异蛋白和总体蛋白的GO分析

图8 差异蛋白质CDC42、F2、PPP1CB的KEGG分析
3 讨论
差异蛋白质组学最初应用于干细胞相关研究,可分析不同来源干细胞、干细胞诱导分化前后蛋白质表达的变化[9-11]。hPDLFs是hDFCs最重要的终末分化细胞之一,二者虽然在细胞形态、免疫荧光及总体蛋白表达谱上均有相似性,但却处于分化不同阶段,是两种具有组织学延续性的不同细胞。本实验借助Label-free蛋白定量技术分析了hPDLFs和hDFCs在蛋白表达水平的相似性和差异性,并观察了hDFCs成纤维分化前后细胞蛋白表达的改变。
为提高蛋白质定性定量的准确性,本实验选择了Label-free蛋白定量技术,该方法区别于双向电泳结合质谱法、稳定同位素标签法,是后基因时代发现蛋白质微量变化中较为精确的方法[12]。同种来源细胞设计样本重复,在蛋白质生物学重复性和定量数据相关性检验良好的前提下(图3),进行两细胞总蛋白谱和差异蛋白质分析。
hDFCs/hPDLFs各蛋白比例的柱状图分析显示,绝大多数蛋白的比值分布于1附近,即hDFCs、hPDLFs虽然处于分化不同阶段,但是两种细胞在蛋白质种类及含量上非常相似,多数蛋白质含量无明显差异。GO分析进一步证实,两种细胞总蛋白质在细胞成分、分子功能、生物过程及其相应term中多数无明显差异。除去个别差异表达蛋白质(图6),以上结果提示hPDLFs与hDFCs总蛋白谱在蛋白种类、含量、功能上具有极大的相似性。
差异蛋白质ALCAM、CTGF、CYR61在hPDLFs中下调表达,可能是hDFCs分化后干性降低的标志。CTGF、CYR61在分子功能上属于胰岛素样生长因子结合蛋白,在胚胎发育期各种结缔组织细胞外基质合成中发挥重要作用。CYR61能促进成骨、成软骨分化,愈合中的骨痂较正常骨高表达[13]。咀嚼力刺激可诱导牙周膜内 CTGF上调[14];CTGF可促进成骨细胞和软骨细胞的增殖分化及骨基质矿化[15]。CYR61和CTGF在胚胎发育早期多种间充质来源的组织中有随组织发育表达降低的趋势[16]。ALCAM的表达蛋白质是活化白细胞粘附因子CD166,作为干细胞标志物在hDFCs和hPDLFs中都有表达[17-18]。由此推测ALCAM、CTGF、CYR61的下调表达,可能是hDFCs逐渐丧失胚胎期干性并最终分化形成hPDLFs的结果,但这需要对牙囊-牙周膜发育过程行进一步免疫组化验证。
差异蛋白质中CDC42、F2、PPP1CB可以对比到细胞骨架调节相关通路。CDC42可以激活蛋白激酶(PAK)通路,从而调控细胞骨架的结构重塑。PPP1CB是蛋白磷酸酶1(PP1)的β催化亚基,后者参与数千种蛋白质的去磷酸化,与细胞分裂密切相关。F2为凝血酶原,是凝血酶的前体,后者与蛋白酶激活受体结合后具有蛋白酶活性,可以刺激成纤维细胞聚集和收缩[19]。由于牙周膜发育过程中伴随咬合力刺激,hDFCs在分化形成hPDLFs的同时,也由牙囊期的细胞无规则散在排列,逐渐获得极性,演变为沿牙周膜胶原纤维改建方向排列,建牙合不同时期成纤维细胞的长轴与牙根间的夹角不同[20];此外在牙周膜发育完成后,机械力刺激可以进一步诱导hPDLFs的细胞骨架沿机械力方向重排[21]。hPDLFs为适应咀嚼力刺激自发进行细胞骨架调节的生物学行为区别于hDFCs,这可能是hDFCs和hPDLFs出现细胞骨架调节相关蛋白表达差异的主要原因。
[1]Osborn JW,Price DG.An autoradiographic study of periodontal development in themouse[J].JDent Res,1988,67(2):455-461.
[2]Kémoun P,Laurencin-Dalicieux S,Rue J,etal.Localization of STRO-1,BMP-2/-3/-7,BMP receptors and phosphorylated Smad-1 during the formation ofmouse periodontium[J].Tissue Cell,2007,39(4):257-266.
[3]Lee HS,Lee J,Kim SO,etal.Comparative gene-expression analysis of the dental follicle and periodontal ligament in Humans[J].PLoSOne,2013,8(12):e84201.
[4]Cho MI,Garant PR.Development and general structure of the periodontium[J].Periodontol2000,2000,24:9-27.
[5]Inoue K,Hara Y,Sato T.Development of the oxytalan fiber system in the ratmolar periodontal ligament evaluated by light-and electron-microscopic analyses[J].Ann Anat,2012,194(5):482-488.
[6]Konstantonis D,Papadopoulou A,Makou M,et al.Senescent human periodontal ligament fibroblasts after replicative exhaustion or ionizing radiation have a decreased capacity towards osteoblastic differentiation[J].Biogerontology,2013,14(6):741-751.
[7]Rentz TJ,Poobalarahi F,Bornstein P,et al.SPARC regulates processing of procollagen Iand collagen fibrillogenesis in dermal fibroblasts[J].JBiol Chem,2007,282(30):22062-22071.
[8]Trombetta JM,Bradshaw AD.SPARC/Osteonectin functions to maintain homeostasis of the collagenous extracellular matrix in the periodontal ligament[J].J Histochem Cytochem,2010,58(10):871-879.
[9]Mrozik KM,Zilm PS,Bagley CJ,et al.Proteomic characterization ofmesenchymal stemcell-like populations derived from ovine periodontal ligament,dental pulp,and bone marrow:analysis of differentially expressed proteins[J].Stem Cells Dev,2010,19(10):1485-1499.
[10]Pivoriuūnas A,Surovas A,Borutinskaite V,et al.Proteomic analysis of stromal cells derived from the dental pulp of human exfoliated deciduous teeth[J].Stem Cells Dev,2010,19(7):1081-1093.
[11]Morsczeck C,Petersen J,Völlner F,et al.Proteomic analysis of osteogenic differentiation of dental follicle precursor cells[J].E-lectrophoresis,2009,30(7):1175-1184.
[12]Trinh HV,Grossmann J,Gehrig P,et al.iTRAQ-based and label-free proteomics approaches for studies of human adenovirus infections[J].Int JProteomics,2013,2013:581862.
[13]Hadjiargyrou M1,AhrensW,Rubin CT.Temporal expression of the chondrogenic and angiogenic growth factor cyr61 during fracture repair[J].JBoneMiner Res,2000,15(6):1014-1023.
[14]Choi JW,Arai C,Ishikawa M,et al.Fiber system degradation,and periostin and connective tissue growth factor level reduction,in the periodontal ligament of teeth in the absence ofmasticatory load[J].JPeriodontal Res,2011,46(5):513-521.
[15]Kanaan RA,Aldwaik M,Al-HanbaliOA.The role of connective tissue growth factor in skeletal growth and development[J].Med SciMonit,2006,12(12):RA277-281.
[16]Ponticos M.Connective tissue growth factor(CCN2)in blood vessels[J].Vascul Pharmacol,2013,58(3):189-193.
[17]Yalvac ME,Ramazanoglu M,Gumru OZ,et al.Comparison and optimisation of transfection of human dental follicle cells,a novel source of stem cells,with different chemicalmethods and electroporation[J].Neurochem Res,2009,34(7):1272-1277.
[18]Nagatomo K,Komaki M,Sekiya I,et al.Stem cell properties of human periodontal ligament cells[J].J Periodontal Res,2006,41(4):303-310.
[19]Chang MC,Chan CP,Wu HL,et al.Thrombin-stimulated growth,clustering,and collagen lattice contraction of human gingival fibroblasts is associated with its protease activity[J].JPeriodontol,2001,72(3):303-313.
[20]Popowics T,Boyd T,Hinderberger H.Eruptive and functional changes in periodontal ligament fibroblast orientation in CD44 wild-type vs.knockout mice[J].J Periodontal Res,2014,49(3):355-362.
[21]Pan J,Wang T,Wang L,et al.Cyclic strain-induced cytoskeletal rearrangement of human periodontal ligament cells via the Rho signaling pathway[J].PLoSOne,2014,9(3):e91580.
The differential proteom ics of human dental follicle cells and human periodontal ligament fibroblasts analysed w ith Label-free technology
GONG Qing-xia*,SONG Yang,LIU Jia,WANG Li-ying,JIN Zuo-lin
(*State Key Laboratory Of Military Stomatology,Department of Orthodontics,School of Stomatology,The Fourth Military Medical University,Shaanxi Key Laboratory of Stomatology,Xi′an 710032,China)
AIM:To identify the differentially expressed proteins of human dental follicle cells(hDFCs)and human periodontal ligament fibroblasts(hPDLFs)with Label-free protein quantification technique.METHODS:Combined with ultramate 3000 nano-UPLC software,Label-free protein quantification was used to examine the total protein extraction samples from hDFCs and hPDLFs.For the proteins identified,the repeatability,the correlation,the relative content of proteins and the differentially expressed proteins of hDFCs and hPDLFs were analyzed.Bioinformatic analysis of GO and KEGG was applied for the analyses of total proteins of hDFCs and hPDLFs,aswell as the differentially expressed proteins.RESULTS:The relative content of proteins and the results of GO analysis of hDFCs swere similar to those of hPDLFs.22 differentially expressed proteinswere identified,ofwhich 15 were down-regulated,7 were up-regulated.CONCLUSION:Label-free quantitative proteomics confirmed the similar proteins profile between hDFCs and hPDLFs.22 differentially expressed proteins provide clues for the transformation mechanism from hDFCs to hPDLFs.
human dental follicle cells(hDFCs);human periodontal ligament fibroblasts(hPDLFs);Labelfree quantitative proteomics
R780.2
A
1005-2593(2015)05-0270-07
10.15956/j.cnki.chin.j.conserv.dent.2015.05.003
2015-01-09;
:2015-03-24
国家自然科学基金面上项目(81271176)
弓青霞(1986-),女,汉族,河南人。硕士生(导师:金作林)
金作林,E-mail:zuolinj@fmmu.edu.cn
