黑腹果蝇线粒体转录终止因子DmTTF蛋白的生物信息学分析
2015-04-18张晓娟左绍远张海洋冯慕华
熊 伟,张晓娟 ,左绍远,张海洋,冯慕华
(1.大理大学基础医学院,云南大理 671000;2.云南省昆虫生物医药研发重点实验室,云南大理 671000;3.大理市第一人民医院,云南大理 671000)
黑腹果蝇Drosophila melanogaster,也称黑尾果蝇,是被人类研究得最彻底的生物之一。其名字源于它喜好腐烂的水果以及发酵的果汁。黑腹果蝇是一种原产于热带或亚热带的蝇种,易于培养,繁殖快,室温条件下,10 d就可以繁殖一代,使用经济,且只有4对染色体,易于遗传操作,有很多突变体可以利用,是遗传、发育、生理和行为等的研究方面最常见的研究对象之一。
线粒体转录终止因子(Mitochondrial Transcription Termination Factor,MTERF)是一类存在于多细胞生物中的高度保守的线粒体DNA结合蛋白,具有相似的亮氨酸拉链结构〔1〕。MTERF蛋白家族不仅在脊椎动物中存在,在昆虫、蠕虫等无脊椎动物中也存在,但目前在真菌中仍未发现与其同源的蛋白质〔1〕。黑腹果蝇线粒体转录终止因子(Drosophila melanogasterMitochondrial Transcription Termination Factor,DmTTF)是在无脊椎动物中克隆到的第一个MTERF蛋白超家族成员,分析和研究DmTTF有助于更好地理解黑腹果蝇线粒体基因转录过程和调控机制〔2〕。
黑腹果蝇属于双翅目,果蝇科。由于繁殖能力强的特点,黑腹果蝇经常被当做生命科学和医学研究的模式物种。到目前为止,对DmTTF的生物学研究已经有一定的进展,但是对其生物信息学分析还未见报道。为此,本研究采用生物信息学方法,对DmTTF的理化性质、亲水性/疏水性、保守结构域、二级结构、三级结构、蛋白质-蛋白质相互作用、与其它物种亲缘关系等方面进行预测和分析,为其后续研究奠定全面的理论基础。
1 材料与方法
1.1 材料 数据资料来源于FlyBase网站已经注册的黑腹果蝇线粒体转录终止因子DmTTF序列。黑腹果蝇CG18124基因编码DmTTF蛋白,其中mRNA序列登陆号为NM_135865.4,蛋白质序列登陆号为NP_609709.1。1.2 方法 利用ProtParam分析蛋白质理化性质;利用ProtScale分析蛋白质亲水性/疏水性;利用TMHMM分析蛋白质的跨膜区〔3〕;蛋白序列同源性、多序列比对及序列系统进化树分别由NCBI protein blast、ClustalX2.0、njplot等软件实现;利用PSORT II Server和Loctree预测蛋白质亚细胞定位;利用Smart在线软件和NCBI Conserved Domains数据库来分析保守区域〔4〕;Jpred和Swiss-Model分别预测蛋白质二级结构和三级结构;String则用来预测蛋白质的相互作用〔5〕。各数据库和软件的相关信息如表1。

表1 预测蛋白质结构和功能应用的网站及软件
2 结果与分析
2.1 DmTTF蛋白的理化性质预测和分析 使用ProtParam蛋白质理化性质预测网站对DmTTF蛋白进行理化性质预测。结果显示,黑腹果蝇CG18124基因编码的DmTTF蛋白质为410个氨基酸,含量最多的氨基酸为亮氨酸和精氨酸;蛋白质分子式C2148H3459N617O607S20,理论分子质量为48 281.0,理论等电点为9.62,说明DmTTF是碱性蛋白质;DmTTF蛋白的半衰期均达到了30 h;DmTTF蛋白的不稳定系数大于40,表明它不是稳定蛋白;脂肪系数为90.78,平均亲水系数(GRAVY)为-0.466,表明它是亲水性蛋白质。
2.2 DmTTF蛋白亲水性/疏水性预测和分析 利用蛋白质亲水性/疏水性在线预测工具ProtScale对DmTTF进行亲水性/疏水性分析(图1)。结果显示,DmTTF亲水性最强位点出现在67位,分值为-2.800,这个位点的氨基酸是谷氨酸(glutamic acid,E);DmTTF疏水性最强位点出现在161位,分值为1.644,这个位置的氨基酸是亮氨酸(leucine,L)。ProtScale的预测结果显示DmTTF的亲水肽链分布在整个氨基酸序列中,且明显多于疏水肽链,预测结果也证明它是一种水溶性蛋白质。
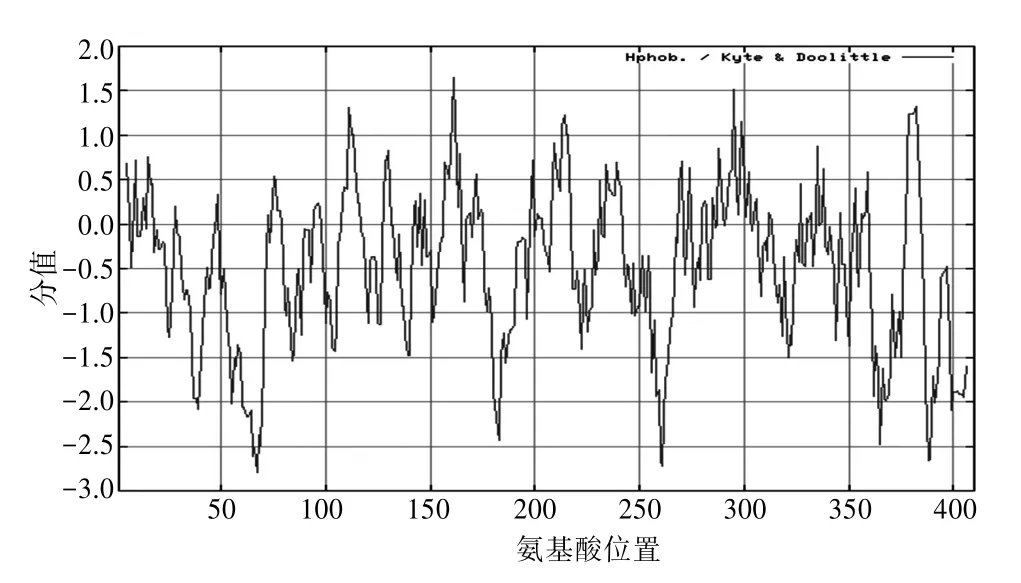
图1 DmTTF蛋白亲水性/疏水性分析(采用Kyte&Doolittle方法)
2.3 DmTTF蛋白的亚细胞定位分析 通过PSORT II工具预测DmTTF蛋白的亚细胞定位,结果表明DmTTF蛋白分布于线粒体、细胞核、细胞质和细胞骨架的可能性分别为56.5%、26.1%、13.0%和4.3%。Loctree在线软件预测结果表明该蛋白质定位于线粒体,所以DmTTF蛋白可能定位于线粒体中发挥作用。
2.4 DmTTF蛋白跨膜区预测和分析 使用在线蛋白质跨膜区分析工具TMHMM软件对DmTTF进行跨膜区分析,见图2。结果显示,DmTTF不含跨膜区,说明DmTTF不是跨膜蛋白质。MTERF蛋白家族是在线粒体DNA转录调控过程中发挥作用,属于线粒体定位,而非膜蛋白,预测结果与这一描述相符。
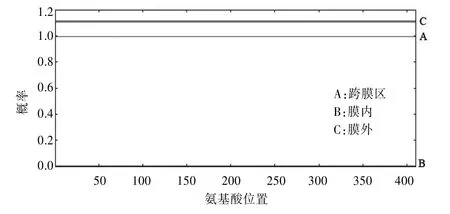
图2 DmTTF蛋白跨膜区预测
2.5 DmTTF蛋白的二级结构预测和分析 蛋白质的二级结构包括等结构组件,对其预测和分析有助于认识蛋白质的空间结构。Jpred软件由Barton Group创建于1998年,目前最新版本为3.0。Jpred采用Jnnet神经网络算法来预测蛋白质二级结构,平均准确率大于81%〔5〕。采用Jpred在线预测软件对DmTTF的二级结构进行预测,结果显示,DmTTF含有大量α-螺旋区和1个β-折叠区。无规则卷曲和β-折叠区氨基酸比率分别占到48.91%和0.80%,说明DmTTF大部分氨基酸(50.29%)处于α-螺旋状态。
2.6 DmTTF蛋白的保守结构域预测和分析 使用Smart在线软件对DmTTF进行保守结构域分析。结果表明DmTTF属于线粒体转录终止因子(MTERF)蛋白超家族,含有3个保守的Mterf结构域,分别位于122-152、314-345、346-376位氨基酸,此结构域由大约30个氨基酸组成,其中氨基酸序列同源性高,并且此保守结构域与NCBI提供的蛋白质保守结构域分析数据库(Conserved domains database)预测的保守区位置基本一致。
2.7 DmTTF蛋白的三级结构预测和分析 蛋白质三级结构预测与分析对了解蛋白质的结构与功能之间的相关性至关重要,也对理解该蛋白质与其他分子的相互作用机理及位点大有帮助。Swiss-Model是一款采用同源建模的方法预测蛋白质三级结构的在线软件,此外它还能够预测蛋白质电荷分布、原子间距离和角度等。将DmTTF蛋白的氨基酸序列提交到Swiss-Model,网站给出了预测结果,如图3。结果显示,DmTTF蛋白主要由α-螺旋和无规则卷曲构成,且与二级结构预测结果基本一致。模型评估使用全局法中的QMEANscore4计分值估算,模板(3mvb.1.A)序列建模方法使用X-ray,分辨率为2.79 Å,比对的范围在73~295aa,覆盖度达到74.0%,分析结果表明预测模型趋近于真实情况。
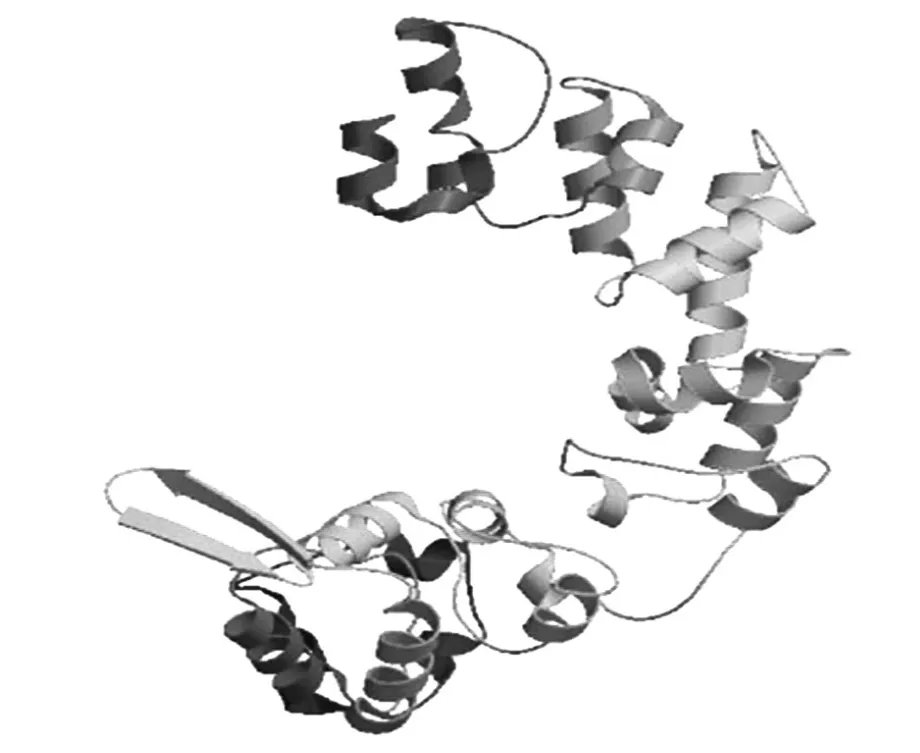
图3 DmTTF蛋白三级结构预测
2.8 DmTTF相互作用蛋白预测和分析 使用String 10.0蛋白质相互作用预测网站对DmTTF进行蛋白质相互作用预测,结果如图4。
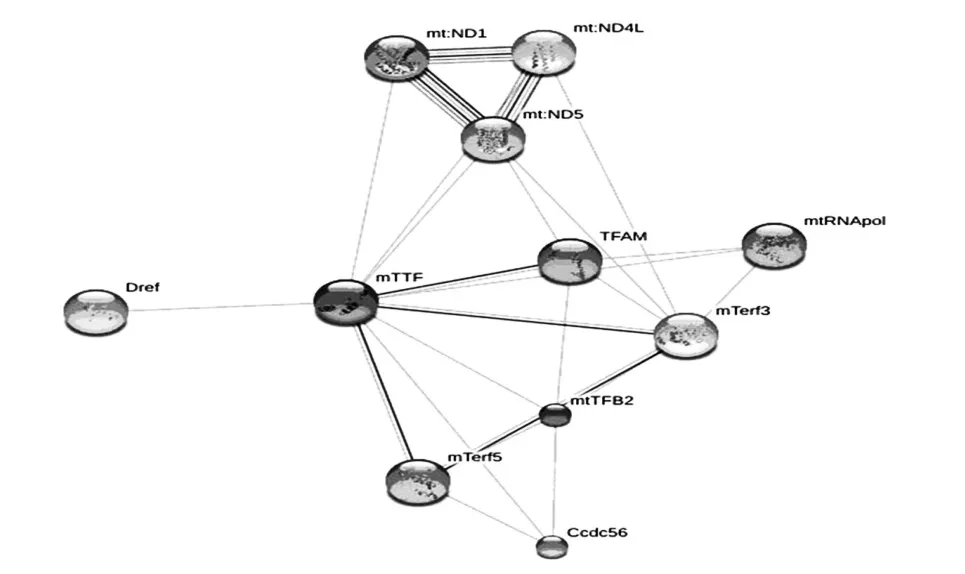
图4 DmTTF蛋白相互作用预测
与DmTTF相互作用最紧密的10个蛋白质及预测结果得分(可靠性)见表2。结果显示,与DmTTF相互作用最紧密的10个蛋白质均为线粒体DNA转录因子或线粒体基因组的组成部分,这与DmTTF参与线粒体基因转录调控的事实相符合。
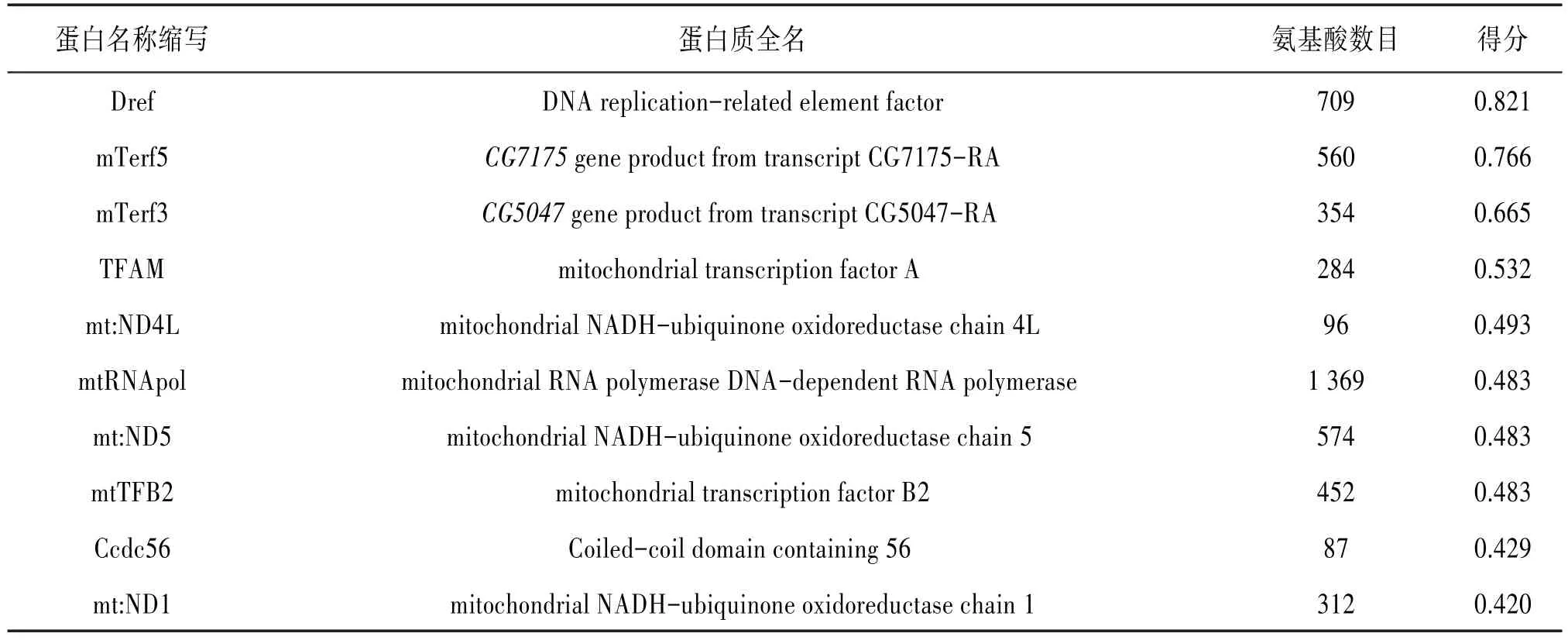
表2 10种最可能与DmTTF相互作用的蛋白质
2.9 DmTTF蛋白的同源性预测和系统进化树分析使用Protein Blast软件对DmTTF蛋白进行同源性分析,数据显示,家蝇Musca demestica、弓背蚁Camponotus flondanus、达氏按蚊Anopheles darlingi、大猩猩Gorilla gorilla gorilla、智人Homo sapiens的线粒体转录终止因子与DmTTF进行比较时,比对序列覆盖范围(Query cover)分别是100%、80%、73%、21%、21%;在此基础上的序列相似性(Identity)分别是54%、24%、41%、32%、30%。利用ClustalX2.1程序对DmTTF于上述物种同源的蛋白序列进行多重比对,进一步使用niprot软件对ClustalX2.1比对结果产生的系统进化树进行可视化分析,结果如图5。结果显示,DmTTF蛋白与家蝇同源蛋白之间进化距离最小,其次是达氏按蚊,与人和大猩猩的进化关系相对较远。另外,人和大猩猩的线粒体转录终止因子蛋白聚为一类,它们之间的进化距离最小。

图5 蛋白质系统进化树
3 讨论
线粒体转录终止因子蛋白主要在线粒体DNA的转录起始和转录终止阶段发挥作用,一方面它能与其它线粒体转录因子相互作用形成转录起始复合物〔6-7〕;另一方面它还能终止线粒体基因组的转录过程〔8-9〕。由于黑腹果蝇繁殖能力强、养殖费用低、突变体多等特点,果蝇被认定为是生命科学和医学研究的理想生物模型〔10〕。
本研究主要从生物信息学角度解析了黑腹果蝇线粒体转录终止因子DmTTF的部分信息。经研究发现,DmTTF蛋白全长410个氨基酸,属于线粒体转录终止因子蛋白超家族,不含跨膜区和信号肽序列,蛋白质亚定位于细胞线粒体。通过Smart软件预测DmTTF蛋白含有3个保守的Mterf基序,每个基序由大约30个氨基酸残基构成。进一步分析DmTTF的二级结构和三级结构,两种分析结果中α-螺旋和β-折叠的数量及出现位置基本一致,说明建模结果可靠,分析结果有助于阐明DmTTF与其他转录因子相互作用的空间结构基础。在研究DmTTF与其他蛋白的相互作用结果中,发现与DmTTF相互作用的蛋白均为线粒体转录因子和线粒体呼吸链蛋白〔11-12〕,其预测结果有文献和数据库两方面的证据,结果真实可靠。通过多重序列比对及构建蛋白质系统进化树发现,DmTTF蛋白与家蝇同源蛋白质的序列相似性最高,且两者之间的进化距离最近,说明该蛋白质的分子进化与物种的进化是一致的。
本研究的分析结果对于深入研究DmTTF蛋白在线粒体基因转录过程中的作用及其调控机制具有一定的指导意义。
〔1〕熊伟,余敏,左绍远.线粒体转录终止因子蛋白家族在线粒体基因表达中的调节作用〔J〕.中国生物化学与分子生物学报,2015,31(3):223-231.
〔2〕ROBERTI M,POLOSA P L,BRUNI F,et al.DmTTF,a novel mitochondrial transcription termination factor that recognises two sequences ofDrosophila melanogastermitochondrial DNA〔J〕.Nucleic Acids Res,2003,31(6):1597-1604.
〔3〕刘洪超,李玉秀,邹先明,等.黑腹果蝇CG18853基因编码蛋白的生物信息学分析〔J〕.生物技术,2014,24(4):42-46.
〔4〕熊伟,杨勇琴,张海洋,等.人线粒体转录终止因子1(hMTERF1)蛋白的生物信息学分析〔J〕.生物信息学,2015,13(1):23-30.
〔5〕杨少斌,冯婧文,赵文铖,等.斑马鱼TATA结合蛋白的生物信息学分析〔J〕.生命科学研究,2015,19(2):119-123.
〔6〕余敏,伍红,谭德勇.线粒体转录终止因子蛋白家族研究进展〔J〕.生命科学,2007,19(5):496-499.
〔7〕ROBERTI M,POLOSA P L,BRUNI F,et al.MTERF factors:a multifunction protein family〔J〕.Bio Mol Concepts,2010,1(2):215-224.
〔8〕ROBERTI M,FERNANDEZ-SILVA P,POLOSA P L,et al.In vitro transcription termination activity of the Drosophila mitochondrial DNA-binding protein DmTTF〔J〕.Biochem Biophys Res Commun,2005,331(1):357-362.
〔9〕ROBERTI M,BRUNI F,POLOSA P L,etal.TheDrosophila termination factor DmTTF regulates in vivo mitochondrial transcription〔J〕.Nucleic Acids Res,2006,34(7):2109-2116.
〔10〕占祖兵,张越,赵若苹,等.黒腹果蝇中嵌合新基因的进化命运和表达模式〔J〕.动物学研究,2011,32(6):585-595.
〔11〕FERNÁNDEZ-MORENO M A,BRUNI F,ADÁN C,et al.The Drosophila nuclear factor DREF positively regulates the expression of the mitochondrial transcription terminationfactorDmTTF〔J〕.BiochemJ,2009,418(2):453-462.
〔12〕BRUNI F,MANZARI C,FILICE M,et al.D-MTERF5 is a novel factor modulating transcription in Drosophila mitochondria〔J〕.Mitochondrion,2012,12(5):492-499.
