合浦珠母贝基因组微卫星富集文库的构建与分析
2015-04-11油九菊范嗣刚黄桂菊郝博飞陈飞飞喻达辉
油九菊 , 范嗣刚 黄桂菊 郝博飞 , 陈飞飞 , 喻达辉
(1.农业部南海渔业资源开发与利用重点实验室, 南海生物资源与开发利用协同创新中心, 中国水产科学研究院 南海水产研究所, 广东 广州 510300; 2.上海海洋大学 水产与生命学院, 上海 201306)
合浦珠母贝(Pinctada fucata), 又名马氏珠母贝(Pinctada martensiiDunker), 隶属于软体动物门(Mollusca)、瓣鳃纲(Lamellibranchia)、珍珠贝目(Pterioida)、珍珠贝科(Pteriidae)、珠母贝属(Pinctada)[1], 主要分布在中国的广东、广西、海南和台湾沿海[2], 是目前我国用于培育海水珍珠的主要贝种之一。合浦珠母贝所产珍珠即为历史上闻名遐迩的南珠, 是名贵装饰品, 在国际上久负盛名。同时,合浦珠母贝肉鲜美可食, 贝壳可加工成珍珠粉, 入药治病。因此, 合浦珠母贝具有重要的经济价值。
中国海水珍珠养殖基地主要集中在广东、广西、海南三省区沿海地带, 并成为其经济发展的支柱性产业之一, 为这些地区的经济发展和国家的出口创汇作出了巨大贡献[3]。然而, 由于在过去数十年的珍珠贝生产中, 大量使用近缘养殖贝作为父母本进行贝苗的培育, 导致了马氏珠母贝的种质急剧退化,养殖性能下降, 养殖成活率低, 个体变小, 育珠质量下降等的问题[3-4]。开展合浦珠母贝的良种选育工作,培育优良的育珠贝品系, 是科研人员当前的重要任务。
微卫星(microsatellite)又称为简单重复序列(simple sequence repeats, SSR), 是以1~6bp的短核苷酸为基本单位, 如(AC)n、(AT)n、(CAG)n、(AGCT)n等, 首尾相连组成的串联重复序列[5]。微卫星标记具有数量多、分布广、共显性遗传、多态性丰富、结果稳定等优点, 已被广泛用于动植物的种群鉴定、选择育种、遗传多样性分析及遗传连锁图谱的构建等方面[6-8]。
迄今, 有关合浦珠母贝微卫星序列的报道并不多, 得到的微卫星标记也很有限[9-12]。本研究拟采用磁珠富集法构建合浦珠母贝微卫星文库, 分离和筛选合浦珠母贝微卫星标记, 为合浦珠母贝的遗传选育、群体遗传学研究和遗传图谱的构建提供基础数据。
1 材料与方法
1.1 样品采集与基因组DNA提取
用于基因组微卫星文库构建的合浦珠母贝闭壳肌组织于2008年12月份采自海南三亚, 2011年3月份取合浦珠母贝三亚养殖群体闭壳肌组织, 用于多态性引物的筛选。样品均于95%酒精保存。按照《分子克隆实验指南》[13]中的方法提取基因组DNA。提取的基因组 DNA 样品用 1%琼脂糖凝胶电泳、EB染色检测, 并测定 OD260和 OD280, 检测 DNA 的质量和计算浓度, 配成50 ng/μL的DNA备用。
1.2 酶切获得目的片段
用限制性内切酶MseⅠ于 37℃烘箱酶切基因组DNA 4 h, 酶切体系 30 μL, 包括 3 μL10×NEB Buffer2, 0.3 μL 100×BSA, 1 μLMseⅠ(10U/μL), 4 μL 50ng/μL基因组DNA, 灭菌双蒸水补足。用1%琼脂糖凝胶电泳检测酶切结果。
1.3 接头连接与PCR扩增
用单链寡核苷酸合成双连接头, 每种寡核苷酸的浓度为 100 μmol/L。MseⅠ接头序列为 Olig A:5′-GACGATGAGTCCTGAG-3′, Olig B: 5′-TACTCA GGACTCAT- 3′。95℃变性10 min, 经4 h缓慢冷却至 10℃, 最终形成双链接头。将酶切产物与双链接头于 16℃连接过夜, 连接体系如下: 10×T4 DNA ligase Buffer (ATP+) 3 μL, 100 μmol/L 接头 3 μL, 上述酶切片段 20 μL, 400 U/μL 的 T4 DNA ligase 0.5 μL,灭菌双蒸水补足 30 μL。连接产物用简并引物MseⅠ-N [5′-GAT GAG TCC TGA GTA A(N)-3′] (N 代表碱基A、G、C、T)进行25 μL体系的PCR预扩增,包括 10×PCR buffer 2.5 μL, 10 mmol/L dNTPs 0.5 μL, 10 mmol/LMseⅠ-N 1 μL, 1 U Taq 酶, 酶切连接产物2 μL, 灭菌双蒸水补足体系。PCR程序为: 95℃变性30 s, 58℃退火1 min, 72℃延伸1 min, 共20个循环; 之后72℃延伸10 min, 4℃。1%琼脂糖电泳检测预扩增效果并扩大体系。去除多余的引物、dNTPs等, 将PCR产物浓缩至30 μL。
1.4 杂交并磁珠富集微卫星序列
1.4.1 杂交
100 μL杂交体系包括0.8 μL SSR 探针A(100 μmol/L)[为生物素标记的(AC)15寡核苷酸: 5′bio-(AC)153′],21 μL 20×SSC, 0.7 μL 10% SDS, PCR 回收产物 3 μL,灭菌双蒸水补足。混匀后95℃变性10 min, 迅速降至58℃, 温育20 min, 然后取出自然冷却到室温。
1.4.2 杂交期间磁珠的准备
取 300 μL 充分摇匀的 Streptavidin MagneSphere®链霉素亲和素顺磁颗粒(Promega)溶液于1.5 mL离心管中, 1 mL TEN100(10 mmol/L Tris-Cl,1 mmol/L EDTA, 100 mmol/L NaCl, pH 7.5)混匀清洗磁珠3~5 min, 于架上1 min, 弃上清, 重复2次, 40 μL TEN100悬浮磁珠。
1.4.3 亲和捕捉
杂交混合液与磁珠悬浮液混合, 加入 300 μL TEN100混匀。室温放置30 min得吸附混合液(其间轻柔混合磁珠)。将吸附混合液于磁架上静置1 min,弃上清, 400 μL松弛性洗涤液TEN1000(10 mmol/L Tris-Cl, 1 mmol/L EDTA, 1000 mmol/L NaCl, pH 7.5)洗涤5 min, 重复2次。400 μL严谨性洗涤液(0.2×SSC,0.1% SDS)洗涤5 min, 重复2次。洗涤后的磁珠悬浮于50 μL TE缓冲液中, 95℃变性5 min。磁架上迅速分离上清, 保存为洗脱液①。再向磁珠中加入12 μL NaOH(0.15 mol/L)混匀室温置 20 min, 分离的上清中加入1.8 μL HCl(1 mol/L), TE缓冲液补足50 μL体积, 保存为洗脱液②。核酸共沉剂沉淀回收两次的洗脱产物, 溶于20 μL灭菌双蒸水中, 即得到富含微卫星序列的单链短基因组DNA片段。以此片段为模板,MseI primer为引物, PCR体系及条件同1.3连接产物的扩增, 获得富含微卫星DNA的双链短基因组DNA片段, 检测富集效果。富集效果好的样品用DNA Gel Extraction Kit(AXYGEN)切胶回收纯化PCR产物, 浓缩至20 μL。1%琼脂糖电泳检测回收效果并定量。
1.5 连接T-载体、克隆
连接反应采用TaKaRa的pMD18-T试剂盒, 连接体系为 10 μL: Ligation Solution I 5 μL, PMD18-T Vector 0.5 μL, DNA 1 μL, DNA 片段 3 μL, 灭菌双蒸水补足。16℃连接 4 h。用E.coliCompetent CellsDH5ą(TaKaRa)进行转化, 得到基因组微卫星文库。
1.6 文库筛选、测序、序列特征分析与引物设计
挑选单克隆, 接种到含有氨苄抗生素的LB培养基中, 37℃振荡培养2 h, 菌液用载体PMD18-T的通用引物 M13-47[5′CGCCAGGGTTTTCCCAGTCACG AC-3′]、M13-48[5′-AGCGGATAACAATTTCACACA GGA-3′]和 寡 核 苷 酸 序 列 SSRm[5′-CACACACAC ACACACACACACAD-3′]进行PCR筛选鉴定阳性克隆, 并送到上海生工生物工程有限公司进行测序。
用 Chromas软件分析测序结果, 去掉载体序列和接头序列。用SSRHunter软件进行微卫星DNA序列查找: 6或6个拷贝以上的2碱基重复序列, 4或4个拷贝以上的3碱基重复序列, 3或3个拷贝以上的4碱基重复序列, 2或2个拷贝以上的6碱基重复序列。获得的微卫星序列根据Weber[14]提出的测序标准,对其核心序列进行类型划分。用Primer Premier 5.0软件对所得的微卫星序列设计引物并进行 PCR 扩增。PCR产物用8%非变性聚丙烯酰胺凝胶电泳检测。
2 结果
2.1 合浦珠母贝微卫星文库的构建
MseⅠ酶切片段主要集中在 200~1000 bp, 表明合浦珠母贝基因组DNA酶切结果理想, 经过PCR扩增、杂交、富集, 得到的洗脱液 PCR扩增产物纯化结果见图1, 利用此富集产物构建合浦珠母贝基因组微卫星文库。
图1 PCR产物纯化结果Fig.1 The purified PCR products
2.2 克隆与测序结果
本研究共获得微卫星文库约2100个菌落。产生两条或两条以上扩增带的插入片段可以推测为含有微卫星序列的阳性克隆。通过 PCR筛选, 得到候选克隆483个(图2)。对其中135个克隆进行测序, 测序结果分析发现有 122个克隆含有重复次数大于或等于3的微卫星序列( 90.37%)。图3为测序得到的合浦珠母贝基因组中一段微卫星特征的(AC)n重复序列。

图2 阳性克隆筛选Fig.2 Screening of positive clones

图3 合浦珠母贝基因组中微卫星特征的(AC)n重复序列Fig.3 Typical (AC)n repeat sequence in Pinctada fucata genomic microsatellite
2.3 微卫星序列分析
根据 Weber[14]提出的测序标准, 合浦珠母贝微卫星序列中完美型70个, 占82.36%; 非完美型6个,占7.59%; 复合型8个, 占9.41%。
合浦珠母贝微卫星序列最大重复次数为 73次,平均重复次数为7.83次, 其中, 重复6~10次的最多,占 57.75%, 其次为重复 2~5次(11.26%), 11~15次(15.49%), 16~20 次(9.86%), 详见图4。
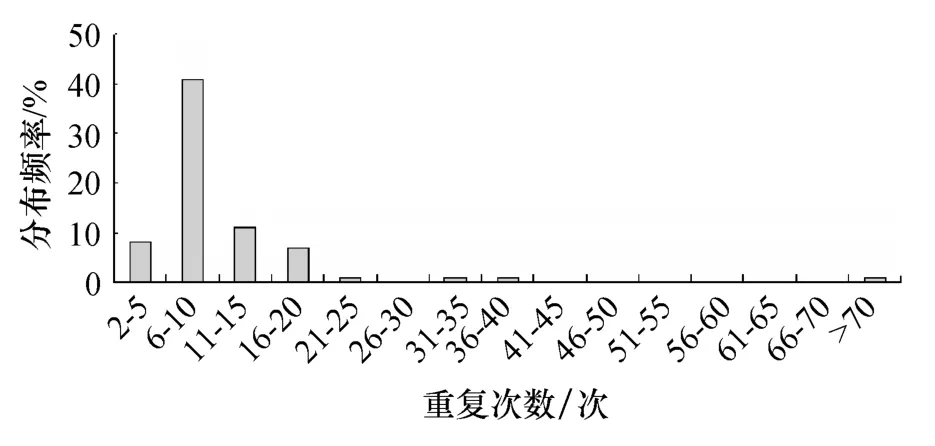
图4 合浦珠母贝微卫星核心序列重复次数分布频率Fig.4 The distribution frequency of the microsatellite corerepetition sequence in Pinctada fucata
2.4 引物设计
利用引物设计软件Primer Premier 5.0对所得的85个微卫星序列进行引物设计, 除去一些侧翼序列较短不能设计引物外, 共设计 64对引物(75.29%)。通过优化 PCR 反应条件(如退火温度, Mg2+浓度),本实验最终获得 11对能稳定有效扩增的合浦珠母贝微卫星多态性引物, 见表1。图5为合浦珠母贝三亚群体在位点PY008(产物片段长度160 bp)的扩增谱带。

图5 合浦珠母贝群体在PY008位点的扩增谱带Fig.5 PCR products amplified from Pinctada fucata population at locus PY008

表1 合浦珠母贝微卫星分子标记及其引物Tab.1 Microsatellite loci and their primers in Pinctada fucata
3 讨论
由于通过经典的小片段DNA克隆法构建和筛选部分基因组文库分离获得的微卫星序列比例很低[15],因此对于大多数研究而言, 寻找一种高效简单的分离方法就成了研究的目标。磁珠富集法是一种快速、高效的分离微卫星分子标记的方法, 自提出后被迅速广泛地应用于动植物的微卫星位点筛选[16-17]。该方法所需时间较短, 获得的微卫星重复序列的比例高[15]。姬长虹等[18]使用磁珠富集法筛选银鲫的微卫星, 阳性克隆为 95.3%, 李小宁等[9]用磁珠富集法筛选合浦珠母贝的微卫星, 阳性克隆为 88.5%, 柳明等[19]利用磁珠富集法进行了大珠母贝微卫星的筛选, 报道的为70.81%, 曲妮妮等[10]用FIASCO法筛选合浦珠母贝的微卫星, 阳性克隆为 83.2%, 匡刚桥等[20]FIASCO法筛选鳜鱼微卫星标记, 阳性克隆为60%。本研究采用磁珠富集法筛选合浦珠母贝的微卫星序列, 在所测135个克隆中有122个含有微卫星序列,阳性克隆率高达 90.37%, 说明磁珠富集法在贝类中也是一种高效快速的微卫星标记分离方法, 可以应用到其他双壳贝类微卫星的分离研究。
与其他水产动物相比, 贝类多态位点的筛选效率相对较低, 需要设计大量的引物才能获得为数不多的多态标记[9-10,21-23]。本研究合成的 65对合浦珠母贝微卫星引物中有20对可以有效扩增, 仅11对具有多态性, 多态性微卫星比例仅有 16.92%。曲妮妮等[10]在筛选合浦珠母贝微卫星标记的研究中, 合成了49对引物只有9对具有多态性, 多态性微卫星比例仅有18.36%。柳明等[19]对大珠母贝微卫星的研究中, 合成的30对引物中仅有10对扩增出具有多态性的目的条带, 多态性微卫星比例仅有 33.33%。所得到有多态性微卫星的比例都很低, 原因可能与贝类个体之间的遗传变异程度较大有关[10,19]。Arias等[24]对扇贝的SNPs检测, 发现大约100个碱基就出现1次 SNP。Sauvage等[25]对太平洋牡蛎(Crassostrea gigas)的研究, 发现在编码区平均每60 bp出现1次SNPs, 在非编码区每40 bp就出现1次SNPs。这些高频率的点突变为微卫星引物的扩增筛选带来了很大困难。
研究表明, 微卫星广泛散在于真核生物基因组中, 特别是二核苷酸微卫星重复(AC/TG)n(n大约为10~60)[26]在基因组中的含量十分丰富。本研究采用(AC)n探针, 获得了85个微卫星序列, 其中(AC/GT)n微卫星引物有64对, 占75.29%。除了大量的双碱基序列(AC、AT、AG)外, 还有其它单碱基(A、C)、三碱基(ACA、ACG、AGG、AAC)、四碱基(AAAC、CTCT)和四碱基以上(ATTTG、AAAAC、ATTGGG、CGTCCG)的微卫星序列, 因此可以考虑采用其他类型的探针, 如(AG)12、(ACA)15、(GATA)8、(GATT)7[9]研究开发合浦珠母贝基因组微卫星, 以便获得更多的微卫星分子标记。
多数学者认为微卫星重复次数与多态性之间存在正相关。Valdes[27]认为重复次数低于5的微卫星几乎检测不出多态性。一般微卫星的核心序列重复次数越高, 其等位基因数也就越多, 即多态性也就越高[28]。Ellegren[29]认为真核生物中微卫星重复碱基重复序列长度大多在30次重复以下。本研究所得序列重复次数在5~20次的占95.74%, 因此理论上讲, 本研究所得引物可用于合浦珠母贝遗传多样性分析。
另外, 与哲罗鱼[12]、中国对虾[30]、剑尾鱼[31]、长牡蛎[17]、鲤[15]相比, 合浦珠母贝微卫星序列中完美型比例最高, 为 82.36%, 而合浦珠母贝微卫星非完美型比例仅为 7.59%, 仅比中国对虾的(7.00%)高(表2)。目前为止, 关于微卫星优势类型的种间差异现象报道较少, 孙效文等认为可能与生物的进化及环境的变迁有关[12]。笔者分析, 可能原因是合浦珠母贝是长期以足丝管附着生活的贝类, 生态环境相比迁徙或浮游生活的水生动物较稳定, 较少有基因组复制过程中的变异。而李琪等[17]报道的固着生活的长牡蛎完美型所占比例并无明显优势, 与本实验所得结论不同。

表2 不同类型的微卫星在水产动物中的分布Tab.2 Distribution of different types of microsatellites in aquatic animals
[1] 王桢瑞.中国动物志无脊椎动物[M].北京: 科学出版社, 2002: 68-98.
[2] Hwang J J, Yamakawa T, Al E.Growth of wild pearl oystersPinctada fucata,Pinctada margaritiferaandPinctada sugillata(Bivalvia: Pteriidae) in Taiwan[J].Fisheries science, 2007, 3(1): 132-141.
[3] 何毛贤.珍珠贝良种培育及产业化的几点思考[J].水产科技, 2004, 6: 1-3.
[4] 喻达辉, 黄桂菊.关于振兴南珠的思考[J].中国宝石,2011, 4: 240-241.
[5] Tautz D.Hypervariabflity of simple sequences as a general source for polymorphic DNA markers[J].Nucleic Acids Research, 1989, 17(16): 6463-6471.
[6] Ning Y, Liu X D, Wang Z Y, et al.A genetic map of large yellow croakerPseudosciaena crocea[J].Aquaculture,2007, 264(1): 16-26.
[7] 罗明坤, 郭金涛, 赵金良, 等.尼罗罗非鱼(♀)×萨罗罗非鱼(♂)F1家系亲权关系微卫星分析[J].南方水产科学, 2014, 1: 9-15.
[8] 孙立元, 郭华阳, 朱彩燕, 等.卵形鲳 育种群体遗传多样性分析[J].南方水产科学, 2014, 2: 67-71.
[9] 李小宁, 张殿昌, 朱彩艳, 等.合浦珠母贝微卫星DNA标记分离与分析[J].福建水产, 2009, 1: 48-54.
[10] 曲妮妮, 龚世园, 黄桂菊, 等.基于 FIASCO 技术的合浦珠母贝微卫星标记分离与筛选研究[J].热带海洋学报, 2010, 29(3): 47-54.
[11] 石耀华, 洪葵, 郭希明, 等.马氏珠母贝 EST微卫星的筛选[J].水产学报, 2008, 32(2): 174-181.
[12] 佟广香, 鲁翠云, 匡友谊, 等.哲罗鱼基因组微卫星富集文库的构建与分析[J].中国水产科学, 2006, 13(2): 181-186.
[13] 萨姆布鲁克, 拉塞尔.分子克隆试验指南(第3版)[M].北京: 科学出版社, 2002: 8.
[14] Weber J L.Informativeness of human (dC/dA) n(dG/dT)n polymorphisms[J].Genomics, 1990, 7: 524-530.
[15] 孙效文, 贾智英, 魏东旺, 等.磁珠富集法与小片段克隆法筛选鲤微卫星的比较研究[J].中国水产科学,2005, 12(2): 126-132.
[16] Edwards K J, Barker J H, Daly A, et al.Microsatellite libraries enriched for several microsatellite sequences in plants[J].Biotechniques, 1996, 20: 758-760.
[17] Li Q, Kijima A.Identification of novel microsatellite loci in the Pacific oyster (Crassastrea gigas) by magnetic bead hybridization selection[J].Tohoku Journal of Agricultural Research, 2002, 53(1-2): 25-32.
[18] 姬长虹, 孙效文.用磁珠富集法快速制备银鲫微卫星标记[J].大连水产学院学报, 2007, 22(6): 460-464.
[19] 柳明, 喻达辉, 黄桂菊.大珠母贝微卫星 DNA 标记的分离与筛选研究[J].海洋科学, 2010, 34(8): 1-5.
[20] 匡刚桥, 刘臻, 鲁双庆, 等.FIASCO法筛选鳜鱼微卫星标记[J].中国水产科学, 2007, 14(4): 608-614.
[21] Evens B S, Knauer J, Taylor J U, et al.Development and characterization of six new microsatellite markers for the silver-or gold-lipped pear oyster,Pinctada maxima(Pteriidae)[J].Molecular Ecology Notes, 2006,6(3): 835-837.
[22] Herbinger C M, Smit H C A, Langy S.Development and characterization of novel tetra- and dinucleotide microsatellite markers for the French Polynesia black-lipped pearl oyster,Pinctada margaritifera[J].Molecular Ecology Notes, 2006, 6(1): 107-109.
[23] 赵莹莹, 朱晓琛, 孙效文, 等.磁珠富集法筛选虾夷扇贝微卫星序列[J].中国水产科学, 2006, 13(5): 749-755.
[24] Arias A, Freire R, Boudry P, et al.Single nucleotide polymorphism for population studies in the scallopsAequipecten opercularisandMimachlamys varia[J].Conserv Genet, 2008, 56: 1000-1007.
[25] Sauvage C, Bieme N, Lapegue S.Single Nucleo-tide polymorphisms and their relationship to codon us-age bias in the Pacific oyster Crassostrea gigas[J].Gene,2007, 406(1-2): 13-22.
[26] 刘萍, 孟宪红, 孔杰, 等.中国对虾部分基因组文库构建和微卫星 DNA序列的筛选[J].高技术通讯,2004, 14(2): 87-90.
[27] Valdes A M.Allele frequencies at microsatellite loci:the stepwise mutation model revisited[J].Genetics,1993, 133(3): 737-749.
[28] Ma Z Q, Roder M, Sorrells.Frequencies and sequence characteristics of di, tri-, and tetra-nucleotide microsatellite in wheat[J].Genome, 1996, 39(1): 123-130.
[29] Ellegren H.Microsatellite evolution: a battle between replication slippage and point mutation[J].Trends in Genetics, 2002, 18: 70.
[30] 徐鹏, 周岭华, 相建海.中国对虾微卫星 DNA 的筛选[J].海洋与湖沼, 2001, 32(3): 255-259.
[31] 李霞, 白俊杰, 吴淑勤, 等.剑尾鱼微卫星DNA的筛选[J].中国水产科学, 2004, 11(3): 196-201.
