基于复杂关联网络的生物医学研究结构的挖掘
2015-03-22,,,,
,, ,,
随着文献数量的急速增长,文本挖掘技术不断应用于大规模文献处理,基于文献的知识发现已经成为文献挖掘领域的重要内容。1986年,Swanson教授提出基于文献的知识发现思想,即对非相关的文献进行整合分析,发现其中隐含的联系,进而形成新的科学假设[1-2]。基于文献的知识发现的核心是通过ABC模型来挖掘概念间的间接关系,即当不相关的实体A与C同时与实体B相关时,A与C也可能相关,这种关联假设的方法在药物发现、药物重定位[3-4]等领域得到了较好的应用。随着大量文献富集,内容相关性会涌现出知识网络,并通过知识网络进行关联挖掘。如通过对文献词语共现网络的研究,总结出当前的研究热点,分析科研结构,发现研究内容的相关性等[5-6]。还有一些研究针对具体实体的关联网络进行分析,如基因调控网络、蛋白质相互作用网络等[7]。此外,部分研究转向系统层面上考察信息间的整合分析,通过多领域多数据源交叉融合,发现间接的隐含联系[8]。然而,面对庞大的关联知识网络,如何从网络微观结构与关联形成的规律,探讨其对文献知识发现的影响,对提高知识发现的效率具有重要作用。
本文基于免费开放的PubMed文献数据集,构建了一个由文献数据衍生出的生物医学实体关联演化网络,从而整合不同时期文献中的关联知识,并利用复杂网络理论分析该关联网络的拓扑特征,从系统层面分析研究大量文献集中于科学知识的结构及相关性,为文献的知识发现引入新的视角与方法,提高知识发现的效率,引导科研人员进行知识发现。
1 网络简介
1.1 网络的定量描述
一个简单的无向无权网络可标记为G=(V,E)。其中集合V称为节点集:V={v1,v2,…,vn},集合E称为边集:E={e1,e2,…,em},任意一条边对应一个节点的二元组:ex=(vi,vj),E是V×V的一个子集。对于用节点和边描述的图,可以用几个定量指标来描述图的性质,包括节点的度、连通性、路径与聚类系数。
节点的度:即节点V在图G的度,指图G中与节点V连接的边数,记为d(v)或k(v)。节点的度主要用于描述节点的连通性。
连通性:若G中每对不同节点U,V之间都存在一条通路,则G是连通的,即G为连通图。
路径:即图的路径,指两个与边交替出现的序列,且所有节点与边都不相同。路径长度是连接两个节点之间边的数量,网络距离可以通过路径长度来描述,一般采用最短路径作为连接两个节点的路径。平均路径长度是网络中所有节点对之间最短路径长度的平均值。
聚类系数:表示图中节点聚集程度的系数,定义为其邻居真实连接数目占邻居最大可能连接数比例的平均。
1.2 网络的拓扑性质
图是一种用来表示实际系统的一种模型。对于图G=(V,E),如果存在一个映射函数f,即
f:E→V×V(公式1)
若将网络中的边映射到节点对,那么图是结构化的,即图存在一定的拓扑结构;如果映射是随机的,那么图就是随机的。通常按度序列分布与熵定义图的结构,其中度序列分布按拓扑对图的分类提供了一种机制,而熵提供了一种对随机性的测量。一般来说,度序列分布表达了图的结构信息,熵则表达了图的结构是否具有规则性。
网络规模很大但平均距离却很小的性质被称为小世界效应。小世界网络一般是指具有相对较小的平均路径长度、相对较大的聚类系数的网络。如果一个图的度序列分布符合幂函数的形式,由于幂函数是标度不变的,通常称这类图为无标度网络。无标度网络同小世界网络类似,很多真实网络都具有无标度特征。
2 生物医学实体关联网络的构建与分析
2.1 基于共现方法的实体关联提取
生物医学文献挖掘研究通常利用共现方法来提取实体的关联,即当两个词语共现于一定的语境中时,词语之间存在一定的语义相关性[9]。对于实体共出现而言,以句子为最大分析单元最常见。本文基于句子共现的实体关联提取的基本步骤如下。
根据基于自然语言的方法识别出句子的实体NP及其位置。
如果在同一个句子中得到的实体按其在句子中的顺序依次为NP1、NP2、NP3,则得到关联(NP1,NP2),(NP1,NP3),(NP2,NP3)。例如,文献标题(PMID: 20856896):β1-syntrophin modulation by miR-222 in mdx mice,提取得到实体及其位置的列表为:
[(β1-syntrophin modulation, 1),(miR-222, 4),(mdx mouse, 6)]
进一步得到关联:(β1-syntrophin modulation, miR-222),(β1-syntrophin modulation, mdx mouse),(miR-222, mdx mouse)。
2.2 网络构建
考虑到PubMed数据库中所有摘要的数据量过大,本文以PubMed中2000-2009年共10年记录的标题数据为实验数据集,抽取其中的实体及关联后,建立关联知识网络。为了观察科学研究的动态结构,构建了按时间(年)增长的演化网络序列,如表1所示。
由于网络过于庞大,本文未能给出关联网络的可视化效果,但从表1的统计结果来看,仍可以观察到一些有用的特征与规律。从网络的演化情况来看,网络的节点与关联每年都在增长,表明整个研究领域的知识量是不断增加的,这与每年文献数量不断增长的情况是一致的。在关联知识网络中,每年都存在新节点新关联的加入,表明在生物医学研究领域每年都有新发现,而且每年新增加的关联数远大于新增加的节点数。这也反映在较短的时间内,真正具有较大创新性的发现相对较少,大部分文献仍然是在已有研究问题基础上的延续研究。总的来说,通过关联知识网络的演化分析,一定程度上反映了知识的形成与发展的规律。关联网络中节点与关联的增长,都能反映出新知识的不断出现。
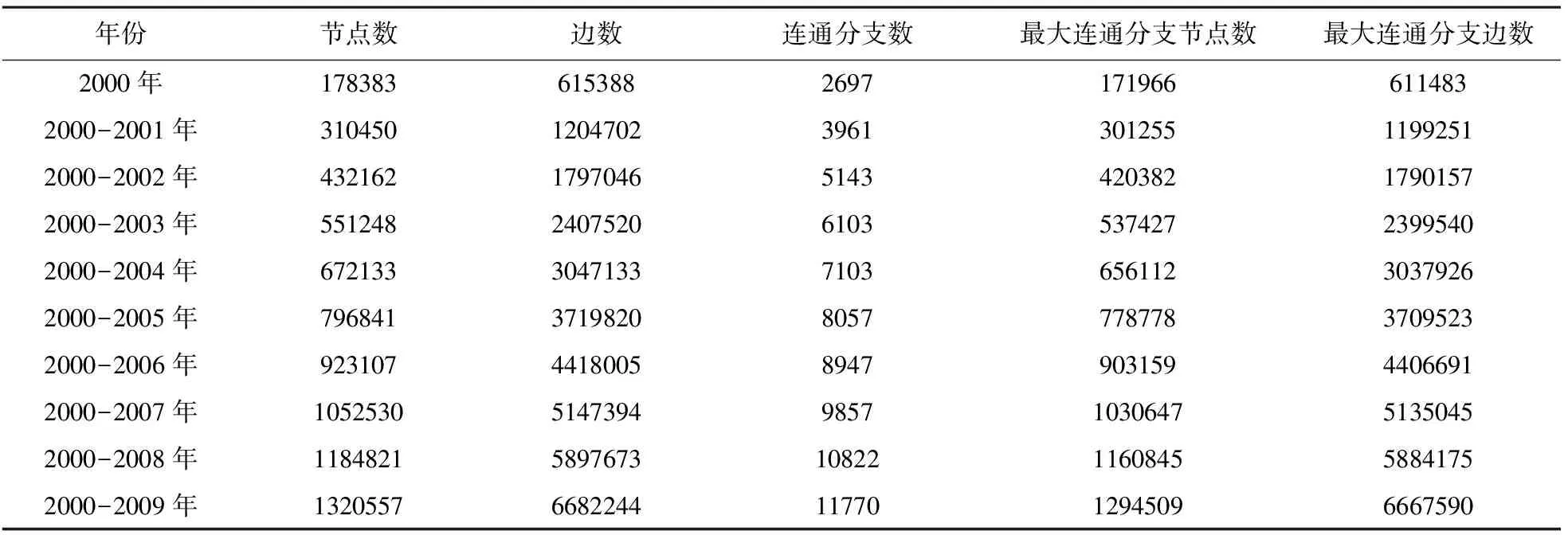
表1 关联演化网络的基本信息
2.3 关联网络的拓扑结构分析
2.3.1 网络的连通性
从表1的计算结果可知,提取到的关联网络是一个非连通网络。从2000年开始,每一年的关联网络都有很多个连通分支,比如2009年的关联网络有11 770个连通分支。尽管存在如此多的大小不一的连通分支,但每个关联网络都有一个最大连通分支,能够覆盖网络的绝对多数的节点与边,比如2009年的关联网络中最大连通分支包含1 294 509个节点与6 667 590条边,分别占整个网络中节点的98.03%以及边的99.78%。因此,主要对最大连通分支进行网络的特征分析。
除了最大的连通分支,关联网络中其他连通分支的规模都很小,表明科学研究的专业化变得更精细,生物医学领域研究内容极具丰富性与多样性;同时也表明在一些特定的领域,领域之间缺乏互通融合,形成了一个个独立的知识“孤岛”。出现大量的相对极小的连通分支,也说明在整个领域存在一些比较“冷门”的研究。
2.3.2 网络的度序列分布
如图1所示,关联知识网络呈现幂函数形式,是一个无标度网络。根据幂律分布的特性,绝大多数节点拥有较少的连接数,而少量的节点拥有极大的连接数。这些拥有极大连接数的节点是关联网络的HUB节点,基本都是一些生物医学研究领域通用的概念。尽管它们无法代表整个领域的研究重点或研究热点,但其他众多概念都围绕它们展开。说明它们在整个生物医学科研体系中起着非常重要的连接桥梁的作用,而一些连接数较少的节点只代表某个具体的研究对象。关联知识网络的无标度特征表明在生物医学领域中研究重点突出,而围绕研究重点开展了很多细致的研究工作。

图1 2000-2009年的关联网络的度序列分布双对数坐标(Log-Log)
2.3.3 计算网络的聚类系数
考虑到计算能力的限制,我们仅以2000年的数据作为测试数据,计算得到网络的平均聚类系数为 0.209390339012,而最大连通分支的平均聚类系数为0.215289709462。接下来构建与原网络、最大连通分支的节点数边数都相同的随机网络,其平均聚类系数分别为3.37415559158e-05与4.98993799995e-05。显然,关联网络的聚类系数远大于随机网络的聚类系数,表明关联网络具有高集群性。
关联网络的高集群性说明围绕一个研究主题所开展的各种研究之间具有很高的相关性,相关研究之间更容易形成连接,而它们之间的连接可以形成新的研究成果,这有助于对研究主题进行更深层次的分析和挖掘。根据综合聚类系数与幂律分布的特征,可推断出关联网络中存在很多集团,集团内部成员之间联系紧密,而集团之间的联系相对疏远,这表明某领域中存在一些研究重点和研究热点。围绕这些重点和热点所展开的大量相关研究之间联系紧密,形成网络结构中的集团,并使得集团内部成员的聚类系数很大,最终使得整个网络的聚类系数较大。
2.3.4 计算网络的平均距离
根据网络距离的定义,当网络不连通时,网络的平均距离是无穷大,该关联网络是不连通的,因此只计算关联网络中最大连通分支的平均距离。以最小的2000年的关联网络的最大连通分支作为测试对象,该连通分支的平均距离长度为3.76923247599,表明关联网络中的节点平均只需经过4步就可到达其他节点。然后根据2000年的关联网络的最大连通分支的大小,建立一个相同大小的随机网络模型。该随机网络的平均路径长度约为5.79725740556,显然,相对于相同大小的关联网络来说其平均路径长度相当小。综合关联网络的聚类系数与平均路径长度,表明该实体关联网络是一个小世界网络。
关联网络的小世界特征表明,在生物医学研究领域,研究主题和研究内容之间关联的紧密程度非常高,而平均路径长度很小则说明主题与内容相互之间存在很强的影响。此外,小世界特征也说明在同一个大的研究领域中,从一个研究对象可以很快转移到另外一个研究对象,二者结合很容易形成新的研究内容。
3 结语
基于自然语言处理方法得到的网络是一个普适的由文献衍生的关联知识网络,它不同于已有的衍生于文献的生物网络,不依赖于任何领域特异性的实体关系。因此,通过该网络可以更好地研究知识本身的发展规律,反映科研问题、概念间的相互关系。
从测试数据衍生而来的关联网络的演化情况来看,网络的节点与关联每年都在增长,表明整个研究领域的知识量在不断增加,每年都有新节点新关联的加入。同时,关联知识网络的小世界无标度特征,表明在生物医学研究领域,研究主题和研究内容之间关联的紧密程度非常高。在同一个研究领域中,从一个研究对象可以很快转移到另外一个研究对象,二者结合很容易形成新的研究内容,这也验证了基于文献的知识发现的思想。
总的来说,关联知识网络的演化分析,一定程度上反映了知识的形成与发展的规律。关联知识网络中节点与关联的增长,反映出新知识的不断出现,而且知识网络的结构与相关性可以更好用于发现有用的关联,提高文献的知识发现效率。
