基于16S rDNA序列分析和BIOLOG快速鉴定方法鉴定杭州地区原料乳中嗜冷菌
2015-03-20叶慧谢海华黄俊刘士旺毛建卫楼坚
叶慧,谢海华,黄俊,刘士旺,毛建卫,楼坚
(1.浙江科技学院生物与化学工程学院/轻工学院,浙江杭州310023;2.浙江省农产品化学与生物加工技术重点实验室,浙江杭州 310023;3.浙江省农业生物资源生化制造协同创新中心,浙江杭州 310023)4.温州医科大学眼视光学和视觉科学国家重点实验室培育基地,浙江温州325027)
基于16S rDNA序列分析和BIOLOG快速鉴定方法鉴定杭州地区原料乳中嗜冷菌
叶慧1,2,3,谢海华4,黄俊1,2,3,刘士旺1,2,3,毛建卫1,2,3,楼坚1,2,3
(1.浙江科技学院生物与化学工程学院/轻工学院,浙江杭州310023;2.浙江省农产品化学与生物加工技术重点实验室,浙江杭州 310023;3.浙江省农业生物资源生化制造协同创新中心,浙江杭州 310023)4.温州医科大学眼视光学和视觉科学国家重点实验室培育基地,浙江温州325027)
以杭州地区四个牧场的原料乳为对象,采用传统平板法分离培养嗜冷菌,得到嗜冷菌的生长曲线。通过形态学观察及镜检分离纯化出15株形态各异的菌种。基于16S rDNA序列分析和BIOLOG微生物快速鉴定方法,最终鉴定为假单胞菌属、类黄假单胞菌、荧光假单胞菌、乳酸菌等种属。其中假单胞菌属有13株,为杭州地区原料乳中嗜冷菌的优势菌。
原料乳;嗜冷菌;BIOLOG鉴定;16S rDNA序列
国际乳品联合会(IDF)把能在20℃以下繁殖,且10~15℃为最适生长温度的微生物定义为嗜冷菌。嗜冷菌可细分为两类,一类是专性嗜冷菌(psychrophiles),最高生长温度不高于20℃,最适生长温度为15℃,严格低温生长;另一类为兼性嗜冷菌(psychrotrophs),最高生长温度高于2℃,最适生长温度高于15℃,在原料乳中更为常见[1]。嗜冷菌具有膜流动性高、tRNA转录后修饰程度低、酶分子柔性高等共同特点,但是不同科、属的嗜冷菌之间形状差异仍然较大。有研究表明,原料乳中嗜冷菌的优势菌为假单胞菌属[2],在乳制品储藏、运输过程中产生极其耐热的蛋白酶和脂肪酶,分解奶制品中的脂肪和蛋白质,出现苦味、腐败味或形成胶凝,从而导致产品品质发生变化。原料乳中嗜冷菌的菌落总数标准测定方法为平板计数法,该法准确,但耗时长,在工业生产中无法广泛使用。目前国内外在原有标准方法的基础上创新,通过与时下新兴技术结合,开发出一些新的基于微生物学、化学、生物化学、生物技术的快速、自动化的分析方法,如酶联免疫吸附法(ELISA)、电阻抗法、流式细胞术(FCM)、脂肪酶活测定[3-4],均适用于对乳制品的检测,与传统方法相比大大缩短了检测时间,但在某种程度上存在一定的不足之处。
研究一种安全有效的原料乳中嗜冷菌的快速鉴定方法十分必要,本文采用的基于16S rDNA序列分析的检测方法及BIOLOG快速鉴定方法,旨在鉴定杭州地区原料乳中嗜冷菌并找出优势菌株,再进行比较,这对控制嗜冷菌繁殖、提高乳品质量、延长乳品货架期具有重要意义。
1 材料与方法
1.1 实验材料
本试验使用的原料乳产自杭州周边地区的四个牧场,取样时间为2014年3月。
用于配制培养基的相关试剂大多购自国药集团化学试剂有限公司;用于镜检的相关试剂购自杭州微生物试剂有限公司;用于16S rDNA序列分析的相关试剂购自上海生工生物工程有限公司;BIOLOG相关试剂及BIOLOG Gen IIIMicrostation购自美国BIOLOG公司;PCR仪(Veriti 96-Well Thermal Cycler)购自美国应用生物系统公司(ABI);凝胶成像分析仪(Tanon2500)购自上海圣科仪器设备有限公司。
1.2 实验方法
1.2.1 培养基的制备[2]
按培养基成分表(酵母提取物2.5 g,脱脂奶粉1.0 g,胰蛋白胨5.0 g,葡萄糖1.0 g,琼脂粉15 g,蒸馏水1 000 mL)称量各组分,并用蒸馏水配成溶液,调节pH至6.8~7.0,置于高压蒸汽灭菌锅内灭菌(121℃,20min)。将灭好菌的培养基从灭菌锅中取出,待冷却至45℃左右时,倒平板。将倒好的平板置于7℃培养箱中备用。
1.2.2 接种与培养
将四个牧场的原料乳依次编号为A、B、C、D,进行梯度稀释。用移液枪均匀吸取原料乳1 mL,加入9mL的无菌蛋白胨水中,稀释至10-1倍,从10-1倍稀释液中取1mL,加入到第二支蛋白胨水试管中,稀释至10-2倍,按此方法将样品稀释至10-8。分别取1 mL 8个稀释度样液均匀涂布在已倒好培养基的平板中,置于7℃的生化培养箱中培养7 d。同时做空白对照组,此过程均为无菌操作。
1.2.3 嗜冷菌的分离纯化
仔细观察平板中各菌的菌落形态,观察菌落大小、形状、色泽、色素产生、表面光滑程度、边缘整齐度,并结合各菌种的革兰氏染色情况将嗜冷菌分离,将不同菌落形态的嗜冷菌菌落编号并进行纯化,转接至斜面,重复纯化3次。
1.2.4 样品镜检
镜检操作参考《微生物学实验(第四版)》[5]。
1.2.5 16S rDNA序列分析
1)DNA提取
细菌总DNA提取参照文献[6]。
2)引物合成及PCR扩增
嗜冷菌16S rDNA序列的PCR扩增采用如下通用引物[7]:16S(F):5'-AGAGT TTGAT CMTGG CTCAG-3';16S(R):5'-CGGYT ACCTT GTTAC GACTT-3'。
PCR扩增采用50μL反应体系,体系组成如表1所示,并根据实际实验需要调整模板的加入量以获得最佳的扩增效果。
反应条件如下:95℃预变性5min,95℃变性30 s,54℃退火30 s,72℃延伸2 min,循环35次,72℃延伸10 min,最后冷却至4℃。其中退火温度需根据不同引物的Tm值进行调整。使用UNIQ-10柱式PCR产物纯化试剂盒对PCR反应产物进行纯化和回收,具体操作按试剂盒说明书进行。
3)PCR反应体系

表1 PCR反应体系组分
4)琼脂糖凝胶电泳
电泳操作参考《分子克隆实验指南(第三版)》[8],所用琼脂糖凝胶浓度为1.00%,Goldview低毒染色剂与琼脂糖充分混匀后倒入制胶膜,插上梳子,室温放置20 min使胶凝固,然后置于电泳槽中进行电泳,工作电压为140 V。
5)16S rDNA序列测定及比对
扩增的16S rDNA的测序和PCR引物的合成委托上海生工生物工程有限公司进行。将获得的序列登陆Genbank(http://www.ncbinlm.nih.gov),应用BLAST程序与数据库中的已有细菌16S rDNA序列进行相似性比较分析,并构建系统发育树。
1.2.6 BIOLOG快速鉴定
在Garland和Mills的方法上稍作修改[9]。
1)菌悬液的制备
初步确定微生物类型后,将纯化后的嗜冷菌接种到BIOLOG专用接种液中,上下移动棉签,将分散的菌落和接种液充分混合,制备成均一的菌悬液,浊度调整为90%~100%。
2)加样和培养
得到的菌悬液倒入储液槽,用微量加样器吸取上述稀释液,加入至BIOLOG ECO微平板中,150μL/孔,加完之后盖上盖子。培养条件为:在7℃恒温培养箱中培养1~2 d,并保持一定的湿度。
3)鉴定与对比
培养24 h后在读数仪上读取菌株的代谢指纹特征,在BIOLOG数据库中进行相似性比对,查找最接近的结果。
2 结果与讨论
2.1 平板计数的生长曲线
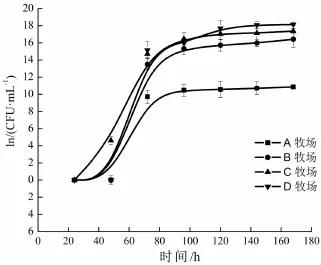
图1 四个牧场的菌落生长曲线
如上图所示,A、B、C、D四个牧场的菌落在开始培养的0~48 h生长缓慢,48~120 h菌落数迅速增加,120~144 h生长趋于平缓。第168 h菌落数达到最高,四个牧场菌落数分别为1.16×108,1.32×108,3.4×108,7.5×108CFU/mL。根据细菌生长曲线,分别对应生长延迟期、对数生长期、稳定期,上述结果与文献较为接近[10-12]。此生长曲线为下一步细菌的分离纯化提供了培养时间的理论依据,大部分嗜冷菌在7℃下培养7 d即达到生长饱和。
2.2 嗜冷菌筛选结果
对产自杭州周边地区四个牧场的原料乳中嗜冷菌进行分离纯化,结合形态学观察,一共筛选出15株菌落形态各异的嗜冷菌,通过镜检对嗜冷菌进行革兰氏染色以及菌落形态描述,结果如表2所示。
筛选菌株的显微观察照片结果如图2所示。分离得到15株菌,随机均匀分成两组,其中7株用PCR快速检测方法鉴定,另外8株采用BIOLOG快速鉴定方法,并作比较。
2.3 PCR快速检测方法结果
摇瓶培养嗜冷菌并收集菌体后,用试剂盒提取基因组,采用细菌16S rDNA通用引物扩增序列。PCR反应体系及反应条件见表1,略作调整以降低非特异性扩增,提高目的片段的纯度和浓度,最终选择的模板加入量为1μL,退火温度为54℃。PCR产物琼脂糖凝胶电泳如图3所示。由电泳图可知,扩增所得该7个菌株的16S rDNA基因片断大小均在1 500 bp左右。

表2 分离出嗜冷菌的革兰氏染色结果及菌落形态描述

图2 筛选菌株的显微观察照片
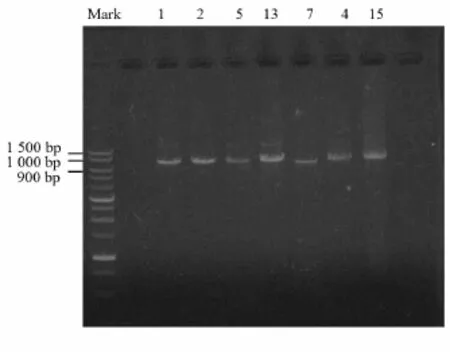
图3 嗜冷菌16S rDNA琼脂糖凝胶电泳图
使用UNIQ-10柱式PCR产物纯化试剂盒对16S rDNA PCR反应产物进行纯化和回收之后送至上海生工生物工程有限公司测序。
较之传统方法,通过16S rDNA序列同源性分析的方法能准确地将未知菌鉴定到种水平。由图4系统发育树可知,与NO.1、15同源性较高的是莓实假单胞菌(Pseudomonas fragi),NO.2与假单胞菌(Pseudomonas gessardii)形成一个分支,NO.7的16S rDNA序列与乳酸菌(Lactobacilluspiscium)的同源性较高,而与NO.4、5、13有较高同源性的是类黄假单胞菌(Pseudomonas synxantha)。通过对7株菌株的16S rDNA序列比对以及系统发育分析,由此鉴定出优势菌为假单胞菌属。吴石金等[13]从155份原料乳样品中分离纯化得到嗜冷菌分离物16株,经微生物形态及生理生化特征鉴定,确定为假单胞菌10株,微球菌4株,产碱杆菌2株。岳喜庆等[14]从原料乳中筛选出气单胞菌、乳球菌等,说明这些菌是普遍存在于原料乳中的,而我们的结果表明假单胞菌较多的存在于杭州地区的原料乳中。
2.4 BIOLOG微生物全自动鉴定结果
2.4.1 革兰氏阴阳性结果

图4 基于16S rDNA基因序列的系统发育树
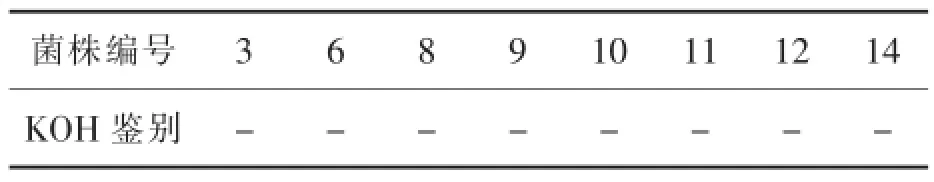
表3 不同菌株的革兰氏阴阳性实验
从表3可以看出,8株待测菌株都呈革兰氏阴性,将其与传统的革兰氏染色法进行比较,可知两种革兰氏阴阳性鉴别方法对8株菌株的鉴定结果是一致的。
2.4.2 BIOLOG鉴定
初步确定微生物类型后,将纯化后的嗜冷菌接种到BIOLOG专用接种液中,制备成均一的菌悬液,浊度调整为90%~100%。菌悬液加至BIOLOG ECO微平板中,置于7℃恒温培养箱中培养16~24 h读取数据,鉴定结果见表4。
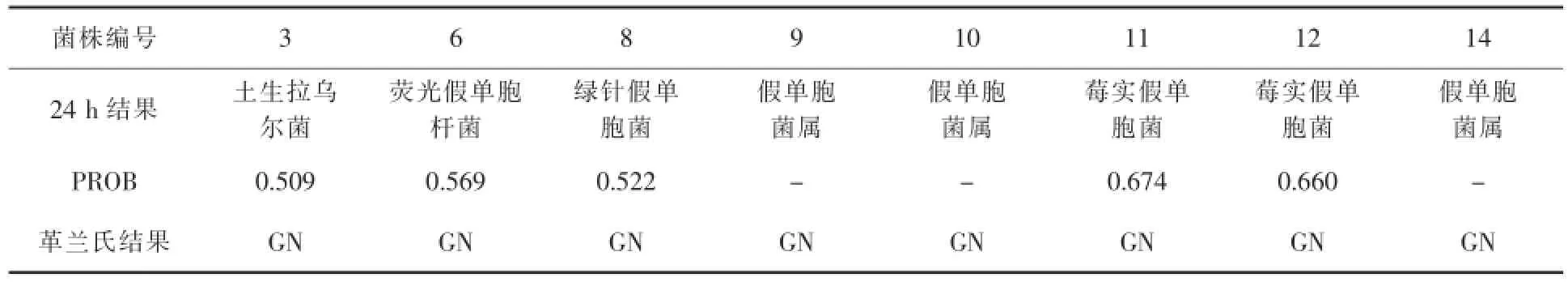
表4 BIOLOG鉴定结果
BIOLOG系统鉴定结果主要有3个参数,即可能性(PROB)、相似性(SIM)和位距(DIST)。其中SIM值和DIST值是两个重要参数,DIST值表示测试结果与数据库相应数据的距离,SIM值表示测试结果与数据库相应数据的相似程度[15]。BIOLOG系统规定:细菌培养4~6 h其SIM≥0.75或培养16~24 h其SIM≥0.5,系统自动给出鉴定结果为种名;反之,当SIM值小于0.75(4~6 h)或0.5(16~24 h),但鉴定结果中属名相同的结果的SIM值之和大于0.75或0.5时,自动给出的鉴定结果为属名,其他情况则不给出结果[17]。
由表4可知,当培养时间为24 h时,NO.3、6、8、11、12的PROB分别为0.509、0.569、0.522、0.674、0.660,均大于0.519,结果给出了种名;而NO.9、10、14只给出了属名,测定结果表示NO.3为拉乌尔菌属,其余7株均为假单胞菌属。
图5给出了BIOLOG鉴定24 h时各菌株对鉴定板上不同碳源的代谢情况。从图中可以看出,8株菌中每一株的代谢指纹特征都不一样,同一种属的菌株代谢情况也有差别。革兰氏结果表明均为阴性菌,这与传统革兰氏染色法和KOH溶液法的鉴定结果一致。采用BIOLOG自动微生物鉴定系统对从原料乳中分离得到的嗜冷菌的鉴定结果是可信的[15]。


图5 菌株的代谢指纹特征图
2.5 16SrDNA序列分析和BIOLOG快速鉴定
方法的比较
原料乳中的嗜冷菌种类繁多,其组成、数量和生态分布因地域和时间不同而有较大差别。在已鉴定出来的嗜冷菌中,细菌是最多的一类,有30多个属,且绝大多数为革兰氏阴性菌。其余种属有假单胞菌、弧菌、无色杆菌、黄杆菌、嗜纤维菌和螺菌等[2]。常用的嗜冷菌检测方法是标准平板计数法,计数准确但过程繁琐而耗时。本文主要采用基于16S rDNA序列分析和BIOLOG快速鉴定方法鉴定杭州地区原料乳中嗜冷菌,总结如下:
1)在低温条件下,原料乳中嗜冷菌占主导地位,成为影响产品质量的主要因素。采用含脱脂奶粉1.0 g/L的培养基,可以较好富集原料乳中的嗜冷菌。镜检结果表明,多数嗜冷菌为革兰氏阴性菌。
2)对产自杭州周边地区四个牧场的原料乳中嗜冷菌进行培养,得到嗜冷菌的生长曲线,四个牧场最高菌落数分别为1.16×108,1.32×108,3.4× 108,7.5×108CFU/mL。
3)对产自四个牧场的嗜冷菌进行分离纯化,筛选得到15株形态各异的菌株,分别用分子生物学法和BIOLOG快速鉴定方法进行鉴定。分子生物学法结果表明,待测7株菌株的鉴定结果为:NO.1、2、4、5、13、15为假单胞菌属,其中NO.4、5、13为类黄假单胞菌,NO.1、15为莓实假单胞菌,而NO.7为乳酸菌。BIOLOG快速鉴定方法,其结果是NO.3、6、8、11、12给出了种名,分别是土生拉乌尔菌、荧光假单胞杆菌、绿针假单胞菌、莓实假单胞菌、莓实假单胞菌;而NO.9、10、14只给出了属名,都是假单胞菌属。
4)分子生物学检测方法操作简单,具有高灵敏度和高特异性,BIOLOG快速鉴定方法快速、准确,两者都是快速检测食品微生物的重要方法。
[1]MORITA R Y.Psychrophilic bacteria[J].Bacteriological Reviews,1975,39(2):144-167.
[2]吕元.原料奶中嗜冷菌的快速检测[D].杭州:浙江大学,2010.
[3]于艳艳,丁甜,刘东红.原料乳中嗜冷菌快速检测新技术研究进展[J].食品工业科技,2014,35(11):359-367.
[4]FRANCIOSI E,SABBATA G,GARDINI F,et al.Changes in psychrotrophic microbial populations during milk creaming to produce Grana Trentino cheese[J].Food Microbiology,2011,28 (1):43-51.
[5]沈萍,陈向东.微生物学实验[M].4版.北京:高等教育出版社,2007:81-84.
[6]CUIXiaolong,MAOPeihong,ZENGMin,etal.Streptimonospora salina gen.nov.,sp.nov.,a new member of the family Nocardiopsaceae[J].International Journal of Systematic and EvolutionaryMicrobiology,2001,51:357-363.
[7]SAKAMOTOM,TAKEUCHIY,UMEDA M,et,al.Application of terminal RFLP analysis to characterize oral bacterial flora in saliva of healthy subjects and patients with periodontitis[J]. Journal of Medical Microbiology,2003,52(1):79-89.
[8]SAMBROOK J,RUSSELL D.分子克隆实验指南[M].3版.北京:科学出版社,2002:27-29.
[9]GARLAND JL,MILLSA.Classification and characterization of heterolrophic microbial communities on the basis of patterns of community-level solecarbon-source utilization[J].Applied and Environmental Microbiology,1991,57(8):2351-2359.
[10]牛会敏,李苗云,赵改名,等.低温条件下冷却猪肉中假单胞菌生长模型的比较分析[J].食品科学,2013,34(5):142-146.
[11]MARGESIN R.Effect of temperature on growth parameters of psychrophilic bacteria and yeasts[J].Extremophiles,2009,13 (2):257-262.
[12]JASNOSL,SLIWA P,KORONA R.Resolution and repeatability of phenotypic assays by automated growth curve analysis in yeast and bacteria[J].Analytical Biochemistry,2005,344(1):138-140.
[13]吴石金,何光华,万常诘,等.原料乳嗜冷菌分离株微生物学特征研究[J].中国乳品工业,2005,33(8):4-7.
[14]岳喜庆,李鹏.原料乳中嗜冷菌的分离鉴定及EDTA对其抑制作用[J].食品研究与开发,2008,29(4):87-91.
[15]张朝正,郭兰珍.利用16S rDNA序列分析和BIOLOG快速鉴定方法鉴定产脂肪酶菌株[J].河北工业大学学报,2009,38(5):52-56.
[16]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
[17]李运,盛慧,赵荣华.BIOLOG微生物鉴定系统在菌种鉴定中的应用[J].酿酒科技,2005(7):84-85.
(责任编辑:朱小惠)
Identification of psychrophilic bacteria in raw m ilk from Hangzhou area based on 16S rDNA sequences analysis and BIOLOG m ethod
YE Hui1,2,3,XIE Haihua4,HUANG Jun1,2,3,LIU Shiwang1,2,3,MAO Jianwei1,2,3,LOU Jian1,2,3
(1.School of Biological and Chemical Engineering/School of Light Industry,Zhejiang University of Science and Technology,Hangzhou 310023,China;2.Zhejiang Provincial Key Lab for Chemical and Biological Processing Technology of Farm Products,Hangzhou 310023,China;3.Zhejiang Provincial Collaborative Innovation Center of Agricultural Biological Resources BiochemicalManufacturing,Hangzhou 310023,China;4.State Key Laboratory Cultivation Base and Key Laboratory of Vision Science,Wenzhou Medical University,Wenzhou 325027,China)
Based on raw milk provided by four pastures in Hangzhou district,15 psychrophile were isolated from culture plate and their growth curves were obtained.According tomorphology,16S rDNA sequences analysis and BIOLOG method,the isolated strains were identified as Pseudomonas sp.,Pseudomonas synxantha,Pseudomonas fluorescens and Lactobacillus sp..Among them,there are 13 species of Pseudomonas sp.,which are the dominant bacteria in raw milk from Hangzhou.
raw milk;psychrophilemicroorganisms;BIOLOG identifieation;16S rDNA sequence
Q93
A
1674-2214(2015)04-0024-06
2015-07-20
国家级大学生创新创业训练计划(201311057009)
叶慧(1994—),女,浙江台州人,本科生,研究方向为生物工程,E-mail:yhuiruc@163.com.通信作者:楼坚,讲师,E-mail:loujianzust@126.com.
