白色念珠菌实时荧光定量聚合酶链反应检测体系的建立*
2015-03-15游亚兰贺湘玲方亦兵邓中平钟礼立朱秀娟邹润英
游亚兰,贺湘玲△,方亦兵,张 兵,邓中平,钟礼立,刘 华,朱秀娟,邹润英,田 鑫,邹 惠
(1.湖南省人民医院,长沙 410005;2.湖南省卫生厅,长沙 410008;3.湖南圣湘生物有限公司,长沙 410205)
·论 著·
白色念珠菌实时荧光定量聚合酶链反应检测体系的建立*
游亚兰1,贺湘玲1△,方亦兵2,张 兵1,邓中平3,钟礼立1,刘 华1,朱秀娟1,邹润英1,田 鑫1,邹 惠1
(1.湖南省人民医院,长沙 410005;2.湖南省卫生厅,长沙 410008;3.湖南圣湘生物有限公司,长沙 410205)
目的 建立白色念珠菌实时荧光定量聚合酶链反应(PCR)检测体系,实现白色念珠菌的早期、快速检测,为临床医生对白色念珠菌感染的诊断提供指导。方法 使用白色念珠菌及其他真菌标准株,利用Primer Premier 5.0和Beacon Designer 7.7设计引物探针。同时对镁离子(Mg2+)、三磷酸脱氧核糖核苷酸(dNTP)、引物及探针浓度等参数进行优化,选出最佳反应体系,并验证反应体系的特异性、重复性,分析反应体系的最低检测限,对临床分离出的45例白色念珠菌进行检测。结果 设计的引物探针仅能对白色念珠菌进行特异性扩增,反应体系不同底物浓度与Ct值之间成良好的线性关系,反应体系的最低检测限为5 CFU/mL,且反应体系稳定性、重复性好,对临床分离菌株的检测阳性率为100%。结论 成功设计了白色念珠菌特异性的引物探针,并建立了实时荧光定量PCR检测体系,能快速、准确地检出白色念珠菌。
白色念珠菌; 感染; 实时荧光定量聚合酶链反应
侵袭性真菌感染(IFI)已经成为继细菌感染之后造成国内外院内感染的重要原因[1-2]。念珠菌为IFI最常见的真菌,其中以白色念珠菌最为常见[3-4]。目前,用于IFI诊断的方法主要包括培养、组织病理学方法、影像学方法、血清学方法等。由于IFI的临床表现常无特异性,给疾病的早期诊断带来了困难。随着聚合酶链反应(PCR)技术的发展,该技术已经应用于真菌的检测[5-6]。本试验通过设计白色念珠菌特异引物和荧光探针,提取白色念珠菌DNA并进行扩增,建立白色念珠菌实时荧光定量PCR检测技术平台,为临床白色念珠菌的实时荧光定量PCR检测奠定实验室基础。
1 材料与方法
1.1 实验菌株及标本来源 白色念珠菌、近平滑念珠菌、热带念珠菌、光滑念珠菌、克柔念珠菌、烟曲霉、黄曲霉、土曲霉、黑曲霉、构巢曲霉、季也蒙假丝酵母菌、清酒假丝酵母、奥黙毕赤假丝酵母、棒曲霉、杂色曲霉、产黄青霉、少根根霉、毛霉菌、尖孢镰孢霉、串珠镰刀菌、马尔尼菲青霉均购自中国普通微生物菌种保藏管理中心;新型隐球菌购自中国普通微生物菌种保藏管理中心。肺炎链球菌、大肠埃希菌、表皮葡萄球菌、产气肠杆菌、肺炎克雷伯菌、鲍曼不动杆菌、铜绿假单胞菌、粪肠球菌、金黄色葡萄球菌均由湖南省人民医院微生物室提供。乙型肝炎病毒(HBV)、丙型肝炎病毒(HCV)、艾滋病毒(HIV)、肺炎支原体(MP)、衣原体、人巨细胞病毒(HCMV)、结核分枝杆菌(TB)、EB病毒(EBV)、柯萨奇病毒A16型(Cox A16)、肠道病毒71型(EV 71)核酸均由湖南圣湘生物有限公司提供。健康人全血采自湖南省人民医院健康志愿者。选取2011年12月至2013年2月湖南省人民医院微生物室从不同临床无菌标本培养中分离出的白色念珠菌45例作为临床检测标本。
1.2 仪器与试剂 Taq酶购自宝生物工程(大连)有限公司。缓冲液、0.1 mol/L三磷酸脱氧核糖核苷酸(dNTP)、1 mol/L氯化镁(MgCl2)、生理盐水、Hex内标反应底物及引物探针、一步裂解法DNA提取试剂盒、浓缩裂解法DNA提取试剂盒均由湖南圣湘生物有限公司提供。血液/细胞/组织基因组DNA提取试剂盒购自天根生化科技(北京)有限公司。美国Applied Biosystems 公司7500荧光定量PCR仪,美国Stratagene Mx3000P实时荧光定量PCR仪。马铃薯葡萄糖琼脂培养基(PDA培养基)高压蒸汽灭菌(121 ℃,30 min)后无菌操作倒入平板。
1.3 方法
1.3.1 标准株培养及制备 霉菌接种于PDA平板,37 ℃培养72 h;念珠菌及新型隐球菌接种于PDA平板,37 ℃培养48 h。均制成浓度约107CFU/mL的混悬液,-80 ℃保存。
1.3.2 核酸提取 (1)标准菌株DNA的提取方法:采用浓缩裂解结合加热法,取200 μL标本,加入等体积的浓缩液,12 000 r/min离心5 min,弃上清液,沉淀中加入100 μL核酸释放剂,充分震荡混匀,95 ℃加热10 min,间隔3~5 min震荡10 s,静置冷却,作为待测标本。(2)人类基因组、细菌、病毒及其他病原菌核酸提取:人类基因组提取按照天根生化科技有限公司血液/细胞/组织基因组DNA提取试剂盒方法提取;细菌及HBV、MP、HCMV、TB、EBV采用一步裂解法核酸提取试剂盒提取DNA;HCV、HIV、Cox A16、EV 71采用由湖南圣湘生物科技有限公司提供的RNA磁珠法提取试剂盒提取。
1.3.3 引物探针设计及产物测序 在基因库(NCBI)中下载多种真菌的基因组序列,找出白色念珠菌相对保守的基因区域,利用Primer Premier 5.0和Beacon Designer 7.7软件设计引物探针,引物探针由美国Invitrogen公司合成。特异性扩增产物送至上海生工测序。
1.3.4 PCR反应体系优化 PCR反应体系内各成分不同含量扩增效果比较,MgCl2溶液分别选取每人份0.1、0.3、0.5、0.7 μL 4个梯度,dNTP分别选取每人份0.1、0.3、0.5、0.7、0.9 μL 5个梯度,引物分别选取每人份5.0、10.0、15.0、20.0、25.0、30.0 pmol,探针分别选取每人份5.0、7.5、10.0、12.5、15.0、17.5、20.0 pmol,在反应程序相同的条件下选取最佳反应条件。PCR反应条件为94 ℃预变性5 min后进入循环,94 ℃ 15 s、57 ℃ 30 s,共45个循环。
1.3.5 特异性与重复性试验 分别对所提取真菌、细菌、病毒及人类基因组等的核酸进行PCR扩增,以检验反应体系对白色念珠菌的特异性,PCR反应条件同前。批内、批间重复性试验选用107~102CFU/mL 6个不同浓度的标准液(记为1~6号)所提取的DNA为模板,批内重复性试验对同一模板、同一反应体系同时进行5次重复测定,批间重复性试验对同一样本在反应体系相同的条件下进行连续5 d重复测定,计算各自循环阈值(Ct)的变异系数(CV)。
1.3.6 线性范围测定和定量标准曲线制备 标准株用生理盐水稀释成107~101CFU/mL,分别按照浓缩裂解结合加热的方法提取DNA进行扩增,并获得标准曲线。
1.3.7 最低检测限分析试验 选取线性范围内能全部测出的最低浓度,用生理盐水将该浓度的标准株混悬液进行1∶1、1∶2、1∶3、1∶4稀释后提取DNA后扩增,重复8次,找出反应全部呈阳性的最低浓度即为反应最低检测限。
1.3.8 Hex内标分析 将标准株稀释成106~103CFU/mL 4个梯度,提取DNA进行扩增,每一反应中分别加入等量的Hex内标反应体系,监测PCR假阴性结果的发生。
1.3.9 临床检测 针对临床分离培养的白色链珠菌标本,采用浓缩裂解结合加热的方法提取DNA进行扩增,PCR反应条件同前。

2 结 果
2.1 引物探针设计与PCR产物测序结果 通过多种真菌基因序列进行比对,最后确定白色念珠菌RSR1基因特异性较好,设计的引物探针如下,上游引物(F):5′-CAGTGCTGGTGGTAGTAGTGGATT-3′;探针(P):FAM-ACGTG TTACATTGATGCTTCTTAATCGAT-3′;反向引物(R):5′-TGCTGTTTTGGT GGGAGTGAT-3′。试验特异性扩增产物测序结果进行Blast分析,序列均在设计引物探针所在目的基因区域。
2.2 反应体系优化结果 通过对不同浓度的Mg2+、dNTPs、引物、探针进行实验,结果显示在MgCl20.1 μL、dNTPs 0.3 μL、F/R 5 pmol、P 7.5 pmol、Taq酶2.0 μL、模板DNA 10 μL,总反应体系50 μL条件下反应体系扩增效率最高、扩增曲线呈典型的“S”形。
2.3 特异性与重复性试验结果 在同一反应条件下,反应体系仅能对白色念珠菌进行扩增,与其他核酸无交叉反应,见图1。表明实验中所建立的反应体系具有很好的特异性。批内重复性试验显示,同一次实验不同反应之间同一浓度扩增曲线基本重合,见图2;批间重复性试验显示,所获得的相同浓度Ct值的平均值CV均小于5%,说明PCR检测体系的重复性好。见表1。
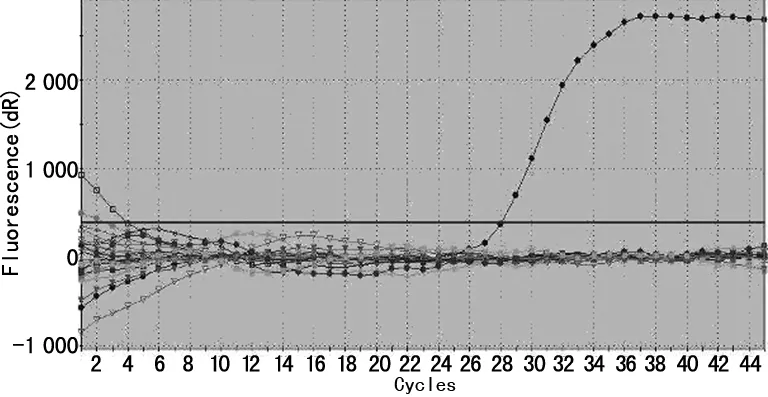
图1 特异性试验结果

图2 批内重复性试验结果
2.4 线性范围测定及标准曲线的建立 分别提取不同浓度的标准株DNA后进行扩增,在107~101CFU/mL浓度能扩增出典型的“S”型曲线,其线性范围为107~101CFU/mL。线性范围与标准曲线,见图3。
2.5 最低检测限分析 将10 CFU/mL标准株混悬液稀释后提取DNA进行PCR扩增,重复8次。经1∶1稀释后能完全扩增,而其他浓度标本未见扩增或仅部分扩增,反应体系能检测的最低浓度可以达到5 CFU/mL。

表1 批内及批间重复性试验Ct值比较

注:A为线性范围;B为标准曲线。
图3 线性范围与标准曲线
2.6 加入内标扩增结果 反应体系中加入Hex荧光标记的内标体系后,PCR反应结果与不加入内标的扩增效果相同,内标在建立的PCR反应中进行扩增与单独进行扩增均有明显的扩增效果,而且Ct值基本一致、扩增曲线基本重合。对Ct值进行方差分析,组间比较差异无统计学意义(P>0.05),说明内标加入对本实验的反应体系无影响。
2.7 临床真菌检测结果 采用本实验建立的实时荧光定量PCR反应体系检测临床分离出的45例白色念珠菌,结果均为阳性,检测率达100%,见图4。

注:不同曲线代表不同标本的扩增曲线。
图4 临床菌株PCR检测结果
3 讨 论
白色念珠菌是IFI最常见的病原菌,目前主要依靠传统的检测方法协助临床诊断,但是传统方法存在耗时长、阳性率低等缺陷,加之真菌感染缺乏特异性的临床表现,早期、快速诊断IFI仍然是困扰临床工作者的问题之一[7-8]。
实时荧光定量PCR是新近发展的检测方法,已广泛应用于多种病毒的临床检测[9-10],而真菌感染的PCR检测目前仍停留在实验室阶段。DNA的提取及引物探针的设计等因素是影响PCR检测广泛应用于白色念珠菌的主要原因[10-11]。真菌DNA的提取方法包括磁珠法、膜吸附法、液氮碾磨法、机械破壁法等[12-13]。本研究选取浓缩裂解法,并在裂解过程中予以加热处理以提高DNA的提取效率,取得了较理想的效果。国内外已经报道了各种可以用于念珠菌特异性检测的基因区域,如ITS区域、FKS1及线粒体基因[14-15]。经过比对,本研究发现RSR1基因区域适宜于设计引物探针,且通过特异性、重复性等试验验证,该引物探针能特异地检测白色念珠菌,其最低检测限可达到5 CFU/mL,并且对临床分离株的检测率达到100%,说明所建立的检测体系不仅可以检测标准株,同样适用于临床标本的检测。此外,实验中Hex内标的加入能有效地避免假阴性结果,提高检测体系的可靠性,给临床检测提供了更好的保障。
综上所述,实时荧光定量PCR用以检测白色念珠菌具有快速、特异的优势,通过本试验获取了白色念珠菌的DNA提取方法,并设计了特异性的引物探针,所建立的PCR检测体系能够准确定量标本中的病原体含量,为临床标本中白色念珠菌的PCR检测奠定了实验基础,可更好地指导临床医生对疾病进行诊断与治疗,具有广阔的应用前景。但是,应用于临床还需要动物及临床试验的进一步验证。
[1]Dereli N,Ozayar E,Degerli S,et al.Three-year evaluation of nosocomial infection rates of the ICU[J].Bras J Anesthesiol,2013,63(1):73-84.
[2]赖基栋,欧阳长安,林新祝,等.新生儿重症监护室医院感染的病原菌分布及耐药性[J].中国当代儿科杂志,2012,14(12):988-990.
[3]Garnacho-Montero J,Diaz-Martin A,Ruiz-Perez De Piappon M,et al.Invasive fungal infection in critically ill patients[J].Enferm Infecc Microbiol Clin,2012,30(6):338-343.
[4]Serrano R,Gimeno A,Plumed L,et al.Epidemiological profile and sensitivity pattern of isolates causing invasive fungal infection vs.colonizing isolates in non-neutropenic critically ill patients[J].Rev Iberoam Micol,2013,30(1):14-20.
[5]Favaro M,Savini V,Favalli C,et al.A multi-target real-time PCR assay for rapid identification of meningitis-associated microorganisms[J].Mol Biotechnol,2013,53(1):74-79.
[6]Sugawara Y,Nakase K,Nakamura A,et al.Clinical utility of a panfungal polymerase chain reaction assay for invasive fungal diseases in patients with haematologic disorders[J].Eur J Haematol,2013,90(4):331-339.
[7]Osmundson TW,Eyre CA,Hayden KM,et al.Back to basics:an evaluation of NaOH and alternative rapid DNA extraction protocols for DNA barcoding,genotyping,and disease diagnostics from fungal and oomycete samples[J].Mol Ecol Resour,2013,13(1):66-74.[8]Yeom SK,Kim HJ,Byun JH,et al.Abdominal aspergillosis:CT findings[J].Eur J Radiol,2011,77(3):478-482.
[9]Hsu CL,Chan SC,Chang KP,et al.Clinical scenario of EBV DNA follow-up in patients of treated localized nasopharyngeal carcinoma[J].Oral Oncol,2013,49(6):620-625.
[10]Burrel S,Fovet C,Brunet C,et al.Routine use of duplex real-time PCR assays including a commercial internal control for molecular diagnosis of opportunistic DNA virus infections[J].J Virol Methods,2012,185(1):136-141.
[11]Poma HR,Davies C,Gutierrez Cacciabue D,et al.Comparison of nucleic acid extraction efficiency using different commercial kits and qPCR.Effect of inhibitors[J].Rev Argent Microbiol,2012,44(3):144-149.
[12]Goldschmidt P,Degorge S,Che Sarria P,et al.New strategy for rapid diagnosis and characterization of fungal infections:the example of corneal scrapings[J].PLoS One,2012,7(7):e37660.
[13]Klimek-Ochab M,Brzezinska-Rodak M,Zymanczyk-Duda E,et al.Comparative study of fungal cell disruption--scope and limitations of the methods[J].Folia Microbiol (Praha),2011,56(5):469-475.
[14]Souza AC,Ferreira RC,Goncalves SS,et al.Accurate identification of Candida parapsilosis (sensu lato) by use of mitochondrial DNA and real-time PCR[J].J Clin Microbiol,2012,50(7):2310-2314.
[15]Garcia-Effron G,Canton E,Peman J,et al.Assessment of two new molecular methods for identification of Candida parapsilosis sensu lato species[J].J Clin Microbiol,2011,49(9):3257-3261.
Establishment of real-time fluorescent quantitative PCR assay system for Candida albicans*
YOUYa-lan1,HEXiang-ling1△,FANGYi-bing2,ZHANGBing1,DENGZhong-ping3,ZHONGLi-li1,LIUHua1,ZHUXiu-juan1,ZOURun-ying1,TIANXin1,ZOUHui1
(1.HunanProvincialPeople'sHospital,Changsha,Hunan410005,China;2.HealthDepartmentofHunanProvince,Changsha,Hunan410008,China;3.HunanSansureBiotechnologyCo.,Ltd.,Changsha,Hunan410205,China)
Objective To establish the real-time fluorescent quantitative polymerase chain reaction(FQ-PCR) assay system for realizing the rapid and accurate detection of Candida albicans to provide the guidance for the clinical doctors making the diagnosis of Candida albicans infection.Methods The standard Candida albicans strains and the other fungal standard strains were used in the experiment.The specific primers and probes for Candida albicans were designed by the Primer Premier 5.0 and Beacon Designer 7.7.At the same time the parameters of Mg2+,dNTPs,primers and probe concentration were optimized for finding out the best reaction system of PCR.The specificity and repeatability were verified and the lowest limit of detection of the reaction system was analyszed.Finally 45 clinically isolated 45 strains of Candida albicans were tested.Results The designed primers and probes only conducted the specific amplification,the different substrate concentrations of the reaction system showed the good linearity with the Ct value,the lowest limit of detection of reaction system was 5 CFU/mL with good stability and good repeatability,its positive detection rate for the clinically isolated strains reached 100%.Conclusion The Candida albicans FQ-PCR assays system may be established based on successfully designing the specific primer and probe,which could identify Candida albicans rapidly and accurately.
candida albicans; infection; real-time fluorescence quantitative polymerase chain reaction
湖南省科技厅科技计划项目(2013FJ6028);湖南省科技厅社会发展支撑计划重点项目(S2012S20321622)。
游亚兰,女,硕士,临床医师,主要从事儿科血液肿瘤研究。△
,E-mail:hexiangl@163.com。
10.3969/j.issn.1672-9455.2015.06.007
A
1672-9455(2015)06-0737-03
2014-08-15
2014-12-19)
