磺胺类耐药菌中抗性基因sul的表达规律
2015-03-14王娜杨晓洪郭欣妍陈彪叶波平葛峰
王娜,杨晓洪,郭欣妍,陈彪,叶波平,葛峰
1. 环境保护部南京环境科学研究所,南京 210042 2. 国家环境保护农药环境评价与污染控制重点实验室,南京 210042 3. 中国药科大学生命科学与技术学院,南京 210009
磺胺类耐药菌中抗性基因sul的表达规律
王娜1,2,杨晓洪3,郭欣妍1,2,陈彪3,叶波平3,葛峰1,*
1. 环境保护部南京环境科学研究所,南京 210042 2. 国家环境保护农药环境评价与污染控制重点实验室,南京 210042 3. 中国药科大学生命科学与技术学院,南京 210009
抗生素环境污染是影响抗生素抗性基因传播和扩散的主要因素,然而关于抗生素耐药菌在抗生素暴露下抗性基因的表达机制研究甚少。本研究利用RT-PCR方法检测了土壤中优势耐药菌菌株炭疽芽孢杆菌(Bacillus anthracis SYN201, G+)和弗氏志贺菌菌株(Shigella flexneri NJJN802, G-)在含有不同浓度磺胺类药物的培养基中生长不同时间后抗性基因(sul1、sul2、sul3)的表达变化。结果发现:无论培养基中是否存在磺胺类药物,菌株SYN201和NJJN802中的3种磺胺类抗性基因均分别在培养的第72小时或36小时出现一个明显的表达峰,而在其他培养时间下不表达或表达量处于相对极低的水平;磺胺嘧啶的存在有助于提高菌株在此特征表达时间下抗性基因的表达水平,与未加磺胺嘧啶组相比,暴露于磺胺嘧啶(60 μg·mL-1)组的sul1、sul2和sul3在菌株SYN201中的相对表达量分别提高了2.5、4.8和3.2倍,而在菌株NJJN802中的相对表达量分别提高了3.7、6.0和5.0倍。通过将耐药菌暴露于不同磺胺嘧啶浓度下考察sul基因相对表达情况,研究发现随着培养基中磺胺嘧啶浓度的升高(0~1 024 μg·mL-1),菌株SYN201和NJJN802中sul基因的表达量整体上呈现出明显的上升趋势。本研究对揭示磺胺耐药菌的抗性表达规律及抗生素暴露对其抗性表达发挥的作用具有重要意义。
磺胺嘧啶;炭疽芽孢杆菌;弗氏志贺菌;sul基因表达;抗性基因
Received 30 April 2015 accepted 5 August 2015
近年来,抗生素在医疗业和养殖业中存在着严重的滥用现象,并且抗生素会通过污水处理系统排水或者农田施用方式进入到环境中[1]。抗生素环境污染是影响抗生素抗性基因(ARGs)传播和扩散的主要因素[2]。作为一类具有广谱抗革兰氏阳性和革兰氏阴性菌活性并且价格低廉的抗菌药物,磺胺类药物在兽医临床、畜牧和水产养殖业中得到广泛应用。然而,随着养殖业中磺胺类药物的大量使用,研究者们在畜禽体内、排泄物及周边环境(土壤、水体)中检测到了多种磺胺类耐药微生物。畜禽体内及畜禽排泄物中检测到的磺胺耐药菌株多为肠球菌属(Enterococcus)和大肠杆菌(Escherichia coli),土壤及水体中多为杆菌[3]。土壤中的耐药菌包括不动杆菌属(Acinetobacter)、土壤杆菌属(Agrobacterium)、节杆菌属(Arthrobacter)、芽孢杆菌属(Bacillus)、肉杆菌属(Carnobacterium)、嗜冷杆菌(Psychrobacter)、志贺氏菌属(Shigella)、丛毛平胞菌属(Comamonas)、棒杆菌属(Corynebacterium)、气球菌属(Aerococcus)、普罗威登斯菌属(Providencia)、假单胞菌属(Pseudomonas)、狭长平胞菌属(Stenotrophomonas)[3]。
迄今为止,尽管很多的报道都显示抗菌药物可诱导微生物产生耐药性,但对于微生物在怎样的抗生素诱导浓度下才能产生抗性或者进行抗性基因的表达这一机制并不清楚。理论上推测:耐药菌的产生可能需要一个合理浓度范围内的抗菌药物进行刺激,当抗菌药物浓度高于这个浓度范围时,菌体在尚未产生抗性的情况下可能就已被药物杀死,而当低于这个浓度范围,抗菌药物所产生的胁迫压力可能并不足以导致菌体因不适而寻求改变,故不会产生抗性[4]。因此,探讨菌株中与耐药相关的抗性基因在不同的抗生素选择压力下的表达规律,将有助于加深对这些耐药菌产生耐药性的认识,同时,相关数据也能为建立针对抗生素的环境安全评估和预警体系提供线索。
在本研究中,我们以在施用过畜禽粪肥土壤中筛选出的常见耐药致病菌炭疽芽孢杆菌菌株(Bacillus anthracis SYN201)和弗氏志贺菌菌株(Shigella flexneri NJJN802)为目标菌株,利用RT-PCR方法探究了它们在含有不同浓度磺胺类药物的培养基中生长不同时间时,3种抗性基因(sul1、sul2、sul3)的表达变化规律。
1 材料与方法(Materials and methods)
1.1 实验材料
革兰氏阳性菌(G+):炭疽芽孢杆菌(Bacillus anthracis SYN201),分离于江苏省宿迁市沭阳县养猪场的磺胺耐药菌。
革兰氏阴性菌(G-):弗氏志贺菌(Shigella flexneri NJJN802),分离于江苏省宿迁市沭阳县养猪场的磺胺耐药菌。
RNA提取试剂盒(RNApro Total RNA Isolation Reagent)购自上海启动元生物科技有限公司,cDNA逆转录试剂盒购自北京全式金生物公司。
1.2 实验方法
1.2.1 磺胺类耐药菌的生长曲线分析
取3 mL在LB液体培养基上活化6 h的测试菌株(SYN201或NJJN802)接种到装有150 mL LB液体培养基或含60 μg·mL-1磺胺嘧啶LB液体培养基的三角瓶中,振荡混匀后,取2 mL于分光光度计上测定OD600,此为培养0 h的读数。将接种菌株后的三角瓶置于摇床上,37 ℃下,180 r·min-1振荡培养,每隔1~2 h取2 mL菌液测定OD600,直至培养后的第96小时。
1.2.2 不同诱导时间对磺胺类耐药菌中抗性基因表达的影响
将待测菌株培养在含60 μg·mL-1磺胺嘧啶的LB液体培养基中,分别培养0、6、12、24、36、48、72、96 h,离心收集菌株后,提取菌株总RNA,逆转录成cDNA第一链后,进行荧光实时定量RT-PCR分析;其间设置平行对照,即同样的菌株在不含抗生素的液体培养基中培养0、6、12、24、36、48、72、96 h,分析抗性基因的表达变化。
1.2.3 不同浓度的磺胺嘧啶对磺胺类耐药菌抗性基因表达的影响
在1.2.2分析结果的基础上,将菌株Bacillus anthracis SYN201和Shigella flexneri NJJN802分别接种在含有不同浓度(0、2、8、16、32、64和1 024 μg·mL-1)磺胺嘧啶的LB液体培养基中,37 ℃分别培养72 h或36 h,离心收集菌株,提取菌株总RNA,-80 ℃保存备用。
根据RNA提取试剂盒说明书提取不同时间及不同浓度下的细菌总RNA,测定总RNA的A260和A280,并结合琼脂糖凝胶电泳检测总RNA的浓度、纯度和质量。
按照逆转录试剂盒说明书,利用TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix逆转录试剂盒对提取的不同时间点或不同浓度下的总RNA样品(4 μg)分别进行逆转录,合成cDNA第一链作为荧光实时定量RT-PCR分析的模板。
qPCR分析:以16S rDNA基因作为内参基因,sul1、sul2和sul3引物序列见表1。参照Ultra SYBR mixture试剂盒说明书在CFX96 TouchTMReal-Time PCR Detection System (Biorad, USA)仪上进行Real-Time PCR,扩增条件为95 ℃热变性10 min,后进行40个循环,每个循环包括95 ℃变性15 s,60 ℃反应1 min,最后60 ℃升到95 ℃,其间每个0.5 ℃采集一次荧光以生成熔解曲线,根据溶解曲线的变化检测

表1 qPCR引物设计Table 1 Primers for qPCR
扩增结果的特异性。将目标菌株培养在不含抗生素的液体培养基中,以此条件培养的菌株总RNA作为对照,以方山土样为阴性对照组,将实验组的△Ct值减去对照组的△Ct值,结果采取2-△△Ct法进行数据处理,分析抗生素胁迫下的抗性基因表达。
1.2.4 细菌培养基中磺胺嘧啶残留量测定
利用高效液相色谱法(HPLC)测定上述培养过程中各个时间点培养基中磺胺嘧啶的残留量。HPLC分析方法参照Lai和Hou等[5]的方法,所用仪器为Waters2695型HPLC,检测波长为270 nm。同时,按照10倍倍比稀释的方法制备磺胺嘧啶标准品,同样条件下分析以获取磺胺嘧啶药物的标准曲线。根据样品检测结果,计算培养基中磺胺嘧啶的残留量。
1.2.4 数据分析
所有测定的数据以“平均值±方差”表示,组间差异采用Students’t-test方法。P<0.05表示组间差异显著,P<0.01表示组间差异极显著。
2 结果与分析(Results and analysis)
2.1 磺胺类药物对耐药菌生长的影响
通过分析不同采样时间菌液的OD600值,分析菌株SYN201和NJJN802在含磺胺嘧啶的培养基和不含磺胺嘧啶的培养基中培养时的生长曲线,分析结果如图1所示。通过HPLC检测2种菌株在生长过程中培养基内磺胺嘧啶浓度的变化,发现菌株201和802培养基中磺胺嘧啶的浓度在培养96 h后分别为培养前的88.5%和96.1%,不同时间的浓度值见表2。由此可知,2株菌的对数生长期均出现在16 h左右,同时60 μg·mL-1磺胺嘧啶的暴露并未对菌株的生长规律产生显著影响。

图1 磺胺类耐药菌株SYN201 (A)和NJJN802 (B)的生长曲线
2.2 耐药菌菌株中抗性基因的表达
2.2.1 不同时间2种细菌的抗性基因表达规律
分别在菌株培养0、6、12、24、36、48、72、96 h后,检测菌株SYN201和NJJN802在空白LB液体培养基及含有60 μg·mL-1磺胺嘧啶的LB液体培养基中3种抗性基因sul1,sul2和sul3的表达量,结果如图2所示。可知,无论菌株是否暴露于磺胺嘧啶,sul基因的表达均在特定时间发生,SYN201在72 h时表达,NJJN802在36 h时表达。然而,暴露于磺胺嘧啶却可以显著提高该表达时间下的sul基因表达量,菌株SYN201在培养72 h时sul1、sul2和sul3的表达量相较于未加磺胺嘧啶的对照组分别提高了2.5、4.8和3.2倍,菌株NJJN802在培养36 h时sul1、sul2和sul3的表达量相较于未加磺胺嘧啶的对照组分别提高了3.7、6.0和5.0倍。与菌株SYN201的sul基因表达水平(图2 a~c)不同的是,菌株NJJN802中3种sul基因的相对表达水平在各组中基本一致(图2 d~f)。
表2 不同时间培养基中磺胺嘧啶的浓度
Table 2 Concentration of sulfadiazine in the culture medium of the resistant bacteria at different times

时间/hTime/h菌株SYN201培养基中磺胺嘧啶的浓度/(μg·mL-1)SulfadiazineconcentrationintheculturemediumofstrainSYN201/(μg·mL-1)菌株NJJN802培养基中磺胺嘧啶的浓度/(μg·mL-1)SulfadiazineconcentrationintheculturemediumofstrainNJJN802/(μg·mL-1)052.1952.41651.6549.591250.4249.322450.2151.313651.0448.914851.3553.036041.1449.837250.2651.399646.2050.39

图2 不同培养时间下菌株SYN201(a~c)和NJJN802(d~f)中sul基因的表达
2.2.2 磺胺嘧啶浓度对耐药菌菌株中sul基因表达的影响
根据抗性基因在不同时间的表达分析结果,将菌株SYN201和NJJN802分别接种在含有不同浓度磺胺嘧啶的LB液体培养基中培养72 h(菌株SYN201)和36 h(菌株NJJN802),提取总RNA,利用RT-PCR方法检测sul基因在不同浓度时相对于未添加药物时的表达量,结果如图3所示。可知,随着培养基中磺胺嘧啶浓度的升高(0~1 024 μg·mL-1),菌株SYN201和NJJN802中sul基因的表达量整体上呈现出明显上升的趋势。通过SPSS软件计算磺胺暴露浓度与sul基因表达量的Pearson’s相关系数(r),所得结果如表3所示。菌株SYN201的sul 2基因表达与磺胺嘧啶暴露浓度相关性良好,r值为0.890,显著性强;菌株NJJN802的sul1和sul2基因表达与磺胺嘧啶暴露浓度相关性极强,r值分别为0.991和0.946,显著性极强。菌株SYN201在磺胺嘧啶暴露浓度为64 μg·mL-1时sul1基因相对无磺胺嘧啶暴露时提高了2 404倍,而在磺胺嘧啶暴露浓度为1 024 μg·mL-1时sul1基因相对无磺胺嘧啶暴露时则提高了1 943倍,并未出现成倍数上升的趋势,这可能因为不同磺胺耐药菌的抗性基因表达与磺胺嘧啶暴露浓度呈现出的线性相关都存在一定的有效浓度范围,超过最高限值的磺胺嘧啶抑制了耐药菌的正常生长和繁殖,从而使得耐药菌表达sul1基因的量减少。由结果可知,在同一株菌内,3种磺胺类抗性基因的表达水平呈现sul1>sul2>sul3的趋势。而sul3基因表达量少,且其表达与暴露浓度的相关性不强。

图3 磺胺耐药菌菌株SYN201(A)和NJJN802 (B)中sul基因在不同磺胺嘧啶浓度下的表达
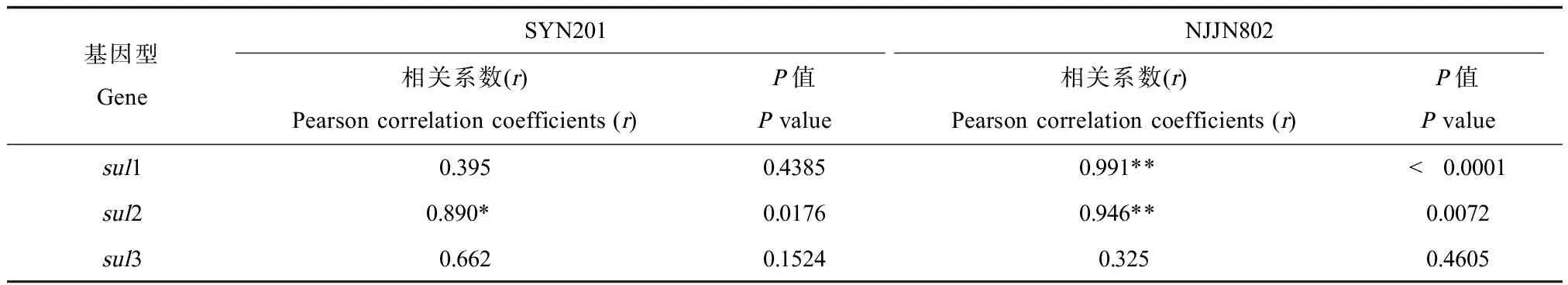
表3 磺胺嘧啶暴露浓度与sul基因表达量的Pearson’s 相关系数Table 3 Pearson correlation coefficients between sulfadiazine concentrations and expression of sul genes
注:*代表显著性强P<0.05,**代表显著性极强P<0.01。
Note: * represents statistic significance P<0.05, ** represents statistic significance P<0.01.
3 讨论(Discussion)
目前,尽管不少研究显示磺胺抗生素暴露会诱导微生物产生耐药性[6-7],但是有关微生物暴露于何种浓度水平的抗生素中会诱导产生抗性基因或使得抗性基因进行表达一直未有定论。本研究初始设计为将无抗性基因的标准菌暴露于磺胺嘧啶中,探讨药物诱导抗性基因产生的规律,但是试验未得到成功的结果。国内外已有不少文献报道了环境介质以及耐药菌中3种磺胺耐药基因的分布水平[8-10],但有关3种磺胺类耐药基因在耐药菌中的表达特征问题却未见报道,于是将试验设计改为培养养殖场采样得到耐药菌,观察耐药菌的抗性表达规律以及抗生素暴露对其抗性表达发挥的作用。
磺胺类抗生素的抗菌机制是由于其化学结构类似于对氨基苯甲酸(PABA),从而可以与PABA竞争二氢叶酸合成酶,阻断细菌中二氢叶酸的生物合成,并进而抑制以二氢叶酸为底物的四氢叶酸的生物合成过程,影响核酸前体物嘌呤和嘧啶的合成,由此抑制细菌的正常生长和繁殖[11]。磺胺抗生素耐药机制主要与DHPS的突变基因sul 1-3相关[11-13],其调控表达的二氢叶酸合成酶与磺胺类的结合能力相较于野生型降低了10 000倍左右[14,15]。然而,本研究结果显示当培养基中的磺胺嘧啶浓度为60 μg·mL-1时,菌株的生长规律并未受到影响,即在2株菌的对数生长期并不是sul基因表达的时间。这可能是因为LB液体培养基的主要成分之一——酵母提取物富含蛋白质、肽、核苷酸、氨基酸以及维生素等成分,可以满足细菌正常生长和繁殖的需要。在对数生长期内,菌株生长环境中存有大量的PABA或者二氢叶酸合成酶,磺胺嘧啶暴露浓度60 μg·mL-1尚不足以完全阻断菌株生长环境中二氢叶酸合成酶对PABA合成四氢叶酸的过程。但是,由于培养基中嘌呤和嘧啶等合成有关成分不断被消耗,因此磺胺嘧啶对菌株的生长抑制作用于16 h后逐渐开始显现,这也可能是菌株在特定时间表达sul基因的原因。
试验发现3种sul基因在微生物体内的表达具有时间特异性,其中可能存在着一种严谨的表达调控模式。无论有无磺胺抗生素的暴露,菌株SYN201和NJJN802中磺胺类抗性基因(sul1、sul2、sul3)分别在第72小时或36小时表达最高。但是,3种sul基因在该特定时间上表达后又迅速下降至一个极低的水平,其中具体机制在本实验中尚无法给出明确的解释,这同时是值得进一步研究的内容。
试验发现在一定浓度范围内,随着磺胺嘧啶暴露浓度的增加,sul基因的表达随之显现出上升趋势,且大多数sul基因呈现出明显的剂量-效应关系。当磺胺抗生素暴露浓度超过最高限值,耐药菌的正常生长和繁殖即受到抑制,从而使得耐药菌表达sul基因的量减少。在同一株菌内,3种磺胺类抗性基因的表达水平呈现sul1>sul2>sul3的趋势。
[1] Kü mmerer K. The presence of pharmaceuticals in the environment due to human use—Present knowledge and future challenges [J]. Journal of Environmental Management, 2009, 90: 2354-2366
[2] Martinez J L. Antibiotics and antibiotic resistance genes in natural environments [J]. Science, 2008, 321: 365-367
[3] 杨晓洪, 王娜, 叶波平. 畜禽养殖中的抗生素残留以及耐药菌和抗性基因研究进展[J]. 药物生物技术, 2014, 21(6): 583-588
Yang X H, Wang N, Ye B P. Review onantibiotic residues, antibiotic resistant bacteria and antibiotic resistant genes in livestock and poultry production [J]. Pharmaceutical Biotechnology, 2014, 21(6): 583-588 (in Chinese)
[4] Tello A, Austin B, Telfer T C. Selective pressure of antibiotic pollution on bacteria of importance to public health [J]. Environmental Health Perspectives, 2012, 120: 1100-1106
[5] Lai H T, Hou J H. Light and microbial effects on the transformation of four sulfonamides in eel pond water and sediment [J]. Aquaculture, 2008, 283(1): 50-55
[6] 陈昦, 董元华, 王辉, 等. 江苏省畜禽粪便中磺胺类药物残留特征[J]. 农业环境科学学报, 2008, 27(1): 385-389
Chen H, Dong Y H, Wang H, et al. Residual characteristics of sulfanilamide in animal feces in Jiangsu Province [J]. Journal of Agro-Environment Science, 2008, 27(1): 385-389 (in Chinese)
[7] 冀秀玲, 刘芳, 沈群辉, 等. 养殖场废水中磺胺类和四环素抗生素及其抗性基因的定量检测[J]. 生态环境学报, 2011, 20(5): 927-933
Ji X L, Liu F, Shen Q H, et al. Quantitative detection of sulfonamides and tetracycline antibiotics and resistance genes in sewage farms [J]. Ecology and Environmental Sciences, 2011, 20(5): 927-933 (in Chinese)
[8] Byrne-Bailey K, Gaze W H, Kay P, et al. Prevalence of sulfonamide resistance genes in bacterial isolates from manured agricultural soils and pig slurry in the United Kingdom [J]. Antimicrobial Agents and Chemotherapy, 2009, 53(2): 696-702
[9] Phuong Hoa P T, Managaki S, Nakada N, et al. Abundance of sulfonamide-resistant bacteria and their resistance genes in integrated aquaculture-agriculture ponds, North Vietnam [J]. Interdisciplinary Studies on Environmental Chemistry—Biological Responses to Contaminants, 2010: 15-22
[10] Phuong Hoa P T, Nonaka L, Hung Viet P, et al. Detection of the sul1, sul2, and sul3 genes in sulfonamide-resistant bacteria from wastewater and shrimp ponds of North Vietnam [J]. Science of the Total Environment, 2008, 405(1): 377-384
[11] Skold O. Sulfonamide resistance: Mechanisms and trends [J]. Drug Resistance Updates, 2000, 3(3): 155-160
[12] Enne V I, King A, Livermore D M, et al. Sulfonamide resistance in Haemophilus influenzae mediated by acquisition of sul2 or a short insertion in chromosomal folP [J]. Antimicrobial Agents and Chemotherapy, 2002, 46(6): 1934-1939
[13] Perreten V, Boerlin P. A new sulfonamide resistance gene (sul3) in Escherichia coli is widespread in the pig population of Switzerland [J]. Antimicrobial Agents and Chemotherapy, 2003, 47(3): 1169-1172
[14] Suzuki S, Phuong Hoa P T. Distribution of quinolones, sulfonamides, tetracyclines in aquatic environment and antibiotic resistance in Indochina [J]. Frontiers in Microbiology, 2012, 3: 67-68
[15] Yun M K, Wu Y, Li Z, et al. Catalysis and sulfa drug resistance in dihydropteroate synthase [J]. Science, 2012, 335(6072): 1110-1114
◆
Expression Patterns ofsulGenes in Sulfonamide-Resistant Bacteria
Wang Na1,2, Yang Xiaohong3, Guo Xinyan1,2, Chen Biao3, Ye Boping3, Ge Feng1,*
1. Nanjing Institute of Environmental Sciences, MEP, Nanjing 210042, China 2. Key Laboratory of Pesticide Environmental Assessment and Pollution Control, MEP, Nanjing 210042, China 3. School of Life Science and Technology, China Pharmaceutical University, Nanjing 210009, China
Antibiotic pollution is the key factor for the transport and spread of antibiotic resistance genes (ARGs). However, there were few reports on the expression mechanism of ARGs in antibiotic resistant bacteria (ARBs) under the exposure of antibiotics. The different expression of sul genes were detected by RT-PCR respectively in Bacillus anthracis (SYN201, G+) and Shigella flexneri (NJJN802, G-) at different times and different sulfadiazine concentrations. It is found that the mRNA levels of sul genes in strain SYN201 and NJJN802 reached climax at 72 h and 36 h respectively regardless of exposure to sulfadiazine, whereas they did not express, or express at a relatively low level at other times. The presence of sulfadiazine was helpful to improve the expression level of resistance gene at 72 h for SYN201 and 36 h for NJJN802. Compared with the group without sulfadiazine, the relative expression of sul1, sul2 and sul3 in strain SYN201 exposed to 60 μg·mL-1sulfadiazine increased as 2.5, 4.8 and 3.2 times, and that in NJJN802 increased as 3.7, 6.0 and 5.0 times. When investigating the relative expression level of sul genes in the resistant strains under the exposure to different concentrations of sulfonamides, we found that the expression levels of sul genes in the resistant strains SYN201 and NJJN802 increased as the exposure concentrations of sulfonamides (0-1 024 μg·mL-1) increased. This study will shed light on the expression characteristics of ARGs of sulfadiazine-resistant bacteria and the role of antibiotic exposure in the expression of ARGs.
sulfadiazine; Bacillus anthracis; Shigella flexneri; Sul gene expression; antibiotic resistance genes
国家自然科学基金(No. 21507037);环保公益专项(201309031,201109038)
王娜(1983—),女,博士,助理研究员,研究方向为有机污染物的生态与健康风险,E-mail: wangna@nies.org;
*通讯作者(Corresponding author), E-mail: gefeng@nies.org
10.7524/AJE.1673-5897.20150430001
2015-04-30录用日期:2015-08-05
1673-5897(2015)5-075-08
X171.5
A
葛峰(1981—),男,环境微生物学博士,副研究员,主要研究方向环境毒理学,发表学术论文40余篇。
王娜,杨晓洪,郭欣妍, 等. 磺胺类耐药菌中抗性基因sul的表达规律[J]. 生态毒理学报,2015, 10(5): 75-81
Wang N, Yang X H, Guo X Y, et al. Expression patterns of sul genes in sulfonamide-resistant bacteria [J]. Asian Journal of Ecotoxicology, 2015, 10(5): 75-81 (in Chinese)
