环境中抗药基因水平转移研究进展
2015-03-14刘苗苗杨敏张昱姚宏
刘苗苗,杨敏,张昱,姚宏
1. 北京交通大学土木建筑工程学院市政环境工程系 水中典型污染物控制与水质保障北京市重点实验室,北京 100044 2. 中国科学院生态环境研究中心 环境水质学国家重点实验室,北京 100085
环境中抗药基因水平转移研究进展
刘苗苗1,杨敏2,*,张昱2,姚宏1
1. 北京交通大学土木建筑工程学院市政环境工程系 水中典型污染物控制与水质保障北京市重点实验室,北京 100044 2. 中国科学院生态环境研究中心 环境水质学国家重点实验室,北京 100085
抗药基因是一种新型环境污染物,一旦被致病菌获得,将导致抗生素的临床使用失效,从而危害公共健康。抗药基因可通过转移因子在细菌间传播,加剧抗药基因的扩散效应,提升了环境健康风险性,因此抗药基因水平转移受到了越来越多的关注。本文介绍了抗药基因水平转移的分子机制,总结了不同环境中各种抗药基因水平转移途径的研究现状,并对该领域将来的研究方向提出了展望,以期为环境中抗药基因的调查评估、污染控制及合理管理提供理论参考。
抗药基因;水平转移;质粒;整合子;噬菌体
自19世纪40年代青霉素首次被发现并成功使用以来,抗生素对疾病治疗和人类健康起到了极大的促进作用[1]。但随着抗生素的大量生产及使用,其在环境中的残留水平逐渐升高,对细菌形成了选择压力,由此显著促进了抗药性的产生和传播[2]。抗药性一旦被致病菌获得,会导致抗生素临床治疗失效,严重威胁人类健康。迄今为止,已在致病菌(如金黄葡萄球菌)中频繁发现抗药基因,并且许多细菌具有多重抗药性,甚至进化成对已有抗生素均具有抗药性的超级细菌[3]。据美国疾病控制研究中心统计,美国每年至少有2 000 000人受到抗药菌感染,其中至少23 000人死于这些感染(http://www.cdc.gov/drugresistance/)。
细菌抗药性通过抗药基因来实现遗传表达。抗药基因编码具有特殊功能的蛋白质,并通过修饰抗生素与核糖体的结合位点、泵出抗生素、或改变抗生素结构等途径使抗生素失去抑菌作用。细菌主要通过3种方式获得抗药性:从环境或其他细菌中通过水平转移因子(如质粒、整合子、基因盒、转座子、病毒等)获得抗药基因;在抗生素及其他物质(如重金属、杀虫剂、纳米材料等)的选择作用下产生抗药性;在遗传过程中细菌自身发生基因突变[2]。其中抗生素及其他选择压力可以促进抗药基因的水平转移和基因突变,但其中基因突变的发生概率较低。在过去的70年当中,抗药基因出现频率大幅增加的主要原因是在残留抗生素的选择作用下,水平转移促进了抗药基因的传播和增殖[2]。
水平转移是指抗药基因通过接合、转化、转导等途径在细菌和细菌之间、环境和细菌之间、病毒和细菌之间传播,最终导致更多细菌获得抗药性[4]。水平转移使抗药基因的传播突破了生物遗传的种属保守限制,抗药基因可在同种属细菌之间转移,也可以在亲缘关系较远的环境细菌和致病菌、或革兰氏阴性菌和革兰氏阳性菌之间传播[3]。致病菌中的抗药基因很可能来自环境细菌,临床上发现的氨基糖苷类、万古霉素类、β-内酰胺类、喹诺酮类抗药基因均和环境中的抗药基因存在直接联系[5]。常见致病菌中由质粒携带的β-内酰胺类抗药基因CTX-M 可以溯源到环境中的Kluyvera spp.菌属(克鲁沃菌属,属于肠杆菌科,γ-变形菌纲)[6]。在临床上分离到的具有环丙沙星抗药性的Klebsiella pneumonia (克雷伯氏肺炎球菌)中发现,其质粒上携带的喹诺酮类抗药基因(qnr 基因)能追溯到几种水生环境细菌[7-8]。致病菌水平转移促进抗药基因的扩散和增殖,提升了环境健康风险,因此环境中抗药基因的水平转移受到了科学家们的广泛关注。
1 抗药基因水平转移的途径、机制及相关基因元件
几乎所有的功能基因都能进行水平转移[9]。介导抗药基因水平转移的转移因子主要包括质粒、整合子、转座子和噬菌体。抗药基因水平转移的途径主要有3种,分别为接合、转化和转导(见图1)[3]。其中,接合涉及到受体细菌和供体细菌之间的生理接触,需要在细胞之间形成通道以供抗药基因传递,抗药基因能通过接合在不同界的生物之间进行遗传物质传递,如细菌和植物之间,以及细菌和酵母菌之间,这种转移方式主要通过可转移的或可移动的质粒实现;转化是指从环境中吸收游离的DNA,成为细菌自身的遗传物质,这种转移方式能够在亲缘关系较远的微生物之间传播;转导是指噬菌体在自我复制的过程中将一个宿主的遗传物质转移到另一个宿主当中(普通转导),或将噬菌体吸附位点附近的DNA转移到宿主当中(特异转导)[4]。
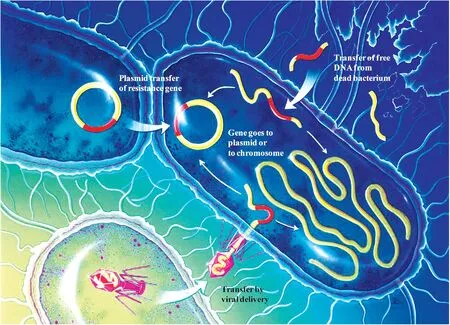
图1 抗药基因的水平转移机制[3]
1.1 整合子(integrons)
整合子通过基因盒(gene cassettes)的位点特异重组系统捕获和转移抗药基因,同时提供启动子以实现基因盒的表达,在抗药基因水平转移中起到非常重要的作用[10]。整合子的主要结构包括3个关键部分:整合酶基因(intI 基因)、特异重组位点(attI )、启动子(Pc)。整合子通常存在2种重组类型:(1)在attI 位点和attC 位点之间,在attI 位点上插入基因盒;(2)在2个attC 位点之间,基因盒被剪切[11]。基因盒既可以是线状形式的整合子,也可以是共价闭合的环状游离基因。基因盒通常不带有启动子,因此需要启动子辅助其实现抗药基因的表达[12]。
根据整合酶基因序列可将整合子分为5类(I型~V型),常见的整合子主要是I~III型[10]。至今,已在整合子上发现了130余种抗药基因,如编码β-内酰胺类、氨基糖苷类、甲氧苄氨嘧啶类、氯霉素类、喹诺酮类、利福平类、大环内酯类等抗生素的抗药基因。携带抗药基因的整合子自身并不能转移,但可以以其他转移因子(如质粒和转座子)为载体实现抗药基因在细菌之间的传播。其中I型整合子在临床和环境细菌,尤其是革兰氏阴性菌中分布最为广泛。I型整合子通常和一些较大的转座子相连,如Tn402等,促进了其在环境中的传播,甚至环境细菌中I型整合子携带的基因盒多样性高于临床细菌[10]。II型整合子的检出频率仅次于I型整合子,但由于大多数II型整合子中含有一个终止子,使得II型整合子的基因盒组成较为稳定,主要包括甲氧苄氨嘧啶类、氨基糖苷类、链丝菌素类抗药基因。II型整合子通常与转座子Tn7相连,因此加强了自身的传播。目前,其他类型的整合子(III型~V型)在临床细菌中检出频率较低,而在环境细菌中关于这些整合子的研究仍然较少。
值得一提的是,由于I型整合子具有如下特征:(1)与抗药基因、抗重金属基因、抗消毒剂基因相连;(2)在许多致病菌和非致病菌中均有检出;(3)其宿主具有较短的世代时间,其丰度变化较快,且能通过水平转移在细菌之间传播;(4)在受人类活动影响的环境中发现特异的I型整合子序列,因此,Gillings等[13]提出I型整合子可以作为人类活动污染的指示基因。
1.2 插入序列共同区域和复杂整合子(insertion sequence common region and complex integrons)
随着自然环境中残留抗生素增加,整合子发生了进化,因此有些整合子在基因盒区域以外增加了一些非基因盒抗性基因。这些非基因盒抗性基因通常与共同区域(CR , common region)相连,且位于I型整合子3’端保守区下游,由于共同区域含有正向插入序列(insertion sequence, IS),因此也被称为“插入序列共同区域”(insertion sequence common region, ISCR s)[14]。ISCR s作为一种新型转移因子,由于其携带β-内酰胺、碳青霉烯、氯霉素、甲氧苄啶和喹诺酮等多种抗生素抗药基因而备受关注。根据保守区域结构对已发现的ISCR s进行分类,可分为20余类,其中1~3类ISCR s(ISCR 1~ISCR 3)上抗药基因出现的频率最高。除了自身携带抗药基因外,ISCR s有一个重要特征,即能与普通整合子形成复杂整合子(complex integrons),实现抗药基因的传播[14]。
1.3 转座子(transponson)
转座子(transposon)又称跳跃基因(jumping gene),是最早发现的可以水平移动的遗传单元,广泛存在于原核生物和真核生物中。Courvalin等[15]观察到一株没有质粒的临床肺链球菌(Streptococcus pneumoniae )具有多药抗性,后来发现该多药抗性是由其染色体上的可连接转座子(Tn1545, 25.3 kb)引起的,该转座子可转移到多种革兰氏阳性菌种,也可转移到重组缺陷的大肠埃希氏菌(Escherichia coli HB101)及芽孢杆菌(Bacillus subtilis )的染色体上。转座子转移的第一步是剪切特定的遗传分子(如抗药基因),形成不可复制的环形中间物;第二步是转座子通过该环形中间物的特异位点插入实现遗传因子(如抗药基因)的水平转移。转座子宿主广泛存在,能在革兰氏阴性菌和阳性菌之间传播。一个细菌细胞内可同时存在多个转座子,转座子既可以存在于质粒上,也可以存在于染色体上,转座子能携带多种抗药基因,如四环素、红霉素、氯霉素、卡那霉素抗药基因。转座子传播抗药基因主要有以下4种机制:(1)最常见的是转座子内部编码的抗药基因直接转移;(2)通过编码抗药基因的质粒或其他转座子的转移而传播抗药基因,如Tn916转座子的剪切和转移有利于同一细胞中其他可接合转座子的转移,而位于不可接合质粒上的可接合转座子也使得该质粒具有转移能力;(3)整合到更大的可连接转座子中,如Tn5251可整合到更大的含有更多抗药基因的Tn5252中;(4)允许供体和受体之间进行染色体上同源基因的重组[16]。
1.4 质粒(plasmids)
质粒(plasmids)是染色体外能够进行自主复制的DNA分子。来自细菌细胞的质粒大多是双链、共价闭合的环状分子,以超螺旋形式存在,是细菌的辅助遗传单位。质粒对宿主生存没有决定性作用,但其存在赋予宿主细菌特殊功能。根据质粒携带的基因和功能不同,可分为抗性质粒、致育因子、降解质粒、侵入性质粒等。抗性质粒可能携带抗生素、重金属等抗性基因,是抗药基因转移的最重要载体,前文所述的转座子、插入序列共同区域、整合子等转移元件都可以在质粒上进行组合,然后通过接合的方式实现转移,因此,质粒介导的传播是抗药基因水平转移的最主要机制[17-18]。质粒的接合转移主要包括以下几种类型:(1)自我传送功能(self-transmissible)接合质粒的转移,如F-质粒和大肠杆菌的RP4质粒。(2)可移动性(mobilizable)质粒的转移。一个不具有自我传送功能但具有一个起始接合转移位点(oriT )的质粒能通过接合质粒进行转移。如IncQ质粒RSF1010可以通过接合IncP1质粒(如RP4)进行转移。(3)共合体(cointegration)的转移。一个没有自我传送功能及移动功能的环状质粒可以与另一个有自我传送功能的环状质粒合为一体,从而被转移[19]。
1.5 噬菌体(bacteriophages)
噬菌体(bacteriophages)是生物圈中丰度最高的生物体,估计总数量范围为1030~1032。噬菌体由DNA或RNA基因组及蛋白质外衣组成,可将自身的基因组注入宿主细胞,之后,这些噬菌体基因组在整合酶的作用下整合到宿主基因组中,或在宿主细胞内复制,该过程称为转导。在转导过程中,噬菌体作为载体实现不同宿主细菌之间的基因交换,从而可能传播抗药基因[20]。近年来年,关于环境中噬菌体介导的抗药基因水平转移受到越来越多的关注。
2 抗药基因水平转移的研究方法
2.1 细菌培养和传统分子生物学技术
在新一代测序技术出现以前,人们主要通过细菌培养、PCR、定量PCR、克隆、基因转移等实验手段来研究抗药基因的水平转移。一方面,使用培养方法从环境中筛选抗药菌,通过药敏试验确定该细菌的抗性谱,并通过DNA提取、PCR检测测定细菌中的转移因子及携带的抗药基因,最后以这些抗药菌作为供体细菌,以抗生素敏感的模式细菌(如大肠杆菌、假单胞菌等)作为受体细菌,在一定条件下共同培养,考察抗药基因从供体菌向受体菌转移的概率[21]。另一方面,也可将环境样品的总基因组作为研究对象,使用PCR、定量PCR等手段定性和定量分析抗药基因及转移因子的存在情况和丰度水平,并通过系统发育多样性分析(克隆)、数据统计分析(如相关性分析)来探讨抗药基因的来源及转移因子在抗药基因传播中的可能作用[5, 22]。
2.2 新一代测序技术
新一代测序技术克服了环境中大多数细菌不可培养的局限性,目前已普遍应用于抗药基因及转移因子的研究[23]。数以千计的宏基因组测序结果已上传到一些公共平台(如MG-RAST、IMG/M)中,这些测序结果可用于探索各种环境中的抗药基因组(resistome)和转移基因组(mobilome)[24-25]。Balcazar等[20]通过MG-RAST平台比较了海洋、土壤、淡水、人类粪便、污水处理系统等不同环境样品的测序结果(共27个宏基因组),发现上述环境中均含有很高水平的转移因子,包括质粒、整合子、噬菌体,人类粪便、污水处理系统中的抗药基因和转移因子均高于其他自然环境。由于宏基因组测序不依赖于细菌培养,且能够同时得到微生物群落组成和功能的信息,并且随着测序方法的不断更新,测序成本逐渐降低,公共数据库和生物信息学分析手段日渐成熟和普及,该方法逐渐成为环境中抗药基因研究的有力工具[26-27]。
3 环境中抗药基因水平转移的研究进展
3.1 环境中质粒介导的抗药基因水平转移
质粒是抗药基因转移的重要媒介,尤其是广谱宿主质粒,能促进抗药基因在不同种属、不同门、甚至不同界之间传播,因此,它在各种环境中的出现、分布、基因型、抗药性表型受到了广泛关注。Binh等[28]对猪粪中的质粒进行了系统研究,使用E. coli 共捕获了15个猪粪样品中的228个转化接合子(通过接合方式获得了其他细菌基因的受体细菌),并通过基因组DNA的PCR和杂交等技术,在猪粪中发现81个质粒,包含了IncN、IncW、IncP-1、pHHV216等类型,并在这些质粒上检测到阿莫西林、磺胺类抗药基因。Bengtsson-Palme等[21]使用测序方法在印度某抗生素污染河流中检测到26种已知质粒和21种新型质粒,表明抗生素污染导致环境中的抗药基因具有很高的水平转移潜力。Szczepanowski等[29]从城市污水厂的活性污泥和出水中分别筛选了140株和123株细菌,通过高通量测序发现细菌质粒上几乎含有所有抗生素的抗药基因。Zhang等[30]从城市污水活性污泥中直接提取总质粒DNA,发现其中含有大量抗药基因、整合子和转座子,表明质粒是污水处理系统中抗药基因的重要传播媒介。Parsley等[31]使用测序和功能宏基因组方法,在城市污水厂活性污泥的质粒基因组中发现了细菌抗药基因,表明污水处理系统中抗药基因可通过质粒(接合)进行转移。在海洋的养殖区域、以及远海(离岸边522 km)、深海(深度达8 200 m)区域均发现抗药性细菌,并在其中检测到质粒携带的抗药基因,且海洋细菌中的抗性质粒存在情况与其抗生素污染程度存在正相关[32]。Jechalke等[33-34]使用PCR、定量PCR等分子生物学手段,在经过100年污水灌溉的土壤中发现了28个IncP-1ε质粒,且这些质粒全部携带I型整合子,表明污水灌溉导致土壤中的质粒、整合子、抗药基因均显著升高。
3.2 环境中整合子及异常插入序列介导的抗药基因水平转移
I型整合子作为最常见的整合子,在城市污水、医疗废水、养殖废弃物、土壤、地表水中均有报导(见表1)。I型整合子在革兰氏阴性菌中分布尤其广泛,在革兰氏阴性人类病原菌、畜牧业病原菌和共生菌中的检出率甚至高达40%~70%[11]。Agerso等[35]在猪粪和猪场土壤中分别筛选出了44和216株细菌,发现其中分别有25%和7%的细菌含有I型整合子,携带多种氨基糖苷类、甲氧苄啶类抗药基因。Li等[36]结合筛菌和分子生物学方法,发现土霉素生产废水处理系统出水异养菌中I型整合子的检出率高达94.2%,但未在其基因盒区域发现相应的四环素类抗药基因。使用定量PCR方法调查发现,四环素和螺旋霉素生产废水中I型整合子的相对丰度高达10-1~100数量级,且与多种四环素类、大环内酯类抗药基因呈显著正相关[37-38]。Wang等[39]发现在猪粪灌溉的土壤中,I型整合子丰度为10-3数量级,并发现I型整合子和四环素类抗药基因tet (G)、磺胺类抗药基因sul (II)之间存在显著正相关关系。Mokracka等[40]从波兰某城市污水厂中筛选了1 832株肠细菌,发现其中207株(11.3%)含有I型整合子,14株(1.0%)含有II型整合子,并在其中发现了多种抗药基因盒。I型整合子在自然水体中也很常见,Drudge等[41]综合使用PCR和基因芯片等方法研究发现,某淡水水域中I型整合子中具有很高的多样性,9种抗生素的抗药基因均被检出。
II型整合子的179位点含有编码终止子的碱基序列,导致其合成的多肽不完整、无活性,因此,环境中II型整合子的检出率和多样性通常显著低于I型整合子[10, 42]。Mokracka等[40]从城市污水厂筛选的肠细菌中发现14株(1.0%)细菌含有II型整合子,并在这些整合子上发现了氨基糖苷类、甲氧苄啶类、链丝菌素类等抗药基因。Marathe等[43]在印度某抗生素生产废水处理系统中发现II型整合子的检出率高达80%,但未在其中发现抗药基因盒。尽管目前II型整合子的检出率较低,且携带的抗药基因盒较少,但关于II型整合子在抗药基因水平转移中的作用仍需要更多的研究。
目前关于异常插入序列及复杂整合子也有一些报导。Wang等[44]从医院环境筛选革兰氏阴性菌,通过PCR检测发现33.3%的细菌含有ISCR 1,并在其上发现了12种喹诺酮类、β-内酰胺类抗药基因。Kristiansson等[45]使用高通量测序的方法在抗生素生产废水受纳河流的排放口和下游检出了ISCR 2。Xia等[46-47]发现从医院废水筛选的细菌中发现ISCR 1,该ISCR 1元件和I型整合子形成的复杂整合子携带多种喹诺酮类抗药基因。
3.3 环境中噬菌体介导的抗药基因水平转移
通过噬菌体转导的抗药基因水平转移可能也是一个重要途径,环境中噬菌体介导的抗药基因水平转移受到越来越多的关注。噬菌体对环境中的细菌具有很高的适应性[49],裂解性噬菌体存在于30%的可培养细菌中;基于非培养方法的估计表明,土壤细菌中4%~68%携带噬菌体。虽然目前发现转导对致病基因岛、病毒特征转移的作用超过对抗药基因转移的作用,但Allen等[50]对使用抗生素的猪粪噬菌体基因组进行研究,发现抗生素的存在能对猪粪中的噬菌体产生选择作用。Colomer-Lluch等[51]使用实时定量PCR方法,在城市污水和屠宰废水的噬菌体基因组中均检出β-内酰胺类、喹诺酮类、磺胺类、甲氧西林抗药基因,在城市污水和河水的病毒基因组中发现了较高水平的β-内酰胺类抗药基因bla (TEM)和bla (CTX-M)及磺胺类抗药基因sul1 (102~104copies·mL-1,与之相比,细菌DNA中的抗药基因丰度为105~107copies·mL-1),并通过转移实验验证了这2种基因能使抗生素敏感的埃希氏大肠杆菌(E. coli )中产生β-内酰胺类抗药性,证实了环境中抗药基因通过噬菌体传播的可能性[52]。此外,该作者在城市污水和动物养殖废水的噬菌体DNA中检出喹诺酮类抗药基因qnr A和qnr S,丰度水平达到101~103copies·mL-1,并发现一些噬菌体诱导因子(如EDTA、柠檬酸钠)导致噬菌体基因组中喹诺酮类、β-内酰胺类抗药基因丰度升高[53]。Marti等[54]使用qPCR方法定量研究发现,医院废水和城市污水处理系统出水的病毒基因组中含有较高水平的β-内酰胺类、喹诺酮类抗药基因,其中qnr S和blaSHV的丰度达到104copies·mL-1。

表1 环境中I型整合子和II型整合子携带的抗药基因盒Table 1 Antibiotic resistance gene cassettes in Class 1 and Class 2 integron from environment
4 抗药基因水平转移的影响因素
首先,抗药基因水平转移受供体细菌和受体细菌浓度的影响。由于接合转移需要细胞和细胞之间接触形成鞭毛或膜孔通道,因此细菌浓度高可能有利于接合转移的发生。Guo等[55]从城市污水厂筛选含有抗药性质粒的大肠埃希氏菌(E. coli )作为供体细菌,使用不具有抗药性的E. coli NK5449作为受体细菌,发现细菌浓度低于104CFU·mL-1时,接合转移受到明显抑制(几乎不发生接合转移),而高于105CFU·mL-1时,不受细菌浓度影响,表明可能存在接合转移的细菌浓度阈值。
其次,环境中的营养元素,如碳源、氮、磷等也对抗药基因水平转移产生显著影响。Guo等[55]发现添加葡萄糖、α-乳糖能将城市污水中抗药基因的水平转移频率提高1~4个数量级,而添加氮和磷也能显著提高接合转化子的数量。
此外,环境中的抗生素、重金属选择压力可能促进抗药基因的水平转移。Zhu等[56]使用高通量定量PCR芯片(high-capacity quantitative PCR arrays)比较了饲喂抗生素和不饲喂抗生素的猪粪处理处置过程中的抗药基因和转移因子,发现土霉素和铜均促进了抗药基因和转移因子水平的升高,且抗生素和重金属同时存在会形成更强的促进作用。医疗废水、制药废水等环境中抗生素浓度高,对生物处理的功能主体-细菌形成较强的选择作用,为抗药基因的水平转移创造了有利条件。城市污水中虽然抗生素浓度较养殖废水、医疗废水、制药废水低,但其中抗生素种类繁多,其生物处理过程也是抗药基因水平转移发生的重要环境。
为了充分利用水资源,常采用三级处理如消毒、膜处理等方法对生物处理出水进行进一步净化,以获得再生水,饮用水通常也会使用氯、臭氧、紫外等消毒方法。消毒过程对抗药基因水平转移的影响受到越来越多的关注。Shi等[57]使用宏基因组测序方法研究了实际饮用水处理过程中的抗药基因,发现氯消毒不仅富集了抗药基因,同时也富集了质粒、插入序列、整合子等转移因子。Guo等[55]通过批量试验比较了紫外消毒和氯消毒对城市污水中抗药基因水平转移的影响,发现在较低的消毒剂使用剂量下,紫外消毒对接合转移没有显著影响,氯消毒使得接合转移频率升高2~5倍,而当消毒剂量升高到一定范围,紫外和氯消毒均能有效抑制抗药基因的水平转移。氯消毒在较低剂量下可能促进抗药基因水平转移,一方面是由于氯消毒剂提高细胞膜通透性,另一方面则是由于抗氯基因可能和抗药基因共存于质粒等转移元件上,因此氯消毒剂会在选择抗氯基因的同时对抗药基因产生共选择作用。
5 展望
抗药菌和抗药基因的在环境中的分布和传播是国际上环境学科、微生物学科、公共卫生领域关注的重点问题,尽管过去几十年国内外很多研究人员致力于探索抗药基因的传播规律,但至今为止抗药基因在环境中的传播风险和控制技术仍存在很多困难。许多研究表明,生物处理过程中的高生物量可能促进抗药基因的扩散,而水处理中常用的消毒方法对抗药基因的控制效果尚不明确,在某些条件下甚至可能起到促进作用[55],给抗药基因的控制带来阻碍。关于抗药基因的行为及影响机制还需要深入研究,随着新一代测序技术日渐成熟,综合使用传统培养法、分子生物学方法、宏基因组测序等方法对环境中抗药基因的传播途径进行深入研究,将为抗生素的合理使用及抗药基因的有效控制提供参考。
[1] Walsh C. Antibiotics: Actions, Origins, Resistance [M]. American Society for Microbiology, 2003: 1-345
[2] Martinez J L. Environmental pollution by antibiotics and by antibiotic resistance determinants [J]. Environmental Pollution, 2009, 157(11): 2893-2902
[3] Rosenblatt-Farrell N. The landscape of antibiotic resistance [J]. Environmental Health Perspectives, 2009, 117(6): A244-A250
[4] Ochman H, Jeffrey G L, Eduardo A G. Lateral gene transfer and the nature of bacterial innovation [J]. Nature, 2000, 405(6784): 299-304
[5] Perry J A, Gerard D W. The antibiotic resistance “mobilome”: Searching for the link between environment and clinic [J]. Frontiers in Microbiology, 2013, 4: 138-144
[6] Humeniuk C, Guillaume A, Valerie G, et al. β-Lactamases of Kluyvera ascorbata , probable progenitors of some plasmid-encoded CTX-M types [J]. Antimicrobial Agents and Chemotherapy, 2002, 46(9): 3045-3049
[7] Poirel L, Jose-Manuel R M, Hedi M, et al. Origin of plasmid-mediated quinolone resistance determinant QnrA [J]. Antimicrobial Agents and Chemotherapy, 2005, 49(8): 3523-3525
[8] Baquero F, José-Luis M R C. Antibiotics and antibiotic resistance in water environments [J]. Current Opinion in Biotechnology, 2008, 19(3): 260-265
[9] Gogarten J, Peter J, Townsend P. Horizontal gene transfer, genome innovation and evolution [J]. Nature Reviews Microbiology, 2005, 3(9): 679-687
[10] Stalder T, Olivier B, Magali C, et al. Integron involvement in environmental spread of antibiotic resistance [J]. Frontiers in Microbiology, 2012, 3: 119-132
[11] Mazel D. Integrons: Agents of bacterial evolution [J]. Nature Reviews Microbiology, 2006, 4(8): 608-620
[12] Collis C, Ruth M, Hall M. Site-specific deletion and rearrangement of integron insert genes catalyzed by the integron DNA integrase [J]. Journal of Bacteriology, 1992, 174(5): 1574-1585
[13] Gillings M R, William H G, Amy P, et al. Using the class 1 integron-integrase gene as a proxy for anthropogenic pollution [J]. The ISME Journal, 2015, 9: 1269-1279
[14] Toleman M A, Bennett P M, Walsh T R. ISCR elements: Novel gene-capturing systems of the 21st century? [J]. Microbiology and Molecular Biology Reviews, 2006, 70(2): 296-316
[15] Courvalin P. Transfer of antibiotic resistance genes between gram-positive and gram-negative bacteria [J]. Antimicrobial Agents and Chemotherapy, 1994, 38(7): 1447-1451
[16] Rice L B. Tn916 family conjugative transposons and dissemination of antimicrobial resistance determinants [J]. Antimicrobial Agents and Chemotherapy, 1998, 42(8): 1871-1877
[17] Bennett P M. Plasmid encoded antibiotic resistance: Acquisition and transfer of antibiotic resistance genes in bacteria [J]. British Journal of Pharmacology, 2008, 153(s1): S347-S357
[18] Norman A, Lars H H, Søren J S. Conjugative plasmids: Vessels of the communal gene pool [J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2009, 364(1527): 2275-2289
[19] Davison J. Genetic exchange between bacteria in the environment [J]. Plasmid, 1999, 42(2): 73-91
[20] Balcazar J L. Bacteriophages as vehicles for antibiotic resistance genes in the environment [J]. PLoS Pathogens, 2014, 10(7): e1004219
[21] Bengtsson-Palme J, Fredrik B, Jerker F, et al. Shotgun metagenomics reveals a wide array of antibiotic resistance genes and mobile elements in a polluted lake in India [J]. Frontiers in Microbiology, 2014, 5: 648-661
[22] Vaz-Moreira I, Nunes O C, Manaia C M. Bacterial diversity and antibiotic resistance in water habitats: Searching the links with the human microbiome [J]. FEMS Microbiology Reviews, 2014, 38(4): 761-778
[23] Metzker M L. Sequencing technologies - The next generation [J]. Nature Reviews Genetics, 2010, 11(1): 31-46
[24] Markowitz V M, Chen I M A, Palaniappan K, et al. IMG: The integrated microbial genomes database and comparative analysis system [J]. Nucleic Acids Research, 2012, 40(D1): D115-D122
[25] Meyer F, Paarmann D, D'Souza M, et al. The metagenomics RAST server-A public resource for the automatic phylogenetic and functional analysis of metagenomes [J]. BMC Bioinformatics, 2008, 9(1): 386-393
[26] Knight R, Jansson J, Field D, et al. Unlocking the potential of metagenomics through replicated experimental design [J]. Nature Biotechnology, 2012, 30(6): 513-520
[27] Schmieder R, Edwards R. Insights into antibiotic resistance through metagenomic approaches [J]. Future Microbiology, 2012, 7(1): 73-89
[28] Binh C T T, Heuer H, Kaupenjohann M, et al. Piggery manure used for soil fertilization is a reservoir for transferable antibiotic resistance plasmids [J]. FEMS Microbiology Ecology, 2008, 66(1): 25-37
[29] Szczepanowski R, Linke B, Krahn I, et al. Detection of 140 clinically relevant antibiotic-resistance genes in the plasmid metagenome of wastewater treatment plant bacteria showing reduced susceptibility to selected antibiotics [J]. Microbiology-SGM, 2009, 155: 2306-2319
[30] Zhang T, Zhang X X, Ye L. Plasmid metagenome reveals high levels of antibiotic resistance genes and mobile genetic elements in activated sludge [J]. PLoS ONE, 2011, 6(10): e26041
[31] Parsley L C, Erin J C, Kavita S K, et al. Identification of diverse antimicrobial resistance determinants carried on bacterial, plasmid, or viral metagenomes from an activated sludge microbial assemblage [J]. Applied and Environmental Microbiology, 2010, 76(11): 3753-3757
[32] Aminov R I. Horizontal gene exchange in environmental microbiota [J]. Frontiers in Microbiology, 2011, 2: 158-176
[33] Jechalke S, Melanie B, Friederike L, et al. Effects of 100 years wastewater irrigation on resistance genes, class 1 integrons and IncP-1 plasmids in Mexican soil [J]. Frontiers in Microbiology, 2015, 6: 163-172
[34] Jechalke S, Schreiter S, Wolters B, et al. Widespread dissemination of class 1 integron components in soils and related ecosystems as revealed by cultivation-independent analysis [J]. Frontiers in Microbiology, 2013, 4: 420-427
[35] Agersø Y, Sandvang D. Class 1 integrons and tetracycline resistance genes in Alcaligenes , Arthrobacter , and Pseudomonas spp. isolated from pigsties and manured soil [J]. Applied and Environmental Microbiology, 2005, 71(12): 7941-7947
[36] Li D, Yu T, Zhang Y, et al. Antibiotic resistance characteristics of environmental bacteria from an oxytetracycline production wastewater treatment plant and the receiving river [J]. Applied and Environmental Microbiology, 2010, 76(11): 3444-3451
[37] Liu M M, Zhang Y, Yang M, et al. Abundance and distribution of tetracycline resistance genes and mobile elements in an oxytetracycline production wastewater treatment system [J]. Environmental Science & Technology, 2012, 46(14): 7551-7557
[38] Liu M M, Ding R, Zhang Y, et al. Abundance and distribution of macrolide-lincosamide- streptogramin resistance genes in an anaerobic-aerobic system treating spiramycin production wastewater [J]. Water Research, 2014, 63: 33-41
[39] Wang F H, Qiao M, Lv Z E, et al. Impact of reclaimed water irrigation on antibiotic resistance in public parks, Beijing, China [J]. Environmental Pollution, 2014, 184: 247-253
[40] Mokracka J, Koczura R, Kaznowski A. Multiresistant Enterobacteriaceae with class 1 and class 2 integrons in a municipal wastewater treatment plant [J]. Water Research, 2012, 46(10): 3353-3363
[41] Drudge C N, Amy V C E, Janina M P, et al. Diversity of integron-and culture-associated antibiotic resistance genes in freshwater floc [J]. Applied and Environmental Microbiology, 2012, 78(12): 4367-4372
[42] Liu M M, Zhang Y, Ding R, et al. Response of activated sludge to the treatment of oxytetracycline production waste stream [J]. Applied Microbiology and Biotechnology, 2013, 97: 8805-8812
[43] Marathe N P, Viduthalai R R, Sandeep A W, et al. A treatment plant receiving waste water from multiple bulk drug manufacturers is a reservoir for highly multi-drug resistant integron-bearing bacteria [J]. PLoS ONE, 2013, 8(10): e77310
[44] Wang F P, Wu K H, Sun J J, et al. Novel ISCR1-linked resistance genes found in multidrug-resistant Gram-negative bacteria in southern China [J]. International Journal of Antimicrobial Agents, 2012, 40(5): 404-408
[45] Kristiansson E, Fick J, Janzon A, et al. Pyrosequencing of antibiotic-contaminated river sediments reveals high levels of resistance and gene transfer elements [J]. PLoS ONE, 2011, 6(2): e17038
[46] Xia R, Guo X, Zhang Y, et al. qnr VC-like gene located in a novel complex class 1 integron harboring the ISCR 1 element in an Aeromonas punctata strain from an aquatic environment in Shandong Province, China [J]. Antimicrobial Agents and Chemotherapy, 2010, 54(8): 3471-3474
[47] Xia R R, Ren Y, Xu H. Identification of plasmid-mediated quinolone resistance qnr genes in multidrug-resistant Gram-negative bacteria from hospital wastewaters and receiving waters in the Jinan Area, China [J]. Microbial Drug Resistance, 2013, 19(6): 446-456
[48] Li D, Yang M, Hu J Y, et al. Determination and fate of oxytetracycline and related compounds in oxytetracycline production wastewater and the receiving river [J]. Environmental Toxicology and Chemistry, 2008, 27(1): 80-86
[49] Vos M, Philip J B, Elizabeth B, et al. Local adaptation of bacteriophages to their bacterial hosts in soil [J]. Science, 2009, 325(5942): 833-833
[50] Allen H K, Torey L, Darrell O B, et al. Antibiotics in feed induce prophages in swine fecal microbiomes [J]. MBio, 2011, 2(6): e00260-11
[51] Colomer-Lluch M, William C C, Sihem J, et al. Antibiotic resistance genes in bacterial and bacteriophage fractions of Tunisian and Spanish wastewaters as markers to compare the antibiotic resistance patterns in each population [J]. Environment International, 2014, 73: 167-175
[52] Colomer-Lluch M, Jofre J, Muniesa M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples [J]. PLoS One, 2011, 6(3): e17549
[53] Colomer-Lluch M, Jofre J, Muniesa M. Quinolone resistance genes (qnr A and qnr S) in bacteriophage particles from wastewater samples and the effect of inducing agents on packaged antibiotic resistance genes [J]. Journal of Antimicrobial Chemotherapy, 2014, 69(5): 1265-1274
[54] Marti E, Balcázar E V. Bacteriophages as a reservoir of extended‐spectrum β‐lactamase and fluoroquinolone resistance genes in the environment [J]. Clinical Microbiology and Infection, 2014, 20(7): O456-O459
[55] Guo M T, Yuan Q B, Yang J. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater [J]. Environmental Science & Technology, 2015, 49(9): 5771-5778
[56] Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms [J]. Proceedings of the National Academy of Sciences, 2013, 110(9): 3435-3440
[57] Shi P, Jia S, Zhang X X, et al. Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water [J]. Water Research, 2012, 47(1): 111-120
Horizontal Transfer of Antibiotic Resistance Genes in Environment: A Review
Liu Miaomiao1, Yang Min2,*, Zhang Yu2, Yao Hong1
1. Department of Municipal and Environmental Engineering, Beijing Jiaotong University, Beijing 100044, China 2. State Key Laboratory of Environmental Aquatic Chemistry, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China
Received 1 June 2015 accepted 24 August 2015
Antibiotic resistance genes are a major emerging environmental pollutant of concern as they reduce the efficacy of antibiotic medical treatment and consequently threaten public health. Horizontal gene transfer of such genes between bacteria greatly accelerates the spread of antibiotic resistance, intensifying the public health risk. Thus, the mobility of antibiotic resistance genes has drawn increasing attention worldwide. This review introduces the molecular mechanisms of the horizontal transfer of antibiotic resistance genes. It is summarized that the variation of these mechanisms amongst different environments. Based on this current progress, future research directions are proposed. Overall, this review aims to provide insight for the future survey and control of antibiotic resistance genes in the environment.
antibiotic resistance genes; horizontal transfer; plasmids; integrons; bacteriophage
国家自然科学基金青年基金(51408032);环境模拟与污染控制国家重点联合实验室(中国科学院生态环境研究中心)专项经费(14K01ESPCR)
刘苗苗(1986-),女,博士,讲师,研究方向为环境微生物,E-mail: lmmhit@163.com;
*通讯作者(Corresponding author), E-mail: yangmin@mails.rcees.ac.cn
10.7524/AJE.1673-5897.20150601001
2015-06-01录用日期:2015-08-24
1673-5897(2015)5-011-09
X171.5
A
杨敏(1964-),男,研究员,博士生导师。主要研究方向为工业废水、城市污水、饮用水处理技术及其中的微生物作用机制。
刘苗苗,杨敏,张昱, 等. 环境中抗药基因水平转移研究进展[J]. 生态毒理学报,2015, 10(5): 11-19
Liu M M, Yang M, Zhang Y, et al. Horizontal transfer of antibiotic resistance genes in environment: A review [J]. Asian Journal of Ecotoxicology, 2015, 10(5): 11-19 (in Chinese)
