阪崎克罗诺杆菌的16S rDNA 鉴定分型
2015-03-09娄彬彬满朝新牛婕婷柴云雷姜毓君
娄彬彬 ,满朝新,费 鹏,牛婕婷,李 然,柴云雷,李 理,姜毓君,
(1东北农业大学食品学院/乳品科学教育部重点实验室,哈尔滨 150030;2东北农业大学/国家乳业工程技术研究中心,哈尔滨 150028)
阪崎克罗诺杆菌是一种含周身鞭毛、能运动并兼性厌氧的革兰氏阴性无芽孢的粗短杆菌。目前克罗诺杆菌属有7 个种[1,2],包括阪崎克罗诺杆菌(C.sakazakii)、丙二酸盐阳性克罗诺杆菌(C.malonaticus)、苏黎世克罗诺杆菌 (C.turicensis)、穆汀斯克罗诺杆菌(C.muytjensii)、都柏林克罗诺杆菌(C.dublinensis)、传奇克罗诺杆菌 (C.universalis)、调味品克罗诺杆菌(C.condimenti)。阪崎克罗诺杆菌是重要的食源性条件致病菌,能够引起严重的新生儿脑膜炎[3]、坏死性小肠结肠炎[4]和菌血症[5]。该菌能分离自广泛的食品来源(包括肉、奶酪、蔬菜、药草、调料和婴幼儿配方粉等)[6,7]、环境、以及昆虫[8,9]。
传统的生化鉴定方法(如API20E),表现出了很大的局限性,如重复性差、分辨力低、易出现假阳性结果等。因此,依据细菌遗传性特征的分子分型(亦称基因分型)技术逐步应用于致病菌的溯源分析及数据库的构建中。本研究利用核糖体分型中以16sRNA 基因序列为研究对象,其在生物体中种类少,含量较多,存在与所有细胞中,功能同源且最为古老,序列变化与进化距离相适应[10]。可通过对保守序列设计通用引物可实现对细菌的鉴定,对可变序列设计特异性引物,用于检测菌种或菌属间的差异并对细菌进行分类。本研究利用16S rDNA 基因测序的方法对37 株阪崎克罗诺杆菌进行鉴定和分型,鉴定结果表明细菌在分子水平上的鉴定较生化鉴定具有更好的准确性和重复性。通过构建系统发育树分析并根据序列同源性比对,可将所有菌株进行分型,这表明16S rDNA 基因测序可以对阪崎克罗诺杆菌进行分型,并进一步揭示其系统发育关系。说明16S rDNA基因测序能够对阪崎克罗诺杆菌进行鉴定和分型。
1 材料与方法
1.1 试验菌株
37 株阪崎克罗诺杆菌,分离自2009—2012年期间不同地区生产的乳粉和婴幼儿配方粉样品及某一湿法加工婴幼儿配方粉生产企业的加工环境和成品的分离菌株,以及来自实验室间菌株交换和相关单位的赠送的标准菌株。
1.2 试剂与仪器
细菌基因组DNA 提取试剂盒,北京天根生物技术有限公司;SeaKem Gold 琼脂糖,美国Amresco 公司;引物,北京invitrogen 公司合成;胰蛋白胨大豆琼脂(TSA),青岛海博生物有限公司;Bcn1360 型生物超净工作台:上海佳胜仪器制造有限公司;9700 PCR 扩增仪:美国Applied Biosystems 公司;DYY-10C 型电泳仪:北京市六一仪器厂;UVP 凝胶成像系统:美国UVP 公司;TGC-16G 型离心机:上海安亭科学仪器厂;高压蒸汽灭菌锅:上海三申医疗器械有限公司;DHP-9272 型电热恒温培养箱:上海一恒科技有限公司。
1.3 试验方法
1.3.1 菌株的培养、活化与纯化
取出-80 ℃甘油保存的含有阪崎克罗诺杆菌的冻存管,将菌株恢复至室温后,按2%的接种量接种于LB液体培养基,37 ℃培养8~12 h 进行活化。用接种环蘸取活化后的菌液于TSA 平板上划线,37 ℃过夜培养。使平板上长出阪崎克罗诺杆菌单菌落,已达到纯化的目的。
1.3.2 基因组总DNA 的提取
挑取TSA 平板中的单菌落,在LB 培养液中过夜培养。取2mL 培养液,进行阪崎克罗诺杆菌全基因组提取,采用细菌基因组DNA 提取试剂盒,具体操作步骤参照试剂盒说明。
1.3.3 16S rDNA 序列的扩增
阪崎克罗诺杆菌16S rDNA 序列扩增:采用16S rDNA 通用引物27f:‘5-AGTCTCTGATCATGC-CTCAG-3’和1492r:‘5-AAGGAGGTGCTCCAGCC-3’。扩增体系为50μL:10 × Buffer (含MgCl2),5.0 μL;dNTPs (10 mmol/L),2.0μL;引物27f (10 μmol/L),1.0 μL;引物1 492r (10 μmol/L),1.0μL;Pyrobest DNA Polymerase (2.5 U/μL),1.0μL;DNA 模板 (20~50 ng),2.0μL,补加ddH2O 至50μL。
PCR 扩增条件:95 ℃预变性5 min;95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸1 min;循环数为30 个;72℃终延伸5 min;将PCR 扩增产物在1 %的琼脂糖凝胶(含EB)上进行电泳检测,100 V 电压和100 A 电流电泳30 min,在凝胶成像系统下观察,检测是否有扩增条带和条带大小。
1.3.4 16S rDNA 序列的测序
将所有菌株的16S rDNA PCR 扩增产物和引物,送到上海立菲生物技术有限公司进行双向测序。测序结果用Chromas 1.45 软件进行查阅,用DNAMAN 5.29 软件将双向测序结果进行剪切与拼接,并将拼接后的序列在NCBI 上进行Blast 序列比对,实现对分离菌株的鉴定。从GenBank 核酸数据库中提取IVERSEN 等递交的克罗诺菌属的相关菌种和亚种的模式菌株及肠杆菌属的相关菌株的16S rDNA 序列作为参比,利用MEGA 4 软件构建系统发育树。
2 结果与分析
2.1 基因组总DNA 的提取与鉴定
将试验菌株参照细菌基因组DNA 提取试剂盒,提取基因组总DNA。将提取后的产物于1%的琼脂糖凝胶(含EB)上进行电泳检测,电泳结果如图1。
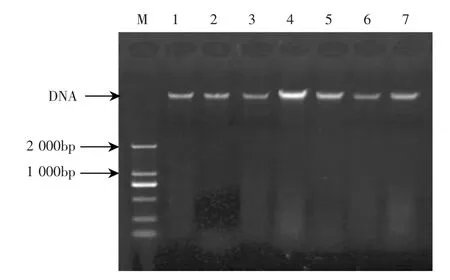
图1 试验菌株基因组总DNA 提取电泳图
由图1 可知,基因组DNA 条带清晰单一、无杂带,可知DNA 提取效果良好,可作为模板进行后续16S rDNA 序列扩增。
2.2 16S rDNA 序列的扩增
采用通用引物27f 和1492r 对菌株16S rDNA 序列进行扩增,扩增产物经1 %琼脂糖凝胶电泳进行检测,检测结果图2 所示。
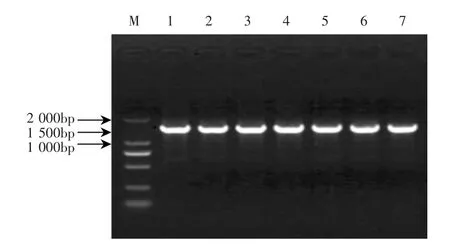
图2 试验菌株16S rDNA PCR 产物电泳图
由图2 可知,扩增产物条带单一明亮,且大小在1 500 bp 左右,与目的条带大小一致。说明产物为16S rDNA 序列扩增产物,可送去测序。
2.3 16S rDNA 序列的测定
将37 株分离菌株的16S rDNA PCR 扩增产物和引物,送到上海立菲生物技术有限公司进行双向测序。测序结果用软件剪切和拼接后,将序列在NCBI 上进行Blast 序列比对。比对结果显示,所有菌株(包括ES59)除了ES4 外,其余菌株与Cronobacter sakazakii 的同源性最高,相似性达99%,而ES4 的比对结果发现它与Citrobacter freundii 的同源性最高,达99%,因此ES4 被鉴定为弗氏柠檬酸杆菌,其余36 株菌株均鉴定为阪崎克罗诺杆菌。这一说明表明除菌株ES4 外,其余66 株分离菌株均鉴定为阪崎克罗诺杆菌。以分离菌株ES1 为例,经Blast 比对后得到的分值较高的菌株为Cronobacter sakazakii,且相似性达99% (附表),其他菌株的同源性分析结果与ES1 相似。
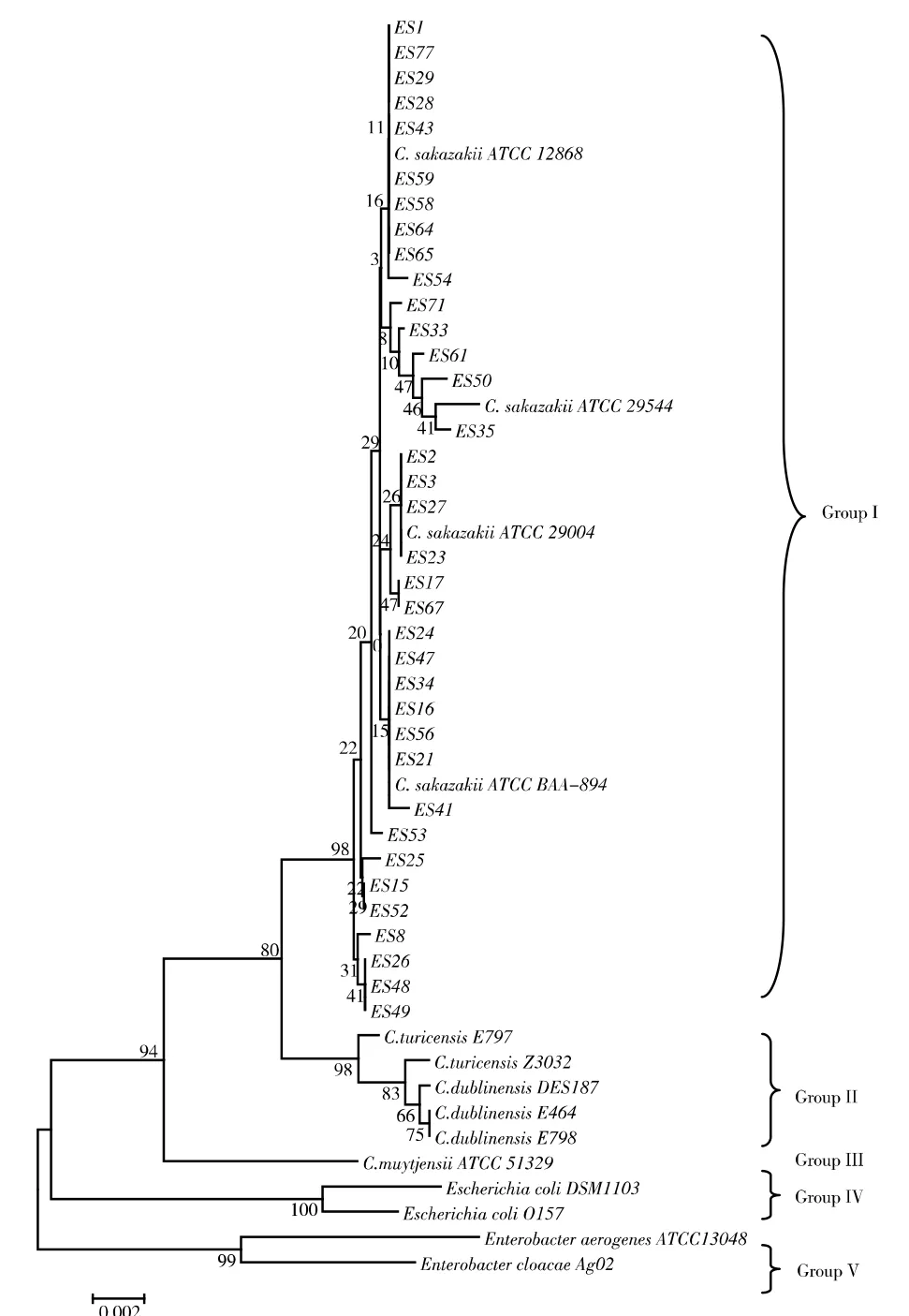
图3 基于16S rDNA 序列构建的系统发育树
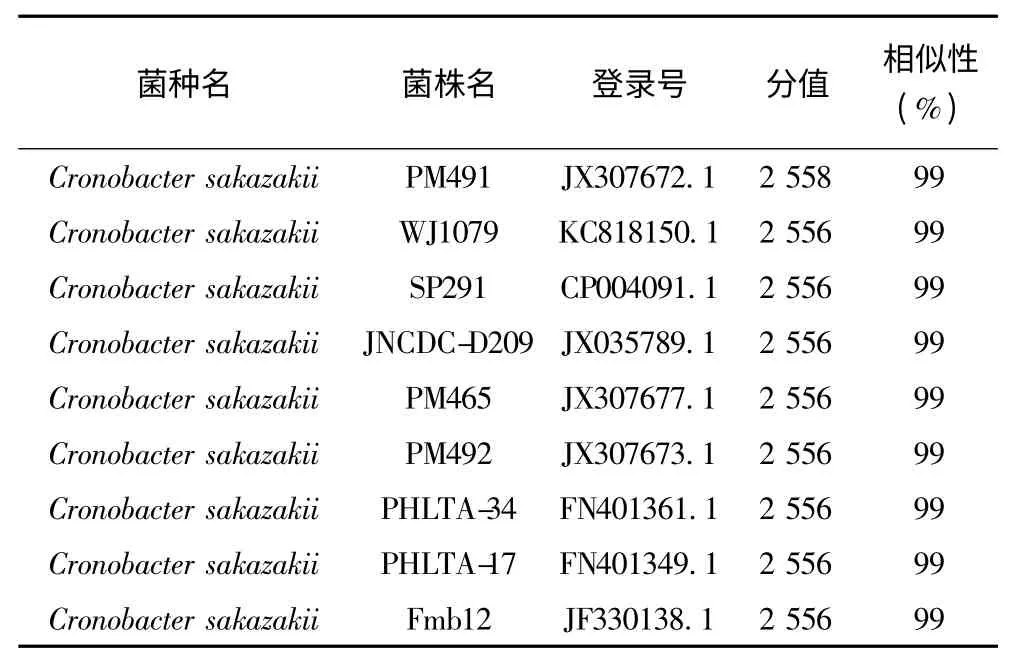
附表 分离菌株ES1 在GenBank 中同部分16S rDNA 序列同源性比对结果
2.4 16S rDNA 序列构建系统发育树及分析
因菌株ES4 被鉴定为弗氏柠檬酸杆菌,所以不参与后续试验分析。利用MEGA 4.1 软件对36 株分离菌株、阪崎克罗诺杆菌标准菌株,克罗诺菌属的相关菌种和亚种的模式菌株(包括苏黎世克罗诺杆菌和都柏林克罗诺杆菌)及肠杆菌属的相关菌株(包括大肠埃希氏菌、产气肠杆菌和阴沟肠杆菌),共计50 株菌株构建系统发育树(图3),参数设置为邻接相邻算法,Kimura 2-parameter 模式,重复次数为1 000 次。
对系统发育树分析,结果显示所有菌株被分成五个类群(Group I~Group V),其中分离菌株与阪崎克罗诺杆菌ATCC 标准菌株(除ATCC51329)在同一系统发育分支Group I,说明亲缘关系最近,这与鉴定结果相符。克罗诺杆菌属的其它菌种组成Group II 类群,ATCC51329 呈单一分支,为Group III。此外,大肠埃希氏菌和大肠埃希氏O157 属于Group IV,产气肠杆菌和阴沟肠杆菌属于Group V。对Group I 中菌株进行多重序列比对分析,发现菌株间的相似度为97.35%,有研究学者认为,当16S rDNA 序列的相似度小于98.3 %时,可以对该组群进一步分型[11]。
在阪崎克罗诺杆菌的分支下可分成7 个子群,子群1 包含10 株分离菌株和1 株ATCC12868,它们之间的相似性为98.51%;子群2 含有5 株分离菌株和1 株ATCC29544,群内相似性为98.30%;子群3 包含6 株分离菌株和1 株ATCC29004,群内相似性为98.37%;子群4 包含8 株分离菌株和1 株ATCCBAA-894,群内相似性为98.82%;子群5 只含有一株菌株ES53;子群6 和7 分别含有3 株分离菌株和4 株分离菌株,它们的群内相似性分别为98.34%和98.46%。
3 结论
本试验结果显示,利用API20E 鉴定为阪崎克罗诺杆菌的ES4 被进一步鉴定为弗氏柠檬酸杆菌(Citrobacter freundii),其余分离菌株的16S rDNA 序列与Cronobacter sakazakii 的同源性最高,相似性达99%,鉴定为阪崎克罗诺杆菌。这表明基因水平的鉴定具有一定的准确性和可靠性,与传统鉴定方法相比,具有更好的灵敏性,较优于传统生化反应的鉴定。此外利用MEGA 4.1 软件构建的系统发育树显示,所有分离菌株位于同一类群中(Group I),依据群内菌株间基因序列的同源性比对,又将Group I 中的菌株分成7 个子群,这说明16S rDNA 测序具有一定的分型能力,可以帮助我们更好的了解不同克罗诺杆菌之间的系统发育关系。
[1]董晓晖,李程思,吴清平,等.食品污染克罗诺杆菌(阪崎肠杆菌)的分离和鉴定[J].微生物学报,2013,53(5):429-436.
[2]JOSEPH S,CETINKAYA E,DRAHOVSKA H,et al.Cronobacter condimenti sp.nov.,isolated from spiced meat and Cronobacter universalis sp.nov.,a novel species designation for Cronobacter sp.genomospecies 1,recovered from a leg infection,waterand food ingredients [J].Int J Syst Evol Microb,2012,62:1277-1283.
[3]Biering G,Karlsson S,Clark N C,et al.Three cases of neonatal meningitis caused by Enterobacter sakazakii in powdered milk [J].Journal of Clinical Microbiology,1989,27(9):2054-2056.
[4]Van Acker J,de Smet F,Muyldermans G,et al.Outbreak of necrotizing enterocolitis associated with Enterobacter sakazakii in powdered milk formula [J].Journal of Clinical Microbiology,2001,39(1):293-297.
[5]Muytjens H L,Zanen H C,Sonderkamp H J,et al.Analysis of eight cases of neonatal meningitis and sepsis due to Enterobacter sakazakii [J].Journal of Clinical Microbiology,1983,18(1):115-150.
[6]TURCOVSK I,KUNIKOV K,DRAHOVSK H,et al.Biochemical and molecular characterization of Cronobacter spp.(formerly Enterobacter sakazakii)isolated from foods [J].Anton Leeuw,2011,99(2):257-269.
[7]王艳,焦彦朝.辣椒制品中阪崎肠杆菌的检测与鉴定[J].贵州农业科学,2012,40(6):198-200.
[8]Hamilton,J.V.,Lehane,M.J.,Braig,H.R.,Isolation of Enterobacter sakazakii from midgut of Stomoxys calcitrans[J].Emerging Infectious Diseases,2003,9:1355-1356.
[9]Kandhai,M.C.,Reij,M.W.,van Puyvelde,K.,Guillaume-Gentil,O.,Beumer,R.R.,van Schothorst,M.,Occurrence of Enterobacter sakazakii in food production environments and households [J].Lancet,2004,363:39-40.
[10]雷正瑜.16S rDNA 序列分析技术在微生物分类鉴定中的应用[J].湖北生态工程职业技术学院学报,2006,4(1):4-7.
[11]Rabeb Miled-Bennour,Timothy C.Ells.Genotypic and phenotypic characterization of a collection of Cronobacter (Enterobacter sakazakii)isolates [J].International Journal of Food Microbiology,2010,139:116-125.
