基于nad5 基因分析黑斑蛙裂头蚴种系发育关系
2015-03-09王先坤邓兴珍刘谭彬
李 晖,王先坤,邓兴珍,刘谭彬,谭 磊,刘 伟*
(1.湘西民族职业技术学院,湖南 吉首 416000;2.湖南农业大学 动物医学学院,湖南 长沙 410128;3.沅陵县畜牧水产局,湖南 怀化 419600)
裂头蚴(Sparganum)属于假叶目,双叶槽科,裂头属。并且是绦虫的幼虫阶段。裂头蚴的感染地区主要以亚洲南部为主,特别是在一些发展中国家感染尤为突出。裂头蚴主要以感染犬、猫为主,也能感染人,是一种人畜共患的寄生虫病。裂头蚴在宿主中的寄生部位因宿主不同而存在差异,例如:在猪、蛇、蛙等宿主主要是寄生在皮下、粘膜和肌肉,偶见于猪的胆管内。而在猫、犬、人等宿主则以寄生在小肠内为主[1-4]。
随着时间的推移,很多寄生虫在形态学上发生了改变,所以传统的鉴定方法已经不能有效的进行分类。而此时,分子标记已经在寄生虫学的分类学中被广泛应用。近年来,线粒体DNA(mtDNA)作为一种核外遗传物质,具有分子量小、结构简单、进化速度快、母性遗传、无组织特异性等特点,使之成为研究寄生虫系统进化的一种理想的分子标记[5-6]。利用PCR 方法并结合DNA 序列分析,首次系统地对蛙裂头蚴线粒体利用线粒体nad5 基因的序列进行多态性研究,并利用nad5重构裂头蚴与其他绦虫蚴的种群遗传关系,从而明确裂头蚴线粒体nad5 基因序列能否成为理想的种间遗传标记,从而为裂头蚴的种群体遗传学研究和其相关疾病的诊断奠定了基础[7-10]。
1 材料和方法
1.1 虫体样品 9 个黑斑蛙裂头蚴样品采自湖南张家界(SeZJJ)、湖南星沙(SeXS)、湖南衡阳(SeHY)、湖南株洲县(SeZZX)、湖南望城(SeWC)、湖南邵阳(SeSY)、湖南浏阳(SeLY)、湖南长沙(SeCS)、湖南醴陵(SeLL),分别保存于70 %酒精中。
1.2 主要试剂 Taq DNA 聚合酶及相关试剂购自TaKaRa 公司;蛋白酶K 购自Merck 公司;WizardTMDNA Clean-up System 试剂盒和基因组DNA 提取试剂盒(Wizard SV GenomicDNA Purification System)购自Promega 公司。引物由上海生工生物工程技术服务有限公司合成。
1.3 目的DNA 片段扩增 利用DNA 提取试剂盒提取裂头蚴基因组DNA,以其为模板,按照文献[11]设计合成一对引物(5'-TCATACTGGGTCTATCA GGTGTT-3'/5'-ACAGCAAAGTTAGGGGGTAATAGG T-3'),进行nad5 基因片段的PCR 扩增,预期扩增片段为510 bp。反应体系为50 μL,扩增条件为:94 ℃5 min;94 ℃30 s、51 ℃30 s、72 ℃30 s,共38 个循环;72 ℃5 min。PCR 产物经1 %琼脂糖凝胶电泳检测。
1.4 nad5 基因片段序列测定和序列分析 PCR 扩增产物由上海生工生物工程技术服务有限公司测序,9 个样品的测序结果采用DNAStar 5.0 软件进行分析,并与GenBank 中登录的代表性的裂头蚴nad5序列进行比较,以胎生网尾线虫(D.viviparus)作为外群。利用Clustal X2.0 及Mega 4.1 软件对扩增的nad5 序列进行比对,并利用Mega 4.1 程序中的临接法(Neighbor-joining method,NJ)构建种系发育树。NJ 法在软件默认设置下进行分析;同时利用DNAStar 5.0 中的Megalign 程序进行相似性分析。
2 结果与讨论
2.1 目的DNA 片段的扩增结果 9 个黑斑蛙裂头蚴样品均扩增出约510 bp nad5,与预期目的片段长度相符,并且无非特异性片段,空白对照为阴性(图1)。
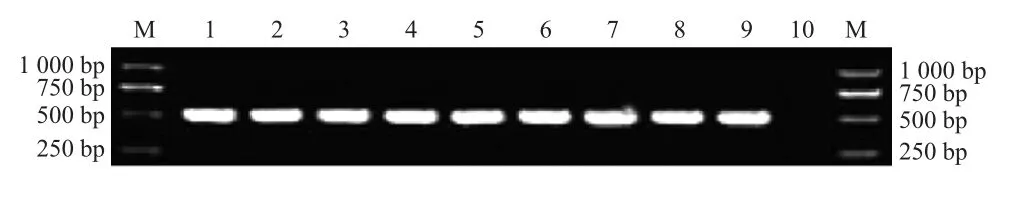
图1 裂头蚴线粒体p nad5 PCR 扩增结果Fig.1 Amplification of mtDNA nad5 gene fragment from frog sparganum by PCR
2.2 测序与序列分析结果 测序结果显示,扩增的蛙裂头蚴nad5 片段大小为510 bp,对9 条黑斑蛙裂头蚴nad5序列进行了种内比较,结果显示,9 个不同个体间的nad5序列的差异为0~3 %,分离株之间的nad5序列相差21 bp,其差异主要是以碱基替换为主。湖南省各分离株之间相应序列的相似性在97 %以上,湖南分离株与基因库中其它裂头蚴nad5序列的相似度均低于89 %。
2.3 nad5 基因序列系统发生树的构建 通过种系发育分析显示,利用NJ 法构建系统发生树。结果显示,湖南9 株分离株中,有5 株(SeCS、SeLL、SeHY、SeZJJ 和SeZZX)位于同一分支,并且同源性为100 %。而其余4 株湖南分离株(SeSY、SeLY、SeXS 和SeWC)与阔节裂头绦虫(D.latum)位于另一分支,这4 株湖南分离株同源性为96 %,而与阔节裂头绦虫的同源性为89 %(图2)。系统发生树中的Bootstrap 值较高。湖南省各分离株之间相隔较近,但与其他绦虫所属分支相隔较远,具有鉴别意义。
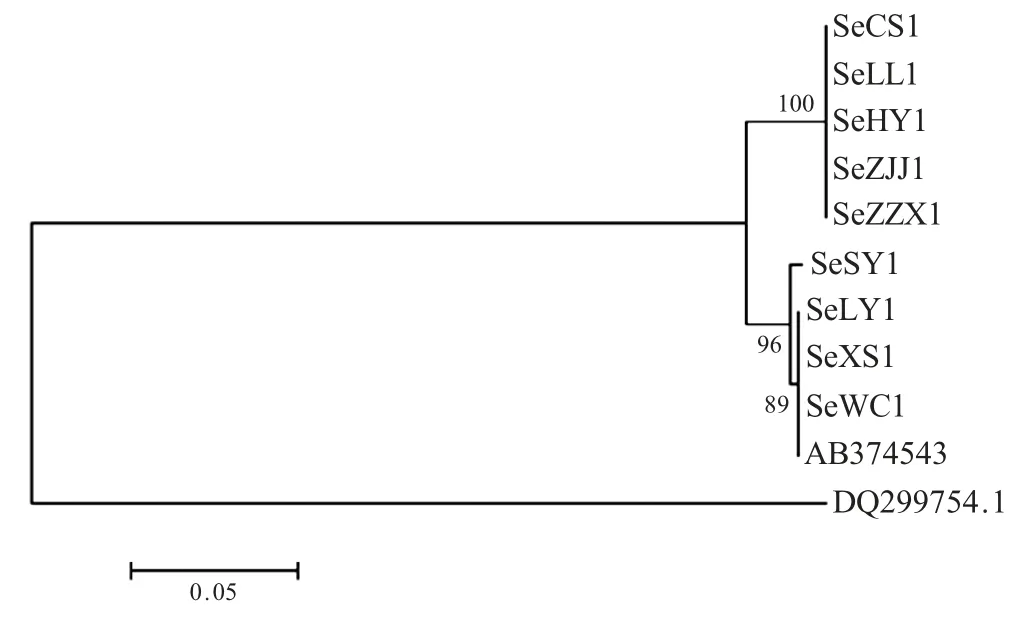
图2 基于nad5 基因序列所构建的系统发生树Fig.2 The Bootstrap consensus phylogram of the stationary tree reconstructed by NJ based on the nad5 sequences
本研究结果表明,蛙裂头蚴湖南省分离株序列与基因库中的D.latum 的序列均有相当高的相似性(p nad5 为96.4 %),9 个蛙裂头蚴湖南省分离株与基因库中其它绦虫序列相比,相似性较低nad5 为89 %。采用NJ 法构建的进化树与其具有一致性,显示出湖南省分离株与D.latum 位于同一大分支,9个分离株所属分支与其他绦虫所属分支相隔较远。本研究结果表明,蛙裂头蚴湖南省分离株nad5 序列的种内相对保守,种间差异相对较大,说明nad5 序列能够作为蛙裂头蚴的遗传分子标记用于蛙裂头蚴的分类学和鉴别鉴定研究。本研究结果填补了国内关于蛙裂头蚴nad5 序列研究的空白,为蛙裂头蚴的分类鉴定以及进一步的分子流行病学调查和群体遗传研究奠定了基础。
[1]Zhu Xing-quan,Gasser R B,Chilton N B,et al.Molecular approaches for studying as caridoid nematodes with zoonotic potential,with an emphasis on Toxocara Species[J].J Helminthol,2001,75(2):101-108.
[2]Zhu Xing-quan,Chilton N B,Jacobs D E,et al.Characterisation of Ascaris from human and pig hosts by nuclear ribosomal DNA sequences[J].Int J Parasitol,1999,29(3):469-478.
[3]郭凯文,牛安欧.线粒体基因组及其在寄生虫学中的应用[J].国外医学寄生虫病分册,2002,29(4):149-153.
[4]Liu Wen-jiang,Zhao Guang-hui,Tan M Y,et al.Survey of Spirometra erinaceieuropaei spargana infection in the frog Rana nigromaculata of the Hunan province of China[J].Vet Parasitol,2010,173:152-156.
[5]Gasser R B,Rossi L,Zhu Xing-quan.Identification of Nematodirus species(Nematoda:Molineidae)from wild ruminants in Italy using ribosomal DNA Markers[J].Int J Parasitol,1999,29(11):1809-1817.
[6]Li Ming-wei,Lin Rui-qing,Song Hui-qun,et al.Electrophoretic analysis of sequence variability in three mitochondrial DNA regions for ascaridoid parasites of human and animal health significance[J].Electrophoresis,2008,29(13):2912-2917.
[7]Ooi H K,Chang S L,Huang C C,et al.Survey of Spirometra erinaceieuropaei in frogs in Taiwan and its experimental infection in cats[J].J Helminthol,2000,74(02):173-176.
[8]Pampiglione S,Fioravanti M L,Rivasi F.Human sparganosis in Italy.Case report and review of the European cases[J].APMIS,2003,111(2):349-354.
[9]Tamura K,Dudley J,Nei M,Kumar S.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599.
[10]Zhao Guang-hui,Mo X H,Zou F C,et al.Genetic variability among Schistosoma japonicum isolates from different endemic regions in China revealed by sequences of three mitochondrial DNA genes[J].Vet Parasitol,2009,162(1-2):67-74.
[11]刘自逵,赵光辉,刘国华,等.湖南省猬迭宫绦虫的线粒体nad5 和rrns 基因的序列测定及种系发育分析[J].中国兽医学报,2012,32(2):258-261.
