细胞质处理小体(P-小体)与基因表达的调控
2015-03-06莫蓓莘符晓婕莫小为徐晓峰岳路明
莫蓓莘,符晓婕,莫小为,刘 丽,2,徐晓峰,岳路明
1)深圳大学生命科学学院,深圳市微生物基因工程重点实验室,深圳 518060;2)加州大学河滨分校植物学与植物科学研究院,综合基因组生物学研究所,美国河滨 92521;3)深圳大学生命科学学院,深圳市海洋生物技术与生态环境重点实验室,深圳 518060

【生物工程 / Bioengineering】
细胞质处理小体(P-小体)与基因表达的调控
莫蓓莘1,符晓婕1,莫小为1,刘 丽1,2,徐晓峰3,岳路明3
1)深圳大学生命科学学院,深圳市微生物基因工程重点实验室,深圳 518060;2)加州大学河滨分校植物学与植物科学研究院,综合基因组生物学研究所,美国河滨 92521;3)深圳大学生命科学学院,深圳市海洋生物技术与生态环境重点实验室,深圳 518060
mRNA处理小体,又称P-小体(processing bodies,P-bodies),是细胞质中含有多种功能蛋白和RNA的聚集体. 评述P-小体的形成和运动特点,以及其与小RNA、外切体相互作用,参与基因表达的调控. 介绍了本实验室在P-小体植物细胞中的最新研究进展. 指出P-小体是mRNA降解和储存的场所,在转录后水平参与基因表达的调控,P-小体在细胞中是动态变化的,P-小体中多样化的功能蛋白在P-小体的组装、功能行使中发挥重要作用.P-小体作为细胞质内mRNA的储存和降解结构在基因表达的转录后调控中起重要作用,其调控机制尚待进一步研究补充,P-小体与小RNA分子(microRNA,miRNA)调控的基因沉默之间也存在关联性.
细胞生物学;P-小体;基因表达调控;mRNA降解;翻译抑制;小RNA分子
2002年,Eystathioy等[1]利用患者血清通过免疫筛选建立cDNA文库,并从该文库中鉴定和克隆出能与mRNA相互作用、大小为182 kD的蛋白质(命名为GW182). GW182位于胞质中的独立区域,该区域不同于其他任何已知的细胞器,这一能被GW182抗体标记的胞质区域被命名为GW body(GWB),随后的研究发现GWB是mRNA在细胞质中储存和降解的位点. 2003年,Sheth等[2]在酵母细胞中发现参与mRNA降解的相关因子,包括mRNA脱帽因子decapping protein(Dcp1和Dcp2)、5′-3′核酸外切酶1(5′-3′exonuclease1,Xrn1)、Sm样蛋白(Sm-like proteins,LSm proteins)以及碳代谢抑制物4(carbon catabolite repression4,Ccr4,一种失活酶)等,这些因子与mRNA降解中间产物同样聚集在细胞质的特定区域,这一聚集区域被命名为细胞质处理小体,即P-小体(processing bodies[2],P-bodies). 有充分证据表明,哺乳动物中的GWB蛋白和酵母中的P-小体蛋白是同源的[1],P-小体不仅是mRNA降解或翻译抑制的场所[3],且在microRNA(miRNA)介导的翻译抑制中起作用. 在miRNA介导的翻译抑制中起重要作用的蛋白Argonaute 1(Ago1)和Argonaute 10(Ago10)均定位于P-小体上,显示P-小体与miRNA调控的翻译抑制之间的关联性[4]. 本文评述P-小体的特点、功能及其与小RNA、外切体互作调控基因表达的方式,展望P-小体今后的研究方向.
1 P-小体的特点
1.1 P-小体的形成
Jain等[5]根据大量实验结果提出P-小体在酵母细胞中的组装模型,其主要组装过程为:首先,P-小体的相关因子组成Pat1、Xrn1、Lsm1-7复合物和Dcp1/Dcp2/Edc3(可能包括Dhh1)复合物,这两种复合物被招募到mRNA上;然后,Edc3、Dcp2与Pat1的相互作用,进而形成一个更大的RNA-蛋白复合物[6-7],由于Pat1-Lsm1-7复合物绑定3′端,而脱帽酶Dcp1/Dcp2偏好结合5′帽子结构,致使该RNA-蛋白复合物形成一个“闭环”结构[8];最终组成可见的细胞质颗粒,即P-小体.
文献[5]证明Edc3、Pat1和Lsm4这3种蛋白在P-小体的装配过程中发挥重要作用,mRNA脱帽和P-小体形成都受Edc3蛋白的调控, Edc3通过3个不同的结构域直接与P-小体中的脱帽酶Dcp2和RNA解旋酶Dhh1/Rck相互作用[9],表明Edc3是P-小体组装过程中重要的支架蛋白,该蛋白还具有促进mRNA脱帽和抑制翻译的功能. 在酵母细胞中,位于Pat1蛋白中间的结构域与Lsm1相互作用,位于C-端的结构域与Edc3、Dcp1相互作用促进P-小体组装,并招募Lsm1进入到P-小体中[10]. 与P-小体形成相关的第3个蛋白质是酵母细胞中的Lsm4[11]. Lsm4 C端延伸区含有一个富含谷氨酰胺和天冬酰胺(Q/N-rich)的功能结构域,研究发现,在Edc3缺失的酵母突变体中,Lsm4C末端的(Q/N-rich)结构域能在一定程度上促使P-小体的形成;而Lsm4 C端(Q/N-rich)结构域的单独缺失对P-小体的形成没有影响,但在Edc3Lsm4双突变株中P-小体数量大幅减少[12]. 综上可见,Edc3、Pat1和Lsm4是P-小体形成过程中必不可少的核心蛋白,除以上核心蛋白外,还有许多相关的蛋白质共同参与P-小体的组装.
1.2 P-小体的运动
P-小体在细胞质中的运动与肌球蛋白有关.
拟南芥肌球蛋白家族包含17个蛋白成员[13],分为VIII和XI两类. VIII类肌球蛋白成员与内源结构内质网及胞间连丝相关[14]. P-小体在细胞质中的运动主要受XI类肌球蛋白调控,XI类肌球蛋白与真菌及动物肌球蛋白V有相似结构域,这类肌球蛋白含有1个与肌动蛋白相连的N-末端马达状结构域[15];1个伸展的可以和钙调蛋白结合的内部颈域[16];以及1个C-末端尾部区域,该区域包含一定程度可变的卷曲螺旋结构域和二分球形结构域[17]. XI类肌球蛋白与各种膜性细胞器和囊泡的运动有关[18-19]. XI类肌球蛋白参与高尔基体、线粒体和过氧化物酶体的快速运输[20],内膜囊泡的长距离运输[21],质体运动[22-23]及ER流动[20]和重塑[24]. 最近研究发现,没有膜结构的RNA聚集体,如P-小体的运动也是由XI类肌球蛋白调控的[18-19].
在哺乳动物、酵母和拟南芥细胞中,Dcp1与肌球蛋白XI -K是共定位的,过量表达显性抑制的肌球蛋白尾部区域可有效抑制P小体的运动;两个XI-K等位基因沉默后,P-小体的运动几乎完全消失,在肌球蛋白XI-K沉默突变体中表达XI-K全长基因可恢复P-小体的运动[25]. 这些结果均表明:P-小体在细胞内沿肌动蛋白的运输是由XI类肌球蛋白介导的,肌球蛋白调控有膜包裹的细胞器的运动需要有衔接蛋白的参与,而调控P-小体的运动则是通过直接与P-小体的核心组分Dcp1相互作用的.
2 P-小体与基因表达的调控
2.1 P-小体与mRNA降解
mRNA的降解通过两条通用且高度保守的途径进行[26],分别是由外切体介导的3′-5′降解途径和由P-小体参与调控的5′-3′降解途径(图1).

图1 mRNA降解途径(外切体介导的3′-5′降解途径和 P-小体参与调控的5′-3′降解途径)及其相关酶Fig.1 The mRNA degradation pathways(exosome mediated 3′-5′ degradation pathway and P-bodies involved in regulation of 5′-3′ degradation pathway)and related enzymes
2.1.1 外切体介导的3′-5′降解途径
外切体(exosome)是一种蛋白质复合物,它的核心是由6个蛋白亚基所组成的环状结构,这6个蛋白质都属于同一类核糖核酸酶(核糖核酸酶PH类似蛋白,含有PH结构域)具有3′-5′核糖核酸外切酶活性,辅助蛋白包括一些因子和RNA解旋酶(Ski2/Ski3/Ski8复合物和Ski7蛋白)[27]. 外切体除可以降解细胞质mRNA外,对细胞内大量RNA的加工、调控和运输也具有重要意义[28].
大多数mRNA进行降解的第1步是脱腺苷酸化,即脱去3′端多聚腺苷酸尾(polyA tail),包括PloyA结合蛋白(PABP)的解离和脱腺苷酸酶Ccr4的降解作用,去腺苷化后再通过由Ski2/Ski3/Ski8复合物和Ski7蛋白作为辅助因子的外切体由3′-5′端方向降解mRNA[29].
2.1.2 P-小体参与调控的5′-3′降解途径
与3′-5′mRNA降解不同,5′-3′mRNA降解从脱帽开始. 这一过程由P-小体的核心蛋白脱帽酶催化. 在酿酒酵母中,脱帽酶是由Dcp1和Dcp2构成的二聚体,Dcp2的MutT结构域提供催化活性[30],一些组装因子也参与到脱帽过程,如七聚Lsm1~Lsm7复合物可结合到脱腺苷酸化mRNA的3′末端,促进mRNA脱帽[31-32]. 参与脱帽过程的蛋白还包括Edc、Dhh1和Pat1[33]等. 脱帽完成后,P-小体中的核酸外切酶Xrn1从5′-3′降解mRNA[25].
Dis3(Rrp44)是外切体复合物中的关键亚基,能从3′-5′方向消化RNA上的核苷酸. 最新研究结果表明,酿酒酵母中Dis3的同源物Dis3l2可以停靠或共定位到P-小体中,参与细胞质中mRNA的降解,且其功能是独立于外切体的[34]. 这一结果表明,外切体介导的3′-5′降解和P-小体介导的5′-3′端方向降解之间有相互作用.
由AU富集元件介导的mRNA降解(ARE(AU-richelement)-mediatedMrnadecay)途径,又称AMD途径,也受P-小体的调控.AMD在调节基因表达的过程中是普遍存在的:5%~8%的mRNA,包括许多细胞因子和原癌基因,mRNA的3′非编码区(UTRs)含有ARE因子[35].ARE因子通过介导mRNA降解从而有效地抑制基因表达,研究表明,超过20种不同的蛋白质,特别是串联锌指蛋白家族,能特异地与ARE结合,诱导AMD途径[36]. 在哺乳动物细胞中,tristetraprolin(TTP),butyrateresponsefactor(BRF)1和BRF2都是十分有效的AMD诱导剂[37-39]. 这些蛋白质的主要功能是作为转接器将含ARE的mRNAs与外切体结合,从而进行脱帽或5′至3′方向的降解,进一步使绑定的mRNA进入常规的降解途径[40-42]. 研究发现,TTP、BRF1和BRF2定位于P-小体中[43-44].P-小体和应激颗粒的相互作用可以增强TT和BRF1的过量表达[45].
2.2P-小体与基因表达
进入P-小体中的mRNA并不是全部被P-小体中的脱帽酶脱帽,然后被核酸外切酶从5′-3′降解;而是部分P-小体中的mRNA可进一步结合活化子而重新进入多核糖体,恢复翻译活化状态. 这部分mRNA进入P-小体中从而被屏蔽于翻译机器(多核糖体)之外, 进一步加强了mRNA的翻译抑制状态[45].
在细胞质中,起储存mRNA作用的结构除P-小体外,还有应激颗粒,它是细胞在受到环境刺激时产生的一种胞浆集合体,该颗粒包括mRNA、转录起始因子、RNA结合蛋白TIA和TIA受体蛋白(TIAR),及40S核蛋白体亚单位[46]. 在不同生理环境下,mRNA可在P-小体、应激颗粒和多核糖体之间运动,从而调节和影响细胞的翻译水平,但mRNA的动态变化及影响翻译进程的机制尚不清楚. 研究发现,PCBP2(poly-Cbindingprotein2)在应激颗粒和P-body之间活跃移动,PCBP2的快速迁移对mRNA进入P-小体的存储和翻译起重要作用[47]. 在酵母(S.cerevisiae)中,葡萄糖缺乏导致多聚核糖体丢失,同时促进P-小体体积增大和数目增多,并促进被抑制的mRNA在P-小体中积累[48]. 这些结果显示,P-小体与mRNA翻译抑制密切相关.
免疫荧光实验研究表明,在丙型肝炎病毒(hepatitisCvirus,HCV)感染过的Huh7(高度分化的肝细胞)细胞中,P-小体和应激颗粒的大部分蛋白与病毒核心蛋白共定位于脂肪滴(病毒RNA的包装位点),HCV通过感染细胞,破坏P-小体和应激颗粒的组分,进而调节病毒基因表达[49]. 由于HCV的RNA丰度是受P-小体的组分(miR-122、Argonaute2和RNA解旋酶RCK/P54)调控的,HCV感染细胞后,P-小体和应激颗粒的数量都发生明显变化,P-小体的丰度明显降低,而应激颗粒的数量增加;敲除P-小体和应激颗粒内特定蛋白后,HCV的RNA丰度明显降低,但是细胞外传染性HCV病毒积累增加. 这充分说明P-小体对HCV基因表达具有重要的调控作用[49].
2.3P-小体与小RNA的协同作用
小RNA包括microRNA(miRNA)和小干扰分子RNA(siRNA),可以通过激活核酸内切酶活性、促进翻译抑制、或通过加速mRNA脱帽,进而在一定程度上影响mRNA的降解和翻译[50].
文献[51]的研究表明,哺乳动物中P-小体的重要组成GW182蛋白家族在miRNA介导的翻译抑制过程起重要作用. 首先,在哺乳动物细胞中,敲除GW182功能基因会减少细胞中P-小体的形成和抑制miRNA介导的翻译抑制[4,51];其次,在哺乳动物细胞中引入GW182显性负等位基因会降低P-小体的形成,并影响miRNA/siRNA介导的基因沉默[51];而且敲除果蝇S2细胞中GW182功能基因会抑制miRNA介导的翻译抑制[52]. 这充分说明小RNA介导的基因沉默和P小体的形成相关.RNA诱导沉默复合体(RNA-inducedsilencingcomplex,RISC)是由siRNA与Argonaute蛋白和Dicer酶复合形成的复合物.RISC在RNA干扰(RNAi)过程起重要作用. 在RNAi中,RISC利用siRNA的反义链切割靶mRNA,达到基因沉默. 研究发现,许多与RISC相关的蛋白都集中在P-小体中,如人体细胞中的4个Argonaute蛋白家族成员,及两个RNA解旋酶Rck和Mov10定位在P-小体中[53].miRNA及其靶mRNAs也定位于P-小体中[53-54]. 植物中也发现Argonaute蛋白家族成员,如AGO1和AGO10定位在P-小体中,显示mRNA的区域化引起的翻译抑制与miRNA调控的翻译抑制之间的关联性[4,55].Bhattacharyya等[46]发现肝细胞中内源性CAT-1(cationicaminoacidtransporter-1)mRNA与miR-122共定位于P-小体中,在翻译被miR-122抑制的条件下,CAT-1mRNA从P-存在于P-小体中,当翻译被激活时,CAT-1mRNA小体中被释放. 当mRNA沉默被阻止时(通过敲除miRNA的生物合成途径的成分)会导致P-小体的解聚[56-57],重新引入siRNA可使P-小体再次出现.这些结果充分说明,P-小体通过聚集和解聚参与小RNA介导的基因沉默.
3 P-小体在植物细胞中的研究进展
P-小体作为细胞质内mRNA储存和降解结构,对基因表达的转录后调控起重要作用,其调控机制尚待研究[58-59]. 对P-小体的研究在哺乳动物细胞中取得令人瞩目的成绩[60-61],在酵母细胞中也开展了较深入的研究,但在植物细胞中的研究才刚起步[62-63].

(Olympus FV1000型激光共聚焦显微镜观察, 青色标记为CFP融合蛋白发光情况,bar=10 μm)图2 胁迫条件下烟草细胞中P-小体的聚集[67]Fig.2 Assembly of P-bodies in tobacco cells under stress[67]
本实验室以植物离体培养悬浮细胞(烟草悬浮细胞)为材料,将特异存在于P-小体中的蛋白Dcp1的基因和青色荧光蛋白的基因融合并转入烟草悬浮细胞中,对转基因烟草细胞进行低温(4 ℃)、高温(41 ℃)和盐胁迫(100mmol/L的NaCl)处理[62,64-66](图2),在激光共聚焦扫描显微镜下观察P-小体形态和数量的变化,发现低温、高温和盐胁迫处理后,转基因烟草活体细胞中P-小体的数量和体积都有所增加,胁迫解除后,P-小体较胁迫条件下数量和体积都明显下降(图3). 说明P-小体的数量和体积受逆境诱导变化,并且这种变化具有可逆性.
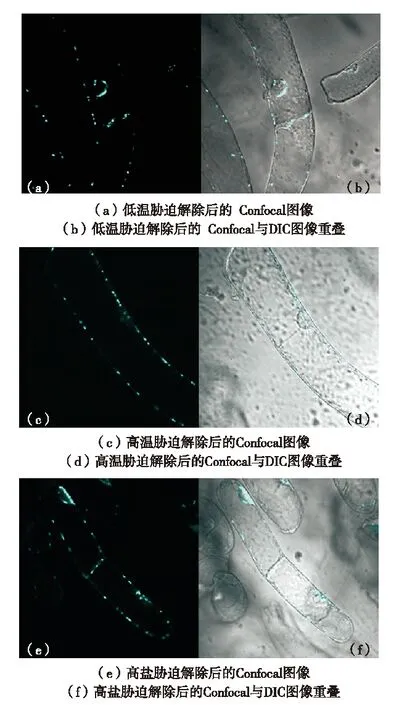
(Olympus FV1000型激光共聚焦显微镜观察, 青色标记为CFP融合蛋白发光情况,bar=10 μm)图3 胁迫解除后P-小体解聚恢复正常水平[67]Fig.3 P-bodies back to normal after stress relieving[67]
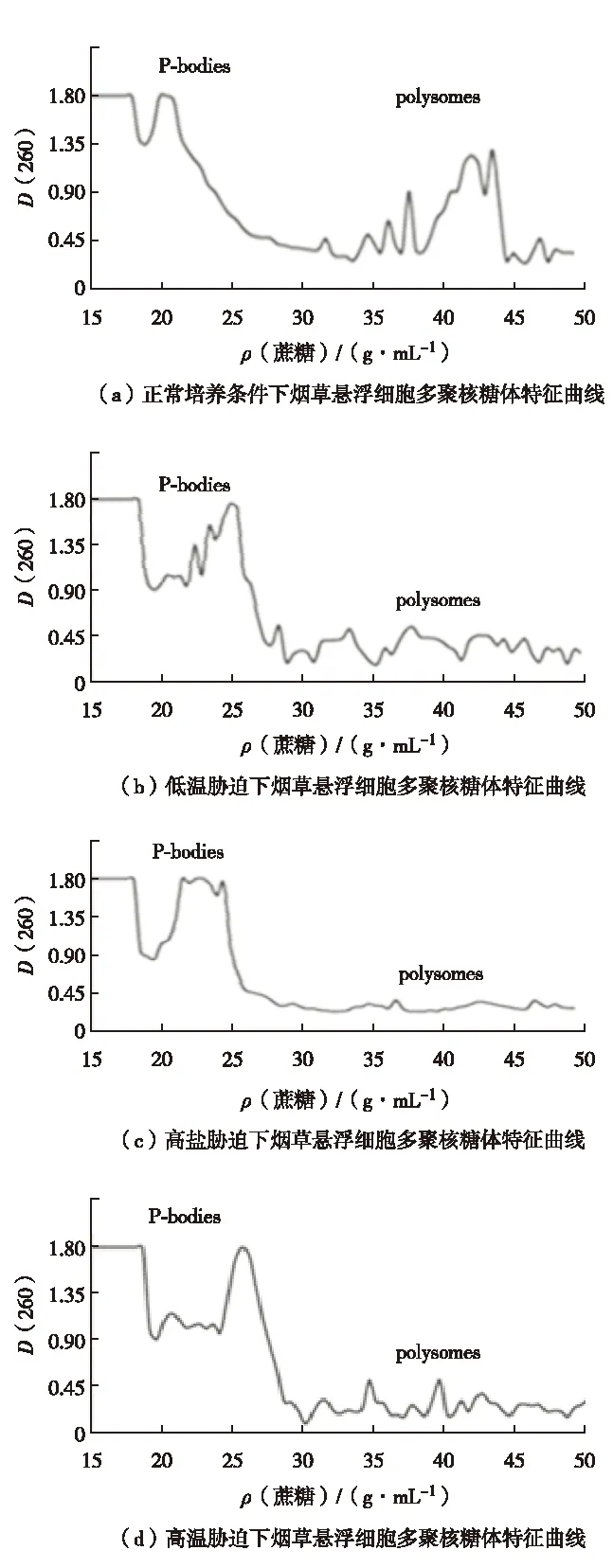
图4 胁迫条件下多聚核糖体解体[67] Fig.4 Polysomes disintegration under stress[67]
本实验室进一步的研究发现,高温和盐胁迫下多聚核糖体的特征峰明显降低(图4)[68],说明细胞内的多聚核糖体在逆境条件下减少. 多聚核糖体是蛋白质翻译的场所,多聚核糖体的解体,是翻译水平受到抑制的体现. 该结果证实了逆境条件下烟草细胞内整体翻译水平受到抑制. 同时,在胁迫条件下P-小体富集区域,即密度为20%~25%的区域,可以看到有明显波峰出现,说明RNA在此富集,逆境下多聚核糖体特征峰的改变与图2中的共聚焦观察结果一致. 说明逆境条件下形成的P-小体为离开多聚核糖体的mRNA提供储存或降解的场所. 胁迫解除后,多聚核糖体恢复正常水平,P-小体特征峰减小甚至消失(图5). 结果进一步验证了P-小体的聚集和多聚核糖体的解体是一种翻译水平的基因表达调控.

图5 胁迫解除后多聚核糖体恢复正常水平[67]Fig.5 Polysomes back to normal after stress relieving[67]
选取β微管蛋白和谷氨酰胺合成酶两种基因,对其mRNA在胁迫处理和解除条件下在细胞内的分布研究发现,在胁迫条件下两种基因的mRNA在多聚核糖体中的量明显下降,在处理小体中的量有所上升;而胁迫解除后又恢复正常水平(图6和图7). 说明胁迫时多聚核糖体解体,RNA离开多聚核糖体后进入处理小体并储存在处理小体中,胁迫解除后回到多核糖体中作为翻译的模板.P-小体通过调控mRNA的亚细胞分布实现转录后水平基因表达的调控.

图6 实时定量PCR检测胁迫和胁迫解除情况下 烟草悬浮细胞两种亚细胞结构中 β微管蛋白mRNA量的变化[67]Fig.6 Changes of β-tubulin mRNA in two sub-cellular structures under stress and stress-relieved situations in tobacco suspension cells detected by real-time PCR[67]
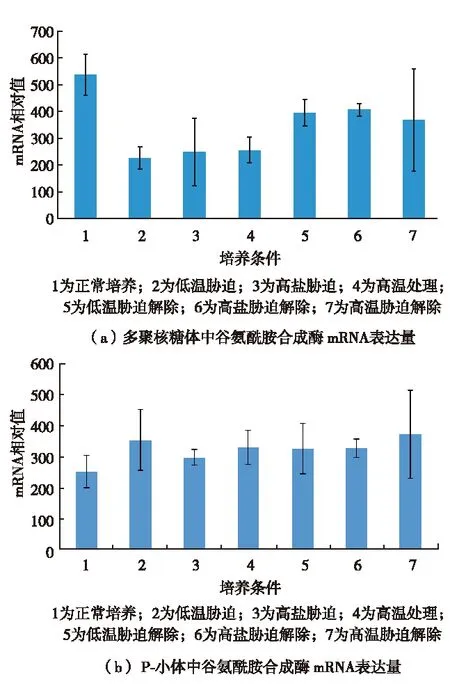
图7 实时定量PCR检测胁迫和胁迫解除情况下 烟草悬浮细胞两种亚细胞结构中 谷氨酰胺合成酶mRNA量的变化[67]Fig.7 Changes in amount of Glutamine synthetase mRNA in two sub-cellular structures under stress and stress-relieved situations in tobacco suspension cells detected by real-time PCR[67]
4 展 望
P-小体的形成能够使细胞应对胁迫环境,P-小体在转录后的基因表达调节过程中发挥作用,对维持细胞内蛋白质水平具有重要意义. 但仍有不少问题有待进一步研究:①P-小体成分是其独有的,还是其他高度有序的细胞结构的一部分?②P-小体中mRNA降解中间体和被抑制的mRNA的空间限制是否是mRNA的翻译调节乃至细胞发挥正常功能所必需的?③P-小体的主要功能在于加强和维持mRNA的抑制状态,还是不可逆转地促使mRNA走向降解?④P-小体的组分在细胞中是如何动态流动的?对这些问题的回答有助于深入了解P-小体在细胞生命活动的作用.
/ References:
[1] Eystathioy T, Chan E K L, Tenenbaum S A, et al. A phosphorylated cytoplasmic autoantigen,GW182,associates with a unique population of human mRNAs within novel cytoplasmic speckles[J]. Molecular Cell, 2002, 13(4):1338-1351.
[2] Sheth U, Parker R. Decapping and decay of messenger RNA occur in cytoplasmic processing bodies[J]. Science, 2003, 300(5620):805-808.
[3] Mo Beixin. RNA localization in plant cells and advances in the research[J]. Journal of Shenzhen University Science and Engineering, 2011, 28(3):229-236.(in Chinese) 莫蓓莘. RNA在植物细胞中的定位及其研究进展[J]. 深圳大学学报理工版,2011, 28(3):229-236.
[4] Liu J, Valencia-Sanchez, Parker R, et al. MicroRNA-dependent localization of targeted mRNAs to mammalian P-bodies[J]. Nature Cell Biology, 2005, 7(7):719-723.
[5] Jain S, Parker R. Ten years of progress in GW/P body research[J]. Advances in Experimental Medicine and Biology, 2013, 768:23-43.
[6] Nissan T, Rajyaguru P, She M, et al. Decapping activators in Saccharomyces cerevisiae act by multiple mechanisms[J]. Molecular Cell, 2010, 39(5):773-783.
[7] Pilkington G R,Parker R.Pat1 contains distinct functional domains that promote P-body assembly and activation of decapping[J]. Molecular and Cellular Biology, 2008, 28(4):1298-1312.
[8] Oishi K, Kurahashi H, Pack C G, et al. A bipolar functionality of Q/N-rich proteins:Lsm4 amyloid causes clearance of yeast prions[J]. Microbiology Open, 2013, 2(3):415-430.
[9] Fromm S A, Truffault V, Kamenz J, et al. The structural basis of Edc3 and Scd6-mediated activation of the Dcp1:Dcp2 mRNA decapping complex[J]. The EMBO Journal, 2012, 31(2):279-290.
[10] Teixeira D, Parker R. Analysis of P-body assembly in Saccharomyces cerevisiae[J]. Molecular Biology of the Cell, 2007, 18(6):2274-2287.
[11] Decker C J, Teixeira D, Parker R. Edc3p and a glutamine/asparagine-rich domain of Lsm4p function in processing body assembly in Saccharomyces cerevisiae[J]. Journal of Cell Biology, 2007, 179(3):437-449.
[12] Reijns M A, Alexander R D, Spiller M P, et al. A role for Q/N-rich aggregation-prone regions in P-body localization[J]. Journal of Cell Science, 2008, 121(15): 2463-2472.
[13] Peremyslov V V, Mockler T C, Filichkin S A, et al. Expression, splicing, and evolution of the myosin gene family in plants[J]. Plant Physiology, 2011, 155(3):1191-1204.
[14] Avisar D, Prokhnevsky A I, Dolja V V. Class VIII myosins are required for plasmodesmatal localization of a closterovirus Hsp70 homolog[J]. Journal of Virology, 2008, 82(6):2836-2843.
[15] Tominaga M, Kojima H, Yokota E, et al. Higher plant myosin XI moves processively on actin with 35 nm steps at high velocity[J]. The EMBO Journal, 2003, 22(6):1263-1272.
[16] Kinkema M, Schiefelbein J A. Myosin from a higher plant has structural similarities to class V myosins[J]. Journal of Molecular Biology, 1994, 239(4):591-597.
[17] Pashkova N, Jin Y, Ramaswamy S, et al. Structural basis for myosin V discrimination between distinct cargoes[J]. The EMBO Journal, 2006, 25(4):693-700.
[18] Sparkes I A. Motoring around the plant cell:insights from plant myosins[J]. Biochemical Society Transactions, 2010, 38(3):833-838.
[19] Sparkes I. Recent advances in understanding plant myosin function: life in the fast lane[J]. Molecular Plant, 2011, 4(5):805-812.
[20] Peremyslov V V, Prokhnevsky A I, Dolja V V, et al. Myosins are required for development, cell expansion, and F-actin organization in Arabidopsis[J]. The Plant Cell, 2010, 22(6): 1883-1897.
[21] Peremyslov V V, Morgun E A, Kurth E G, et al. Identification of myosin XI receptors in Arabidopsis defines a distinct class of transport vesicles[J]. BMC Plant Cell, 2013, 25(8):3022-3038.
[22] Natesan S K, Su llivan J A, Gray J. Myosin XI is required for actin-associated movement of plastid stromules[J]. Molecular Plant, 2009, 2(6):1262-1272.
[23] Sattarzadeh A, Krahmer J, Germain A D, et al. A myosin XI tail domain homologous to the yeast myosin vacuole-binding domain interacts with plastids and stromules in Nicotiana benthamiana[J]. Molecular Plant, 2009, 2(6):1351-1358.
[24] Ueda H, Yokota E, Kutsuna N, et al. Myosin-dependent endoplasmic reticulum motility and F-actin organization in plant cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(15):6894-6899.
[25] Steffens A, Jaegle A, Trech A, et al. Processing body movement inArabidopsisthalianadependsonaninteractionbetweenmyosinsandDCP1[J].PlantPhysiology, 2014, 164(4):1879-1892.
[26]ParkerR,SongH.TheenzymesandcontrolofeukaryoticmRNAturnover[J].NatureStructural&MolecularBiology, 2004, 11(2):121-127.
[27]HouseleyJ,LaCavaJ,TollerveyD.RNA-qualitycontrolbytheexosome[J].NatureReviewsMolecularCellBiology, 2006, 7(7):529-539.
[28]SchaefferD,ClarkA,KlauerAA,etal.Functionsofthecytoplasmicexosome[J].AdvancesinExprimentalMedicineandBiology, 2011, 702:79-90.
[29]AndersonJS,ParkerRP.The3′to5′degradationofyeastmRNAsisageneralmechanismformRNAturnoverthatrequirestheSKI2DEVHboxproteinand3′to5′exonucleasesoftheexosomecomplex[J].TheEMBOJournal, 1998, 17(5):1497-1506.
[30]SteigerM,Carr2SchmidA,SchwartzDC.Analysisofrecombinantyeastdecappingenzyme[J].RNA-APublicationofTheRNASociety, 2003, 9(2):231-238.
[31]TharunS,HeW,MayesAE,etal.YeastSm-likeproteinsfunctioninmRNAdecappinganddecay[J].Nature, 2000, 404(6777):515-518
[32]TharunS,ParkerR.TargetinganmRNAfordecapping:displacementoftranslationfactorsandassociationoftheLsm1p27pcomplexondeadenylatedyeastmRNAs[J].MolecularCell, 2001, 8(5):1075-1083.
[33]CollerJ,ParkerR.GeneraltranslationalrepressionbyactivatorsofmRNAdecapping[J].Cell, 2005, 122(6):875-886.
[34]MaleckiM,ViegasSC,CarneiroT,etal.TheexoribonucleaseDis3L2definesanoveleukaryoticRNAdegradationpathway[J].TheEMBOJournal, 2013, 32(13):1842-1854.
[35]BakheetT,WilliamsBR,KhabarKS.AnupdateofAU-richelementmRNAdatabase[J].NucleicAcidsResearch, 2003, 31(1):421-423.
[36]ShawG,KamenR.AconservedAUsequencefromthe3′untranslatedregionofGM-CSFmRNAmediatesselectivemRNAdegradation[J].Cell, 1986, 46(5):659-667.
[37]CarballoE,LaiWS,BlackshearPJ.Feedbackinhibitionofmacrophagetumornecrosisfactor-alphaproductionbytristetraprolin[J].Science, 1998, 281(5379):1001-1005.
[38]LaiWS,CarballoE,ThornJM,etal.InteractionsofCCCHzincfingerproteinswithmRNA:bindingoftristetraprolin-relatedzincfingerproteinstoAu-richelementsanddestabilizationofmRNA[J].JournalofBiologicalChemistry, 2000, 275(23):17827-17837.
[39]StoecklinG,ColombiM,RaineriI,etal.FunctionalcloningofBRF1,aregulatorofARE-dependentmRNAturnover[J].TheEMBOJournal, 2002, 21(17):4709-4718.
[40]ChenCY,GherziR,OngSE,etal.AUbindingproteinsrecruittheexosometodegradeARE-containingmRNAs[J].Cell, 2001, 107(4):451-464.
[41]LykkeAJ,WagnerE.RecruitmentandactivationofmRNAdecayenzymesbytwoARE-mediateddecayactivationdomainsintheproteinsTTPandBRF-1[J].Genes&Development, 2005, 19(3):351-361.
[42]StoecklinG,MayoT,AndersonP.ARE-mRNAdegradationrequiresthe5′-3′decaypathway[J].TheEMBOJournal, 2006, 7(1):72-77.
[43]KedershaN,StoecklinG,AyodeleM,etal.StressgranulesandprocessingbodiesaredynamicallylinkedsitesofmRNPremodeling[J].JournalofCellBiology, 2005, 169(6):871-884.
[44]StoecklinG,AndersonP.Inatightspot:ARE-mRNAsatprocessingbodies[J].Genes, 2007, 21:627-631.
[45]BhattacharyyaSN,HabermacherR,MartineU,etal.ReliefofmicroRNA-mediatedtranslationalrepressioninhumancellssubjectedtostress[J].Cell, 2006, 125(6):1111-1124.
[46]KedershaNL,GuptaM,LiW,etal.RNA-bindingproteinsTIA-1andTIARlinkthephosphorylationofeIF-2alphatotheassemblyofmammalianstressgranules[J].JournalofCellBiology, 1999, 147(7):1431-1441.
[47]FujimuraK,KanoF,MurataM.IdentificationofPCBP2,afacilitatorofIRES-mediatedtranslation,asanovelconstituentofstressgranulesandprocessingbodies[J].RNA-APublicationoftheRNASociety, 2008, 14(3):425-431.
[48]TeixeiraD,ShethU,Valencia-SanchezMA,etal.ProcessingbodiesrequireRNAforassemblyandcontainnontranslatingmRNAs[J].RNA-APublicationoftheRNASociety, 2005, 11(4):371-382.
[49]CaraTP,SylviaS,TeresaMA,etal.ModulationofhepatitisCvirusRNAabundanceandvirusreleasebydispersionofprocessingbodiesandenrichmentofstressgranules[J].Virology, 2013, 435(2):472-484.
[50]Valencia-SanchezMA,LiuJD,HannonGJ,etal.ControloftranslationandmRNAdegradationbymiRNAsandsiRNAs[J].Genes&Development, 2006, 20(5):515-524.
[51]JakymiwA,LianS,Eystathioy,etal.DisruptionofGWbodiesimpairsmamalianRNAinterference[J].NatureCellBiology, 2005, 7(12):1167-117.
[52]RehwinkelJ,Behm-AnsmantI,GatfieldD,etal.AcrucialroleforGW182andtheDCP1:DCP2decappingcomplexinmiRNA-mediatedgenesilencing[J].RNA-APublicationOfTheRNASociety, 2005, 11(11):1640-1647.
[53]PillaiRS,BhattacharyyaSN,ArtusCG,etal.InhibitionoftranslationalinitiationbyLet-7microRNAinhumancells[J].Science, 2005, 309(5740):1573-1576.
[54]ChuCY,RanaTM.TranslationrepressioninhumancellsbymicroRNA-inducedgenesilencingrequiresRCK/p54[J].PLOSBiology, 2006, 4(7):1122-1136.
[55]BrodersenP,Sakvarelidze-AchardL,Bruun-RasmussenM,etal.WidespreadtranslationalinhibitionbyplantmiRNAsandsiRNAs[J].Science, 2008, 320(5880):1185-1190.
[56]PauleyKM,EystathioyT,JakymiwA,etal.FormationofGWbodiesisaconsequenceofmicroRNAgenesis[J].TheEMBOJournal, 2006, 7(9):904-910.
[57]EulalioA,Behm-AnsmantI,SchweizerD,etal.P-bodyformationisaconsequence,notthecause,ofRNA-mediatedgenesilencing[J].MolecularandCellBiology, 2007, 27(11):3970-3981.
[58]BeckhamCJ,ParkerR.Pbodies,stressgranulesandvirallifecycles[J].CellHost&Microbe, 2008, 3(4):206-212.
[59]MandalMN,VasireddyV,ReddyGB,etal.CTRP5isamembrane-associatedandsecretoryproteinintheRPEandciliarybodyandtheS163RmutationofCTRP5impairsitssecretion[J].InvestigativeOphthalmology&VisualScience, 2006, 47(12):5505-5513.
[60]KulkarniM,OzgurS,StoecklinG.OntrackwithP-bodies[J].BiochemicalSocietyTransactions, 2010, 38:242-251.
[61]KedershaN,AndersonP.Mammalianstressgranulesandprocessingbodies[J].MethodsinEnzymology, 2007, 431: 61-81.
[62]NilssonD,SunnerhagenP.CellularstressinducescytoplasmicRNAgranulesinfissionyeast[J].RNA-APublicationoftheRNASociety, 2011, 17(1):120-133.
[63]YangL,WuG.PoethigRS.MutationsintheGW-repeatproteinSUOrevealadevelopmentalfunctionformicroRNA-mediatedtranslationalrepressioninArabidopsis[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2012, 109(1): 315-320.
[64]NakaminamiK,MatsuiA,ShinozakiK,etal.RNAregulationinplantabioticstressresponses[J].BiochimicaetBiophysicaActa(BBA)-GeneRegulatoryMechanisms, 2012, 1819(2):149-153.
[65]KoukalováB,KovaríkA,FajkusJ,etal.Chromatinfragmentationassociatedwithapoptoticchangesintobaccocellsexposedtocoldstress[J].FEBSLetters, 1997, 414(2):289-292.
[66]MoBeixin,YeHao,OuZhonghua,etal.Stress-inducedprocessingbodyformationintobaccosuspensioncells[J].JournalofShenzhenUniversityScienceandEngineering, 2012, 29(1):80-84.(inChinese) 莫蓓莘,叶 浩,欧忠华,等.逆境诱导烟草悬浮细胞内处理小体的形成[J]. 深圳大学学报理工版, 2012, 29(1):80-84.
[67]YeHao.FunctionstudyofmRNA-store/decayprocessingbodiesinplantresponsetostresses[D].Shenzhen:ShenzhenUniversity, 2013.(inChinese) 叶 浩.储存/降解mRNA的处理小体在植物胁迫应答中的功能研究[D]. 深圳:深圳大学, 2013.
[68]LacsinaJR,LaMonteG,NicchittaCV,etal.PolysomeprofilingofthemalariaparasitePlasmodiumfalciparum[J].MolecularandBiochemicalParasitology, 2011, 179(1):42-46.
【中文责编:晨 兮;英文责编:艾 琳】
Cytoplasmic processing bodies (P-bodies) and the regulation of gene expression
Mo Beixin1, Fu Xiaojie1, Mo Xiaowei1, Liu Li1,2,Xu Xiaofeng3†, and Yue Luming3
1) College of Life Science, Shenzhen University, Shenzhen Key Laboratory of Microbial Genetic Engineering,Shenzhen 518060, P.R.China 2) Department of Botany and Plant Sciences, University of California, Riverside,Institute of Integrative Genome Biology, Riverside, CA 92521, USA 3) College of Life Science, Shenzhen University, Shenzhen Key Laboratory of Marine Biological Resources and Ecological Environment, Shenzhen 518060, P.R.China
mRNA processing bodies (P-bodies) are cytosolic aggregates containing a variety of functional proteins and RNAs. We briefly review the features of P-bodies formation and movement, the interaction of P-bodies with small RNAs and exosomes in the regulation of gene expression. We also summarize the latest P-bodies related research progress in our lab. P-bodies where mRNA degradation and storage take place play an important role in the posttranscriptional regulation of gene expression. They undergo dynamic changes inside the cell. A variety of functional proteins located in P-bodies are involved in the cytoplasmic assembly and biological functioning of P-bodies. The regulatory mechanism of P-bodies still needs to be investigated futher. P-bodies also participate in microRNA(miRNA)-mediated gene silencing.
cell biology; P-bodies; regulation of gene expression; mRNA degradation; translation inhibition; microRNA(miRNA)
:Mo Beixin,Fu Xiaojie,Mo Xiaowei,et al. Cytoplasmic processing bodies(P- bodies)and the regulation of gene expression[J]. Journal of Shenzhen University Science and Engineering, 2015, 32(1): 48-57.(in Chinese)
Q 75
A
10.3724/SP.J.1249.2015.01048
国家自然科学基金资助项目(31210103901)
莫蓓莘(1966—),女(汉族),江西省修水县人,深圳大学教授.E-mail:bmo@szu.edu.cn
Received:2014-09-03;Accepted:2014-11-09
Foundation:National Natural Science Foundation of China(31210103901)
† Corresponding author:Associate professor Xu Xiaofeng. E-mail: xxf@szu.edu.cn
引 文:莫蓓莘,符晓婕,莫小为,等.细胞质处理小体(P-小体)与基因表达的调控[J]. 深圳大学学报理工版,2015,32(1):48-57.
