AtDPBF4基因及其编码蛋白的生物信息学分析
2015-03-02李文新马建忠兰州理工大学兰州730050
文 / 李文新 马建忠(兰州理工大学,兰州730050)
AtDPBF4基因及其编码蛋白的生物信息学分析
文 / 李文新 马建忠(兰州理工大学,兰州730050)
ABI5转录因子参与了植物种子胚胎发育晚期的基因表达调控以及ABA信号的转导。对ABI5亚家族中一个最小的成员AtDPBF4基因序列进行生物信息学分析,推测其编码蛋白质的分子生物学特征,了解AtDPBF4基因编码蛋白质的基本性质和功能,为进一步了解其在ABA信号转导途径中发挥的作用提供参考。
AtDPBF4,生物信息学,分析
1、引言
生物信息学是一门利用计算机技术研究生物系统的规律的新学科,通过综合运用生物学、计算机科学和信息技术,揭示大量而复杂的生物数据包含的生物学奥秘。作为一个新的学科领域,生物信息学把基因组核苷酸序列信息分析作为开始,在获得蛋白质编码区的信息后进行蛋白质的空间结构模拟和预测。
ABI5(AtDPBFs/ABFs)家族是一类亮氨酸拉链型转录因子。这类转录因子参与了植物种子胚胎发育晚期的基因表达调控以及ABA信号的转导。在拟南芥中,ABI5至少有八个成员,他们被分别命名为AtDPBF1/ABI5、AtDPBF2、AtDPBF3/AREB3、AtDPBF4、AtDPBF5/ABF3、ABF1、ABF2、ABF4/ABRE2。
2、分析方法
利用ExPASy中的ProtParam软件分析AtDPBF4基因编码蛋白的分子式、分子质量、氨基酸组成、理论等电点、稳定性等。运用NetPhos 2.0 Server对AtDPBF4基因编码蛋白进行分析,判断是否含有磷酸化位点。蛋白质的二级结构利用在线软件SSPro4.0进行预测。蛋白质的进化单元和功能结构主要存在于结构域中,分析结构功能域对于蛋白质结构的预测和分类有着重要作用。利用SMART分析结构域。使用NCBI的CDD数据库对AtDPBF4的蛋白序列的保守结构进行分析。集中NCBI中AtDPBF4同源性95%以上的相关序列,利用软件MEGA6.05进行多重序列比对绘制系统进化树。
3、结果与分析
3.1 蛋白质性质分析
ProtParam分析AtDPBF4基因编码的氨基酸序列的理化性质,结果显示,该序列的总分子量为39617.6u,氨基酸数为262,理论等电点为8.89。总负电荷残基(Asp+Glu)数目为35,总正电荷残基(Arg+Lys)数目为38。分子式为C1279H2108N384O404S9,不稳定指数为56.24,属于不稳定蛋白,脂肪指数为79.92。
Portscale对AtDPBF4基因编码的氨基酸序列进行疏水性分析,结果如下图。结果表明:145位谷氨酰胺疏水性最弱,疏水指数为-2.978,117位丙氨酸疏水性最强疏水指数为1.778。

横坐标:氨基酸序列号 纵坐标:+:疏水性;-:亲水性。
3.2 蛋白质磷酸化位点分析
NetPhos 2.0 Server软件分析结果显示,AtDPBF4基因编码的蛋白可能含有11个磷酸化位点,其中包括8个丝氨酸磷酸化位点(S13,S21,S24,S37,S74,S170,S203,S223)和3苏氨酸磷酸化位点(T32,T77,T99)。
3.3 蛋白质结构预测
3.3.1 二级结构预测分析
SSPro 4.0软件预测结果显示AtDPBF4所编码蛋白,有14个α螺旋、6个β折叠、1个β转角和10个无规卷曲。
3.3.2 结构域的预测
SMART软件预测结果显示在188到260位氨基酸之间的组成结构域是基本区域的亮氨酸拉链(basic region leucin zipper,BRLZ)结构域,含有一个锌指结构。
3.3.3 保守结构域分析

利用NCBI的CDD数据库对AtDPBF4氨基酸序列保守结构域进行分析,结果如图,该序列在192-237之间是一个bZIP结构域。长度55,E-value∶3.54e-22 。
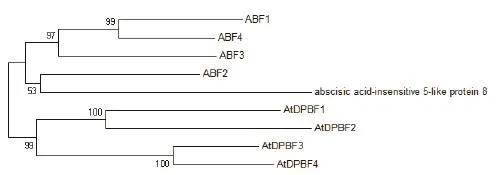
3.4 系统发育分析
软件MEGA6.05对NCBI数据库中AtDPBF4同源性95%以上的基因序列进行多重序列比对并利用近邻结合法绘制基因进化树。
4、结论
基因通过其编码的蛋白质发挥作用,本论文通过生物信息学分析AtDPBF4编码的蛋白质属于ABI5家族,包含一个BRLZ结构域。AtDPBF4编码蛋白存在多个磷酸化位点,介导蛋白与蛋白间的相互作用,在细胞内信号转导过程中发挥重要作用。对AtDPBF4核苷酸序列及其编码蛋白质的分析为进一步研究其在ABA信号转导途径发挥的作用提供了参考。
[1]杨华,杨风琴,廖国玲,刘学英。SH2D7基因及其编码蛋白的生物信息学分析。科技导报,2015,33(2)
[2] Bensmihen S, Rippa S, Lambert G,Jublotb D,Pautotb V,Granierc F,Giraudat J,Parcy F,(2002)The Homologous ABI5 and EEL Transcription Factors Function Antagonistically to Fine-Tune Gene Expression during Late Embryogenesis. The PlantCell,14(10):1391-1403.
[3] Bensmihen S,To A, Lambert G,Kroj T,Giraudat J,Parcy F,(2004) Analysis of an Activated ABI5 Allele Using a New Selection Method for Transgenic Arabidopsis Seeds. FEBS Lett.561(1-3):127-131.
[4] Bensmihen S, Giraudat J,Parcy F,(2005) Characterization of Three Homologous Basic Leucine Zipper Transcription Factors (bZIP) of the ABI5 Family during Arabidopsis thaliana Embryo Maturation. Joumal of Experimental botany 56(412):597-603.
[5] Finkelstein RR, Lynch TJ,(2000) The Arabidopsis Abscisic Acid Response Gene ABI5 Encodes a Basic Leucine Zipper Transcription Factor. The Plant Cell 12 (4) :599-610.
