中华野海棠遗传多样性分析
2015-02-27洪震傅冰傅金尧陈盛专季国华
洪震,傅冰,傅金尧,陈盛专,季国华
(1. 丽水市林业科学研究院,浙江 丽水 323000;2.丽水职业技术学院,浙江 丽水 323000; 3.浙江省平阳林场,浙江 平阳 325406;4.遂昌九龙山自然保护区,浙江 遂昌 323312)
中华野海棠遗传多样性分析
洪震1,傅冰2,傅金尧2,陈盛专3,季国华4
(1. 丽水市林业科学研究院,浙江 丽水 323000;2.丽水职业技术学院,浙江 丽水 323000; 3.浙江省平阳林场,浙江 平阳 325406;4.遂昌九龙山自然保护区,浙江 遂昌 323312)
利用SRAP-PCR方法研究了8个不同种源中华野海棠的遗传多样性。结果表明:8个中华野海棠种质资源被划分为2大类群,其中遂昌、景宁、庆元、泰顺、武夷山种源聚成1个类群;平阳、松阳、云和种源聚成另外个类群;遗传相似性系数界于0.4230~0.9615,平均相似性系数为0.71。
SRAP-PCR;中华野海棠;遗传多样性
中华野海棠(Brediasinensis),为野牡丹科野海棠属常绿灌木,花瓣粉红色至紫红色,优美艳丽,叶形美观,叶色翠绿有光泽,叶与花均有很高的观赏价值[1]。同时,该种具有较高的药用价值,全株供药用,在浙江民间常用其治小儿腹泻、感冒、头痛以及疟疾等病症[2]。中华野海棠主要分布于两广、江西、浙江、福建等省,浙江省则分布于丽水、温州等浙南山区,通常在海拔400~1400m的山谷、山坡、林下等地有生长。
目前,对于中华野海棠的研究较少,主要集中在移栽培养[3]、抗逆性研究、化学成分分析[4]、生物学特性[5]研究等方面,而对于遗传育种、分类鉴定等方面,现有报道均未涉及。利用SRAP技术分析不同种源的中华野海棠的遗传多样性,可为分类鉴定以及良种选育等工作提供一定的理论依据。
1 材料与方法
1.1 材料

表1 供试材料
1.1.1 中华野海棠种植资源。供试8种中华野海棠种质资源(表1)采集于浙江及福建各地。采集鲜嫩叶片后,-70℃超低温冰箱保存。
1.1.2 SRAP引物及筛选。共选择了6个正向引物,8个反向引物用于实验筛选,具体如表2,引物由北京全式金生物技术有限公司按照相关序列合成。

表2 引物序列
1.2 方法
1.2.1 基因组DNA的提取。用全氏金公司PlanZol植物基因组提取试剂盒抽提中华野海棠叶片组织的基因组DNA。利用琼脂糖凝胶电泳检测所提取基因组DNA的完整性,并用Lambda 650紫外分光光度计(PE,美国)测定浓度及质量。
1.2.2 SRAP-PCR反应体系及程序。参考前期对蓝莓SRAP-PCR反应体系及参数的研究[6],初步确定反应体系及反应程序。初步确定反应体系为:dNTPs 100 μmol/ L、Taq 酶0.8U、MgCl22.5 mmol/ L、DNA 40 ng、正、反向引物各0.2 μmol/ L、1×Reaction Buffer(总体积20 μL)。PCR反应程序为: 95 ℃预变性5 min;95 ℃变性45 s,35 ℃退火60 s,72 ℃延伸60 s,5个循环;95 ℃变性45 s,50 ℃退火60 s,72 ℃延伸90 s,30个循环; 72 ℃延伸10 min;4 ℃保存。
1.2.3 SARP引物筛选。以松阳种源中华野海棠基因组DNA为模板,随机组合正、反向引物,进行引物筛选。
1.2.4 电泳及染色。将筛选得到的引物分别以所有供试中华野海棠基因组DNA为模板进行PCR反应,并进行非变性聚丙烯酰胺凝胶电泳,用Dolphin-DOC Plus凝胶成像系统(Wealtec,美国)分析、记录数据。DNA标准分子量为MarkerIII(北京全式金生物技术有限公司)。
1.2.5 数据处理和统计方法。用Dolphin-DOC Plus凝胶成像系统自带软件结合人工方法读带,并记录扩增结果,其中有带记为1,无带记为0,并导入Excel表格。用NTSYS-pc2.10e分析软件分析所得Excel表格,计算遗传相似性系数,同时用UPGMA对不同种源进行聚类分析,绘制聚类图。
2 结果与分析
2.1 引物筛选及多态性分析
以松阳种源中华野海棠基因组DNA为模板,验证PCR反应体系及反应程序,同时筛选合适的SRAP引物组合。共随机组合了20对引物组合,电泳结果如图1所示,从中筛选出4对能够扩增出清晰的条带,并且多态性好、稳定性高的引物组合,分别为:ME4-EM17、ME4-EM18、ME5-EM13、ME6-EM10。筛选结果同样证明该PCR体系是适用的。4对组合PCR后经3.5%丙烯酰胺凝胶电泳,共扩增出99条条带,其中多态性条带为60条,多态性比率为60.61%(表3)。
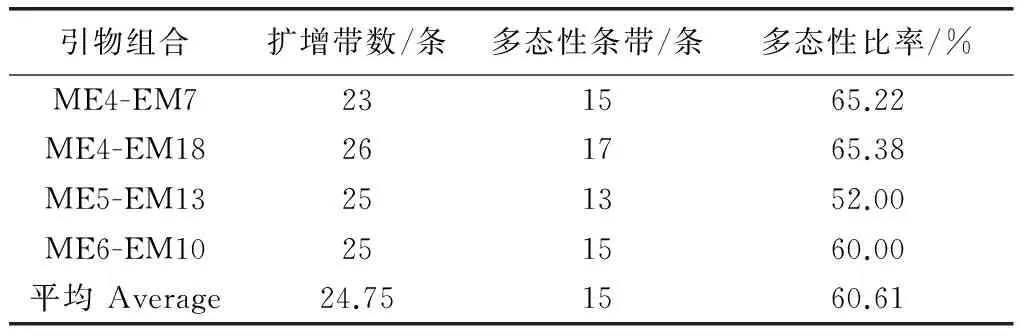
表3 9个引物组合的扩增结果
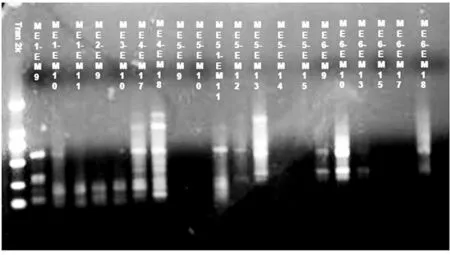
图1 引物筛选电泳图
2.2 种质资源相似性系数分析
根据4对组合PCR电泳结果(图2为ME6-EM10引物组合电泳结果),利用NTSYS-pc2.10e分析软件计算样品间的Jaccard遗传相似性系数,结果见表4。8个中华野海棠种质资源的遗传相似性系数分布于0.4230~0.9615之间,平均相似性系数为0.71。其中,最高相似性系数出现在遂昌和景宁种源之间,为0.9615;最低相似性系数出现在福建和云和种源之间,为0.4230。

表4 8个中华野海棠种质资源的Jaccard相似性系数矩阵
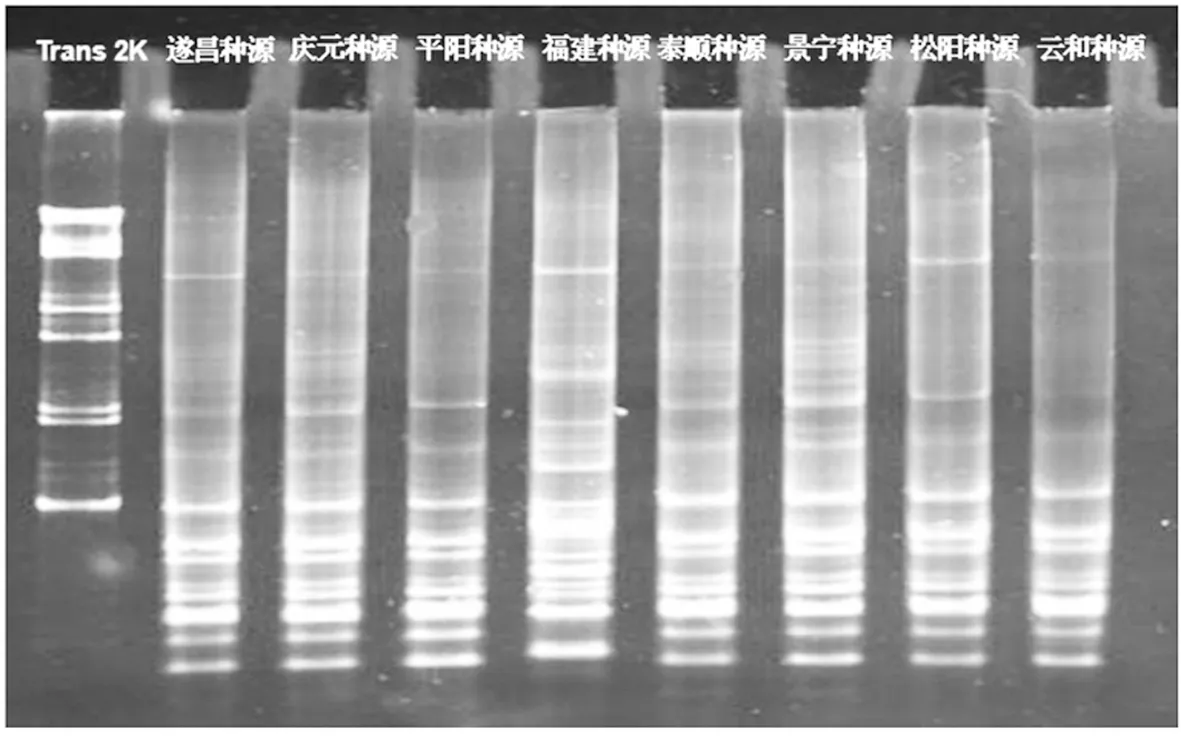
图2 ME6-EM10 3.5%丙烯酰胺凝胶电泳图
2.3 种质资源的聚类分析
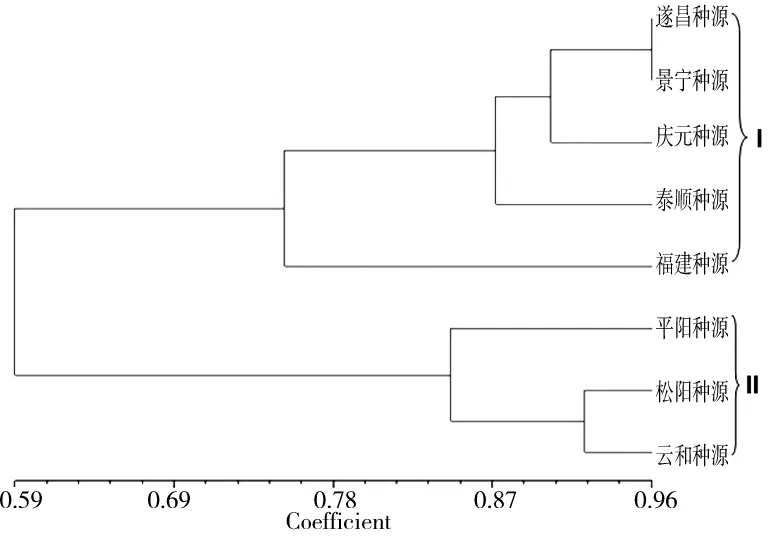
图3 8个中华野海棠种质的UPGMA聚类图
基于Jaccard遗传相似系数的UPGMA聚类图(图3)显示,8个中华野海棠种质资源被划分为两大类群:其中遂昌、景宁、庆元、泰顺、福建种源聚成一个类群(I);平阳、松阳、云和种源聚成另一个类群(II)。类群I又可以细分成2个小的类群,其中,福建种源自成一类类群,其它4个种源为一个类群。类群II同样也可细分成2个小的类群,分别为松阳、云和种源为一个类群;平阳种源为另一个类群。
3 讨论
通过对试验结果的分析,8种供试种质资源遗传相关性并不是很高,说明中华野海棠具有较丰富的遗传多样性。虽然除了福建武夷山种源的中华野海棠外,其它7个种质资源均采集自浙江省丽水市及温州市,但是这7个种质资源之间并没有因为分布较近而表现出高的亲缘关系,这可能跟种质资源的人工移栽、自然迁移[7]等因素有关。
[1]洪震,李根有,马丹丹,等.野生花卉中华野海棠的抗逆性研究[J].北方园艺,2012(5):62-66.
[2]屠娟丽,周紫球,吴小华.3野生灌木中华野海棠的发芽试验[J].浙江林业科技,2003 (2):24-26.
[3]张作焕,李林,陶正明.中华野海棠野生苗移栽技术初探[J].浙江林业科技,2003(1):54-55.
[4]沈双玲.中华野海棠化学成分及药效学分析[D].福州:福建农林大学,2013.
[5]马丹丹,金清.3种野海棠属野生花卉的光合特性研究[J].河南林业科技,2011(3):1-3.
[6]傅冰,洪震,刘跃钧,等.蓝莓SRAP-PCR反应体系的建立和优化及其遗传多样性分析[J].北方园艺,2013(17):95-99.
[7]钟永德,李迈和.Norbert Kraeuchi,地球暖化促进植物迁移与入侵[J].地理研究,2004(3):347-353.
Analysis the Genetic Diversity ofBrediasinensis
Hong Zhen1,Fu Bing2,Fu Jinyao2,Chen Shengzhuan3,Ji Guohua4
(1. Lishui Academy of Forestry, Lishui ,Zhejiang 323000;2.Lishui vocational & technical college, Lishui , Zhejiang,323000; 3.Pingyang Forest Farm of Zhejiang Province,Pingyang,Zhejiang 325406;4. Jiulongshan Nature Reserve of Suichang,Suichang, Zhejiang 323312)
SRAP-PCR method was used to study the genetic diversity of 8Brediasinensisprovenances.The results indicated that: the 8Brediasinensisprovenances were divided into two groups,which provenances of Suichang County, Jingning County, Qingyuan County, Taishun County,Wuyishan City together into one group; and the other group include provenances of Pingyang County, Songyang County, Yunhe County.The GS coefficients among the 8Brediasinensisprovenances were ranged from 0.4230 to 0.9615 with an average of 0.71.
SRAP-PCR;Brediasinensis;Genetic diversity
2015-08-25
浙江省科技计划项目(2013C32102);丽水市科技计划项目(20120310)
洪震(1969-),男,高级工程师,主要从事观赏植物开发利用研究,E-mail:452451651@qq.com。
S661.4
B
DOI.:10.13268/j.cnki.fbsic.2015.06.001
