地中海贻贝贝壳肌棱柱层蛋白质组学分析
2015-02-22王新星鲍林飞范美华
王新星,高 鹏,鲍林飞,范美华,廖 智
(浙江海洋学院海洋科学与技术学院,海洋生物资源及分子工程实验室,浙江舟山 316022)
地中海贻贝贝壳肌棱柱层蛋白质组学分析
王新星,高 鹏,鲍林飞,范美华,廖 智
(浙江海洋学院海洋科学与技术学院,海洋生物资源及分子工程实验室,浙江舟山 316022)
贻贝贝壳与后闭壳肌形成紧密连接并由此介导了贝壳的闭合。为了解贻贝后闭壳肌-贝壳之间连接界面以及蛋白质分子组成,利用扫描电子显微镜、傅里叶红外光谱对地中海贻贝贝壳的微观结构、碳酸钙晶体构型进行分析;同时,采用酸抽提法获取地中海贻贝后闭壳肌痕部位肌棱柱层的贝壳基质蛋白,对酸可溶性蛋白和酸不溶性蛋白分别采用液质联用技术结合转录组数据库搜索,共鉴定61种贝壳基质蛋白。上述研究为深入了解贝壳的生物矿化机制以及肌肉-贝壳界面的无机相-有机相连接机理奠定了基础。
地中海贻贝;肌棱柱层;扫描电子显微镜;傅里叶红外光谱;蛋白质组学
Key words:Mytilus galloprovincialis;myostracum;scanning electronic microscopic;fourier transform infrared spectroscopy;proteomics
贝类贝壳是由碳酸晶体在贝壳基质蛋白(shell matrix protein,SMP)指导下形成的有序的纳米结构,具有极佳的力学性能,因而在生物材料,生物医学工程等方面具有较好的仿生学意义[1-3]。贻贝(Mytilus)贝壳微观结构属于典型的“珍珠质-棱柱模型”(nacro-prismatic model)[4],主要包括两种不同晶体构型的碳酸钙晶体结构,分别为位于贝壳外侧的纤维棱柱层(fibrous prism)和位于贝壳内侧的珍珠质层(nacre)。其中,纤维棱柱层赋予贝壳应力分散(crack propagation)和抗冲压性能(puncture-resistance),而内侧的珍珠质层则赋予了贝壳抗断裂性能(fracture-resistance)[5-7]。肌棱柱层(myostracum)是一种特殊的贝壳微观结构,在不同的双壳贝类贝壳中均有发现[8,9]。肌棱柱层在贝壳的微观结构中所占比例较小,然而,肌棱柱层在贻贝贝壳的后闭壳肌痕(Adductor Muscle Scar,AMS)部位直接与后闭壳肌产生紧密连接,因而在控制贝壳的闭合方面具有重要的意义[10]。GALTSOFF曾报道,太平洋牡蛎Crassostrea virginica中由后闭壳肌介导的贝壳闭合力超过10 kg,且在强行拉开时,断裂部位通常位于后闭壳肌中间,而非后闭壳肌与贝壳的连接部位[11]。这表明后闭壳肌-贝壳的连接界面极为牢固,为人造骨骼的移植以及人工肌腱的研发提供了很好的参考价值。
贝壳的形成及其力学性能与其所含的贝壳基质蛋白密切相关。贝壳基质蛋白指导或参与了贝壳碳酸钙晶体的成核,晶体生长方向控制,晶体构型选择等过程[12],同时,部分贝壳基质蛋白,如丝状蛋白等,与贝壳内其他有机质,如几丁质等,参与了贝壳内有机制框架的形成,并赋予了贝壳优异的机械性能[13]。目前对于贝壳基质蛋白的研究主要集中于珍珠质层或者纤维棱柱层[14],而对于肌棱柱层的贝壳基质蛋白研究尚未见报道。为深入了解贝壳肌棱柱层的微观结构以及蛋白质分子组成,我们以地中海贻贝Mytilus galloprovincialis为研究对象,采用扫描电子显微镜以及傅里叶红外光谱等手段分析其后闭壳肌痕部位的微观结构,同时,采取蛋白质组学结合转录组数据库搜索策略,分析了后闭壳肌痕部位肌棱柱层的贝壳基质蛋白组成。上述研究为深入研究肌棱柱层的生物矿化机制以及肌棱柱层在后闭壳肌-贝壳连接界面中的作用奠定了基础。
1 材料与方法
1.1 贝壳的采集和样品处理
成年地中海贻贝(体长3~4 cm)采自浙江省舟山市东极岛海域。去除体内软组织后,将贝壳浸没于5%氢氧化钠溶液30 min以去除贝壳表面附着的各种杂质,之后以纯水清洗并晾干备用。
1.2 扫面电子显微镜观察
将贝壳沿纵向于后闭壳肌痕部位断裂,断裂后的贝壳样品以去离子水清洗并进行超声处理以去除横断面表面的杂质。断面经喷金处理后,置于扫描电子显微镜(TESCAN,VEGA3型,捷克)进行观察,扫描电压为20 kV。
1.3 傅里叶红光谱分析
肌棱柱层粉末状样品以不锈钢手术刀从后闭壳肌痕部位表层刮取。所刮取的粉末样品于玛瑙研钵中进一步研磨至粒径200目。粉末样品和磨细的KBr粉末按质量比约1∶100在玛瑙研钵中研磨混合均匀,采用透射模式测量;分析仪器为美国Nicolet Nexus 670 FTIR分光光度计,扫描范围400~4 000 cm-1,扫描次数32,分辨率为4 cm-1。红外谱图采用仪器附带的Omnic软件(版本8.2.387)进行分析处理。
1.4 肌棱柱层贝壳基质蛋白的提取
取自肌棱柱层的贝壳粉末样品溶于5%醋酸,充分溶解1 h后,离心(12,000×g,4℃)20 min,上清液(酸可溶性贝壳基质蛋白)经透析处理(截留分子量1 kDa)后冷冻干燥,于-20℃储存备用;沉淀(酸不溶性贝壳基质蛋白)以去离子水洗涤6次后冷冻干燥后于-20℃储存备用。
1.5 蛋白质酶解及质谱分析
提取的贝壳基质蛋白样品溶于8 M尿素溶液,超声处理30 min,加入事先溶解于50 mM碳酸氢铵溶液的二硫苏糖醇(dithiothreitol,DTT),终浓度为10 mM,于57°C条件下还原1 h;之后再加入事先溶解于50 mM碳酸氢铵溶液的碘乙酰胺溶液(iodoacetamide,IAA)至终浓度20 mM;室温,黑暗条件下进行烷基化反应45 min。还原烷基化反应后,将样品溶液稀释10倍,按1:50比例加入胰蛋白酶(Proteomics grade,Sigma),37°C条件下孵育18 h。酶解完成后,样品溶液经离心(12,000×g,4℃)20 min,上清液冷冻干燥后复溶于30 L 4%乙腈溶液(含0.1%三氟乙酸)准备进行质谱分析。
质谱分析在四级杆飞行时间质谱仪(QSTAR-Elite,Applied Biosystems,USA)上进行。酶解后的样品溶液首先上样质谱仪配备的高效液相色谱系统(20AD HPLC system,Shimadzu,日本);分离柱为Zorbax 300SB-C18反相柱(0.1×150 mm,5 μ,300 Å;Microm,Auburn,CA);洗脱液分别为含0.1%甲酸的5%乙腈溶液(buffer A)和95%乙腈(buffer B);洗脱梯度为90 min内buffer B的比例由5%上升到35%,流速为0.3mL/min。质谱数据的采集由软件Analyst QS 1.1(Applied Biosystems,美国)自动完成;一级质谱扫描范围m/z 350~1 500;分辨率分别为60 000(全扫描))和2 000(MS/MS扫描);强度最大的8个峰(m/z 100~2 000)经动态排除法收集后进行序列分析。
1.6 蛋白质鉴定与序列分析
蛋白质鉴定采用MSCOT软件(版本2.1,Matrix Science,London,英国)进行。蛋白质数据库采用贻贝EST序列衍生数据库,包括来自加利福尼亚贻贝M.californianus的EST 42 354条、地中海贻贝M.galloprovincialis的EST 19 617条、紫贻贝M.edulis的EST 5 300条、以及厚壳贻贝M.coruscus的EST 719条等,共计68 209条EST序列(下载自美国国家医学图书馆数据库)。上述EST数据按照六种可能的翻译框翻译成蛋白质氨基酸序列,经MSCOT软件转化格式后构成自定义本地数据库,二级质谱数据用以检索该蛋白质序列数据库;质谱质量容差(mass tolerance)分别设定为20 ppm(一级质谱)和0.3 Da(二级质谱);固定修饰设定为乙酰化,可变修饰设定为氧化;蛋白质得分在100分以上且最佳离子得分(ion score)为39分以上为可信鉴定。
鉴定后的所匹配的EST序列采用lasergene软件分析其开放阅读框;同原序列搜索在http://www.ncbi. nlm.nih.gov/BLAST/在线进行;结构域预测采用SMART软件在线http://smart.embl-heidelberg.de/进行;氨基酸组成分析采用ProtParam软件在线http://web.expasy.org/protparam/进行。
2 结果
2.1 地中海贻贝后闭壳肌痕部位的断面及表面微观结构
由图1a可见,地中海贻贝贝壳内侧后闭壳肌紧密粘附于贝壳后背端靠近生长边缘处,经5%氢氧化钠溶液处理后,该肌肉组织可被去除,原所在部位为近椭圆形的深色区域,即后闭壳肌痕(图1b)。将贝壳沿图1b白线所示方向断开后,后闭壳肌痕部位的断面结构经扫描电子显微镜观察,可发现有3种不同形貌的层次结构,从上至下分别肌棱柱层(Myostracum)、珍珠质层(Nacre)和纤维棱柱层(Fibrous prism)。与肌肉组织直接连接的为肌棱柱层。此外,后闭壳肌痕的表面覆盖一层光滑的膜样组织且分布有少量纳米级的小孔(图1d)。
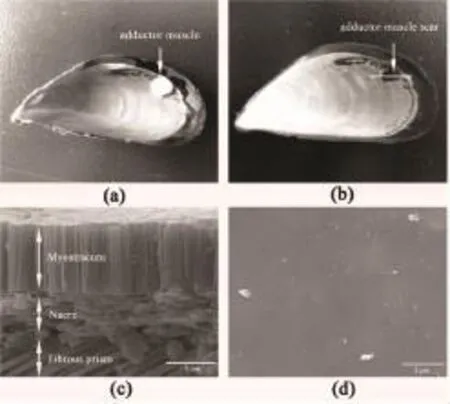
图1 地中海贻贝贝壳后闭壳肌痕(AMS)断层的扫描电子显微镜图谱(a)地中海贻贝贝壳内表面,箭头示后闭壳肌(adductor muscle);(b)地中海贻贝贝壳内表面经5%氢氧化钠溶液处理后,后闭壳肌去除,箭头所示为后闭壳肌痕;白色线代表贝壳断裂位置;(c)地中海贻贝后闭壳肌痕部位断面结构,展示三种不同的层次结构,分别为肌棱柱层(myostracum)、珍珠质层(nacre)和纤维棱柱层(fibrous prism);(d)后闭壳肌痕部位的表面结构,显示该部位表面覆盖有一层膜样组织且分布少量纳米级小孔.Fig.1 The photograph of interior surface and SEM images of cross-section of adductor muscle scar in M.galloprovincialis shell (a)the photograph of inner surface of an adult M.galloprovincialis.The adductor muscle was denoted by an arrow;(b)the photograph of inner surface of M.galloprovincialis shell after the muscle was removed by 5%NaOH;(c)the SEM image of section of the adductor muscle scar.The myostracum,nacre and fibrous prism were shown;(d)the SEM image of surface of adductor muscle scar.The membrane-like material with micro holes can be observed.
2.2 肌棱柱层傅里叶红外光谱分析
地中海贻贝贝壳肌棱柱层的傅里叶红外光谱展示了1个位于酰胺一带的特征峰(1 784.80 cm-1)和4个碳酸钙晶体特征峰(图2),分别为CO32-的反对称伸缩振动峰(1 437.60 cm-1,υ3)、CO32-的对称伸缩振动峰(1 082 cm-1,υ1)、CO32-的面外弯曲振动峰(854.60 cm-1,υ2)和CO32-的面内弯曲振动峰(712.09 cm-1和699.13 cm-1,υ4)。根据DAUPHIN等[15]的描述,上述4个碳酸钙晶体特征峰属于文石型碳酸钙晶体的特征峰,表明地中海贻贝贝壳肌棱柱层的碳酸钙晶体构型为文石型;而酰胺一带出现的特征峰表明在肌棱柱层内含有少量有机质。

图2 地中海贻贝贝壳肌棱柱层粉末样品的FTIR光谱Fig.2 FTIR spectra of myostracum layer from M.galloprovincialis shell
2.3 肌棱柱层贝壳基质蛋白的鉴定
利用LC-MS/MS质谱技术结合贻贝EST数据库搜索,从地中海贻贝贝壳肌棱柱层可溶性蛋白样品中共获得12个高可信度的鉴定结果(表1);从不可溶性蛋白样品中共计获得49个高可信度的鉴定结果(表2)。对所匹配的61条EST序列进行开放阅读框分析并进行序列同源性搜索、结构域预测以及氨基酸组成分析。代表性二级质谱图见图3,其匹配的EST为gi|145900307,该EST编码的蛋白与来自牡蛎 C.gigas的贝壳基质蛋白 Whirlin(gb| EKC33408.1)的序列一致性为61%,匹配的肽段数为四条,蛋白质得分208分(表1)。
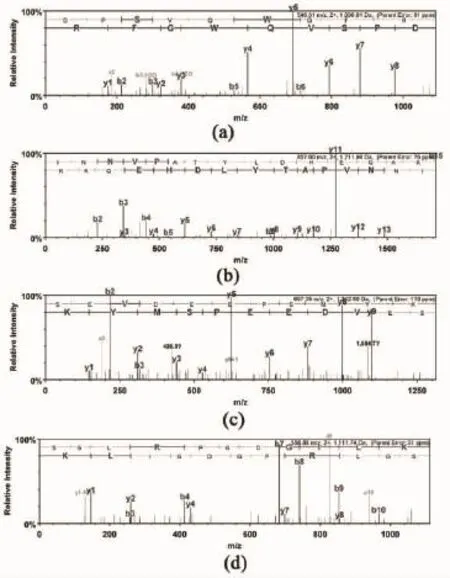
图3 匹配结果为gi|145900307的代表性二级质谱图(a)质荷比为1090.61Da的蛋白质片段序列-DPSVQWGFR-;(b)质荷比为1711.98Da的蛋白质片段序列-INNVPATYLDHEQAK-;(c)质荷比为1312.69Da的蛋白质片段序列-SEVDEEPSMYK-;(d)质荷比为1111.74Da的蛋白质片段序列-SGLRPGDGILK-.Fig.3 Representative MS/MS spectra for four peptides matching with gi|145900307 (a)MS/MS spectrum of the“-DPSVQWGFR-”with m/z of 1090.61Da; (b)MS/MS spectrum of the“-INNVPATYLDHEQAK-”with m/z of 1711.98 Da;(c)MS/MS spectrum of the“-SEVDEEPSMYK-”with m/z of 1312.69 Da;(d)MS/MS spectrum of the“-SGLRPGDGILK-”with m/z of 1111.74 Da.
不溶性蛋白样品中所鉴定的蛋白质种类远多于可溶性蛋白样品,表明多数贝壳基质蛋白具有较强的疏水性。在上述已鉴定的蛋白中,有6种蛋白与已报道的来自其他贝类的贝壳基质蛋白同源,如MUSP-3(EST gi|212814580)、NSPI-like protein(EST gi|238643545)、Shellmatrix protein (EST gi| 145887968)、Perlucin -like protein (EST gi| 58306883)、Nacrein-like protein (gi|223025603)和Whirlin(EST gi|145900307)。其余为此前未曾报道的新型贝壳基质蛋白。其中,Calponin蛋白家族的种类最多,共鉴定到14种,其次是胶原类(Collagenlike)蛋白、含Calponin同源结构域的Transgelin类蛋白、Filamin蛋白、酶类(包括精氨酸激酶和醛缩酶)、以及具有特殊氨基酸组成的蛋白(如富含丙氨酸、甘氨酸和亮氨酸的蛋白)等。
3 讨论
贝壳基质蛋白研究不仅有助于了解贝壳的形成机制,同时也有助于从中开发具有生物医药应用价值的新型蛋白药物,如骨生长促进因子等。目前数据库中已报到的贝壳基质蛋白种类已经超过100种,主要来自贝壳的珍珠质层或者棱柱层,物种来源主要为鲍鱼(Haliotis)、牡蛎(Crassostrea)、以及珠母贝(Pinctada)。贻贝的贝壳基质蛋白目前研究尚不多见,已报到的贻贝贝壳基质蛋白目前仅十余种[16],肌棱柱层特异的贝壳基质蛋白目前尚未见报道。地中海贻贝贝壳肌棱柱层在后闭壳肌痕部位直接与后闭壳肌形成牢固连接。扫描电镜观察结果表明,肌棱柱层在形貌上与珍珠质层和纤维棱柱层有较大的区别,主要表现为垂
直柱状结构,这一结构特点有利于分散来自闭壳肌的拉力,从而使得肌棱柱层与后闭壳肌之间连接较为紧密;傅里叶红外光谱分析表明,地中海贻贝贝壳肌棱柱层主要采取了文石型的碳酸钙晶型。该结果类似于牡蛎的肌棱柱层[8],表明肌棱柱层的碳酸钙晶体构型在不同贝类贝壳中基本一致。

表1 地中海贻贝贝壳肌棱柱层可溶性蛋白的鉴定结果Tab.1 Protein identification of acid-soluble matrix of mytostracum from M.galloprovincialis shell
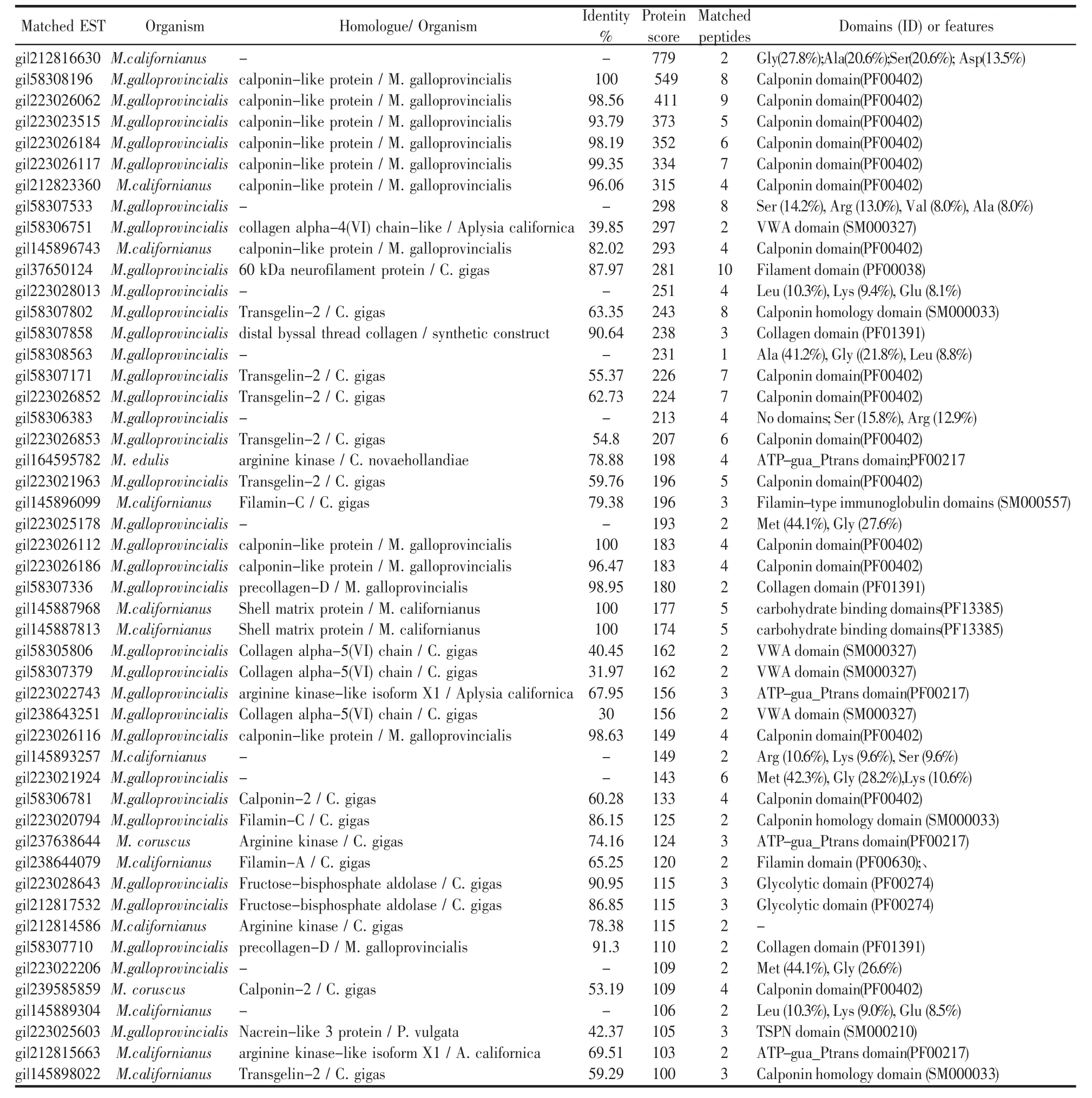
表2 地中海贻贝贝壳肌棱柱层不可溶性蛋白的鉴定结果Tab.2 Protein identification of acid-insoluble matrix of mytostracum from M.galloprovincialis shell
贝壳基质蛋白通常具有较强的疏水性且在贝壳中形成交联结构,因而常规的蛋白质分离纯化和鉴定策略在贝壳基质蛋白研究中遭遇不少困难。目前,利用质谱的高通量、高灵敏度、高精确度特点,结合贻贝转录组数据进行贝壳基质蛋白的鉴定是当前研究生物矿化相关分子组成的主流方法[17-19]。由于肌棱柱层不仅是贝壳的一种组成结构,同时还参与了与后闭壳肌的紧密连接,因而在所鉴定的贝壳基质蛋白中,不仅存在参与生物矿化的贝壳基质蛋白,还存在可能参与肌肉-碳酸钙连接的蛋白。在本次研究中,我们鉴定到6种已知贝壳基质蛋白的同源蛋白,分别为MUSP-3、Shell matrix protein、Perlucin-like protein、Nacrein-like protein、NSPI-like protein和Whirlin-like protein。其中MUSP-3和Shell matrix protein最早发现于加利福尼亚贻贝的贝壳[8],MUSP-3贝壳形成中的功能尚不清楚,而Shell matrix protein是一种具有钙离子结合活性的蛋白,被认为在贝壳形成过程中参与了与碳酸钙的结合[8];Perlucin鉴定自鲍鱼(H.cumingii)贝壳,可促进碳酸钙的结晶并对碳酸钙晶体的生长方向具有调节作用[20]。Nacrein是第一种被报道的贝壳基质蛋白,最早发现于珠母贝(P.fucata),且在珍珠质层和棱柱层均有发现[21,22];此外,Nacrein序列中含有碳酸酐酶结构域,在贝壳形成中具有促进碳酸钙形成的作用,对生物矿化过程具有重要意义[23]。NSPI(Nacre serine protease inhibitor)是一种珠母贝(P.maxima)珍珠质层的贝壳基质蛋白,具有蛋白酶抑制剂活性[24];本次从地中海贻贝贝壳肌棱柱层鉴定到的NSPI类似蛋白序列中含有两个Kunitz型(KU)蛋白酶抑制剂结构域,预示着该蛋白可能在贝壳中起到保护贝壳基质蛋白防止被降解的作用。Whirlin是一种鉴定自牡蛎的贝壳基质蛋白,其序列中含有PDZ结构域[25]。该结构域参与蛋白质相互作用,可选择性的与其他蛋白的C端产生结合[26,27]。推测该蛋白可能参与了贝壳基质蛋白的分子交联。
鉴定结果中丰度最高的为含calponin结构域(Calponin domain)或者calponin同源结构域(Calponin homology domain)的蛋白,包括Calponin-like protein、Transgelin-like protein和Filamin-like protein。calponin结构域以及calponin同源结构域均具有肌动蛋白结合活性[28];此外,我们还鉴定到一种含有Thymosin(THY)结构域的蛋白(EST gi|238649869),THY结构域同样具有肌动蛋白结合活性[29];上述蛋白的发现暗示着这些蛋白可能通过与肌动蛋白的相互作用从而使得肌棱柱层与后闭壳肌的肌肉组织之间形成连接。
鉴定结果中还包括7种胶原蛋白(表2)。尽管此前的研究已从贝壳的分泌组织外套膜中鉴定到分泌型胶原蛋白[30],但贝壳中胶原蛋白的存在一直未获证实。本次研究中所鉴定的7种胶原蛋白均来自酸不溶性蛋白样品,多数含有VWA(von Willebrand factor type A)结构域,含VWA结构域的蛋白通常是构成细胞外基质的成分,多与细胞粘附有关[31]。Galtsoff早期的一项研究表明,胶原蛋白酶处理能显著降低闭壳肌与贝壳之间的连接力[11]。因此,胶原蛋白可能在肌棱柱层与闭壳肌的连接中发挥了重要作用。此外,在地中海贻贝贝壳肌棱柱层中还鉴定到细丝蛋白(filamin)和粗丝蛋白(filament),两者序列中分别含有filamin和filament结构域。Filamin结构域通常以串联形式存在于骨架蛋白,具有肌动蛋白结合功能,同时也参与了蛋白质相互作用[32];Filament则是构成中间纤维的主要成分,主要发现于细胞骨架蛋白[33]。上述两种蛋白在肌肉组织中含量丰富。细丝蛋白和粗丝蛋白在地中海贻贝贝壳肌棱柱层中的存在暗示着这两种蛋白可能贯穿于肌肉组织和肌棱柱层;同时,结合图1D中肌棱柱层表面膜结构中的小孔,推测在肌肉组织和肌棱柱层之间存在纤维状有机质,使得闭壳肌和肌棱柱层之间的连接更为紧密。
此外,地中海贻贝贝壳肌棱柱层中还鉴定到部分未知蛋白,这些未知蛋白在序列同源性搜索以及结构域预测中均未获得超过阈值的结果;而氨基酸组成分析表明,这些未知蛋白往往具有特殊的氨基酸组成。例如,EST gi|58308563编码的蛋白序列中含有高达41.2%的丙氨酸;EST gi|212830099编码的蛋白序列中含有丰富的亮氨酸(11.3%);EST gi|58307533编码的蛋白序列中含有丰富的丝氨酸(14.2%)等(表1)。此前的研究表明,部分贝壳基质蛋白表现出对某种氨基酸的偏好性(如高含量的丙氨酸、甘氨酸、丝氨酸、天冬氨酸等)[17-19],特殊的氨基酸组成有利于贝壳基质蛋白形成特殊的空间结构以便与碳酸钙分子产生结合或者与其他基质蛋白(或几丁质等其他有机物)形成框架结构用以指导碳酸钙分子的结晶和沉积[34]。
上述研究结果表明,地中海贻贝贝壳后闭壳肌与肌棱柱层形成紧密连接。肌棱柱层碳酸钙晶体采取了文石型的晶体构型且具有复杂的蛋白质分子组成。从中所鉴定的61种贝壳基质蛋白对肌棱柱层的形成以及与后闭壳肌的连接具有重要意义,同时也为深入探讨肌棱柱层的生物矿化机制以及碳酸钙-肌肉之间的连接机制奠定了基础。
致谢:感谢上海复旦大学蛋白质组研究中心周新文博士提供质谱分析。
[1]ALBECK S,ADDADI L,WEINER S.Regulation of calcite crystal morphology by intracrystalline acidic proteins and glycoproteins[J].Connective Tissue Research,1996,35:365-370.
[2]SELLINGER A,WEISS P M,NGUYEN A,et al.Continuous selfassembly of organic-inorganic nanocomposite coatings that mimic nacre[J].Nature,1998,394:256-260.
[3]LEE S W,JANG Y N,RYU K W,et al.Mechanical characteristics and morphological effect of complex crossed structure in biomaterials:fracture mechanics and microstructure of chalky layer in oyster shell[J].Micron,2011,42:60-70.
[4]KOUCHINSKY A.Shell microstructures in Early Cambrian mollusks[J].Acta Palaeontol Pol,2000,45(2):119-150.
[5]JACKSON A P,VINCENT J F V,TURNER R M.The mechanical design of nacre[J].Proc R Soc Lond B Biol Sci,1988,234: 415-440.
[6]TAYLOR J,LAYMAN M.The mechanical properties of bivalve(Mollusca)shell structures[J].Paleontology,1972,15(1):73-87.
[7]LI X,CHANG W C,YUH J C,et al.Nanoscale structural and mechanical characterization of a natural nanocomposite material: The shell of red abalone[J].Nano Lett,2004,4:613-617.
[8]LEE S W,JANG Y N,KIM J C.Characteristics of the Aragonitic Layer in Adult Oyster Shells,Crassostrea gigas:Structural Study of Myostracum including the Adductor Muscle Scar[J].Evid Based Complement Alternat Med,2011,2011:742 963.
[9]LOWENSTAM H A,WEINER S.On Biomineralization[M].New York:Oxford University Press,1989.
[10]CARTER J G.Shell microstructural data for the Bivalvia[Z]//Skeletal Biomineralization:Patterns,Processes and Evolutionary Trends.New York:Van Nostrand Reinhold,1990,1:297-411.
[11]GALTSOFF P A.The American oyster.Fish and Wildlife Service[M].U.S.Government Printing Office,1964.
[12]ALBECK S,ADDADI L,WEINER S.Regulation of calcite crystal morphology by intracrystalline acidic proteins and glycoproteins[J].Connective Tissue Research,1996,35:365-370.
[13]SUZUKI M,SAKUDA S,NAGASAWA H.Identification of chitin in the prismatic layer of the shell and a chitin synthase gene from the Japanese pearl oyster,Pinctada fucata[J].Bioscience,biotechnology,and biochemistry,2007,71:1 735-1 744.
[14]MARIE B,JOUBERT C,TAYAL A,et al.Different secretory repertoires control the biomineralization processes of prism and nacre deposition of the pearl oyster shell[J].Proc Natl Acad Sci,2012,109(51):20 986-20 991.
[15]DAUPHIN Y.Infrared spectra and elemental composition in recent biogenic calcites:relationships between the υ4 band wavelength and Sr and Mg concentrations[J].Appl Spectrom,1999,53:184-190.
[16]MARIE B,LE RN,ZANELLA-CLEON I,et al.Molecular evolution of mollusc shell proteins:insights from proteomic analysis of the edible mussel Mytilus[J].J Mol Evol,2011,72:531-546.
[17]CAROLINE J,DAVID P,BENJAMIN M.Transcriptome and proteome analysis of Pinctada margaritifera calcifying mantle and shell:focus on biomineralization[J].BMC Genomics,2010,11:613-625.
[18]MARIE B,MARIE A,JACKSON D J.Proteomic analysis of the organic matrix of the abalone Haliotis asinina calcified shell [J].Proteome Science,2010,8:54-64.
[19]MARIE B,TRINKLER N,ZANELLA-CLEON I.Proteomic identification of novel proteins from the calcifying shell matrix of the Manila clam Venerupis philippinarum[J].Marine Biotechnology,2011,13:955-962.
[20]LIN J Y,MA K Y,BAI Z Y,et al.Molecular cloning and characterization of perlucin from the freshwater pearl mussel,Hyriopsis cumingii[J].Gene,2013,526(2):210-216.
[21]MIYASHITA T,TAKAGI R,MIYAMOTA H,et al.Identical carbonic anhydrase contributes to nacreous or prismatic layer formation in Pinctada fucata(Mollusca:Bivalvia)[J].Veliger,2002,45:250-255.
[22]MARIE B,MARIN F,MARIE A,et al.Evolution of nacre:Biochemistry and proteomics of the shell organic matrix of the cephalopod Nautilus macromphalus[J].Chembiochem,2009,10:1 495-1 506.
[23]MIYAMOTO H,YANO M,MIYASHITA T.Similarities in the structure of nacrein,the shell-matrix protein,in a bivalve and a gastropod[J].J Molluscan Stud,2003,69:87-89.
[24]JACKSON D J,MCDOUGALL C,WOODCROFT B,et al.Parallel evolution of nacre building gene sets in molluscs[J].Molecular Biology Evolution,2010,27:591-608.
[25]ZHANG G,FANG X,GUO X,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature, 2012,490:49-54.
[26]KHAN Z,LAFON M.PDZ domain-mediated protein interactions:therapeutic targets in neurological disorders[J].Curr Med Chem,2014,21(23):2 632-2 641.
[27]CHI C N,BACH A,STR?MGAARD K,et al.Ligand binding by PDZ domains[J].Biofactors,2012,38(5):338-348.
[28]FERJANI I,FATTOUM A,MANAI M,et al.Two distinct regions of calponin share common binding sites on actin resulting in different modes of calponin-actin interaction[J].Biochimica et Biophysica Acta,2010,1804:1 760-1 767.
[29]XUE B,LEYRAT C,GRIMES J M,et al.Structural basis of thymosin-β4/profilin exchange leading to actin filament polymerization[J].Proc Natl Acad Sci,2014,111(43):4 596-4 605.
[30]RAMAN M,MATHEW S.Study of chemical properties and evaluation of collagen in mantle,epidermal connective tissue and tentacle of Indian Squid,Loligo duvauceli Orbigny[J].J Food Sci Technol,2014,51(8):1509-16.
[31]COLOMBATTI A,BONALDO P,DOLIANA R.Type A modules:interacting domains found in several non-fibrillar collagens and in other extracellular matrix proteins[J].Matrix,1993,13(4):297-306.
[32]RUSKAMO S,YLNNE J.Structure of the human filamin A actin-binding domain[J].Acta Crystallogr D Biol Crystallogr,2009, 65(11):1 217-1 221.
[33]HERRMANN H,B?R H,KREPLAK L,et al.Intermediate filaments:from cell architecture to nanomechanics[J].Nat Rev Mol Cell Biol,2007,8(7):562-573.
[34]EVANS J S.“Tuning in”to mollusk shell nacre-and prismatic-associated protein terminal sequence.Implications for biomineralization and the construction of high performance inorganic-organic composites[J].Chemical Reviews,2008,108:4 455-4 462.
Proteomic Analysis of Myostracum of Mytilus galloprovincialis Shell
WANG Xin-xing,GAO Peng,BAO Lin-fei,et al
(Laboratory of Marine Biological Resources and Molecular Engineering,School of Marine Science and Techonology,Zhejiang Ocean University,Zhoushan 316022,China)
The attachment of adductor muscle-shell in Mytilus mediates the closing of shells.For understanding the mechanism and the molecular composition of this attachment,Scanning Electronic Microscopic and Fourier Transform Infrared Spectroscopy were used to explore the micro-structure and polymorph of muscle-shell attachment of Mytilus galloprovincialis.Furthermore,the protein composition of shell matrix from myostracum layer from adductor muscle scar of M.galloprovincialis was detected by a combination of LC-MS/ MS analysis with the Mytilus EST dataset search,which resulted in the identification of a total of 61 proteins from acid-soluble and acid-insoluble shell matrix proteins form myostracum of M.galloprovincialis shell.From this protein set,many novel shell proteins were identified which included proteins with possible link to biomineralization and certain uncharacterized proteins with unusual amino acid composition.This data would be useful in understanding the role of SMPs associated with the formation of myostracum.Further,the identified protein set from the myostracum layer could provide a clue for exploring the mechanism of adductor muscle-shell attachment.
Q67
A
1008-830X(2015)05-0444-08
2015-02-10
浙江省自然科学基金项目(LY14C100001);蛋白质化学与发育生物学教育部重点实验室开放课题(2015DF02);国家大学生创新创业训练计划项目(01410340002)
王新星(1995-),女,研究方向:海洋生物学.E-mail:1421861484@qq.com
廖智.E-mail:liaozhi@zjou.edu.cn
