植物长链非编码RNA研究进展
2015-02-04黄小庆李丹丹吴娟
黄小庆,李丹丹,吴娟
植物长链非编码RNA研究进展
黄小庆,李丹丹,吴娟
东北林业大学盐碱地生物资源环境研究中心,东北油田盐碱植被恢复与重建教育部重点实验室,哈尔滨 150040
长链非编码RNA(Long non-coding RNA,lncRNA)长度大于200个核苷酸,大量存在于生物体中并具有多种生物学功能。目前,植物中发现的lncRNA大多由RNA聚合酶Ⅱ转录,并通过目标模仿、转录干扰、组蛋白甲基化和DNA甲基化等多种机制介导基因的表达,在植物开花、雄性不育、营养代谢、生物和非生物胁迫等生物过程中起着调节因子的作用。文章综述了近年来发现的植物lncRNA数据库、预测方法、表达及可能的生物学功能。
长链非编码RNA;数据库;基因表达调控;生物学功能
生物体内存在着两种不同的RNA:能翻译成蛋白质的编码RNA(Coding RNA,即mRNA)和不翻译成蛋白质的非编码RNA(Non-coding RNA, ncRNA)[1]。ncRNA种类繁多,目前尚无规范的命名方法。根据其表达特征,ncRNA分为持家非编码RNA(House- keeping non-coding RNA)和调控非编码RNA(Regulatory non-coding RNA)[2]。持家非编码RNA包括rRNA、tRNA、snRNA(Small nuclear RNA)和snoRNA (Small nucleolar RNA)等,这些 RNA在所有类型细胞中均要表达并且受环境因素影响较小,对维持细胞的基本功能是必不可少的。调控非编码RNA在生物体的特定组织器官和发育阶段表达,或者对应激环境产生应答反应后特异表达,这种特异表达调控着各种生物过程[3]。调控非编码RNA按照长度可分为短链非编码RNA(Small ncRNA)和长链非编码RNA(Long non-coding RNA,lncRNA)两大类[2]。短链非编码RNA长度小于200 nt,包括miRNA、siRNA和piRNA(Piwi-interacting RNA)等[4, 5]。短链非编码RNA的转录调控和作用机制已经比较清晰,如miRNA首先经RNA聚合酶Ⅱ转录形成pri-miRNA,然后经RNA核酸酶的剪切作用后成为20~30nt的成熟miRNA,它们在基因转录后水平通过与靶mRNA互补结合,在RNA加工、转录及转录后的基因沉默、应激应答、发生及发育等生物过程中起着调节因子的作用。
lncRNA是指长度在200nt以上的调控非编码RNA[6],没有长的开放阅读框(ORF),不具备编码蛋白质的功能,但是在特定的条件下有些lncRNA可以编码功能性寡肽[7]。lncRNA与mRNA有许多共同特征,例如由RNA聚合酶Ⅱ进行转录,并有5′加帽、3′聚腺苷酸化结构等[8]。研究表明,哺乳动物基因组中80%的转录产物为lncRNA[9]。lncRNA的作用机制和生物学功能极其多样,通过影响其他RNA或蛋白质的稳定性来调节基因的表达,参与剂量补偿、基因组印记、X染色体失活等生物过程[10, 11]。哺乳动物中超过1000个lncRNA参与了基因表达调控,在细胞周期调控、免疫监视和胚胎干细胞多能性等过程发挥作用[12]。大量研究证明,多种疾病与lncRNA的异常表达或突变相关[13]。lncRNA的功能已经引起了人们广泛的关注。
与人类和动物相比,植物lncRNA研究仍处于起步阶段。全基因组阵列分析和RNA-seq结果显示,植物体内存在大量lncRNA,一些通过目标模仿、转录干扰、与多梳蛋白抑制复合体2(Polycomb repressive complex 2, PRC2)有关的组蛋白甲基化和DNA甲基化等机制介导基因表达,在植物开花、雄性不育、营养代谢、生物和非生物胁迫等生物过程中起着调节因子作用。本文主要对植物lncRNA的最新研究进展进行了综述。
1 长链非编码RNA概述
1.1 长链非编码RNA的来源和分类
真核生物基因组中非编码部分所占比例较大,例如酿酒酵母()为29%,拟南芥()为71%,而人类基因组的非编码部分则占98%[14]。高等生物体内编码蛋白质的基因数目是相对保守的,并且一些基因的同源性很高,例如人类和小鼠有99%的蛋白质编码基因都是同源的[15],基因组测序发现人类不同个体之间的蛋白质编码序列可变性约为整个基因组的0.3%[16]。据上述研究结果推测,个体间和物种间的差异可能是由于非编码序列的不同导致基因表达的不同而造成的[15]。
由于限制ncRNA进化的因素较少,导致物种间lncRNA序列保守性低、不能形成大的同源基因家族。Ponting等[2]认为lncRNA可能通过以下5种方式形成:(1)蛋白质编码基因发生阅读框的插入,插入的阅读框和之前的编码序列形成新的功能性lncRNA,如在哺乳动物进化过程中,与雌性动物肌肉和成骨细胞分化和发育有关的基因的部分序列形成了X染色体失活相关的lncRNA,基因启动子区域和4个外显子与基因的部分外显子同源,而其余6个外显子则来自不同的转座子[17~19];(2)染色体重排后,两个不转录的并且之前相隔很远的序列区域并列形成一个多外显子的lncRNA,如犬科动物经染色体重排形成了一个含有ESTs为BM537447、C0597044和DN744681的lncRNA[2];(3)非编码基因通过反转录转座进行复制,形成有功能的非编码逆基因或非功能性的非编码反转录假基因,如小鼠的一个lncRNA基因家族的形成[20];(4)在ncRNA内部出现邻近的序列重复复制,两个重复序列串联形成新的lncRNA,如的5′区域和部分序列的形成;(5)插入转座因子产生有功能的lncRNA,如分别由不同的转座因子形成的存在于啮齿动物大脑细胞质中的lncRNA和类人猿大脑细胞质中的lncRNA。
Ponting等[2]根据lncRNA在基因组中相对于蛋白质编码基因的位置将其分为5种类型:(1)正义或(2)反义lncRNA,这种lncRNA分别与相同链或相反链的另一个蛋白质编码基因的一个或多个外显子相重叠。小鼠lncRNA与相同链的蛋白质编码基因的几个外显子部分序列重叠,lncRNA形成于蛋白质编码基因的反义链上;(3)双向(Bidirectional) lncRNA,它的转录起始位点与相反链上编码蛋白质基因的转录起始位点非常接近,但转录方向相反。拟南芥中最近发现的lncRNA形成于编码高尔基体运输复合物相关蛋白基因的反义链上,两者的转录方向相反[7];(4)内含子(intronic) lncRNA,它来源于次级转录物的内含子区域(有时可能为mRNA前体序列);(5)基因间(intergenic) lncRNA,它产生于两个基因之间区域。小鼠lncRNA即在编码基因和之间生成;酵母的lncRNA也产生于和编码基因之间。
1.2 长链非编码RNA的功能
lncRNA起初被认为是基因组转录的“噪音”,是RNA聚合酶Ⅱ转录的副产物,不具有生物学功能。然而,研究表明,许多lncRNA具有保守的二级结构及剪接形式[21~25]。有些lncRNA在特异的组织、细胞、发育阶段表达并且表达受到调节。Dinger等[21]发现小鼠的胚胎干细胞中多个lncRNA有差异表达,并且具有多能性;Mercer等[23]发现成年小鼠大脑不同区域特异表达的多个lncRNA;Sone等[24]发现lncRNA在小鼠神经系统的神经元中特异表达。这些特征预示着lncRNA是一种以RNA形式存在于生物体内的功能性分子。
lncRNA功能分析表明,它们在转录层面上调控编码基因的表达机制很大程度上是由其在基因组上的转录位点决定的。例如,来源于蛋白质编码位点的顺式-NAT通常采取“转录干扰”调控目标基因表达,因为目标基因的启动子和lncRNA很接近,两个启动子转录起始可以被共调节;同时一些lncRNA可以与目标基因启动子DNA结合,形成RNA-dsDNA三联体,阻断转录起始复合物的形成。其他一些lncRNA也可以通过控制转录因子的亚细胞定位或抑制RNA聚合酶活性在转录水平调节目标基因[8]。
lncRNA还可以通过调节pre-mRNA的可变剪接、运输、翻译和降解等,在转录后层面调节编码基因的表达。例如,lncRNA通过反式作用方式调节目标mRNA的稳定性。当lncRNA与目标mRNA存在碱基互补时,它就可以与目标mRNA形成双链RNA复合体,RNA复合体可以被加工成endo-siRNA,使目标mRNA降解[26]。
有些lncRNA能够招募染色质重构复合物到特定位点从而介导相关基因的表达沉默,进而在表观遗传控制基因表达中发挥重要作用,如、、和等。在人类基因组中,来源于基因座的能够招募染色质重构复合物PRC2并将其定位到位点,介导位点的表观遗传变化[27]。
Wilusz等[28]总结了生物体内lncRNA的8种作用机制,包括:(1)在蛋白质编码基因上游的启动子区域发生转录,上调或下调下游基因的表达。酵母lncRNA在基因的启动子区域发生转录,使的表达受到抑制[29];(2)通过抑制RNA聚合酶Ⅱ活性或介导染色质重构以及组蛋白修饰,影响下游基因表达。酵母中反义RNA的表达使Hda1组蛋白产生脱乙酰作用,引起基因的表达沉默[30];(3)lncRNA与蛋白质编码基因的转录物形成互补双链,阻断剪接体对于剪接位点的识别,从而形成可变剪接转录物。如哺乳动物的A型利尿钠肽前体(NPPA)的反义链可能与NPPA mRNA形成双链RNA,影响NPPA mRNA的剪接[31];(4)与正义或者反义转录物杂交形成互补双链,在Dicer酶作用下产生内源性的siRNA,调控基因的表达水平。在雌性哺乳动物中,与X染色体失活相关的lncRNA和其反义链转录物可形成互补双链,并被Dicer酶加工为siRNA,参与X染色体失活[32];(5)通过结合到特定蛋白质上,lncRNA能够调节相应蛋白质的活性。如可与转录因子Dlx2形成复合物,使Dlx2的活性增强[33];(6)作为RNA-蛋白质复合体结构的组成成分。Fox等[34]发现lncRNA与RNA结合蛋白PSP1、PSP2和p54/nrb共同构成了一种新型核质结构域——paraspeckles[25, 34~36];(7)结合到特定蛋白上,改变蛋白质在细胞中的定位。如转录因子NFAT位于细胞质中,钙依赖信号使其进入细胞核,从而激活靶基因的转录,而当lncRNA与核质运输有关的蛋白结合后,抑制了NFAT向细胞核的运输[37, 38];(8)lncRNA能被加工产生小分子RNA,如miRNA、piRNA和其他小RNA[39, 40]。
2 植物中长链非编码RNA研究
植物在应对环境变化的过程中显示出非凡的发育可塑性。为了最大程度地减小环境的不利影响,植物与环境相互作用,逐渐形成了许多内在生理和外在形态方面的适应对策,出现了不同的植物性状,这些特殊生理现象的形成与其体内一些基因的表达调控息息相关。
植物ncRNA的研究起步较晚,且多数研究集中在短链非编码RNA上,lncRNA的识别更是处于起步阶段,因此植物lncRNA的研究可能揭示控制植物生长和分化的未知新机制。
2.1 植物长链非编码RNA相关的数据库
在ncRNA研究过程中,随着lncRNA的分子特征、表达模式和功能数据增多,建立了一些针对人类和动物lncRNA的数据库,但这些数据库所含的植物lncRNA信息非常少。Yang等[41]首次通过收集人类、小鼠、狗、鸡、果蝇和线虫不同组织和细胞的543个ChIP-seq实验数据构建了ChIPBase数据库,主要包含了848 834个TF-lncRNA(transcription factor- lncRNA)和TF-miRNA(transcription factor-miRNA)调控关系,提供了TF-lncRNA和TF-miRNA调控关系图谱以及转录因子结合位点(TFBSs:transcription factor binding sites)等信息。Mattick等[42]于2009年通过芯片和原位杂交数据构建了哺乳类动物和人类的NRED(Noncoding RNA Expression Database)数据库,提供了lncRNA的二级结构特征、进化保守性和表达等信息。中国科学院计算技术研究所Zhao等[43]通过人类和小鼠芯片数据构建了包含73 370个lncRNA表达和功能注释的NONCODEv3数据库。2014年,Zhao等[44]进一步收集RNA-seq数据,并更新NONCODEv3数据库到NONCODEv4版本,其中lncRNA数量已经扩增到210 831个,有56 018和46 475个lncRNA分别来自人类和小鼠。LNCipedia[45]是一个专门提供人类lncRNA序列和结构全面注释的数据库。LncRNABase[46, 47]提供了miRNA调控lncRNA的互作信息。LncRNADisease[48]提供了疾病相关的lncRNA信息。
近年随着新的高通量测序技术的开发和运用,植物lncRNA的研究已经取得显著成果。拟南芥[7, 49~52]、苜蓿()[53]、玉米()[54]和小麦(L)[55]等植物全基因组范围内ncRNA的研究中发现了大量lncRNA。与人类和动物相比,植物lncRNA相关的专门数据库非常少,同时一些最新研究成果在公共数据库中并没有进行记录和注释[56]。
TAIR(TheInformation Resource)是专门的拟南芥基因数据库,是进行拟南芥生物学和生物模型研究的重要信息来源[57]。TAIR记载了拟南芥基因组序列及基因组图谱、各种基因的序列、结构、表达模式和功能注释及详尽的各种代谢途径,同时还有各种拟南芥种子库存数据等信息[58]。从2000~2010年,TAIR已经更新了10个版本。最新TAIR10中记载了拟南芥478条lncRNA信息,遗憾的是它们的生物学信息没有被记载。
2011年,Mattick等[59]构建了所有真核生物的lncRNAdb(lncRNAs Database)数据库,2014年,Quek等[60]将其更新为第二版,这个数据库包含lncRNA的序列及结构特征、进化保守性、表达、基因组序列、亚细胞定位、功能证据和文献链接等其他相关信息,其中记载了拟南芥(6个)、水稻(L,2个)、大豆(,1个)、苜蓿(3个)、葡萄(,1个)、芜菁(,1个)、番茄(,1个)和杨树(,1个)共计16个lncRNA表达情况和可能的功能等相关信息。
PlantNATsDB[61](Plant Natural Antisense Transcripts DataBase)是首个专门用于预测、查询植物天然反义转录物(Natural antisense transcript,NAT;一种产生于编码基因或非编码基因反义链的ncRNA,参与调节各种生物和非生物胁迫响应过程[62~64])及其调控功能的数据库,这个数据库大约包含69种植物的200万个NAT,其中包括拟南芥的7788个NAT(3005个正义-NAT,4783个反义-NAT)。但是,PlantNATsDB仅列出所有NAT对,不提供全基因组查询。
PLncDB[56](Plant long non-coding RNA database)是目前唯一公开发表的植物lncRNA专用数据库,该数据库收集了EST分析、RepTAS 分析、芯片、RNA-seq分析及 TAIR中发现的16 227个拟南芥lncRNA的信息,记载了在不同组织、发育阶段、突变体和应激处理条件下这些lncRNA的表达特性,介绍了编码lncRNA的基因座及其侧翼基因组区域的DNA甲基化和组蛋白修饰等表观遗传变化和可能的功能,同时也收集了全基因组siRNA信息。PLncDB是目前较为全面的植物lncRNA查询数据库。
以上lncRNA数据库的相关信息见表1。
2.2 植物长链非编码RNA的预测
植物lncRNA的预测方法主要分为计算RNA组学方法(Computational RNomics)和实验RNA组学方法(Experimental RNomics)[65](表2)。计算RNA组学方法是指采用计算机与生物学相结合的生物信息分析(Bioinformatic analyse)进行lncRNA预测的方法。计算预测lncRNA方法中使用的原始数据可以是公开发表的cDNA序列、表达序列标签(EST)、各种全长cDNA克隆、tiling arrays数据、RNA-seq数据等。首先将这些数据与基因组序列进行比对,去除蛋白质编码基因重叠区域,通过GeneMark.hmm[66]、GenScan[67]、ESTScan2[68]、ANGLE[69]和ORF-Predictor[70]等软件对剩余序列进行ORF预测,根据明显开放阅读框(ORF)存在与否,区分mRNA和ncRNA,保留下来的ncRNA序列长度>200 nt的被认为是lncRNA。此外,预测RNA序列是否具有编码蛋白质能力的生物信息学软件(如CRITICA[71]、DIANA- EST[72]、CSTminer[73]、CONC[74]、Coding Potential Calculator[75]、integrated ncRNA finder[76]和RNAcode[77])也已经被用于动、植物lncRNA探索研究中。
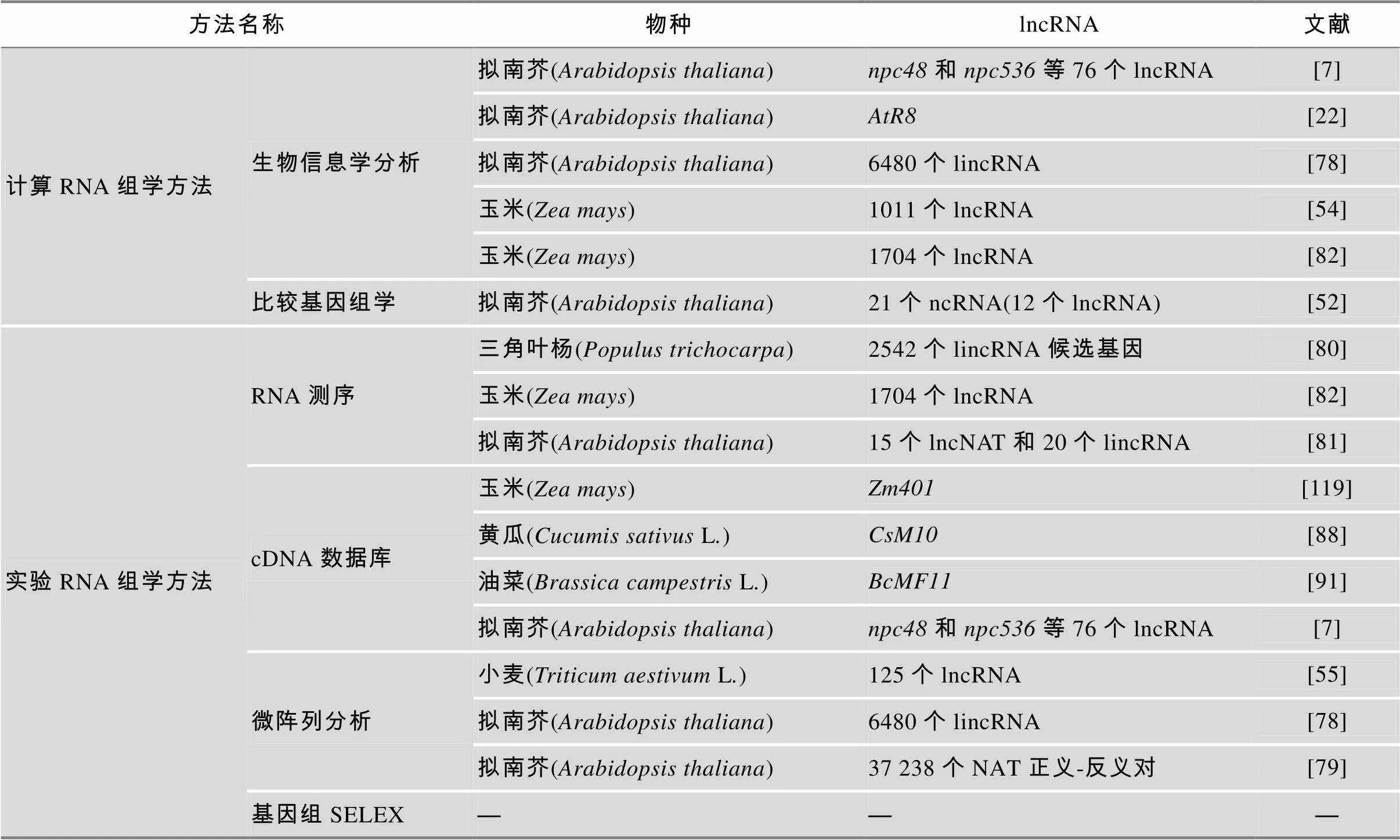
表2 植物lncRNA的预测
实验RNA组学方法是通过RNA-seq、构建cDNA数据库、微阵列分析和基因组SELEX等发现lncRNA[65]。高通量测序技术特别是RNA-seq技术是发现lncRNA的有效方法,可以直接、快速地发现低丰度、新的lncRNA。目前研究中更广泛采用了计算与实验相结合的方法,这种结合可以更准确、有效地预测lncRNA。
Ben等[7]通过对拟南芥基因组全长cDNA数据库进行生物信息学分析,发现了76个ncRNA基因,其中14个为NAT,5个是siRNA前体;22个lncRNA与非生物应激应答相关,其中2个lncRNA在非生物应激环境中异常表达影响了拟南芥的生长和分化。
最近,Liu等[78]开发了RepTAS(Reproducibility-based tiling array analysis strategy)生物信息学方法,并对公开发表的200个拟南芥tiling array数据进行了全基因组范围综合分析,发现了6480个基因间长链非编码RNA(Long intergenic non-coding RNA, lincRNA),后续的RNA-seq结果表明其中的2708个表现出组织特异性和非生物胁迫应答特性。Wang等[79]利用RepTAS生物信息学方法分析了公开发表的来自拟南芥不同器官、组织、不同激素处理、生物和非生物胁迫的200个tiling array数据,发现大量的新lncRNA。这些lncRNA平均长度约731nt,包括基因内转录物和37 238个NAT正义-反义对,实验分析证明大量光应答NAT对与组蛋白修饰有关。
Xin等[55]利用微阵列分析和高通量SBS测序相结合的方法确定了小麦中125个lncRNA(其中部分为小RNA前体),它们具有组织特异性表达特征并对白粉病和热胁迫产生应激应答反应。
Shuai等[80]采用RNA测序方法在三角叶杨()中识别出2542个lincRNA候选基因,其中504个对干旱胁迫产生应激应答。
为了研究拟南芥对尖孢镰刀菌感染的应激应答反应,Zhu等[81]对来自感染细菌的拟南芥RNA进行深度测序,发现15个长链非编码NAT和20个新的lincRNA的表达受尖孢镰刀菌感染的调节,部分基因对真菌感染产生防御作用,表明某些lncRNA是拟南芥中抗真菌网络中的重要节点。
Wu等[22]侧重于Pol Ⅲ的Ⅲ型启动子结构特点,对拟南芥基因组进行计算检索,发现了20种可能的ncRNA候选基因,通过体外无细胞转录系统活性检测,发现了lncRNA(约260nt),其在拟南芥幼苗中表现了低氧应激应答特性。
Boerner等[54]开发并优化了Python Pipeline计算机语言,采用Python Pipeline与SVM(Support Vector Machine)相结合的生物信息分析方法筛选了玉米全长cDNA序列,发现2492个lncRNA候选基因,其中19个为miRNA前体,237个为shRNA(short hairpin)前体,1225个siRNA前体,1011个为lncRNA,这些lncRNA中572个位于编码基因内,439个位于基因间区域。
Li等[82]对已经发表的30个不同实验组玉米EST数据和玉米全基因组序列注释及RNA-seq数据进行生物信息学分析,首次发现玉米中的1704个lncRNA,平均长度为463nt,表达分析显示近50%的lncRNA具有组织特异性,其中一些来自于、和(与玉米生长发育和农业性状相关的关键基因)的调控区域。
研究表明,一些重要的ncRNA在许多生物基因组中是保守的。基于序列保守性的比较基因组学方法也是预测ncRNA的生物信息学方法[52, 83]。Song等[52]运用比较基因组学方法比对了拟南芥、水稻、三角叶杨、木瓜()、葡萄的全基因组序列,确定了拟南芥中16个基因家族的21个新ncRNA基因。
基因组SELEX方法已成功运用在mRNA结合蛋白筛选中,但ncRNA研究中的应用目前未见报道[84]。
关于植物lncRNA的预测总结于表2。
2.3 植物长链非编码RNA的表达及可能的生物学功能
2.3.1 植物长链非编码RNA的转录
真核生物细胞核中的RNA聚合酶分为三类:RNA聚合酶Ⅰ(Pol Ⅰ:RNA polymeraseⅠ)、RNA聚合酶Ⅱ(Pol Ⅱ:RNA polymeraseⅡ)和RNA聚合酶Ⅲ(Pol Ⅲ:RNA polymeraseⅢ)。Pol Ⅰ位于核仁中,合成rRNA,如28S rRNA、18S rRNA和5.8S rRNA,这些rRNA是细胞中主要的核糖核酸之一,与多种核糖体蛋白质共同构成核糖体,在蛋白质合成过程中起重要作用。除上述几种持家非编码RNA外,目前还没有发现Pol Ⅰ合成的其他ncRNA。Pol Ⅱ位于核质中,除合成编码蛋白质的mRNA外,还合成许多ncRNA,如snRNA、snoRNA和microRNA。此外,一些lncRNA也由Pol Ⅱ合成,如大豆、苜蓿和水稻中保守的[85~87]、黄瓜(L)中的[88]、玉米中的[89]、拟南芥中的[90]、及[7]和油菜(L)中的[91]等。Pol Ⅲ位于核质中,合成tRNA、5S rRNA、snoRNA及microRNA等ncRNA。最近研究发现,Pol Ⅲ也合成调节编码基因表达的新ncRNA[92]。另外拟南芥中Pol Ⅲ的Ⅲ型启动子合成了低氧应激应答型lncRNA[22]。Wierzbicki等[93]发现,拟南芥中Pol Ⅱ进化而来的RNA聚合酶IV和V(Pol IV/V)参与了短链非编码RNA的合成;同时Pol V也合成一类拟南芥lncRNA,它们可以和一些抑制染色质修饰的沉默因子结合导致基因沉默[94]。此外,拟南芥和玉米中Pol IV和Pol V可能也合成一类与RNA介导的DNA甲基化有关的lncRNA[54, 95]。
2.3.2 植物长链非编码RNA的表达及可能的生物学功能
lncRNA的研究成果主要集中在人类和动物中。近年,随着对植物ncRNA研究的不断深入,发现了一些植物特有的lncRNA。
是一个磷酸盐饥饿条件下表达被高度诱导的lncRNA家族[8]。这类lncRNA除了具有短的、不保守的开放阅读框外,还具有23nt长度的保守核苷酸序列,根据上述特征推测它们可能作为核糖核酸调节子或编码一些信号肽在生物体内起作用[96, 97]。番茄的是家族中最早被发现的成员,其在基因组中为单拷贝,并且磷酸盐饥饿条件下在根和叶中被迅速诱导,表明可能与番茄中早期磷酸盐饥饿反应机制有关[98]。苜蓿[99~101]序列中含有许多短的开放阅读框,其中一个开放阅读框的部分序列与番茄中的开放阅读框重合。在磷酸盐饥饿条件下在苜蓿的根中被大量诱导,但在磷酸盐充足的条件下几乎不被转录。是在水稻中发现的家族成员,磷酸盐饥饿时在根和叶部被诱导表达,但在根中的诱导尤为显著[102]。(Induced by phosphate starvation 1)是拟南芥中发现的又一个家族成员,在高度磷酸盐饥饿条件下,在叶和根部大量积累[96]。是拟南芥中同样被磷酸盐饥饿诱导积累于地上部分和根中的miRNA,它通过与的5′UTR区域的部分序列碱基互补配对介导()mRNA的断裂,在转录后水平降低的表达[97, 103]。研究发现,序列中23个核苷酸保守区域与的部分序列也存在碱基互补,通过模仿的靶基因,与竞争结合,抑制的活性,使mRNA的积累增加,维持磷酸盐饥饿条件下拟南芥的正常生长[96, 97]。通过“目标模仿”机制抑制miRNA的活性[8],是首个明确作用机制的家族成员。作用机制表明,一些lncRNA可以通过自身部分序列与功能性miRNA碱基互补配对,从而模仿miRNA的靶基因,减弱miRNA的调节因子作用,在生物学过程中发挥重要作用。这为今后未知功能的lncRNA作用靶元件预测提供了一个新思路。
染色质修饰对动、植物发育过程中组织特异性基因的表达和基因组重组具有重要作用。lncRNA和染色质重构复合物之间的相互作用是动物基因表观遗传沉默的普遍机制[104]。植物开花调控基因(FLOWERING LOCUS C)可以通过染色质修饰影响植物开花时间。研究表明,冷处理诱导(Vernalization insensitive 3)的表达,可以招募染色质重构复合物PRC2到基因座产生抑制型H3K27me3组蛋白修饰,从而改变染色质活性状态,引起基因的表观遗传沉默,调控植物开花时间[8]。Swiezewski等[105]采用Single Nucleotide Resolution Array技术对双链及侧翼50 kb区域进行研究,发现了多个来自基因座的冷诱导反义转录物,即(Cold induced antisense intragenic RNA)。具有5¢端加帽及3¢端聚腺苷酸化结构,并可进行剪接。的冷应激反应比发生得更早,并且表达不受缺失影响。冷诱导条件下对的表达有抑制作用,但环境条件由寒冷变为温暖时,的抑制作用变得可逆,这表明只能暂时抑制表达,而基因失活的表观遗传模式是调控植物开花的必要途径。因为和的表达水平呈相反趋势,并且在发生转录情况下,基因座上的Pol II含量减少,这意味着通过转录干扰的方式抑制表达。所以冷胁迫下的表达调控可能通过下面模式进行:冷胁迫诱导的表达,瞬时抑制的转录,进一步冷胁迫处理诱导了的表达,招募染色质重构复合物PRC2到基因上,产生抑制型H3K27me3组蛋白修饰,引起基因的表观遗传沉默,进而调控植物开花[8]。另外,新的研究表明不是抑制表达所必须的ncRNA[106]。
(Cold assisted intronic noncoding RNA)是来自拟南芥基因第一个内含子的正义转录物,是不同于的又一类Pol Ⅱ转录的lncRNA[107]。长约1100nt,具有Pol IV和V转录的lncRNA结构特点,如5¢端加帽,但3¢端没有聚腺苷酸化等特征。冷处理诱导表达,冷处理20 d后的表达达到峰值,冷处理30 d后其表达降到冷处理前水平。虽然的表达模式和相似,但它的表达峰值比滞后10 d。研究表明,缺失型拟南芥植株表现出冷处理后开花延迟现象,这可能是由于的缺失导致基因的染色质没有富集抑制型H3K27me3组蛋白引起的。RNA结合实验也进一步表明,特异地与CLF(Curly Leaf,与H3K27me3修饰有关的PRC2复合体的重要组成成分)相互作用。因此,冷处理条件下,可能通过募集PRC2到染色质引起表观遗传修饰,构建了基因沉默机制,即通过Polycomb-依赖模式负调控的表达。是首次发现的参与基因染色质表观遗传学修饰的植物lncRNA[8]。
Nongken 58S(NK 58S)是水稻Nongken 58N (NK 58N)的自然突变品种,其光敏感引起雄性不育(Photoperiod-sensitivemalesterility, PSMS)。NK 58S在长日照条件下花粉完全不育,短日照则不会引起不育。Ding等[108]采用位置克隆方法(Position cloning strategy)发现长约1236nt的lncRNA,即(Long-day-specific male-fertility-associated RNA)。长日照条件下,NK 58N和NK 58S花粉的发育需要大量,而调查发现长日照条件下NK 58S的积累量少于NK 58N,并且NK 58S启动子区域甲基化程度高于NK 58N。进一步研究表明,这个现象是由于NK 58S序列的一个碱基C突变成G引起了二级结构产生变化,导致启动子区域的甲基化,抑制了NK 58S表达,使长日照条件下NK 58S雄花不育。同时,来自启动子区域正义链的siRNA,即也对启动子区域的甲基化起调节作用[109]。Zhou等[110]证明中136nt长度的小RNA是引起PSMS的重要序列。遗憾的是,基因和小RNA是否调控雄性花粉不育目前仍未阐明[111]。
是最初在豆科植物中发现的根瘤特异性表达并与共生固氮根瘤形成有关的lncRNA[86,112],之后分别在水稻及苜蓿中发现了的同源基因[87]和[113]。在水稻茎中特异表达,对水稻的器官分化和维管束组织发育发挥关键作用[87]。酵母三杂交实验发现苜蓿中与一种持续表达的RNA结合蛋白MtRBP1产生相互作用,使细胞质中MtRBP1产生移位,这种RNA结合蛋白的亚细胞定位改变可能代表lncRNA在细胞中的某种新功能[85]。最近,酵母三杂交实验进一步发现与两个结瘤素酸性RNA结合蛋白MtSNARP1和MtSNARP2相互作用[114]。上述结果表明,酵母三杂交技术可以有效应用于与lncRNA相互作用的生物大分子筛选研究。番茄ENOD40-GUS转基因植株的GUS表达模式显示,番茄中可能与消除乙烯的刺激反应有关[115]。此外,在大豆中编码12和24个氨基酸长度的短肽,这些短肽特定地与蔗糖合成酶结合,表明与根瘤中蔗糖利用率的调节有关。苜蓿编码13和27个氨基酸长度的短肽,这两个短肽与生物活性相关[116, 117]。但是,的作用机制可能只是通过的RNA分子本身而不是短肽起作用[27]。
(1517nt)是在黄瓜中发现的一种lncRNA[88],其在黄瓜的不同组织、不同发育阶段和不同光照周期都有差异表达,可能与黄瓜的雌雄分化有关。以前的研究表明,、和这3种ncRNA是生物胁迫信号中至关重要的基因[118]。与、和有179nt的保守区域,推测可能调节某些生物应激应答基因的表达。
Dai等[119]发现玉米中(1149nt)是一种花粉特异性表达的lncRNA,在单子叶植物中有高度保守的序列和稳定的RNA二级结构。能调节、、这3种与玉米花粉发育有关的关键基因的表达,抑制或者过表达该基因都会影响雄蕊和花药的生长发育,引起玉米小孢子空瘪、变形,绒毡层出现退化延迟,使花粉形态异常,可用于玉米雄性不育植株的制作[89]。在烟草(L.)的研究中也得到了相似的结果,烟草中的异常表达扰乱了花的生长发育,约有80%转基因烟草的花蕾在开花前花轴变黄,最终大多数脱落[120]。
Song等[91]发现了在油菜花粉发育的各个阶段持续表达的花粉特异基因(828nt)。在转基因油菜中,雌花发育正常,而雄花产生不育的花粉。研究结果显示过表达植株的绒毡层和小孢子发育异常,使花药发育不全,颜色变为褐色或白色,形态干瘪,甚至不产生花粉,引起油菜雄花不育[91,121]。
Ben等确定了拟南芥中76个lncRNA[7]:14个为蛋白质编码基因的反义转录物,它们可能起着反式调控作用;5个是24nt siRNA的前体;经DCL4加工后形成;22个lncRNA受非生物胁迫调节。生物学功能研究发现,过量表达改变了积累并导致拟南芥出现锯齿状叶和开花延迟现象;盐胁迫下过量表达调节了拟南芥根的生长。
Wu等[22]用计算检索方法发现了Pol Ⅲ合成的lncRNA,其在拟南芥根中及拟南芥由来的MM2d细胞的细胞质中特异地大量表达,且在甘蓝()和萝卜(L.)等十字花科植物中发现了它的同源物。低氧应激处理抑制表达,表明它是低氧应激应答型lncRNA。目前,的作用机制尚不清楚。
植物中的lncRNA总结于表3。
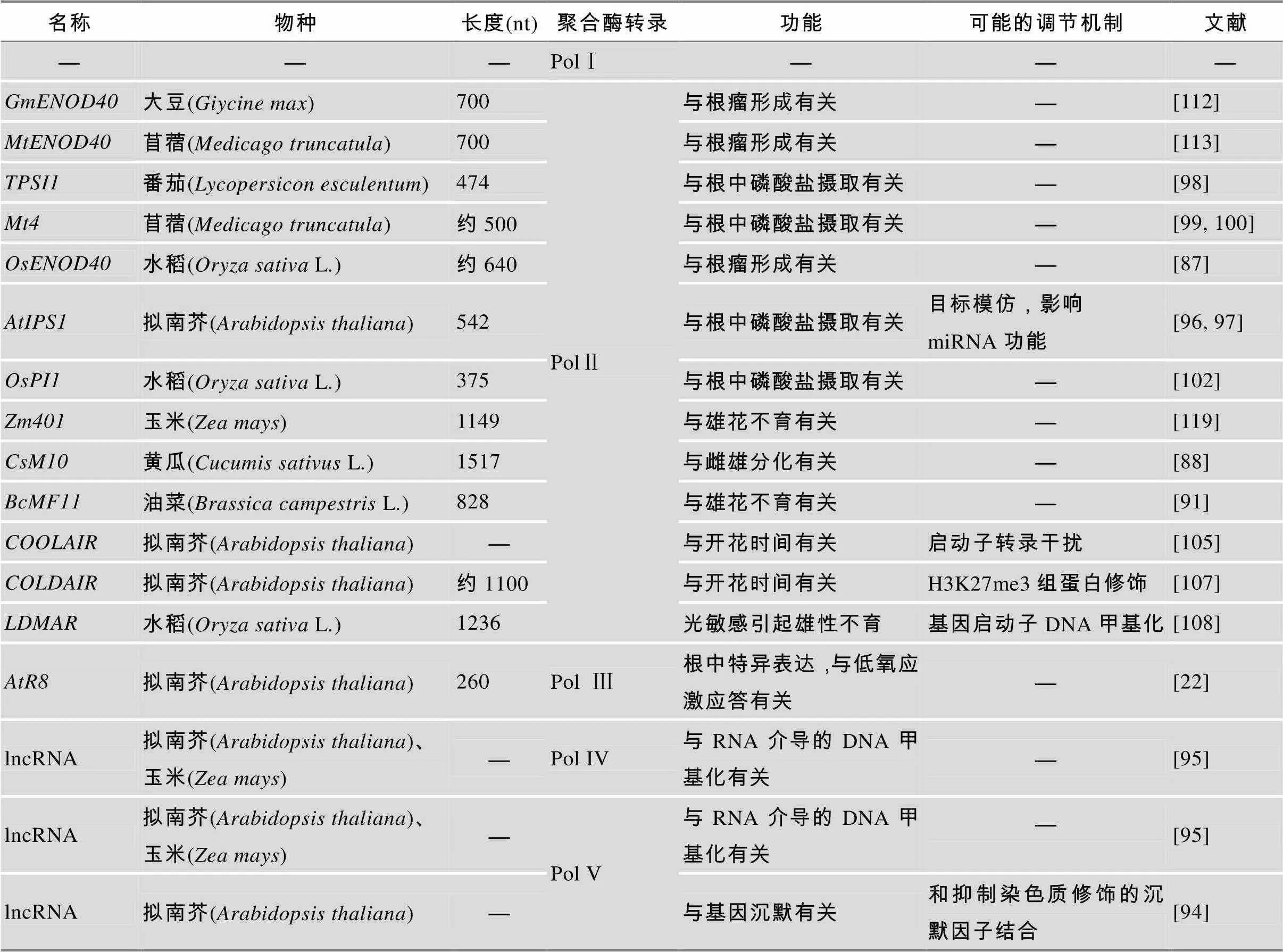
表3 植物中的lncRNA
3 结语与展望
ncRNA具有明确的生理功能,但却不编码任何蛋白质,仅以RNA分子形式发挥作用。从ncRNA在不同生物体细胞的所有转录物中所占的比例可以看出,RNA的数量和多样性似乎与物种的复杂性密切相关,其普遍性及重要性远远超出人们的想象,在生物体的多种生命活动中起着至关重要的作用。到目前为止,人们只看到部分ncRNA所具有的功能,如果能进一步确定ncRNA的结构特征将会有更大的发现。
随着被发现的ncRNA增多,越来越多的相关研究涉及到植物lncRNA,但目前植物lncRNA的研究仍处在初级阶段,且研究主要集中在拟南芥、水稻、小麦和玉米这几种植物中,已经发现的部分lncRNA不能被确定是功能性转录物还是转录“噪音”,未知的lncRNA的详细数量仍不清楚。同时,预测植物lncRNA作用靶元件的生物信息学工具相对较少,这为准确推测其可能的作用机制增加了难度。目前,已经证明了某些lncRNA是microRNA的前体,部分lncRNA可以调节microRNA或蛋白质编码基因的功能,但对其详细作用机制所知甚少,而且不同lncRNA适用的研究方法很少可以相互借鉴。这些因素限制了植物lncRNA研究进展。在以后的研究中,有必要开发针对植物lncRNA的研究方法,从而更好地预测植物中lncRNA和其作用靶目标,识别更多符合已知功能模式或未知功能模式的lncRNA,并阐明其作用机制,为植物ncRNA研究提供新思路。
[1] Eddy SR. Non-coding RNA genes and the modern RNA world., 2001, 2(12): 919–929.
[2] Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs., 2009, 136(4): 629–641.
[3] Stoughton RB. Applications of DNA microarrays in biology., 2005, 74: 53–82.
[4] Carthew RW, Sontheimer EJ. Origins and Mechanisms of miRNAs and siRNAs., 2009, 136(4): 642–655.
[5] Malone CD, Hannon GJ. Small RNAs as guardians of the genome., 2009, 136(4): 656–668.
[6] Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions., 2009, 10(3): 155–159.
[7] Ben Amor B, Wirth S, Merchan F, Laporte P, d'Aubenton-Carafa Y, Hirsch J, Maizel A, Mallory A, Lucas A, Deragon JM, Vaucheret H, Thermes C, Crespi M. Novel long non-protein coding RNAs involved in Arabidopsis differentiation and stress responses., 2009, 19(1): 57–69.
[8] Zhang J, Mujahid H, Hou YX, Nallamilli BR, Peng ZH. Plant long ncRNAs: a new frontier for gene regulatory control., 2013, 4: 1038–1045.
[9] Kapranov P, Cheng J, Dike S, Nix DA, Duttagupta R, Willingham AT, Stadler PF, Hertel J, Hackermüller J, Hofacker IL, Bell I, Cheung E, Drenkow J, Dumais E, Patel S, Helt G, Ganesh M, Ghosh S, Piccolboni A, Sementchenko V, Tammana H, Gingeras TR. RNA maps reveal new RNA classes and a possible function for pervasive transcription., 2007, 316(5830): 1484– 1488.
[10] Prasanth KV, Spector DL. Eukaryotic regulatory RNAs: an answer to the 'genome complexity' conundrum., 2007, 21(1): 11–42.
[11] Amaral PP, Dinger ME, Mercer TR, Mattick JS. The eukaryotic genome as an RNA machine., 2008, 319(5871): 1787–1789.
[12] Cao XW, Yeo G, Muotri AR, Kuwabara T, Gage FH. Noncoding RNAs in the mammalian central nervous system., 2006, 29: 77–103.
[13] Wapinski O, Chang HY. Long noncoding RNAs and human disease., 2011, 21(6): 354–361.
[14] 郑晓飞. 非编码RNA. 北京: 化学工业出版社, 2008: 2–3.
[15] Mattick JS. Non-coding RNAs: the architects of eukaryotic complexity., 2001, 2(11): 986–991.
[16] Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, Smith HO, Yandell M, Evans CA, Holt RA, Gocayne JD, Amanatides P, Ballew RM, Huson DH, Wortman JR, Zhang Q, Kodira CD, Zheng XH, Chen L, Skupski M, Subramanian G, Thomas PD, Zhang JH, Gabor Miklos GL, Nelson C, Broder S, Clark AG, Nadeau J, McKusick VA, Zinder N, Levine AJ, Roberts RJ, Simon M, Slayman C,Hunkapiller M, Bolanos R, Delcher A, Dew I, Fasulo D, Flanigan M, Florea L, Halpern A, Hannenhalli S, Kravitz S, Levy S, Mobarry C, Reinert K, Remington K, Abu-Threideh J, Beasley E, Biddick K, Bonazzi V, Brandon R, Cargill M, Chandramouliswaran I, Charlab R,Chaturvedi K, Deng ZM, Di Francesco V, Dunn P, Eilbeck K, Evangelista C, Gabrielian AE, Gan WN, Ge WM, Gong FC, Gu ZP, Guan P, Heiman TJ, Higgins ME, Ji RR, Ke ZX, Ketchum KA, Lai ZW, Lei YD, Li ZY, Li JY, Liang Y, Lin XY, Lu F, Merkulov GV, Milshina N, Moore HM, Naik AK,Narayan VA, Neelam B, Nusskern D, Rusch DB, Salzberg S, Shao W, Shue B, Sun JT, Wang ZY, Wang AH, Wang X, Wang J, Wei MH, Wides R, Xiao CL, Yan CH, Yao A, Ye J, Zhan M, Zhang WQ, Zhang HY, Zhao Q, Zheng LS, Zhong F, Zhong WY, Zhu SC, Zhao SY, Gilbert D, Baumhueter S, Spier G, Carter C, Cravchik A, Woodage T, Ali F, An HJ, Awe A, Baldwin D, Baden H, Barnstead M, Barrow I, Beeson K, Busam D,Carver A, Center A, Cheng ML, Curry L, Danaher S, Davenport L, Desilets R, Dietz S, Dodson K, Doup L, Ferriera S, Garg N,Gluecksmann A, Hart B, Haynes J, Haynes C, Heiner C, Hladun S, Hostin D, Houck J, Howland T, Ibegwam C, Johnson J, Kalush F, Kline L, Koduru S, Love A, Mann F, May D, McCawley S, McIntosh T, McMullen I, Moy M, Moy L, Murphy B, Nelson K, Pfannkoch C, Pratts E, Puri V, Qureshi H, Reardon M, Rodriguez R, Rogers YH, Romblad D, Ruhfel B, Scott R, Sitter C, Smallwood M, Stewart E, Strong R,Suh E, Thomas R, Tint NN, Tse S, Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J,Zaveri K, Abril JF, Guigó R, Campbell MJ, Sjolander KV, Karlak B, Kejariwal A, Mi H, Lazareva B, Hatton T, Narechania A, Diemer K,Muruganujan A, Guo N, Sato S, Bafna V, Istrail S, Lippert R, Schwartz R, Walenz B, Yooseph S, Allen D, Basu A, Baxendale J, Blick L,Caminha M, Carnes-Stine J, Caulk P, Chiang YH, Coyne M, Dahlke C, Mays A, Dombroski M, Donnelly M, Ely D, Esparham S, Fosler C,Gire H, Glanowski S, Glasser K, Glodek A, Gorokhov M, Graham K, Gropman B, Harris M, Heil J, Henderson S, Hoover J, Jennings D,Jordan C, Jordan J, Kasha J, Kagan L, Kraft C, Levitsky A, Lewis M, Liu XJ, Lopez J, Ma D, Majoros W, McDaniel J, Murphy S, Newman M, Nguyen T, Nguyen N, Nodell M, Pan S, Peck J, Peterson M, Rowe W, Sanders R, Scott J, Simpson M, Smith T, Sprague A, Stockwell T, Turner R, Venter E, Wang M, Wen M, Wu D, Wu M, Xia A, Zandieh A, Zhu XH. The sequence of the human genome., 2001, 291(5507): 1304–1351.
[17] Duret L, Chureau C, Samain S, Weissenbach J, Avner P. The Xist RNA gene evolved in eutherians by pseudogenization of a protein-coding gene., 2006, 312(5780): 1653–1655.
[18] Elisaphenko EA, Kolesnikov NN, Shevchenko AI, Rogozin IB, Nesterova TB, Brockdorff N, Zakian SM. A dual origin of the Xist gene from a protein-coding gene and a set of transposable elements., 2008, 3(6): e2521.
[19] Flynn M, Saha O, Young P. Molecular evolution of the LNX gene family., 2011, 11: 235.
[20] Ravasi T, Suzuki H, Pang KC, Katayama S, Furuno M, Okunishi R, Fukuda S, Ru K, Frith MC, Gongora MM, Grimmond SM, Hume DA, Hayashizaki Y, Mattick JS. Experimental validation of the regulated expression of large numbers of non-coding RNAs from the mouse genome., 2006, 16(1): 11–19.
[21] Dinger ME, Amaral PP, Mercer TR, Pang KC, Bruce SJ, Gardiner BB, Askarian-Amiri ME, Ru K, Soldà G, Simons C, Sunkin SM, Crowe ML, Grimmond SM, Perkins AC, Mattick JS. Long noncoding RNAs in mouse embryonic stem cell pluripotency and differentiation., 2008, 18(9): 1433–1445.
[22] Wu J, Okada T, Fukushima T, Tsudzuki T, Sugiura M, Yukawa Y. A novel hypoxic stress-responsive long non-coding RNA transcribed by RNA polymerase III in Arabidopsis., 2012, 9(3): 302–313.
[23] Mercer TR, Dinger ME, Sunkin SM, Mehler MF, Mattick JS. Specific expression of long noncoding RNAs in the mouse brain., 2008, 105(2): 716–721.
[24] Sone M, Hayashi T, Tarui H, Agata K, Takeichi M, Nakagawa S. The mRNA-like noncoding RNA Gomafu constitutes a novel nuclear domain in a subset of neurons., 2007, 120(Pt 15): 2498–2506.
[25] Clemson CM, Hutchinson JN, Sara SA, Ensminger AW, Fox AH, Chess A, Lawrence JB. An architectural role for a nuclear noncoding RNA: NEAT1 RNA is essential for the structure of paraspeckles., 2009, 33(6): 717–726.
[26] Golden DE, Gerbasi VR, Sontheimer EJ. An inside job for siRNAs., 2008, 31(3): 309–312.
[27] Zhu QH, Wang MB. Molecular functions of long non- coding RNAs in plants., 2012, 3(1): 176– 190.
[28] Wilusz JE, Sunwoo H, Spector DL. Long noncoding RNAs: functional surprises from the RNA world., 2009, 23(13): 1494–1504.
[29] Martens JA, Laprade L, Winston F. Intergenic transcription is required to repress the Saccharomyces cerevisiae SER3 gene., 2004, 429(6991): 571–574.
[30] Camblong J, Iglesias N, Fickentscher C, Dieppois G, Stutz F. Antisense RNA stabilization induces transcriptional gene silencing via histone deacetylation in., 2007, 131(4): 706–717.
[31] Annilo T, Kepp K, Laan M. Natural antisense transcript of natriuretic peptide precursor A (): structural organization and modulation ofexpression., 2009, 10: 81.
[32] Ogawa Y, Sun BK, Lee JT. Intersection of the RNA interference and X-inactivation pathways., 2008, 320(5881): 1336–1341.
[33] Feng JC, Bi CM, Clark BS, Mady R, Shah P, Kohtz JD. Thenoncoding RNA is transcribed from the Dlx-5/6 ultraconserved region and functions as a Dlx-2 transcriptional coactivator., 2006, 20(11): 1470– 1484.
[34] Fox AH, Lam YW, Leung AKL, Lyon CE, Andersen J, Mann M, Lamond AI. Paraspeckles: a novel nuclear domain., 2002, 12(1): 13–25.
[35] Sasaki YTF, Ideue T, Sano M, Mituyama T, Hirose T. MENε/β noncoding RNAs are essential for structural integrity of nuclear paraspeckles., 2009, 106(8): 2525–2530.
[36] Sunwoo H, Dinger ME, Wilusz JE, Amaral PP, Mattick JS, Spector DL. MEN epsilon/beta nuclear-retained non-coding RNAs are up-regulated upon muscle differentiation and are essential components of paraspeckles., 2009, 19(3): 347–359.
[37] Hogan PG, Chen L, Nardone J, Rao A. Transcriptional regulation by calcium, calcineurin, and NFAT., 2003, 17(18): 2205–2232.
[38] Willingham AT, Orth AP, Batalov S, Peters EC, Wen BG, Aza-Blanc P, Hogenesch JB, Schultz PG. A strategy for probing the function of noncoding RNAs finds a repressor of NFAT., 2005, 309(5740): 1570–1573.
[39] Reinhart BJ, Weinstein EG, Rhoades MW, Bartel B, Bartel DP. MicroRNAs in plants., 2002, 16(13): 1616–1626.
[40] Hirsch J, Lefort V, Vankersschaver M, Boualem A, Lucas A, Thermes C, d'Aubenton-Carafa Y, Crespi M. Characterization of 43 non-protein-coding mRNA genes in, including the-derived transcripts., 2006, 140(4): 1192–1204.
[41] Yang JH, Li JH, Jiang S, Zhou H, Qu LH. ChIPBase: a database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP- Seq data., 2013, 41(Database issue): D177–D187.
[42] Dinger ME, Pang KC, Mercer TR, Crowe ML, Grimmond SM, Mattick JS. NRED: a database of long noncoding RNA expression., 2009, 37(Database issue): D122–D126.
[43] Bu D, Yu K, Sun S, Xie C, Skogerbø G, Miao R, Xiao H, Liao Q, Luo H, Zhao G, Zhao H, Liu Z, Liu C, Chen R, Zhao Y. NONCODE v3.0: integrative annotation of long noncoding RNAs., 2012, 40(Database issue): D210–D215.
[44] Xie C, Yuan J, Li H, Li M, Zhao G, Bu D, Zhu W, Wu W, Chen R, Zhao Y. NONCODEv4: exploring the world of long non-coding RNA genes., 2014, 42(Database issue): D98–D103.
[45] Volders PJ, Helsens K, Wang XW, Menten B, Martens L, Gevaert K, Vandesompele J, Mestdagh P. LNCipedia: a database for annotated human lncRNA transcript sequences and structures., 2013, 41(Database issue): D246–D251.
[46] Li JH, Liu S, Zhou H, Qu LH, Yang JH. starBase v2.0: decoding miRNA-ceRNA, miRNA-ncRNA and protein- RNA interaction networks from large-scale CLIP-Seq data., 2014, 42(Database issue): D92–D97.
[47] Yang JH, Li JH, Shao P, Zhou H, Chen YQ, Qu LH. starBase: a database for exploring microRNA-mRNA interaction maps from Argonaute CLIP-Seq and Degradome- Seq data., 2011, 39(Database issue): D202–D209.
[48] Chen G, Wang ZY, Wang DQ, Qiu CX, Liu MX, Chen X, Zhang QP, Yan GY, Cui QH. LncRNADisease: a database for long-non-coding RNA-associated diseases., 2013, 41(Database issue): D983–D986.
[49] MacIntosh GC, Wilkerson C, Green PJ. Identification and analysis of Arabidopsis expressed sequence tags characteristic of non-coding RNAs., 2001, 127(3): 765–776.
[50] Marker C, Zemann A, Terhörst T, Kiefmann M, Kastenmayer JP, Green P, Bachellerie JP, Brosius J, Hüttenhofer A. Experimental RNomics: identification of 140 candidates for small non-messenger RNAs in the plant Arabidopsis thaliana., 2002, 12(23): 2002–2013.
[51] Rymarquis LA, Kastenmayer JP, Hüttenhofer AG, Green PJ. Diamonds in the rough: mRNA-like non-coding RNAs., 2008, 13(7): 329–334.
[52] Song D, Yang Y, Yu B, Zheng B, Deng Z, Lu BL, Chen X, Jiang T. Computational prediction of novel non-coding RNAs in Arabidopsis thaliana., 2009, 10(Suppl 1): S36.
[53] Wen J, Parker BJ, Weiller GF. In Silico identification and characterization of mRNA-like noncoding transcripts in Medicago truncatula., 2007, 7(4–5): 485–505.
[54] Boerner S, McGinnis KM. Computational identification and functional predictions of long noncoding RNA in., 2012, 7(8): e43047.
[55] Xin MM, Wang Y, Yao YY, Song N, Hu ZR, Qin DD, Xie CJ, Peng HR, Ni ZF, Sun QX. Identification and characterization of wheat long non-protein coding RNAs responsive to powdery mildew infection and heat stress by using microarray analysis and SBS sequencing., 2011, 11: 61.
[56] Jin JJ, Liu J, Wang H, Wong L, Chua NH. PLncDB: plant long non-coding RNA database., 2013, 29(8): 1068–1071.
[57] Swarbreck D, Wilks C, Lamesch P, Berardini TZ, Garcia-Hernandez M, Foerster H, Li D, Meyer T, Muller R, Ploetz L, Radenbaugh A, Singh S, Swing V, Tissier C, Zhang P, Huala E. The Arabidopsis Information Resource (TAIR): gene structure and function annotation., 2008, 36(Database issue): D1009–D1014.
[58] Lamesch P, Berardini TZ, Li DH, Swarbreck D, Wilks C, Sasidharan R, Muller R, Dreher K, Alexander DL, Garcia-Hernandez M, Karthikeyan AS, Lee CH, Nelson WD, Ploetz L, Singh S, Wensel A, Huala E. The Arabidopsis Information Resource (TAIR): improved gene annotation and new tools., 2012, 40(Database issue): D1202–D1210.
[59] Amaral PP, Clark MB, Gascoigne DK, Dinger ME, Mattick JS. lncRNAdb: a reference database for long noncoding RNAs., 2011, 39(Database issue): D146–D151.
[60] Quek XC, Thomson DW, Maag JL, Bartonicek N, Signal B, Clark MB, Gloss BS, Dinger ME. lncRNAdb v2.0: expanding the reference database for functional long noncoding RNAs., 2015,43(Database issue): D168–D173.
[61] Chen DJ, Yuan CH, Zhang J, Zhang Z, Bai L, Meng YJ, Chen LL, Chen M. PlantNATsDB: a comprehensive database of plant natural antisense transcripts., 2012, 40(Database issue): D1187–D1193.
[62] Lavorgna G, Dahary D, Lehner B, Sorek R, Sanderson CM, Casari G. In search of antisense., 2004, 29(2): 88–94.
[63] Werner A. Natural antisense transcripts., 2005, 2(2): 53–62.
[64] Charon C, Moreno AB, Bardou F, Crespi M. Non-protein- coding RNAs and their interacting RNA-binding proteins in the plant cell nucleus., 2010, 3(4): 729–739.
[65] Hüttenhofer A. RNomics: identification and function of small non-protein-coding RNAs in model organisms., 2006, 71: 135–140.
[66] Lukashin AV, Borodovsky M. GeneMark.hmm: new solutions for gene finding., 1998, 26(4): 1107–1115.
[67] Burge CB, Karlin S. Finding the genes in genomic DNA., 1998, 8(3): 346–354.
[68] Lottaz C, Iseli C, Jongeneel CV, Bucher P. Modeling sequencing errors by combining Hidden Markov models., 2003, 19(Suppl 2): ii103-ii112.
[69] Shimizu K, Adachi J, MuraokaY. ANGLE: a sequencing errors resistant program for predicting protein coding regions in unfinished cDNA., 2006, 4(3): 649–664.
[70] Jia H, Osak M, Bogu GK, Stanton LW, Johnson R, Lipovich L. Genome-wide computational identification and manual annotation of human long noncoding RNA genes., 2010, 16(8): 1478–1487.
[71] Badger JH, Olsen GJ. CRITICA: coding region identification tool invoking comparative analysis., 1999, 16(4): 512–524.
[72] Hatzigeorgiou AG, Fiziev P, Reczko M. DIANA-EST: a statistical analysis., 2001, 17(10): 913–919.
[73] Mignone F, Grillo G, Liuni S, Pesole G. Computational identification of protein coding potential of conserved sequence tags through cross-species evolutionary analysis., 2003, 31(15): 4639–4645.
[74] Liu J, Gough J, Rost B. Distinguishing protein-coding from non-coding RNAs through support vector machines., 2006, 2(4): e29.
[75] Kong L, Zhang Y, Ye ZQ, Liu XQ, Zhao SQ, Wei LP, Gao G. CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine., 2007, 35(Web Server issue): W345– W349.
[76] Lu ZJ, Yip KY, Wang GL, Shou C, Hillier LW, Khurana E, Agarwal A, Auerbach R, Rozowsky J, Cheng C, Kato M, Miller DM, Slack F, Snyder M, Waterston RH, Reinke V, Gerstein MB. Prediction and characterization of noncoding RNAs inby integrating conservation, secondary structure, and high-throughput sequencing and array data., 2011, 21(2): 276–285.
[77] Washietl S, Findeiβ S, Müller SA, Kalkhof S, von Bergen M, Hofacker IL, Stadler PF, Goldman N. RNAcode: robust discrimination of coding and noncoding regions in comparative sequence data., 2011, 17(4): 578–594.
[78] Liu J, Jung C, Xu J, Wang H, Deng S, Bernad L, Arenas-Huertero C, Chua NH. Genome-wide analysis uncovers regulation of long intergenic noncoding RNAs in Arabidopsis., 2012, 24(11): 4333–4345.
[79] Wang H, Chung PJ, Liu J, Jang IC, Kean MJ, Xu J, Chua NH. Genome-wide identification of long noncoding natural antisense transcripts and their responses to light in., 2014, 24(3): 444–453.
[80] Shuai P, Liang D, Tang S, Zhang ZJ, Ye CY, Su YY, Xia XL, Yin WL. Genome-wide identification and functional prediction of novel and drought-responsive lincRNAs in., 2014, 65(17): 4975–4983.
[81] Zhu QH, Stephen S, Taylor J, Helliwell CA, Wang MB. Long noncoding RNAs responsive toinfection in., 2014, 201(2): 574–584.
[82] Li L, Eichten SR, Shimizu R, Petsch K, Yeh CT, Wu W, Chettoor AM, Givan SA, Cole RA, Fowler JE, Evans MM, Scanlon MJ, Yu J, Schnable PS, Timmermans MC, Springer NM, Muehlbauer GJ. Genome-wide discovery and characterization of maize long non-coding RNAs., 2014, 15(2): R40.
[83] McCutcheon JP, Eddy SR. Computational identification of non-coding RNAs in Saccharomyces cerevisiae by comparative genomics., 2003, 31(14): 4119–4128.
[84] Hüttenhofer A, Vogel J. Experimental approaches to identify non-coding RNAs., 2006, 34(2): 635–646.
[85] Campalans A, Kondorosi A, Crespi M., a short open reading frame-containing mRNA, induces cytoplasmic localization of a nuclear RNA binding protein in., 2004, 16(4): 1047–1059.
[86] van de Sande K, Pawlowski K, Czaja I, Wieneke U, Schell J, Schmidt J, Walden R, Matvienko M, Wellink J, van Kammen A, Franssen H, Bisseling T. Modification of phytohormone response by a peptide encoded byof legumes and a nonlegume., 1996, 273(5273): 370–373.
[87] Kouchi H, Takane K, So RB, Ladha JK, Reddy PM. Rice: isolation and expression analysis in rice and transgenic soybean root nodules., 1999, 18(2): 121–129.
[88] Cho J, Koo DH, Nam YW, Han CT, Lim HT, Bang JW, Hur Y. Isolation and characterization of cDNA clones expressed under male sex expression conditions in a monoecious cucumber plant (L. cv. Winter Long)., 2005, 146(3): 271–281.
[89] Ma JX, Yan BX, Qu YY, Qin FF, Yang YT, Hao XJ, Yu JJ, Zhao Q, Zhu DY, Ao GM., a short-open reading-frame mRNA or noncoding RNA, is essential for tapetum and microspore development and can regulate the floret formation in maize., 2008, 105(1): 136–146.
[90] Shin H, Shin HS, Chen RJ, Harrison MJ. Loss offunction impacts phosphate distribution between the roots and the shoots during phosphate starvation., 2006, 45(5): 712–726.
[91] Song JH, Cao JS, Yu XL, Xiang X. BcMF11, a putative pollen-specific non-coding RNA fromssp. chinensis., 2007, 164(8): 1097–1100.
[92] Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A. The expanding RNA polymerase III transcriptome., 2007, 23(12): 614–622.
[93] Wierzbicki AT, Haag JR, Pikaard CS. Noncoding transcription by RNA polymerase Pol IVb/Pol V mediates transcriptional silencing of overlapping and adjacent genes., 2008, 135(4): 635–648.
[94] Rowley MJ, Böhmdorfer G, Wierzbicki AT. Analysis of long non-coding RNAs produced by a specialized RNA polymerase in., 2013, 63(2): 160–169.
[95] Haag JR, Pikaard CS. Multisubunit RNA polymerases IV and V: purveyors of non-coding RNA for plant gene silencing., 2011, 12(8): 483–492.
[96] Martín AC, del Pozo JC, Iglesias J, Rubio V, Solano R, de La Peña A, Leyva A, Paz-Ares J. Influence of cytokinins on the expression of phosphate starvation responsive genes in., 2000, 24(5): 559–567.
[97] Franco-Zorrilla JM, Valli A, Todesco M, Mateos I, Puga MI, Rubio-Somoza I, Leyva A, Weigel D, García JA, Paz-Ares J. Target mimicry provides a new mechanism for regulation of microRNA activity., 2007, 39(8): 1033–1037.
[98] Liu CM, Muchhal US, Raghothama KG. Differential expression of TPS11, a phosphate starvation-induced gene in tomato., 1997, 33(5): 867–874.
[99] Burleigh SM, Harrison MJ. Characterization of the Mt4 gene from., 1998, 216(1): 47–53.
[100]Burleigh SH, Harrison MJ. The down-regulation of-like genes by phosphate fertilization occurs systemically and involves phosphate translocation to the shoots., 1999, 119(1): 241–248.
[101]Burleigh SH, Harrison MJ. A novel gene whose expression in Medicago truncatula roots is suppressed in response to colonization by vesicular-arbuscular mycorrhizal (VAM) fungi and to phosphate nutrition., 1997, 34(2): 199–208.
[102]Wasaki J, Yonetani R, Shinano T, Kai M, Osaki M. Expression of thegene, cloned from rice roots using cDNA microarray, rapidly responds to phosphorus status., 2003, 158(2): 239–248.
[103]Fujii H, Chiou TJ, Lin SI, Aung K, Zhu JK. A miRNA involved in phosphate-starvation response in., 2005, 15(22): 2038–2043.
[104]Nagano T, Fraser P. Emerging similarities in epigenetic gene silencing by long noncoding RNAs., 2009, 20(9–10): 557–562.
[105]Swiezewski S, Liu FQ, Magusin A, Dean C. Cold-induced silencing by long antisense transcripts of anPolycomb target., 2009, 462(7274): 799–802.
[106]Helliwell CA, Robertson M, Finnegan EJ, Buzas DM, Dennis ES. Vernalization-repression of Arabidopsis FLC requires promoter sequences but not antisense transcripts., 2011, 6(6): e21513.
[107]Heo JB, Sung S. Vernalization-mediated epigenetic silencing by a long intronic noncoding RNA., 2011, 331(6013): 76–79.
[108] Ding JH, Lu Q, Ouyang YD, Mao HL, Zhang PB, Yao JL, Xu CG, Li XH, Xiao JH, Zhang QF. A long noncoding RNA regulates photoperiod-sensitive male sterility, an essential component of hybrid rice., 2012, 109(7): 2654–2659.
[109] Ding JH, Shen JQ, Mao HL, Xie WB, Li XH, Zhang QF. RNA-directed DNA methylation is involved in regulating photoperiod-sensitive male sterility in rice., 2012, 5(6): 1210–1216.
[110]Zhou H, Liu QJ, Li J, Jiang DG, Zhou LY, Wu P, Lu S, Li F, Zhu LY, Liu ZL, Chen LT, Liu YG, Zhuang CX. Photoperiod- and thermo-sensitive genic male sterility in rice are caused by a point mutation in a novel noncoding RNA that produces a small RNA., 2012, 22(4): 649–660.
[111]Zhu DM, Deng XW. A non-coding RNA locus mediates environment-conditioned male sterility in rice., 2012, 22(5): 791–792.
[112]Yang WC, Katinakis P, Hendriks P, Smolders A, de Vries F, Spee J, van Kammen A, Bisseling T, Franssen H. Characterization of, a gene showing novel patterns of cell-specific expression during soybean nodule development., 1993, 3(4): 573–585.
[113]Crespi MD, Jurkevitch E, Poiret M, d'Aubenton-Carafa Y, Petrovics G, Kondorosi E, Kondorosi A. enod40, a gene expressed during nodule organogenesis, codes for a non-translatable RNA involved in plant growth., 1994, 13(21): 5099–5112.
[114]Laporte P, Satiat-Jeunemaître B, Velasco I, Csorba T, Van de Velde W, Campalans A, Burgya J, Arevalo-Rodriguez M, Crespi M. A novel RNA-binding peptide regulates the establishment of the-nitrogen-fixing symbiosis., 2010, 62(1): 24–38.
[115]Vleghels I, Hontelez J, Ribeiro A, Fransz P, Bisseling T, Franssen H. Expression ofduring tomato plant development., 2003, 218(1): 42–49.
[116]Rohrig H, Schmidt J, Miklashevichs E, Schell J, John M. Soybean ENOD40 encodes two peptides that bind to sucrose synthase., 2002, 99(4): 1915–1920.
[117]Sousa C, Johansson C, Charon C, Manyani H, Sautter C, Kondorosi A, Crespi M. Translational and structural requirements of the early nodulin gene, a short-open reading frame-containing RNA, for elicitation of a cell-specific growth response in the alfalfa root cortex., 2001, 21(1): 354–366.
[118]Erdmann VA, Szymanski M, Hochberg A, de Groot N, Barciszewski J. Non-coding, mRNA-like RNAs database Y2K., 2000, 28(1): 197–200.
[119]Dai XY, Yu JJ, Zhao Q, Zhu DY, Ao GM. Non-coding RNA for, a pollen-specific gene of., 2004, 46(4): 497–504.
[120]Ma JX, Zhao Q, Yu JJ, Ao GM. Ectopic expression of a maize pollen specific gene,, results in aberrant anther development in tobacco., 2005, 144(1-2): 133–140.
[121]Song JH, Cao JS, Wang CG., a novel non-coding RNA gene from, is required for pollen development and male fertility., 2013, 32(1): 21–30.
(责任编委: 张宪省)
Long non-coding RNAs in plants
Xiaoqing Huang, Dandan Li, Juan Wu
Long non-coding RNAs (lncRNAs), which are longer than 200 nucleotides in length, widely exist in organisms and function in a variety of biological processes. Currently, most of lncRNAs found in plantsare transcribed by RNA polymerase Ⅱ and mediate gene expression through multiple mechanisms, such as target mimicry, transcription interference, histone methylation and DNA methylation, and play important roles in flowering, male sterility, nutrition metabolism, biotic and abiotic stress and other biological processes as regulators in plants. In this review, we summarize the databases, prediction methods, and possible functions of plant lncRNAs discovered in recent years.
long non-coding RNAs (lncRNAs); database; gene expression regulation; biological function
2014-12-04;
2015-01-27
东北林业大学中央高校基本科研业务费专项资金项目(编号: DL12BA38),长江学者和创新团队发展计划资助(编号: IRT13053),东北林业大学中央高校基本科研业务费专项资金项目(编号: DL13EA04 - 02)和教育部留学回国人员科研启动基金(第47批)资助
黄小庆,硕士研究生,专业方向:生物化学与分子生物学。E-mail: huangxiaoqing85@163.com
吴娟,博士,副教授,研究方向:植物生理学和RNA分子生物学。E-mail: wuj1970@163.com
10.16288/j.yczz.14-432
2015-3-3 16:58:43
http://www.cnki.net/kcms/detail/11.1913.R.20150303.1658.002.html
