43份菊芋种质资源遗传多样性的ISSR分析
2015-01-07赵孟良孙雪梅王丽慧
赵孟良,孙雪梅,王丽慧,李 莉
(青海省农林科学院菊芋研发中心 青海省蔬菜遗传与生理重点实验室,青海 西宁 810016)
43份菊芋种质资源遗传多样性的ISSR分析
赵孟良,孙雪梅,王丽慧,李 莉
(青海省农林科学院菊芋研发中心 青海省蔬菜遗传与生理重点实验室,青海 西宁 810016)
【目的】 探明国内外菊芋(HelianthustuberosusL.)资源间的亲缘关系远近及遗传多样性。【方法】 以40份国外菊芋资源与3个国内栽培品种为材料,选用14条引物进行ISSR分子标记分析,分析他们的遗传多样性,利用POPGENE32软件计算多态位点比率(PPB)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)、Shannon’s信息指数(I)。采用NTSYS 2.10软件计算品种间的相似系数,进行聚类分析,并对不同类群的形态学主要特点进行分析。【结果】 采用14条引物对43份菊芋资源进行PCR扩增,共获得203个标记,其中多态性标记199个,多态性比率为98.0%。采用POPGENE32软件分析,供试的43份菊芋资源平均有效等位基因数为1.894 8,平均 Nei’s基因多样性指数为0.467 7,平均 Shannon’s 信息指数为0.659 0。43份菊芋资源间的相似系数为0.443~0.955。聚类结果显示,供试的43个菊芋资源可聚为2个类群,能很好地将国外菊芋资源与国内品种区分开来。不同类群菊芋的块茎形态及颜色有明显差异。【结论】 国内外菊芋资源间遗传关系较远,且国外资源间存在丰富的遗传多样性。
菊芋;种质资源;ISSR;遗传多样性
菊芋(HelianthustuberosusL.)又名洋姜、鬼子姜,为菊科(Compositae)向日葵属(Helianthus)多年生草本植物,18世纪末从欧洲传入中国,之后被人们当作蔬菜栽培食用。菊芋具有极强的抗旱、抗寒、耐盐碱能力,能够适应恶劣的生长环境。菊芋还具有极大的利用价值,国内众多单位已经开展了关于菊芋的生物质能源利用[1]、果聚糖代谢调控机理[2]、菊芋秸秆造纸[3]及饲草料加工应用[4]等方面的研究。目前,国内对于菊芋品种的选育主要是采用人工定向的方式,因此丰富的种质资源必将是菊芋育种的先决条件。青海省农林科学院菊芋研发中心历经十余年的育种研究,已经审定通过了青芋1号、青芋2号、青芋3号3个菊芋品种[5-7],是目前国内审定品种最多的菊芋研发单位,已收集、保存国内外菊芋资源400余份。
ISSR(Inter-simple sequence repeat)是Zietkeiwitcz等于1994年发展起来的一种微卫星基础上的分子标记,目前已广泛应用于居群遗传多样性分析[8-9]、杂交后代鉴定[10-11]及分子生态学[12]研究中。在菊芋方面,本课题组已成功建立了菊芋ISSR-PCR扩增条件及程序,并筛选出了特异性好的引物,同时利用该技术对国内青芋1号、2号、3号3个菊芋品种进行了快速鉴定[13-14]。因此,ISSR分子标记在菊芋方面的应用已较为成熟,可以利用该技术研究不同来源的菊芋资源遗传多样性。
本研究从青海省农林科学院菊芋研发中心种质资源库中挑选了40份国外菊芋种质资源及国内3个审定菊芋品种为供试材料,利用ISSR技术,对国外与国内菊芋品种的亲缘关系及遗传多样性进行了分析,旨在为菊芋种质资源的进一步开发利用提供参考。
1 材料与方法
1.1 材 料
试验材料为40份国外菊芋资源与3个国内审定菊芋品种(表1),均种植于青海省农林科学院菊芋研发中心资源圃内。于2012-04选取幼嫩菊芋叶片,硅胶干燥后在-20 ℃保存备用。43份菊芋资源分别于2012-10及2013-10采收时按照菊芋观察记载标准进行统计。
表1 43份菊芋种质资源及来源Table 1 43 Helianthus tuberosus L.and their exporting countries
1.2 方 法
试验于2012-04-2013-09在青海省蔬菜遗传与生理重点实验室内完成。
1.2.1 DNA提取与检测 菊芋叶片DNA提取及检测参照文献[15]的方法进行。
1.2.2 引物筛选及PCR扩增 参照韩睿等[14]的菊芋 ISSR 引物筛选结果,从中选择扩增条带清晰、多态性好的14条引物进行PCR反应。引物名称及序列已在表2中列出。ISSR-PCR反应体系和扩增程序参照赵孟良等[13]的报道进行,PCR反应体系(20 μL)包括:1.5 mmol/L Mg2+,200 μmol/L dNTPs,0.5 μmol/L引物,1.0 UTaqDNA 聚合酶和50 ng模板DNA;PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火40 s,72 ℃延伸75 s,循环40次;72 ℃延伸10 min,4 ℃保存。
1.3 数据统计分析
凝胶图像用Image Lab 3.0软件进行分析,电泳图谱的每条带(DNA片段)均为1个分子标记(Marker),代表1个引物的结合位点。根据各分子标记的迁移率及其有无统计所有的二元数据,有带(显性)记作1,无带(隐性)记为0,强带和弱带均赋值,根据0,1矩阵建立43个菊芋资源的DNA指纹图谱。利用POPGENE32软件计算多态位点比率(PPB)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)、Shannon’s信息指数(I)。用NTSYS 2.10版软件计算样品间的相似系数,然后利用SAHN Clustering软件的算术平均法(Unweighted pair-group method arithmetie average,UPGMA)进行聚类分析。
2 结果与分析
2.1 菊芋资源的ISSR分子标记的多态性
由表2可知,14条引物对43份菊芋模板共扩增出位点203个,其中多态性位点199个,多态性位点比率(PPB)达到98.0%,引物847的多态位点比率最低,为91.7%;14条引物扩增的条带数为9~18条,平均为14.5条,其中扩增条带数最少的为引物844,只有9条。扩增片段长度大多为300~2 000 bp。引物844的扩增结果见图1。

表2 基于14条引物的43份菊芋PCR扩增结果Table 2 PCR amplification of 43 Jerusalem artichoke varieties based on 14 primers
注:*Y 代表简并碱基C 或者 T,R 代表简并碱基A 或者 G。
Note:Y represents degenerate base C or T,and R represents degenerate base A or G.
由表3可知,供试的43份菊芋资源平均有效等位基因数(Ne)为1.894 8,平均Nei’s 基因多样性指数为0.467 7,平均 Shannon’s信息指数为0.659 0。各位点遗传多样性程度也存在较大差异,Nei’s基因多样性指数最大值为0.499,最小值为0.430 8。Shannon’s 信息指数最大值为0.693,最小值为0.308。
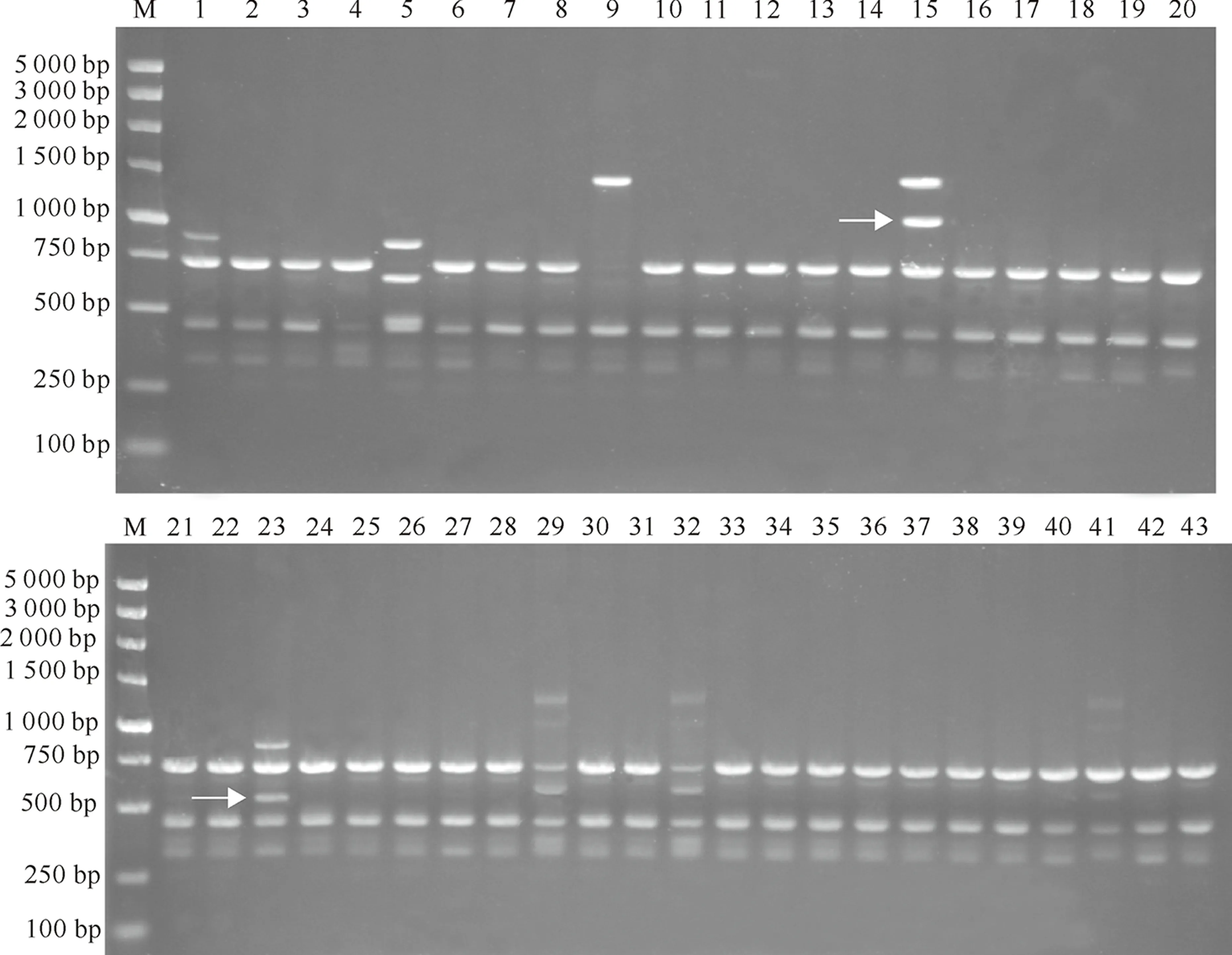
图1 引物844对43份菊芋的PCR扩增结果M.DNA Marker DS5000;1~43.分别为表1中的43份菊芋资源;箭头所示为多态性位点位置Fig.1 PCR amplification of 43 Jerusalem artichoke varieties based on primer 844 M.DNA Marker DS5000;1-43.the 43 of Helianthus tuberosus L.as in Table 1;The position indicated by arrows are polymorphism loci
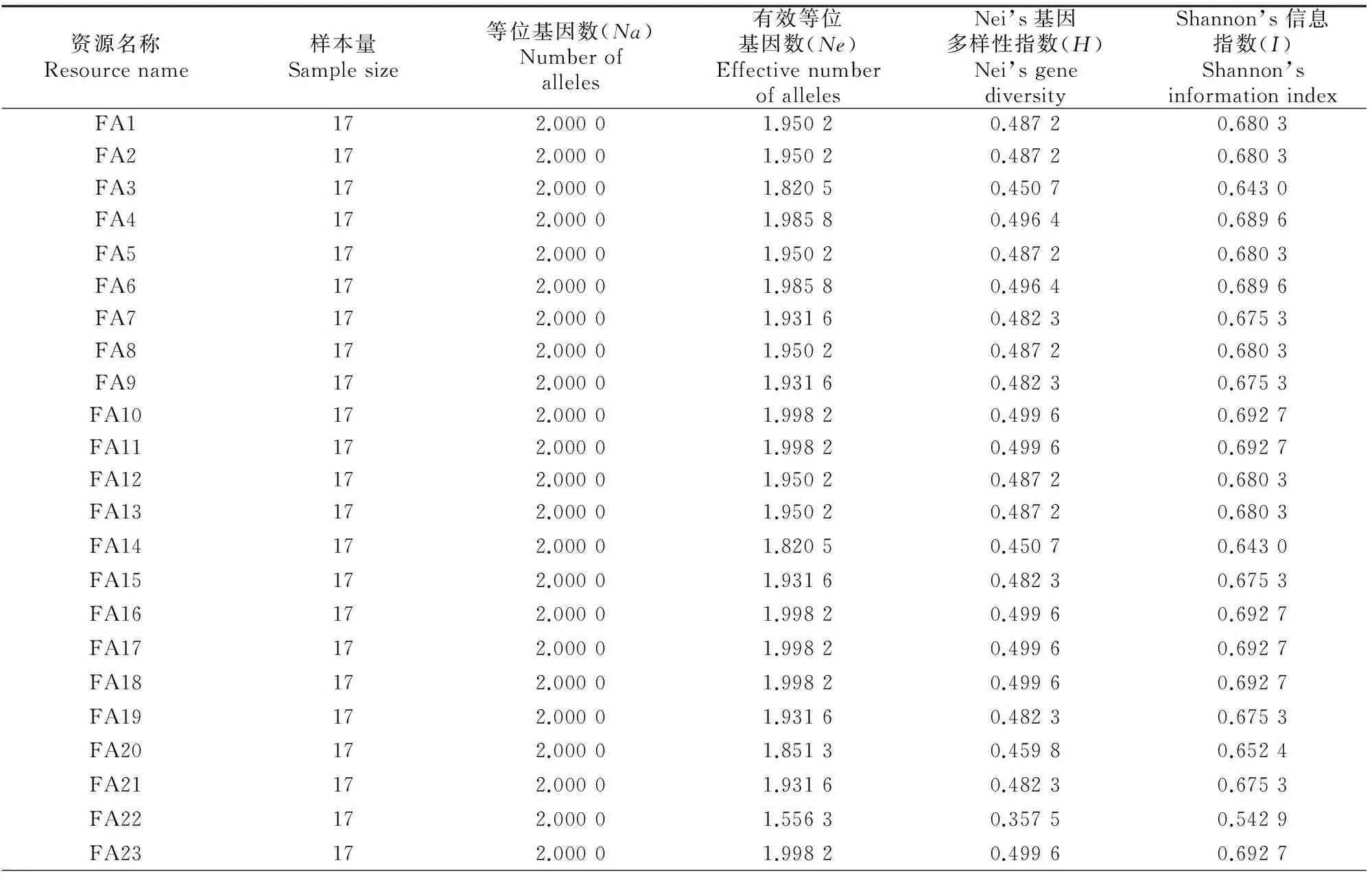
表3 供试43份菊芋资源的Nei’s基因多样性指数和Shannon’s信息指数Table 3 Nei’s gene diversity index and Shannon’s index of the tested 43 Jerusalem artichoke resources
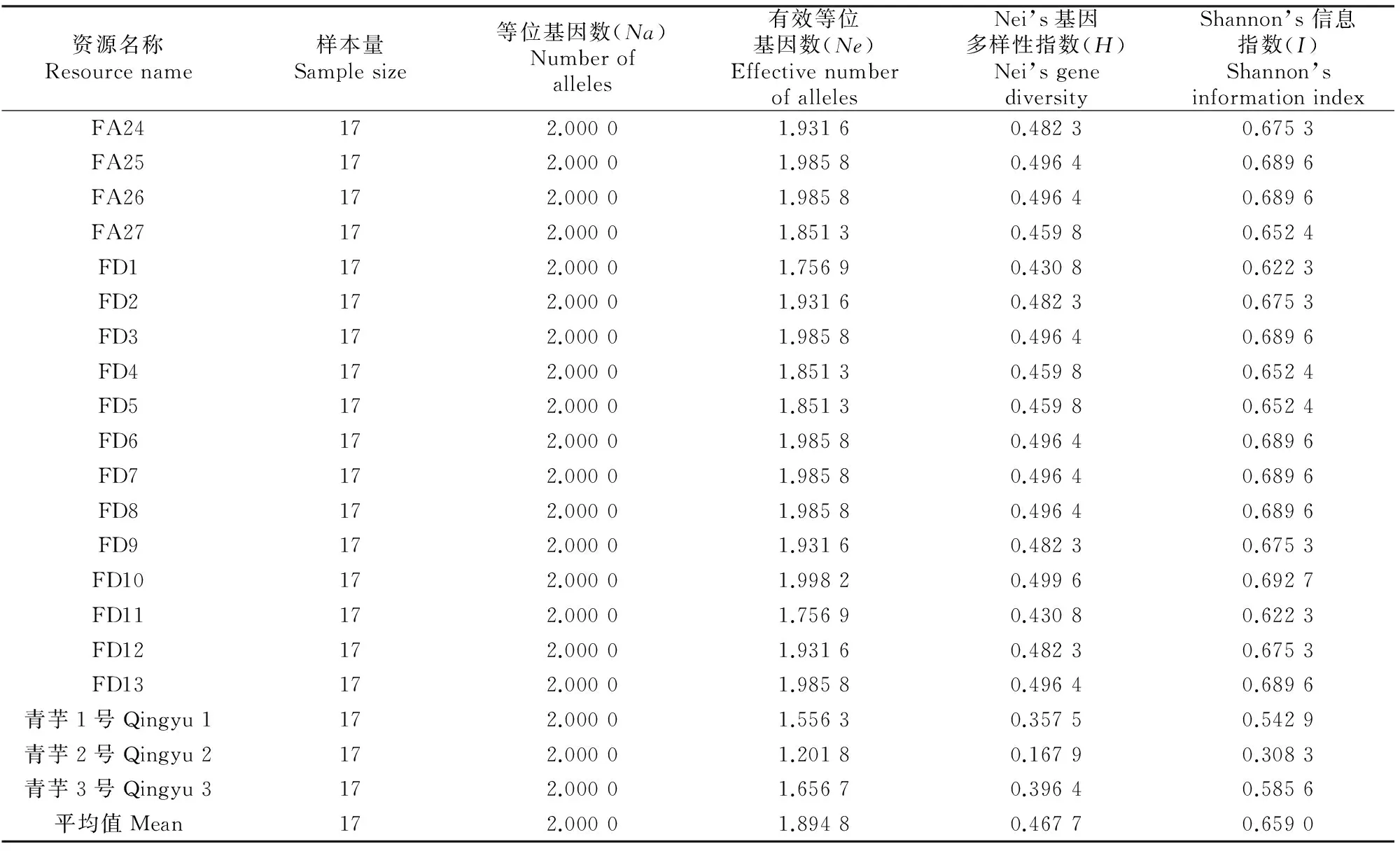
续表3 Continued table 3
2.2 基于相似系数的供试菊芋资源ISSR聚类分析
以供试的43份菊芋资源扩增出的203个位点数据为原始矩阵,共获得903个两两不同的遗传相似系数,其中FA25与FA26的相似系数最大,为0.955,说明这2个菊芋资源亲缘关系很近;FA2与青芋2号的相似系数最小,为0.443,说明二者亲缘关系较远。从聚类图(图2)可以看出,以0.64为阈值可以将供试菊芋资源分为2大类群。第Ⅰ类群:40份资源,均为国外资源(占所有资源的93.02%),对其进一步分析,又可将第Ⅰ大类在阈值为0.784处分为7个亚类。第1亚类有30个资源,占第Ⅰ类群的75.0%;第2亚类有3个资源,分别是FA4、FA6和FA12,占第Ⅰ类群的7.5%;第3亚类有2个菊芋资源,分别是FA15和FA20,占第Ⅰ类群的5.0%;第4亚类有2个菊芋资源,分别是FD2和FD5,占第Ⅰ类群的的5.0%;第5亚类有1个菊芋资源,即FA5,占第Ⅰ类群的2.5%;第6亚类有1个菊芋资源,即FA7,占第Ⅰ类群的2.5%;第7亚类有1个菊芋资源,即FA9,占第Ⅰ类群的2.5%。第Ⅱ类群有3个菊芋品种,均为国内审定品种,包括青芋1号、2号、3号3个菊芋品种。
由以上分类可知,国外40份菊芋资源与国内3个菊芋品种之间的亲缘关系较远。同时,聚类图显示同一地区的种质资源优先聚在一起,如来自中国的青芋1号、2号、3号聚为一类,体现出一定的地域性。
2.3 不同类群菊芋种质资源的形态学主要特点
在实际生产中,通常是通过观察对比菊芋全生育期内的植物学性状,如株高、茎粗、叶形、块茎形状、块茎颜色等来区分不同的菊芋资源,但最常用的最直观的方法是通过块茎的形状及颜色来进行区分。因此本研究主要从块茎形状及颜色对菊芋种质资源进行分析。
第1亚类30份菊芋种质资源中块茎形状主要有纺锤形、球形、瘤状3种,其中纺锤形的包括FA1、FA23、FD3、FD6、FD9、FD10、FD13 等7种,球形的包括FA2、FA3、FA8、FA11、FA14、FA17、FA18、FA19、FA25、FA26、FD1、FD4等12种,瘤状的包括FA10、FA13、FA16、FA21、FA22、FA24、FA27、FD7、FD8、FD11、FD12等 11种;块茎颜色主要有白色、红色、紫色、褐色、白褐色5种,其中白色的包括FA1、FA3、FA8、FA10、FA13、FA16、FA17、FA18、FA21、FA22、FA23、FA24、FA25、FA26、FA27、FD4、FD7、FD8、FD12等19种,红色的包括FA2、FA11、FA14、FD1 等4种,紫色的包括FD3、FD6 2种,褐色的包括FD13 1种,白褐色的包括FA19、FD9、FD10、FD11等 4种。第2亚类3份菊芋资源块茎形状:FA4与FA12为纺锤形,FA6为球形;块茎颜色:FA4为紫红色,FA6为白褐色,FA12为红色。第3亚类的2份菊芋资源块茎形状:FA15为纺锤形,FA20为球形;块茎颜色:FA15为白褐色,FA20为红色。第4亚类中的2份菊芋资源块茎形状均为瘤状,块茎颜色均为白色。第5亚类中FA5块茎为纺锤形,颜色为白色。第6亚类中FA7块茎为棒状,颜色为红色。第7亚类中FA9块茎为纺锤形,颜色为白褐色。

图2 基于相似系数的43份菊芋资源的UPGMA聚类结果
Fig.2 Similarity coefficient based UPGMA clustering of the 43HelianthustuberosusL.resources
3 讨 论
3.1 国外菊芋资源遗传多样性分析
丰富的菊芋种质资源是菊芋育种的基础,而探明亲本材料的遗传多样性程度和亲缘关系的远近是育种的关键[16]。目前,ISSR标记技术已被广泛应用到种质遗传多样性和遗传差异检测的研究中[17-18]。本试验采用ISSR分子标记技术研究了国外40份菊芋资源与国内3个菊芋品种之间的遗传多样性及亲缘关系。结果表明,供试的43份菊芋资源平均有效等位基因数为1.894 8,平均Nei’s基因多样性指数为0.467 7,平均Shannon’s 信息指数为0.659 0;43份菊芋资源间的相似系数为0.443~0.955,说明ISSR标记技术完全能够应用于菊芋资源遗传多样性分析研究。
因此,根据本研究的结果,在今后菊芋育种过程中可以利用遗传距离相对较远的资源进行杂交,创制新的菊芋种质。此外本研究还发现,来源相同的菊芋资源如FA5、FA7和FA9单独聚为一类,下一步可以通过增加ISSR引物、优化试验条件来深入探讨这些菊芋资源间的遗传多样性[19]。
3.2 菊芋遗传距离与地理距离的相关性
国外40份菊芋资源与国内的3个菊芋资源间的遗传距离较国内品种间的遗传距离大,具有较高的遗传多样性,其中部分资源可以作为新的育种材料加以利用。另外,各供试材料在种质遗传距离与来源地的关系上表现出了很强的相关性,聚类结果显示,国内与国外菊芋种质资源未聚为一类,说明国外菊芋资源与国内菊芋品种亲缘关系较远。
3.3 菊芋聚类结果与形态学分类结果的差异
对40份国外菊芋资源的聚类分析结果与根据形态性状进行分类的结果部分吻合,说明二者具有一定的相关性[20]。供试的一部分菊芋资源从形态学上分类不属于一类,但是在基于ISSR 标记的聚类分析中被划分到同一类中,例如第Ⅰ大类中的第2亚类FA6与FA12,二者块茎形状一致但颜色不同,在基于ISSR标记分析的聚类树状图中二者亲缘关系较近并聚在一起,在第1亚类中同样有类似的情况出现。这一方面说明传统的形态分类方法不能完全反映出菊芋资源间的亲缘关系,必须结合分子标记等其他生物学方法进行完善;另一方面说明可以适当增加ISSR引物做进一步的筛选分析。
本研究首次利用ISSR分子标记技术对来自国外的菊芋群体资源与国内审定的3个菊芋品种进行了亲缘关系及遗传多样性分析,结果表明,国内与国外菊芋资源间遗传关系较远,且国外资源间存在丰富的遗传多样性。因此,在今后的育种工作中可以依据遗传多样性分析和聚类结果进行亲本选择,避免人力物力资源的浪费,为后续菊芋资源的可持续性开发利用提供科学依据。
[1] 刘祖昕,谢光辉.菊芋作为能源植物的研究进展 [J].中国农业大学学报,2012,17(6):122-132.
Liu Z X,Xie G H.An overview of researches on Jerusalem artichoke as a biofuel crop [J].Journal of China Agricultural University,2012,17(6):122-132.(in Chinese)
[2] 钟启文,王丽慧,李 屹,等.全生育期菊芋不同器官果聚糖积累分配特征研究 [J].北方园艺,2012(17):1-4.
Zhong Q W,Wang L H,Li Y,et al.Jerusalem artichoke fructan accumulation and distribution characteristics in different organs of the whole growth [J].Northern Horticulture,2012(17):1-4.(in Chinese)
[3] 薛崇昀,贺文明,张睿玲.菊芋秆资源在造纸工业的利用 [J].中华纸业,2007,28(2):67-70.
Xue C Y,He W M,Zhang R L.The utilization of Jerusalem artichoke stalk resources in paper industry [J].China Pulp & Paper Industry,2007,28(2):67-70.(in Chinese)
[4] 孔 涛,张 楠,林凤梅,等.不同品种菊芋块茎及茎叶营养成分分析比较 [J].广东农业科学,2013(6):108-109,113.
Kong T,Zhang N,Lin F M,et al.Analysis and comparison of stem leaf and tuber nutritional composition in different Jerusalem artichoke [J].Guangdong Agricultural Sciences,2013 (6):108-109,113.(in Chinese)
[5] 李 莉,马本元,侯全刚.青芋1号菊芋 [J].中国蔬菜,2004(4):59.
Li L,Ma B Y,Hou Q G.The Jerusalem artichoke of Qingyu 1 [J].China Vegetables,2004(4):59.(in Chinese)
[6] 侯全刚,马本元,李 莉,等.加工专用型菊芋青芋2号 [J].中国蔬菜,2006(2):56.
Hou Q G,Ma B Y,Li L,et al.Processing special Jerusalem artichoke Qingyu 2 [J].China Vegetables,2006(2):56.(in Chinese)
[7] 李 屹,孙雪梅,钟启文,等.加工型菊芋新品种青芋3号的选育 [J].中国蔬菜,2011(10):100-102.
Li Y,Sun X M,Zhong Q W,et al.A new processing variety of Jerusalem artichoke-‘Qingyu No.3’ [J].China Vegetables,2011(10):100-102.(in Chinese)
[8] 林 立,胡仲义,李纪元,等.10 个山茶岛屿天然居群的遗传多样性分析 [J].园艺学报,2012,39(8):1531-1538.
Lin L,Hu Z Y,Li J Y,et al.Analysis on genetic diversity of ten insular populations ofCamelliajaponica[J].Acta Horticulturae Sinica,2012,39(8):1531-1538.(in Chinese)
[9] 李 明,王树香,高宝嘉.不同群落类型油松居群的遗传多样性 [J].应用与环境生物学报,2013,19(3):421-425.
Li M,Wang S X,Gao B J.Genetic diversity ofPinustabulaeformisCarr.populations in different forest types [J].Chin J Appl Environ Biol,2013,19(3):421-425.(in Chinese)
[10] 葛亚英,张 飞,王炜勇,等.丽穗凤梨杂交后代ISSR鉴定 [J].分子植物育种,2013,11(1):85-89.
Ge Y Y,Zhang F,Wang W Y,et al.Identification ofVrieseahybridsby ISSR. [J].Molecular Plant Breeding,2013,11(1):85-89.(in Chinese)
[11] 管 洁,焦雪辉,吴锦娣,等.用ISSR分子标记鉴定亚洲百合杂种F1代 [J].分子植物育种,2013,11(3):415-420.
Guan J,Jiao X H,Wu J D,et al.Identification of Asian lily hybrid F1 by Using ISSR [J].Molecular Plant Breeding,2013,11(3):415-420.(in Chinese)
[12] 程燕林,梁 军,吕 全,等.葡萄座腔菌科研究进展:鉴定,系统发育学和分子生态学 [J].生态学报,2011,31(11):3197-3207.
Cheng Y L,Liang J,Lü Q,et al.Advances in Botryosphaeriaceae:Identification,phylogeny and molecular ecology [J].Acta Ecologica Sinica,2011,31(11):3197-3207.(in Chinese)
[13] 赵孟良,韩 睿,李 莉.菊芋ISSR-PCR反应体系的建立与优化 [J].西南农业学报,2012,25(1):243-246.
Zhao M L,Han R,Li L.Establishment and optimization of ISSR-PCR reaction system forHelianthustuberosusL. [J].Southwest China Journal of Agricultural Sciences,2012,25(1):243-246.(in Chinese)
[14] 韩 睿,赵孟良,李 莉.3个菊芋品种的ISSR引物筛选及分子鉴别 [J].西南农业学报,2013,26(1):290-293.
Han R,Zhao M L,Li L.Primers screening and identification of three samples inHelianthustuberosusL.by ISSR molecular marker [J].Southwest China Journal of Agricultural Sciences,2013,26(1):290-293.(in Chinese)[15] 赵孟良,韩 睿,马胜超,等.菊芋基因组DNA提取方法的比较 [J].中国蔬菜,2012(14):41-44.
Zhao M L,Han R,Ma S C,et al.Comparison of genomic DNA extraction fromHelianthustuberosusL. [J].China Vegetables,2012(14):41-44.(in Chinese)
[16] 陈 英,邱 琳,涂升斌,等.用SSR标记检测杂交籼稻三系亲本的遗传差异 [J].应用与环境生物学报,2009,15(5):585-590.
Chen Y,Qiu L,Tu S B,et al.Genetic variation of three-line parents of indica hybrid rice detected with SSR markers [J].Chin J Appl Environ Biol,2009,15(5):585-590.(in Chinese)
[17] 魏小玲,曹福祥,陈 建.海南木莲遗传多样性的ISSR及亲缘关系的分析 [J].生物技术通报,2013(8):74-77.
Wei X L,Cao F X,Chen J.Studies on the genetic fiversity and relationship ofManglietiahainanensisdandy by ISSR [J].Biotechnology Bulletin,2013(8):74-77.(in Chinese)
[18] 王 波,祁丽萍,鲜 灵,等.金针菇单核原生质体菌株遗传差异的ISSR分析 [J].西南农业学报,2013,26(3):1126-1131.
Wang B,Qi L P,Xian L,et al.ISSR analysis of genetic diversity of protoplasted mononuclear strains ofFlammulinavelutipes. [J].Southwest China Journal of Agricultural Sciences,2013,26(3):1126-1131.(in Chinese)
[19] 石颜通,周 波,张秀新,等.牡丹89个不同种源品种遗传多样性和亲缘关系分析 [J].园艺学报,2012,39(12):2499-2506.
Shi Y T,Zhou B,Zhang X X,et al.Assessment of genetic diversity and relationship of 89 tree peony cultivars from different provenances [J].Acta Horticulturae Sinica,2012,39(12):2499-2506.(in Chinese)
[20] 赵孟良,韩 睿,李 莉.24个菊芋品种(系)遗传多样性的ISSR标记分析 [J].植物资源与环境学报,2013,22(4):44-49.
Zhao M L,Han R,Li L.ISSR marker analysis on genetic diversity of twenty-four cultivars (lines) ofHelianthustuberosus[J].Journal of Plant Resources and Environment,2013,22(4):44-49.(in Chinese)
ISSR based genetic diversity of 43HelianthustuberosusL.
ZHAO Meng-liang,SUN Xue-mei,WANG Li-hui,LI Li
(ResearchandDevelopmentCenterofJerusalemArtichoke,QinghaiKeyLaboratoryofVegetableGeneticsandPhysiology,QinghaiAcademyofAgricultureandForestry,Xining,Qinghai810016,China)
【Objective】 This study aimed to clarify genetic relationship and genetic diversity of Jerusalem artichoke resources at home and abroad. 【Method】 A total of 40 foreignHelianthustuberosusL. resources and 3 domestic varieties were used as experimental material,and 14 primers were adopted for ISSR molecular marker analysis to analyze their genetic diversity.POPGENE32 software was used to calculate ratio of polymorphic loci (PPB),effective number of alleles (Ne),Nei’s genetic diversity index (H),and Shannon’s information index (I) while NTSYS 2.10 software was used to calculate similarity coefficient between varieties,conduct clustering analysis,and analyze the morphological characteristics of different groups.【Result】 A total of 203 marks were obtained when using 14 primers to amplify the 43 Jerusalem artichoke resources by PCR amplification.199 polymorphism markers were polymorphic markers with the polymorphism rate of 98.0%.POPGENE32 software analysis showed that averaged effective number of alleles,Nei’s genetic diversity index,and Shannon’s information index,of the 43HelianthustuberosusL.resources were 1.894 8,0.467 7,and 0.659 0,respectively,while NTSYS showed that and the similarity coefficient was 0.443 to 0.955.Clustering analysis showed that 43HelianthustuberosusL.【Conclusion】 The genetic relationship ofHelianthustuberosusL.resources between domestic and abroad was far,and the foreign resources had abundant genetic diversity.
HelianthustuberosusL.;germplasm resources;ISSR;genetic diversity
时间:2015-08-05 08:56
10.13207/j.cnki.jnwafu.2015.09.021
2014-03-05
农业部“948”项目(2013-Z72);青海省科技厅基础研究项目(2012-H-809)
赵孟良(1986-),男,河南商丘人,硕士,助理研究员,主要从事菊芋遗传育种及生理生化研究。 E-mail:zhaomengliang@qhu.edn.cn
李 莉(1959-),女,江苏丰县人,研究员,硕士生导师,主要从事蔬菜遗传育种及栽培生理研究。 E-mail:yyslili@163.com
S330
A
1671-9387(2015)09-0150-07
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20150805.0856.042.html
