miR-520a靶基因PIK3CA结合位点多态性与中国汉族人群大肠癌易感性的关联研究
2015-01-04丁丽芳姜藻陈巧云秦蓉方悦李皓
丁丽芳,姜藻,陈巧云,秦蓉,方悦,李皓
1.东南大学附属中大医院肿瘤科,江苏 南京 210009;
2.江苏省丹阳市人民医院肿瘤科,江苏 丹阳 213000;
3.江苏大学附属人民医院中心实验室,江苏 镇江 212002;
4.江苏大学附属人民医院肿瘤科,江苏 镇江 212002;
5.泰兴人民医院检验科,江苏 泰兴 225400
miR-520a靶基因PIK3CA结合位点多态性与中国汉族人群大肠癌易感性的关联研究
丁丽芳1,2,姜藻1,陈巧云3,秦蓉4,方悦3,李皓5
1.东南大学附属中大医院肿瘤科,江苏 南京 210009;
2.江苏省丹阳市人民医院肿瘤科,江苏 丹阳 213000;
3.江苏大学附属人民医院中心实验室,江苏 镇江 212002;
4.江苏大学附属人民医院肿瘤科,江苏 镇江 212002;
5.泰兴人民医院检验科,江苏 泰兴 225400
背景与目的:越来越多的研究表明,微小RNA(microRNA,miRNA,miR)靶基因位点的多态性可能会影响miRNA的转录或生成能力,影响个体对肿瘤的易感性。本研究旨在探讨大肠癌患者miR-520a靶基因PIK3CA 3’端单核苷酸多态性(single nucleotide polymorphism,SNP)位点rs141178472与中国汉族人群大肠癌易感性之间的关系。方法:选取386例大肠癌标本作为实验组,394名年龄及性别比例匹配的非肿瘤个体作为对照组。病例对照研究检测rs141178472的分布频率。统计分析大肠癌易感性与多态性之间的关系。结果:携有rs141178472 CC基因型或C等位基因显著增加了大肠癌的发病风险(CC vs TT,OR=1.716,95%CI:1.084~2.716,P=0.022;C vs T,OR=1.258,95%CI:1.021~1.551,P=0.033)。通过检测大肠癌患者外周血单个核细胞PIK3CA的表达发现,PIK3CA mRNA的表达与SNP基因型rs141178472具有关联性。结论:miR-520a与PIK3CA 3’UTR的SNP位点rs141178472相互作用可能与大肠癌易感性有关。
大肠癌;PIK3CA基因;单核苷酸多态性
大肠癌是最常见的消化道恶性肿瘤之一,其发病率和病死率在世界范围内都位于胃肠道肿瘤的前列[1]。近几十年,国内对于大肠癌发生、发展的研究越来越多[2]。大肠癌的发生 是由环境因素和遗传易感性等多因素共同作用所导致[3-4]。大肠癌的病因尚未明确。尽管环境因素被认为是大肠癌的主要风险因素,然而在面临同样风险因素的个体中,只有极少数最终罹患大肠癌,这意味着大肠癌的发生还与其他因素有关。近几年大量的研究表明,基因多态性所调控的肿瘤易感性在人类癌症的发生中起重要作用[5-6]。
微小RNA(microRNA,miRNA,miR)是一组内源性长约21~23个核苷酸大小的非编码RNA分子,在抑制转录后基因表达和促进靶miRNA降解的过程中起重要的负性调节作用[7]。编码miRNA的基因及多数蛋白编码基因的3’UTR也存在大量的单核苷酸多态性(single nucleotide polymorphism,SNP)位点,这些SNP位点可能会影响miRNA的转录加工或影响miRNA与靶基因3’UTR结合能力的变化甚至是结合位点的改变,从而导致所调控的靶基因表达或功能改变,影响个体对肿瘤的易感性。PIK3CA是Volinia等[8]于1994年发现的一种癌基因,定位于3q2613。研究发现,约1/3的人类实体肿瘤中存在PIK3CA基因突变,尤其在大肠癌中,约32%患者存在PIK3CA基因突变[9]。生物信息学分析SNP rs141178472位点位于PIK3CA基因的3’UTR区,存在于miR-520a潜在结合区,提示PIK3CA基因表达可能受到miR-520a调控。目前,关于此区域SNP与肿瘤的关系少见报道。仅Arcaroli等[10]发现,一个新的PIK3CA基因3’UTR区SNP(19T>C)与塞卡替尼的药物敏感性相关。此SNP位于miR-520a和miR-525结合位点。然而,尚未有研究证实这种SNP与miR-520a之间的相互作用是否与患者对大肠癌的易感性有关。
综上所述,我们提出假设,PIK3CA 3’UTR的基因多态性可能与个体对大肠癌的肿瘤易感性有关。因此,在本研究中,我们采用病例对照试验来探讨大肠癌患者miR-520a及其靶基因PIK3CA 3’端SNP位点rs141178472与中国汉族人群大肠癌易感性之间的关系。
1 材料和方法
1.1 实验对象
研究人群由386例大肠癌病例(年龄23~72岁)和394名健康对照人群(年龄21~73岁)组成。大肠癌病例来源于丹阳市人民医院和镇江市第一人民医院的住院患者,均经过病理组织学检查确诊;健康对照人群来源于丹阳市人民医院和镇江市第一人民医院体检中心的正常人群,与患者组性别和年龄的差异无统计学意义(P>0.05)。所有受检者相互间无血缘关系,对照组人群无家族遗传史和肿瘤史。所有受检对象均知情同意,研究经医院伦理委员会通过。
1.2 基因分型
按照人全血基因组提取试剂盒说明书提取人全血中有核细胞基因组DNA,以大肠癌病例和健康对照人群的血基因组DNA作为模板进行PCR扩增。根据Gene Bank检索的序列设计E rs141178472野生型及突变型的特异性检测引物,引物序列见表1。PCR反应条件如下:95 ℃3 min,95 ℃ 30 s,58 ℃ 45 s,72 ℃ 30 s,共35个循环;72 ℃ 10 min。产物经1.2%琼脂糖凝胶电泳鉴定扩增效果,野生型引物管出现272 bp条带,基因型为CC;突变型引物管出现272 bp条带,基因型为TT;两管均出现272 bp条带,基因型为CT。所有PCR产物均送上海生工生物技术有限公司测序。

表1 引物序列Tab. 1 Primer sequences
1.3PIK3CA3’-UTR荧光素酶报告基因质粒的构建与转染
引物由广州锐博生物科技有限公司合成,其序列如下:PIK3CA 3’UTR-F:5’-TCATGGTGGCTGGACAACAA-3’;PIK3CA 3’UTR-R:5’-TCCAAAGCTTTACT GGTGTGAGCCACTGTG-3’。将合成的引物进行PCR扩增,按照凝胶纯化试剂盒说明书进行纯化产物,酶切后回收纯化酶切产物,将目的基因及载体进行连接反应后得到连接产物。将连接产物转化入感受态细胞中,扩增、提取质粒酶切鉴定,并对质粒进行扩增和提取。293T细胞进行常规传代培养后铺种于24孔板中,24 h后细胞密度达50%~80%,细胞贴壁伸展良好,即可用于后续转染操作。按照转染说明书加入miR-520a mimics质粒LipofectamineTM2000混合液,培养24 h后,以Promega公司双荧光报告系统检测试剂盒检测荧光素酶活性。
1.4 实时荧光定量聚合酶链反应(quantitative real-time polymerase chain reaction,qRTPCR)检测PIK3CA mRNA表达水平
按照人外周血基因组提取试剂盒说明书提取人外周血单核细胞总DNA。采用Takara的荧光定量逆转录试剂盒进行检测。qRT-PCR引物序列如下:5’-GGAGCCTGGAAGAGCCC-3’(F);5’-CGTGGAGGCATTGTTCTGAT-3’(R)。qRTPCR检测方法依照参考文献[11]所述。
1.5 统计学处理
所有资料运用SPSS 12.0软件进行统计分析。对照组SNP rs141178472位点的Hardy-Wenbger平衡检验利用χ2检验进行分析。采用χ2检验或确切概率法分析病例组和对照组rs141178472位点的分布差异与大肠癌风险之间的关系,用OR及95%CI表示相对危险度。P<0.05为差异有统计学意义。
2 结 果
2.1 研究对象的一般特征
本研究共纳入386例大肠癌病例和394名健康对照人群,人口统计学特征及一些已知的大肠癌危险因素等在病例组和对照组间分布见表2。病例组和对照组间的年龄分布均衡可比(P=0.506),两组吸烟史人群分布差异也无统计学意义(P=0.536)。但病例组和对照组在肿瘤家族史分布上存在差异,大肠癌病例组有肿瘤家族史的比例明显高于健康对照组(P=0.023)。在386例病例中,212例(54.9%)患有结肠癌,其余174例(45.1%)患有直肠癌。另外,在临床分期上,大部分病例为Ⅱ期 (44.8%)和Ⅲ期(35.0%)。
2.2 病例组与对照组中rs141178472基因型分布频率的比较
rs141178472基因型AS-PCR分型结果见图1。rs141178472基因型在健康对照组中的分布均符合Hardy-Weinberg平衡规律(χ2=0.139,P=0.709)。rs141178472多态性位点与肠癌易感性的基因型分布情况见表3。在rs141178472 C>T基因型中,大肠癌病例组基因型分布频率明显高于对照组(C:37.3% vs 32.1%;T:62.7% vs 67.9%)。与携带rs141178472野生纯合子(TT)的个体相比,携带rs141178472突变纯合子(CC)的个体患大肠癌的风险明显上升(OR=1.716,95%CI:1.084~2.716,P=0.022)。此外,相对于携带至少1个T等位基因者,携带至少1个C等位基因者的人群更易患结肠癌(OR=1.258,95%CI:1.021~1.551,P=0.033)。
2.3 SNP rs141178472影响miR-520a对PIK3CA表达的调控
通过生物信息学分析发现,S N P rs141178472位于PIK3CA潜在的miR-520a结合区,T等位基因可与PIK3CA 3’UTR互补配对形成一个新的miR-520a结合位点(图2A)。因此,我们假设miR-520a可以调控PIK3CA基因的表达,而SNP rs141178472通过增加T等位基因可以影响此调控过程。为了验证SNP rs141178472是否影响PIK3CA的表达,我们构建了rs141178472 T或C基因型的荧光素酶报告基因载体,然后分别将这两种质粒与miR-520a mimics共转染到293T细胞中,结果发现与rs141178472 C基因型相比,T基因型荧光素酶活性明显下降(图2B)。此外,我们检测了大肠癌患者外周血单个核细胞PIK3CA mRNA的表达水平。qRT-PCR结果显示,与TT基因型相比,CC基因型的大肠癌患者PIK3CA mRNA的表达明显增加(图2C)。

表2 研究对象的一般情况在病例组和对照组中的分布Tab. 2 General characteristics of colorectal cancer case group and control group
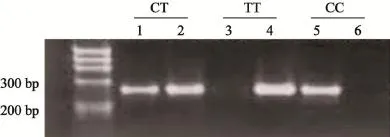
图1 rs141178472基因型AS-PCR分型结果Fig. 1 The results of AS-PCR assay for rs141178472

表3 rs141178472与大肠癌易感性的关系Tab. 3 The association between rs141178472 and colorectal cancer risk

图2 SNP rs141178472影响miR-520a对PIK3CA表达的调控Fig. 2 Effect of the PIK3CA 3’-UTR Polymorphism rs141178472 on PIK3CA expression
3 讨 论
在本研究中,我们发现PIK3CA 3’UTR的SNP位点rs141178472与大肠癌易感性密切相关。此外,rs141178472可调控PIK3CA的表达。因此,我们认为miR-520a与PIK3CA的交互作用可能在大肠癌的发生、发展中扮演重要的角色。
越来越多的研究证实,miRNA可通过靶向调控肿瘤相关基因的表达从而促进或抑制肿瘤的发生和增殖。成熟的miRNA通过与其靶基因的3’UTR结合,降解靶基因的mRNA或抑制其翻译,从而调控基因的表达[12-13]。由于miRNA依赖碱基互补配对与靶基因的3’UTR结合,因此,3’UTR的SNP或seed区可能通过影响miRNA与mRNA的结合从而引起靶基因表达水平的变化[14]。TNFAIP2的miR-184结合区多态性位点(rs8126 T>C)可以通过调控TNFAIP2的表达增加患者对颈部鳞状细胞癌的患病风险率[15]。Hikami等[16]研究发现SPI1的一个3’UTR区SNP位点能够增加SPI1的mRNA水平和对SLE的易感性。一系列在卵巢癌和乳腺癌中的研究也发现了类似的结果[17-18]。本研究选取了780例中国汉族研究对象,证实PIK3CA SNP位点rs141178472与大肠癌易感性有关。rs141178472可能是一个具有汉族人群大肠癌相关性的SNP位点。另外,通过对大肠癌患者外周血单个核细胞的检测,PIK3CA mRNA的表达水平与SNP位点 rs141178472密切相关。
磷脂酰肌醇激酶-3家族是一类调节信号通路的酯酶,具有重要生物学活性,与调解肿瘤细胞的存活、增殖、黏附和迁移等密切相关。PIK3CA是PI3Ks家族的关键成员之一,编码ⅠA型磷脂酰肌醇-3-激酶(P13K)的p110催化亚基,致癌性的PIK3CA突变可通过激活PI3K通路参与恶性肿瘤的发生、发展[19-20]。PI3K通过调控下游PI3K/AKT通路发挥其生物学效应,体外研究证实,血管内皮生长因子(vascular endothelial growth factor,VEGF)、表皮生长因子(epidermal growth factor,EGF)、分类血小板衍生因子(platelet derived growth factor,PDGF)等可通过调控PI3K通路从而促进肿瘤细胞增殖[21]。PTEN是一种分布广泛的抑癌基因,能够负向调控PI3K通路的活性[22]。PIK3CA基因突变与PTEN蛋白缺失呈负相关,通过诱导PI3K通路的异常激活进而促进肿瘤的发生和增殖[23]。研究发现,PIK3CA在包括胃癌、肠癌、脑癌、乳腺癌、肝癌、卵巢癌和肺癌等恶性肿瘤组织中均有异常的过度表达[24-25]。PIK3CA突变在多个外显子中均有发现,但主要存在于两个热点突变区:一个是位于9号外显子的E542K和E545K突变,另一个是位于20号外显子的H1047R突变。其中,外显子9突变率为7%,外显子20突变率为32%,这两个突变热点都能增强PI3K的脂质激酶活性。本研究中,我们发现PIK3CA 3’UTR的miRNA结合区rs141178472与大肠癌的易感性相关,并且在大肠癌的发生、发展过程中可能起着重要的作用。
综上所述,本研究发现miR-520a与PIK3CA的相互作用与大肠癌的发生、发展可能存在相关性,PIK3CA的SNP位点rs141178472可能通过影响miR-520A的调节作用,从而影响大肠癌的患病风险。然而,由于本研究样本量较小,对miR-520a/PIK3CA通路在结肠癌发生、发展过程中的具体作用需要进一步大样本的研究。
[1] JEMAL A, TIWAFI R, MURRY T, et al. Global cancer statistics[J]. CA Cancer J Clin, 2011, 61(2): 69-90.
[2] ZHAO P, DAI M, CHEN W, et al. Cancer trends in China[J]. Jpn J Clin Oncol, 2010, 40(4): 281-285.
[3] ARENDS M J. Pathways of colorectal carcinogenesis[J]. Appl Immunohistochem Mol Morphol, 2013, 21(2): 97-102.
[4] TANNAPFEL A, NEID M, AUST D. The origins of colorectal carcinoma: specific nomenclature for different pathways and precursor lesions[J]. Dtsch Arztebl Int, 2010, 107(43): 760-766.
[5] MA Z B, LI K, WANG J, et al. Role of KAI1/CD82 polymorphisms in colon cancer risk in Han Chinese population[J]. Med Oncol, 2013, 30(3): 668.
[6] HU J J, SMITH T R, MILLER M S, et al. Symposium overview: genetic polymorphisms in DNA repair and cancer risk[J]. Toxicol Appl Pharmacol, 2002, 185(1): 64-73.
[7] SHI T Y, CHEN X J, ZHU M L, et al. A pri-miR-218 variant and risk of cervical carcinoma in Chinese women[J]. BMC Cancer, 2013, 13: 19.
[8] VOLINIA S, HILES I, ORMONDROYD E, et al. Molecular cloning, cDNA sequence, and chromosomal localization of the human phosphatidylinositol 3-kinase p110 alpha (PIK3CA) gene[J]. Genomics, 1994, 24(3): 472-477.
[9] CATHOMAS G. PIK3CA in Colorectal Cancer[J]. Front Oncol, 2014, 4: 35.
[10] ARCAROLI J J, QUACKENBUSH K S, POWELL R W, et al. Common PIK3CA mutants and a novel 3’UTR mutation are associated with increased sensitivity to saracatinib[J]. Clin Cancer Res, 2012, 18(9): 2704-2714.
[11] GARG R, KAPOOR V, MITTAL M, et al. Abnormal expression of PI3K isoforms in patients with tobacco-related oral squamous cell carcinoma[J]. Clin Chim Acta, 2013, 416: 100-106.
[12] CHO W C. MicroRNAs in cancer-from research to therapy[J]. Biochim Biophys Acta, 2010, 1805(2): 209-217.
[13] CHO W C. MicroRNAs: potential biomarkers for cancer diagnosis, prognosis and targets for therapy[J]. Int J Biochem Cell Biol, 2010, 42(8): 1273-1281.
[14] WANG Y, LONG L, LI T, et al. Polymorphisms of microRNA-binding sites in integrin genes are associated with oral squamous cell carcinoma susceptibility and progression[J]. Tohoku J Exp Med, 2014, 233(1): 33-41.
[15] LIU Z, WEI S, MA H, et al. A functional variant at the miR-184 binding site in TNFAIP2 and risk of squamous cell carcinoma of the head and neck[J]. Carcinogenesis, 2011, 32(11): 1668-1674.
[16] HIKAMI K, KAWASAKI A, ITO I, et al. Association of a functional polymorphism in the 3’-untranslated region of SPI1 with systemic lupus erythematosus[J]. Arthritis Rheum, 2011, 63(3): 755-763.
[17] WYNENDAELE J, BÖHNKE A, LEUCCI E, et al. An illegitimate microRNA target site within the 3’UTR of MDM4 affects ovarian cancer progression and chemosensitivity[J]. Cancer Res, 2010, 70(23): 9641-9649.
[18] ZHANG L, LIU Y, SONG F, et al. Functional SNP in the microRNA-367 binding site in the 3’UTR of the calcium channel ryanodine receptor gene 3 (RYR3) affects breast cancer risk and calcification[J]. Proc Natl Acad Sci U S A, 2011, 108(33): 13653-13658.
[19] MILED N, YAN Y, HON W C, et al. Mechanism of two classes of cancer mutations in the phosphoinositide 3-kinase catalytic subunit[J]. Science, 2007, 317(5835): 239-242.
[20] BADER A G, KANG S, VOGT P K. Cancer-specific mutations in PIK3CA are oncogenic in vivo[J]. Proc Natl Acad Sci U S A, 2006, 103(5): 1475-1479.
[21] ROSTY C, YOUNG J P, WALSH M D, et al. PIK3CA activating mutation in colorectal carcinoma: associations with molecular features and survival[J]. PLoS One, 2013, 8(6): e65479.
[22] FURNARI F B, HUANG H J, CAVENEE W K. The phosphoinositol phosphatase activity of PTEN mediates a serum-sensitive G1 growth arrest in glioma cells[J]. Cancer Res, 1998, 58(22): 5002-5008.
[23] JABBOUR E, OTTMANN O G, DEININGER M, et al. Targeting the phosphoinositide 3-kinase pathway in hematologic malignancies[J]. Haematologica, 2014, 99(1): 7-18.
[24] SAMUELS Y, WANG Z, BARDELLI A, et al. High frequency of mutations of the PIK3CA gene in human cancers[J]. Science, 2004, 304(5670): 554.
[25] CAMPBELL I G, RUSSELL S E, CHOONG D Y, et al. Mutation of the PIK3CA gene in ovarian and breast cancer[J]. Cancer Res, 2004, 64(21): 7678-7681.
A functional variant at miR-520a binding site in PIK3CA alters susceptibility to colorectal cancerin a Chinese Han population
DING Lifang1,2, JIANG Zao1, CHEN Qiaoyun3, QIN Rong4, FANG Yue3,LI Hao5(1.Department of Oncology, the Affiliated Zhongda Hospital of Southeast University, Nanjing Jiangsu 210009, China; 2.Department of Oncology, the Danyang People’s Hospital, Danyang Jiangsu 213000, China; 3.Department of Central Laboratory, the Affliated People’s Hospital of Jiangsu University, Zhenjiang Jiangsu 212002, China; 4.Department of Oncology, the Affliated People’s Hospital of Jiangsu University, Zhenjiang Jiangsu 212002, China; 5.Department of Clinical Laboratory, the Taixing People’s Hospital, Taixing Jiangsu 225400, China)
JIANG Zao E-mail: jiangzao@126.com
Background and purpose:Increasing evidence has indicated that polymorphisms in the microRNA (miRNA, miR) binding site of target gene can alter the ability of miRNA and modulate the risk of cancer. We aimed to investigate the association between a miR-520a binding site single nucleotide polymorphism (SNP) rs141178472 in the PIK3CA 3’UTR and the risk of colorectal cancer in a Chinese Han population.Methods:The polymorphism rs141178472 was analyzed in a case-control study, including 386 colorectal cancer patients and 394 ageand sex-matched controls. The relationship between the polymorphism and the risk of colorectal cancer was examined by statistical methods.Results:Individuals carrying the rs141178472 CC genotype or C allele had an increased risk of developing colorectal cancer (CC vs TT, OR=1.716, 95%CI: 1.084-2.716, P=0.022; C vs T, OR=1.258, 95%CI: 1.021-1.551, P=0.033). Furthermore, the expression of PIK3CA was detected in the peripheral blood mononucleated cell of colorectal cancer patients, suggesting that mRNA levels of PIK3CA might be associated with SNP rs141178472.Conclusion:These fndings provide evidence that a miR-520a binding site polymorphism rs141178472 in the PIK3CA 3’UTR may play crucial roles in the etiology of colorectal cancer.
Colorectal cancer; PIK3CA gene; Single nucleotide polymorphism
10.3969/j.issn.1007-3969.2015.03.005
R735.3+4
A
1007-3639(2015)03-0184-06
2014-08-23
2014-11-19)
姜藻 E-mail:jiangzao@126.com
