奶牛α-1,3-岩藻糖基转移酶基因的克隆表达
2014-12-25张阳旸吴正钧
姚 晶 , 张阳旸 , 吴正钧 , 任 婧 *
(1.乳业生物技术国家重点实验室,光明乳业股份有限公司 技术中心,上海 200436;2.上海海洋大学 食品学院,上海 201306)
α -1,3- 岩 藻 糖 基 转 移 酶 (alpha-(1,3)-fucosyltransferase,FT)参与Lewis糖链合成最后一步的岩藻糖化反应,FT有6类,包括FT-Ⅲ、FT-Ⅳ、FT-Ⅴ、FT-Ⅵ、FT-Ⅶ和FT-Ⅸ,其主要功能是催化GDP-岩藻糖的岩藻糖基转移至糖链中N-乙酰氨基乳糖单位的N-乙酰氨基葡萄糖(GlcNAc)上。Lewis是一组存在于细胞表面糖脂和糖蛋白糖链(主要在O-糖链,也可在N-糖链)上的糖类抗原[1-3]。唾液酸化Lewis(Slex)则是一种细胞粘附分子——选择素(selectin)的共同糖配体,选择素主要参与了细胞粘附和迁移,这与细胞的生长发育、组织正常功能的维持、炎症的发生及肿瘤的形成和转移等多种生理过程密切相关,但其中的作用机制,如Slex的表达与癌细胞的增殖、转移、凋亡之间的关系等,并不十分清楚[4-5]。FT是Slex合成过程中必不可少的糖基转移酶,催化合成反应的最后一步,调节和控制着Slex的表达。由于FT与免疫应答反应、肿瘤的发生发展及转移等体内生理活动密切相关,国内外学者对于FT的研究多集中在FT的酶活、表达量、参与合成产生的Slex的结构与体内这些生理活动的关系,以及FT基因的调控作用等方面[6-9]。为了研究FT基因的作用,相关学者已经成功克隆了人、鼠、兔等的FT基因,并分别在人表皮癌细胞系A431、小鼠脾细胞、原核表达系统pET30中成功表达;而牛的FT相关基因的克隆表达尚未见报道[10-12]。作者通过对牛的其中一种FT基因开展相关的分子生物学研究,为进一步理解其在体内的生理功能和作用机理,及相关疾病新型药剂的研发奠定基础。
1 材料与方法
1.1 菌株和质粒
大肠杆菌Escherichia coli DH5α,为光明乳业股份有限公司技术中心实验室保存,基因型:supE44ΔlacU169(φ80 lacZΔM15)hsdR recA1 endA1 gurA1 thi-1 relA1。毕赤酵母 GS115,为 Invitrogen公司产品,由复旦大学惠赠。
质粒pMD19-T,为TaKaRa公司产品。毕赤酵母分泌型表达载体pPIC9K,为 Invitrogen公司产品,由上海生物工程有限公司惠赠。
1.2 试剂和仪器
1.2.1 主要试剂 限制性内切酶NotⅠ-HF、AvrⅡ、BalⅡ、Sac I-HF ,NEB 公司产品;T4 DNA 连接酶、溴酚蓝指示剂(6x buffer)、X-Gal、硫酸卡纳霉素、氨苄青霉素、血液组织细胞基因组提取试剂盒、蛋白loading buffer、标准蛋白Marker,天根公司产品;Taq DNA聚合酶,probest DNA聚合酶,Wide range DNA Marker,DL2000 DNA Marker,TAKARA公司产品;其余常规试剂均为国产分析纯。
1.2.2 主要仪器设备 TC-25/H型PCR仪,杭州博日科技有限公司制造;EPS-300A型数显双稳电泳仪,上海天能科技有限公司制造;412951型凝胶成像仪,BIO-RAD公司制造。
1.3 FT基因片段的生物信息学分析
1.3.1 基本理化性质预测分析 根据NCBI(www.ncbi.nlm.nih.gov)上公布的FTⅨ的蛋白质序列(获取 号 :NP_777160.1), 登 录 http://www.expasy.ch/tools/protparam.html,在线对其相对分子质量、氨基酸组成、等电点及疏水性等基本理化性质进行分析[13]。
1.3.2 蛋白质组学预测分析 利用网站工具软件PROF 软件(http://www.predictprotein.org/)和 HNN软件(http://npsa-pbil.ibcp.fr)预测 FT 的二级结构。利 用 网 站 工 具 软 件 Scan Prosite (http://www.predictprotein.org/)对FT进行结构域分析。利用网站工具软件 SWISS-MODEL 软件(http://swissmodel.expasy.org/)进行建模,预测该酶的三级结构,结果用蛋白质三维图象软件java 2.0查看[14-16]。
1.3.3 蛋白质序列的同源性分析与系统进化树的构建 将氨基酸序列提交至NCBI数据库中,利用网站工具软件BLAST软件(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源性比对分析。进而利用Clustal W软件进行多序列同源性比对和聚类分析,系统进化树使用MEGA 5.0软件的邻接归并法(NJ法)构建[17]。 供试物种为:家犬(Canis lupus familiaris,NP_001005380.1),大熊猫(Ailuropoda melanoleuca,XP_002927989.1),黑 猩猩(Pan troglodytes,NP_001 008978.1), 家 兔 (Oryctolagus cuniculus,XP_002714 636.1),家鼠(Mus musculus,NP_034373.1),马(Equus caballus,XP_001503875.1),褐鼠(Rattus norvegicus,NP_445917.1),鸭嘴兽 (Ornithorhynchus anatinus,XP_001506677.1),短尾猊(Monodelphis domestica,XP_001367612.1),红原鸡(Gallus gallus,NP_00107 2970.1),火鸡(Meleagris gallopavo,XP_003204387.1),安乐蜥(Anolis carolinensis,XP_003225370.1)。 还有人(Homo sapiens,CAB41890.1)。
1.4 FT基因片段的扩增
由于奶牛的FT基因不含有内含子,因此可以直接从基因组DNA直接扩增获得。从荷斯坦奶牛静脉采血5 mL,分装置至含有抗凝剂的1.5 mL的离心管中,液氮速冻,后置于-80℃的超低温冰箱贮存。用血液、组织、细胞基因组DNA抽提试剂盒抽提基因组,并电泳检测,保存基因组DNA于TE缓冲液中备用,主要操作按照试剂盒说明书。
参考 GenBank(www.ncbi.nlm.nih.gov)数据库荷斯坦奶牛 α-1,3-岩藻糖基转移酶Ⅸ(alpha-(1,3)-fucosyltransferase,FTⅨ)编码区序列,设计引物:
P1:ACTGAGCCTAGGATGACCTCAGCATCCAA AGG;P2:AAGGAAAAAAGCGGCCGCTTAATTCCAA AACCATTTCTCTAAATTACC,进行 PCR,用质量分数0.8%琼脂糖凝胶电泳检测PCR产物,并对目的DNA回收纯化。
1.5 重组质粒pMD19-FT的构建
将上述PCR产物溶液于质量分数0.8%琼脂糖凝胶上电泳后,以Marker为参照估算扩增产物的物质的量。在与T载体连接前,首先进行一个加A反应。然后将目的基因片段和载体pMD19-T按摩尔比1∶3~1∶10进行连接反应并转化。对转化的重组子进行蓝白斑筛选,挑选出目的重组子,并对重组子进行PCR鉴定和双酶切鉴定。将PCR鉴定与双酶切鉴定均为阳性的转化子送至Invitrogen公司进一步测序鉴定。送至Invitrogen公司测序,测序引物为M13(+)、M13(-),对测序结果进行比对分析。
1.6 表达质粒pPIC9K-FT的构建
制备具有双黏性末端的重组质粒pMD19-FT以及pPIC9K质粒片段,按一定比例加入到含有T4连接酶缓冲液和T4连接酶的体系中,16℃连接过夜,转化。并按上述方法进行重组子的筛选及鉴定,测序引物为α-factor/3’AOX,并根据测序结果,对目的基因的序列及位置进行验证。
1.7 重组酵母GS115-FT转化子制备及诱导表达
抽提鉴定正确的重组质粒pPIC9K-FT,电转化至P.pastoris GS115感受态细胞,电转化电压1.5 kV,电击时间5 ms。电击结束后取900 μL山梨醇溶液混匀菌体并移至1.5 mL离心管中,30℃静置培养1h。取200 μL转化液涂布至MD平板,30℃培养2~3 d至出现His+转化子。
待转化子长至肉眼可见时,将转化子菌落编号并一一对应地点在含有G418硫酸盐质量浓度为2 mg/mL的MD和MM平板上,30℃培养2~3 d,筛选在MD、MM平板上都正常生长的转化子His+Mut+。随机挑取5个His+Mut+转化子,用Lysis buffer for microorganism to direct PCR试剂进行菌落PCR,PCR 引物为 α-factor/3’AOX。
取鉴定His+Mut+转化子接种至100 mL的BMGY培养基中,30℃ 250 r/min,培养至OD600=2.0~6.0。3 000 g离心5 min收集菌体,用BMMY培养基重悬并稀释至OD600=1.0,分别进行诱导培养24、48、72 h,以 1%(体积分数)甲醇为诱导剂进行诱导表达。蛋白质样品15 000 r/min离心2 min收集上清液,取1 mL置液氮中速冻并保存于-80℃。添加甲醇至 V(上清)∶V(甲醇)=1∶1,4 ℃静置 1 h,12 000 r/min 离心 5 min,沉淀复溶于 100 μL ddH2O,质量分数12%SDS-PAGE检测蛋白质表达情况。
2 结果与分析
2.1 FT基因的生物信息学分析
2.1.1 FT基因基本理化性质 FT cDNA为1 101 bp,有1个ORF(碱基序列9-1088),不含有内含子。通过其序列的在线分析发现,FT的化学式为C1930H2885N489O535S15,原子总数为5 854,相对分子质量为41 978.9,包括359个氨基酸残基,等电点理论值为7.61,负电荷残基(Asp+Glu)总数为34,正电荷残基(Arg+Lys)总数为35,不稳定系数为45.20,故认为该蛋白质为不稳定蛋白质。脂肪系数为80.33,亲水性评估为-0.275。依据氨基酸分值越低亲水性越强和分值越高疏水性越强的规律,可见该酶为亲水性蛋白质。该酶的氨基酸组成如表1所示,其中相对含量比较多的氨基酸为Ser(31个,占8.6%),含量比较少的氨基酸是Tyr(6个,占1.7%)。其在哺乳动物体内的半衰期预测值为30 h,在酵母菌中则>20 h,在大肠杆菌中为>10 h。
2.1.2 FT基因的蛋白质组学分析 通过PROF和HNN软件对FT进行二级结构的预测,结果如表2所示。从预测的结果可以看出,两种方法所得出的二级结构中,包含的α螺旋基本一致,而β折叠、自由卷曲则有一定差别。利用ScanPROSITE对FT的功能域分析发现,它有3个N糖基化位点,1个cAMP和cGMP依赖的蛋白激酶磷酸化位点,6个蛋白激酶C磷酸化位点,3个酪蛋白激酶Ⅱ磷酸化位点,1个酪氨酸激酶磷酸化位点以及2个N豆蔻酰基化位点,如表3所示。利用SWISS-MODEL软件预测的FT的三维结构如图1所示。
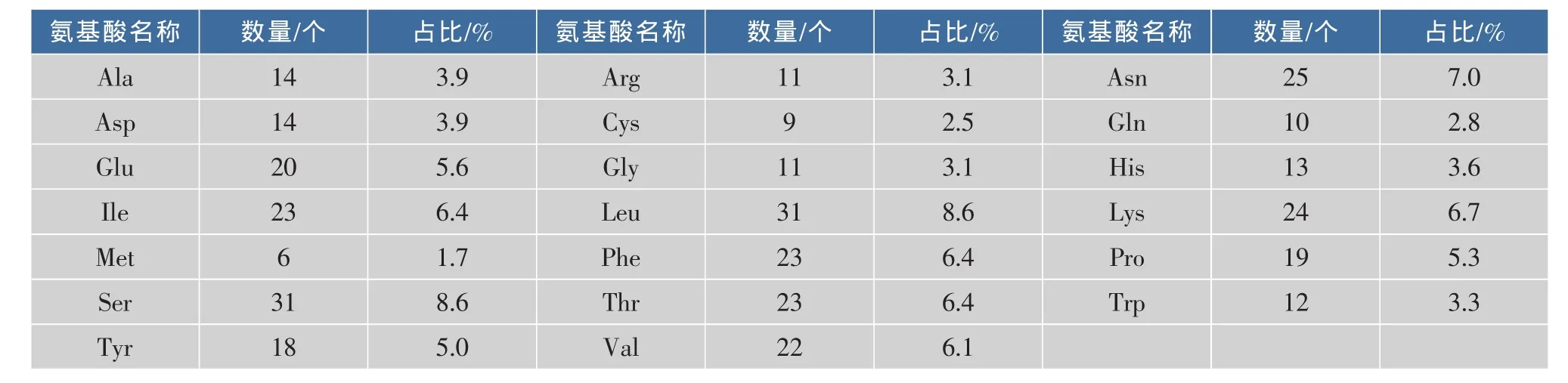
表1 FT的氨基酸组成Table 1 Amino acid composition of alpha-(1,3)-fucosyltransferase Ⅸ

表2 FT二级结构分析Table 2 Predicted secondary structure of alpha-(1,3)-fucosyltransferaseⅨ

表3 FT蛋白功能域分析Table 3 Predicted protein function of alpha-(1,3) -fucosyltransferaseⅨ
2.1.3 蛋白序列同源性分析与系统进化树的构建将蛋白质序列在NCBI上进行BLAST,通过多重序列比对,发现和供试物种的同源度都非常高。通过MEGA 5.0建立的系统进化树如图2所示,可以看出,供试的14个物种的FT被聚成2大类:红原鸡、火鸡和安乐蜥的FT为一类。其中雉科的红原鸡、火鸡为一支。褐鼠、家鼠等其他11个哺乳动物的FT聚为另一类。其中,短尾猊的FT自为一支,其他10个的FT聚为一支。在此10个物种的FT中,鸭嘴兽的FT为一分支,其他9种聚为另一分支。这9个物种FT又分为2个分支:褐鼠、家鼠、黑猩猩和家兔,以及人的FT聚为一个分支,其中家兔又自成一分支,褐鼠和家鼠聚为一组,黑猩猩和人聚为一组,两两组成另一个分支;大熊猫、家犬、马和牛的FT聚为另一分支,其中大熊猫自成一支,马和牛的FT组成的小分支和家犬聚为另外一分支。

图1 SWISS-MODEL预测的FT三维结构Fig.1 Predicted 3D structure of alpha-(1,3) -fucosyltransferaseⅨby SWISS-MODEL

图2 FT的系统发育树Fig.2 Phylogenetic tree of alpha-(1,3)-fucosyltransferaseⅨ
2.2 荷斯坦奶牛基因组DNA的提取和目的基因的扩增
为得到奶牛α-1,3-岩藻糖基转移酶的相关基因,根据GenBank数据库中报道的岩藻糖基转移酶Ⅸ基因设计PCR引物,并结合表达载体引入相关酶切位点。用血液、组织、细胞基因组DNA抽提试剂盒按照试剂盒说明书抽提基因组,并进行电泳检测,如图3所示,可以看出,基因组DNA条带清晰、整齐,说明该试剂盒提取的基因组DNA纯度较高,品质较好。保存基因组DNA于TE缓冲液中,作为扩增目的基因的模板备用。
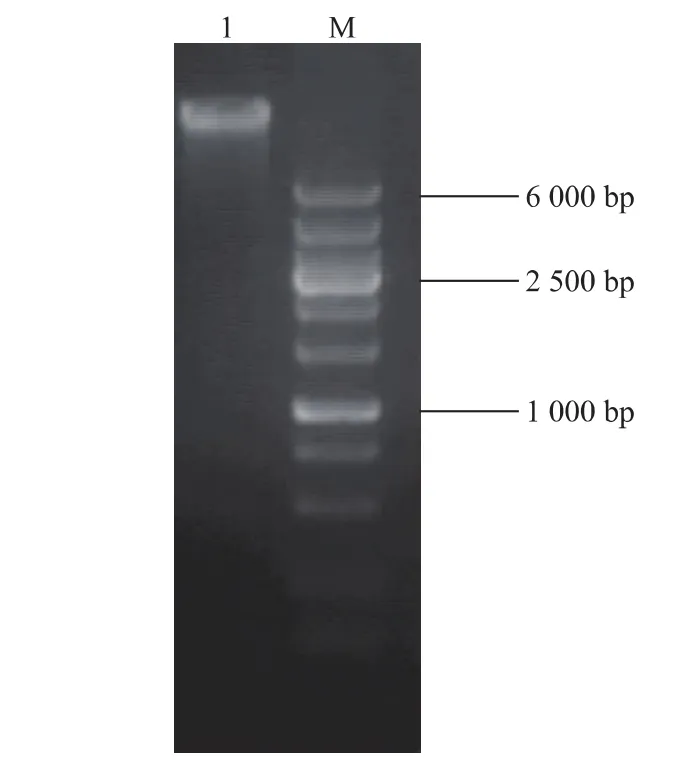
图3 基因组DNA电泳结果Fig.3 Electrophoresis result of genomic DNA
将上步抽提的基因组DNA作为模板,加入到50 μL PCR体系中进行PCR反应,电泳检测在约1.1 kb处出现目的条带,如图4所示。
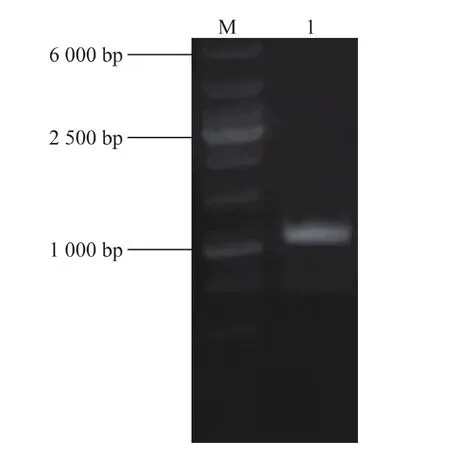
图4 PCR产物电泳图Fig.4 Electrophoresis result of PCR product
2.3 重组质粒阳性克隆筛选及鉴定
上步PCR扩增产物经回收试剂盒纯化后,进行加A反应。按照载体与插入片段摩尔比约为1∶3~1∶10,进行T载体连接。连接产物转化至E.coil DH5α感受态细胞,利用蓝白斑筛选转化子。经过夜培养,平板上可明显观察到蓝白斑。对平板上正常生长的白色菌斑随机挑取5个,进行PCR鉴定,结果如图5所示,发现①号、②号菌落为阳性克隆。继而将②号菌落进行双酶切鉴定,可以得到较好的酶切结果(图6)。将②号菌保种后送样测序分析。将测序结果与NCBI公布的FT基因比对,结果发现PCR产物发生了一个碱基突变。通过多次重复实验,发现在同一位点均发生相同的碱基突变,由于本实验中PCR模板为中国荷斯坦乳牛,而genebank中公布的序列来源为美国乳牛,推测这个位点的突变属于个体差异。至此,构建出重组质粒并命名为pMD19-FT。

图5 重组质粒的PCR鉴定Fig.5 PCR analysis of the DH5α

图6 ②号菌质粒的双酶切图Fig.6 Restriction endonuclease of pMD19-FT
2.4 表达载体的构建及鉴定
将含有FT编码区序列的重组质粒pMD19-FT和表达载体pPIC9K双酶切后,取适量于16℃过夜连接,转化到E.coil DH5α。用通用引物α-factor与3’AOX对挑取的菌落进行PCR鉴定,如图7所示,①号菌、②号菌可能为阳性克隆。
将①号菌质粒进行双酶切鉴定,测序验证,结果为阳性,证明目的基因FT已正确连接到表达载体pPIC9K中,说明已成功构建表达载体pPIC9K-FT。

图7 重组质粒的PCR鉴定Fig.7 PCR analysis of the DH5α
2.5 重组酵母的筛选鉴定及其诱导表达
大量制备pPIC9K-FT,用限制性内切酶Sac IHF将其线性化并浓缩至0.5~1.0 μg/μL。将线性化质粒电转化至宿主菌P.pastoris GS115中,筛选有G418抗性的His+Mut+转化子,并进行PCR鉴定。挑选在MD、MM平板上长得最快的G418抗性的His+Mut+转化子,经G418终质量浓度为2 mg/mL的BMGY培养至OD600=2.0~6.0时,收集菌体用G418终质量浓度为2 mg/mL的BMMY稀释至OD600=1.0,再进行诱导培养。并对目的基因蛋白质进行诱导表达,结果如图8所示。在约45.0 kDa处,有一条明显的蛋白质条带,其相对分子质量与预测值相符,而此处阴性对照无此条带,因此可以判断此条带为目的基因FT可以在宿主菌P.pastoris GS115中成功表达,并可以分泌至胞外,而且随着培养时间的增加,目的蛋白质含量也不断积累。
3 结语
依据NCBI上的相关序列,对奶牛FT基因进行了生物信息学的分析,通过在线预测可知,目的蛋白质FT为一种可溶性稳定蛋白质,具有糖基化、磷酸化、酰胺化等多个功能位点。利用蛋白质分析软件SWISS-MODEL对其三维结构进行了建模预测。通过对FT基因的生物信息学分析,对主要理化性质及蛋白质结构进行了预测,这对其克隆表达、分离纯化等后续相关工作的开展具有一定的指导意义。将FT编码区序列连接到重组质粒pMD19-T载体上并转化至E.coil DH5α中,对转化子进行PCR鉴定、双酶切鉴定及测序鉴定,证明了此段克隆基因的正确性。在此基础上进一步构建了真核表达系统P.pastoris GS115-FT,并对其进行诱导表达,通过SDS-PAGE进行了目的检测,结果显示目的蛋白质的相对分子质量大小与预测值相符,且能够成功分泌到胞外并随之累积,证实了之前生物信息学分析的部分结果是可靠的。但是,有蛋白质的产生并不代表蛋白质就是有活性的。如果在表达过程中,重组蛋白质由于糖基化等后修饰需要或者经过折叠等导致了酶学性质的变化或者丧失,对其今后的开发利用都会产生巨大的影响。因此,后续的工作将重点研究所表达重组蛋白质的酶学性质,进一步证实其中一个点突变来源于个体差异的推测,及其他生物信息学的分析结果的可靠性。在证实重组蛋白质相关酶学性质后,进一步优化发酵条件,以期在发酵周期内获得最大的生物量,为后续的开发利用创造条件。

图8 GS115-GT不同时间的SDS-PAGE图谱Fig.8 SDS-PAGE analysis of expression by GS115-FT
作者利用分子生物学技术成功克隆了奶牛的FT基因,并在真核系统中可溶性表达,这为FT的大量制备提供了可能,为后续的科研工作,如酶学性质、Slex的生物合成及相关生理、病理机理等方面的深入研究打下了良好的基础。
[1]陈惠黎.糖复合物的结构和功能[M].上海:上海医科大学出版社,1997:54-270.
[2]Becker D J,Lowe J B.Fucose:Biosynthesis and biological function in mammals[J].Glycobiology,2003,13(7):41-53.
[3]Javaud C,Dupuy F,Maftah A,et al.The fucosyltransferase gene family:an amazing summary of the underlying mechanisms of gene evolution[J].Genetica,2003,118(2-3):157-170.
[4]Munro J M.Expression of sialyl-lewis X an E-selectin ligand in flammation,immune processes and lymphoid tissues[J].American Journal of Pathology,1992,141:1397-1408.
[5]Wang Q Y,Guo P,Duan L L,et al. α1,3-Fucosyltransferase-Ⅶ stimulates the growth of hepatocarcinoma cells via cyclin dependent kinase inhibitor p27Kip1[J].Cell Mol Life Sci,2005,62:171-178.
[6]Miyashiro M,Furuya S,Sugita T.Development of a sensitive separation and quantification method for sialyl Lewis X and Lewis X involving anion-exchange chromatography:biochemical characterization of alpha 1,3 fucosyltransferase Ⅶ [J].J Biochem,2004,136(5):723.
[7]吴兴中,陈迎风.岩藻糖糖链与肝癌细胞的迁移作用[J].生物化学与生物物理进展,2002(6):932-937.WU Xingzhong,CHEN Yingfeng.Fucosylated ogligosaccharides in the migration of hepatoma cells[J].Progress in Biochemistry and Biophysics,2002(6):932-937.(in Chinese)
[8]李文桦,张文.α1,3岩藻糖基转移酶Ⅶ和糖蛋白CD24与肿瘤转移的研究进展[J].中国癌症杂志,2008,18(10):790-795.LI Wenhua,ZHANG Wen.Advance in the study of α1,3 fucosy1transferase Ⅶ and glycoprotein CD24 in tumor metastasis[J].China Oncology,2008,18(10):790-795.(in Chinese)
[9]Elmgren A,Mollicone R,Costache M,et al.Significance of individual point mutations,T202C and C314T,in the human Lewis(FUT3) gene for expression of Lewis antigens by the human alpha (1,3/1,4)-fucosyltransferase,Fuc-TIII[J].The Journal of Biological Chemistry,1997,272(35):21994-21998.
[10]杨雪松,贾爽,张震波,等.人α1,3-岩藻糖基转移酶IV荧光真核表达载体的构建及鉴定[J].世界华人消化杂志,2008,16(10):1064-1068.YANG Xuesong,JIA Shuang,ZHANG Zhenbo,et al.Construction and identification of human fucosyltransferase 4 eukaryotic fluorenscent expression vector[J].World Chinese Journal of Digestology,2008,16(10):1064-1068.(in Chinese)
[11]Elizabeth M S, Frances I S, Gherman W,et al.Cloning of a rat α1,3-fucosyltransferase gene:a member of the fucosyltransferase IV family[J].Glycoconjugate Journal,1997,14(2):249-258.
[12]哲名家,张淼涛,云涛,等.兔岩藻糖基转移酶基因的克隆与原核表达[J].西北农林科技大学学报:自然科学版,2012,40(4):21-24.ZHE Mingjia,ZHANG Miaotao,YUN Tao,et al.Cloning and prokaryotic express of rabbit fucosyltransferases[J].Journal of Northwest A&F University:Nat Sci Ed,2012,40(4):21-24.(in Chinese)
[13]Baxevanis A D,Ouellette B F F.生物信息学[M].李衍达,孙之荣,译.北京:清华大学出版社,2000:231-250.
[14]Arnold K,Bordoli L,Kopp J,et al.The SWISS-MODEL workspace:A Web-Based environment for protein structure homology modelling[J].Bioinformatics,2006,22:195-201.
[15]Schwede T,Kopp J,Guex N,et al.SWISS-MODEL:An automated protein homology-modeling server[J].Nucleic Acids Research,2003,31:3381-3385.
[16]Guex N,Peitsch M C.SWISS-MODEL and the Swiss-PdbViewer:An environment for comparative protein modelling[J].Electrophoresis,1997,18:2714-2723.
[17]Mount D W.生物信息学[M].钟扬,王莉,张亮,译.北京:高等教育出版社,2003:301-345.
