旱麦草LEA3基因的克隆与序列分析
2014-11-02孙黎黄先忠祝建波吕新华邹恵娟安向向李晨瑕周晓霞
孙黎,黄先忠,祝建波,吕新华,邹恵娟,安向向,李晨瑕,周晓霞
(石河子大学生命科学学院农业生物技术重点实验室,石河子,832003)
干旱胁迫是自然界最主要的非生物胁迫之一,干旱严重影响植物的生长发育和作物产量。经过多年的研究,人们已经在干旱对植物产生的危害和植物耐干旱的机制方面取得了许多进展[1-3]。进一步加强具有抗旱基因资源的发掘和创新,开展抗旱生理和遗传学研究,采用基因工程技术提高作物的耐旱能力是目前研究的热点之一。
植物晚期胚胎发生丰富蛋白 (Late embryogenesis abundant protein,LEA)是植物种子发育后期产生的一类小分子特异多肽,这类蛋白与植物耐脱水性密切相关,具有强的亲水性和热稳定性,即使在煮沸的情况下也可以保持水溶状态[4]。植物种子在脱水和休眠的过程中面临的水分胁迫使细胞组成成分晶体化,使细胞的有序结构遭受破坏,而LEA蛋白具有高度的亲水性,能把足够的水分捕获到细胞内,从而保护细胞免受水分胁迫的伤害[2-6]。此外许多营养组织在外源脱落酸(ABA)、低温或干旱胁迫的诱导下也能产生特异的LEA蛋白[6]。自1981年首次报道植物LEA蛋白[7]以来,已从小麦、大麦、高粱、玉米等多种植物中发现了新的LEA蛋白[7-10]。根据LEA蛋白中氨基酸序列的同源性及一些特殊基元序列,可将LEA蛋白分为6组[11]。其中第3组LEA蛋白在生物体内具有非常重要的生理功能,它们的显著特点是含有一段由11个氨基酸组成的基元序 列(T/AA/TO/EA/TA/TK/RQ/EDK/RA/TXE/DQ)串联重复排列,该基元序列可以形成亲水的α-螺旋结构,在植物受到干旱胁迫时能避免细胞内高浓度离子的积累引起的损伤,同时可以防止组织过度失水[12]。近年来,第3组LEA蛋白在植物干旱诱导蛋白的研究方面受到普遍关注,已成为植物抗旱研究的重点[13-15]。
旱麦草(Eremopyrum triticeum)是禾本科小麦族旱麦草属的早春短命植物,小麦的野生近源种,在我国仅分布在新疆北部的天山北麓和准噶尔盆地及其周围干旱的荒漠地区,具有生育期短、抗旱、耐盐、光和效率高等优良性状[16]。在国内外诸多LEA基因的相关报道中未见有关旱麦草LEA基因的文献资料。本研究根据小麦LEA第3组基因的编码区全长cDNA设计引物,从干旱胁迫的旱麦草叶片中分离LEA3的同源序列,并利用一系列生物信息学软件分析了该蛋白的理化性质、跨膜特性、三级结构和系统进化等,旨在为进一步研究该基因的功能奠定基础。
1 材料与方法
1.1 材料
旱麦草(E.triticeum)种子为本实验室保存,在生命科学学院智能型温室播种。M-MLV反转录酶、Ribonuclease inhibitor等购自 Invitrogen公司,Ex Taq DNA 聚 合 酶 、dNTP、DNA marker、pMD18-T Vector等购自 TaKaRa公司,RNase-free DNase等购自Promega公司。其它试剂均为国产分析纯。引物合成和克隆测序由北京六合华大生物工程技术服务有限公司完成。
1.2 方法
1.2.1旱麦草RNA的提取和cDNA的合成
旱麦草幼苗于温室中培养2周后,用20%的PEG 6000模拟干旱胁迫8 h,采用TIANGEN公司的RNA prep pure Plant Kit试剂盒 (离心柱型)提取总RNA,参照说明书中的方法进行。cDNA模板第1链的制备,根据Promega公司M-MLV Reverse Transcriptase合成。
1.2.2 EtLEA3基因的克隆与测序
由于旱麦草是小麦的野生近源种,而且通过对已克隆的小麦、大麦等单子叶植物LEA3基因核苷酸序列进行比对发现,其开放阅读框(ORF)区域的5’和3’末端较为保守,因此我们根据小麦LEA3基因的cDNA序列(GenBank登录号为AY148492)设计了1对特异引物用于扩增旱麦草LEA3基因的ORF。
正向引物 P1:5’-ATGGCCTCCAACCAGAACCA-3’; 反向引物 P2:5’-CTAGTGATTCCTGGTGGTGG-3’。以合成的单链cDNA为模板,P1,P2为引物进行PCR扩增。
PCR 扩增体系为:cDNA 1.0 μL,10 × Ex Taq Buffer 2.0 μL,2.5 mmol/L dNTP Mixture 1.5 μL,10 μmol/L 的上下游引物各 0.5 μL,Ex Taq (5 U/μL)0.2 μL,加水补足至 25 μL。
扩增条件:94℃ 3 min后;94℃ 30 s,53℃30 s,72 ℃ 1 min,35个循环;72 ℃延伸 10 min。
PCR扩增产物经1.0%琼脂糖凝胶电泳分离,凝胶回收试剂盒回收目的片段。将目的片段连接到pMD18-T载体上,转化大肠杆菌Top10,通过蓝白斑筛选,挑选白色菌落过夜摇菌,菌液PCR验证后,提取质粒鉴定后送北京六合华大基因科技股份有限公司测序。
1.2.3生物信息学分析
使用DNAMAN软件分析DNA序列,用NCBI(http://www.ncbi.nlm.nih.gov)数据库中的BLAST分别进行核苷酸与蛋白质序列比对。利用http://www.expasy.org/tools/网站中的相关软件进行蛋白质的性质分析。利用ProtParam在线分析蛋白分子量、等电点、氨基酸组成;protscale(http://cn.expasy.org/tools/protscale.html)分析亲水性/疏水性。PSORTⅡPrediction分析亚细胞定位。利用TMHMM软件(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白的跨膜结构。在线软件http://npsa-pbil.ibcp.fr/进行二级结构的预测。提交EtLEA3基因所编码的蛋白序列到 SWISSMODEL服务器(http://swissmodel.expasy.org/)进行自动建模,得到蛋白的三维结构。MEGA5.0软件对EtLEA3蛋白与已知的部分LEA3蛋白进行聚类分析并绘制系统进化树[17-19]。
2 结果与分析
2.1 EtLEA3基因的克隆与测序
根据设计的引物,以旱麦草干旱胁迫的叶片cDNA为模板进行扩增,获得约为600 bp的目标片段,电泳检测结果如图1。将该片段克隆、测序,结果表明目的基因片段具有完整的CDS区,长度为600 bp,命名为 EtLEA3。将该序列在 NCBI上进行BLAST比对,结果显示与已知的小麦LEA3基因(GenBank登录号为AY148492和AB297680)和大麦LEA3基因(GenBank登录号:FJ026801)有较高的相似性。
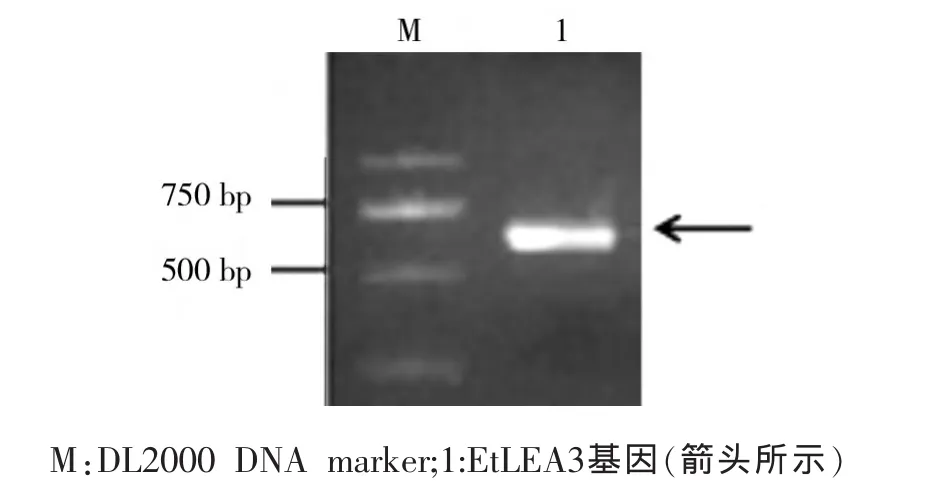
图1 旱麦草EtLEA3基因的PCR扩增Fig.1 PCR amplification of EtLEA3 gene fromE.triticeum
2.2 EtLEA3基因的序列分析
序列分析表明,克隆得到的EtLEA3基因序列包含1个完整的开放阅读框(ORF),编码199个氨基酸(图2)。EtLEA3蛋白与已知的禾本科植物小麦TaLEA3蛋白 (BAF79928)、大麦 HvLEA3蛋白(ACH89913)和山羊草 AtLEA3 蛋白(EMT21661)的相似性分别为83%、82%和81%(图3)。
旱麦草EtLEA3蛋白含丙氨酸(21.6%)、苏氨酸(16.1%)、赖氨酸(12.6%)、谷氨酰胺(9.5%)、谷氨酸(8.5%)等亲水性氨基酸,并且不含脯氨酸、色氨酸、异亮氨酸、半胱氨酸和苯丙氨酸,这与大多数LEA3蛋白质中不含有色氨酸和半胱氨酸的结果[11]一致。另外,旱麦草EtLEA3基因开放阅读框编码的199个氨基酸残基中包含5个由11个氨基酸组成的串联重复基元序列(图2方框部分表示),这属于第3组LEA蛋白的典型结构特征[11-12]。

图2 旱麦草EtLEA3基因核苷酸序列及其所编码的氨基酸序列Fig.2 Nucleotide and deduced amino acid sequences of EtLEA3 gene fromE.triticeum
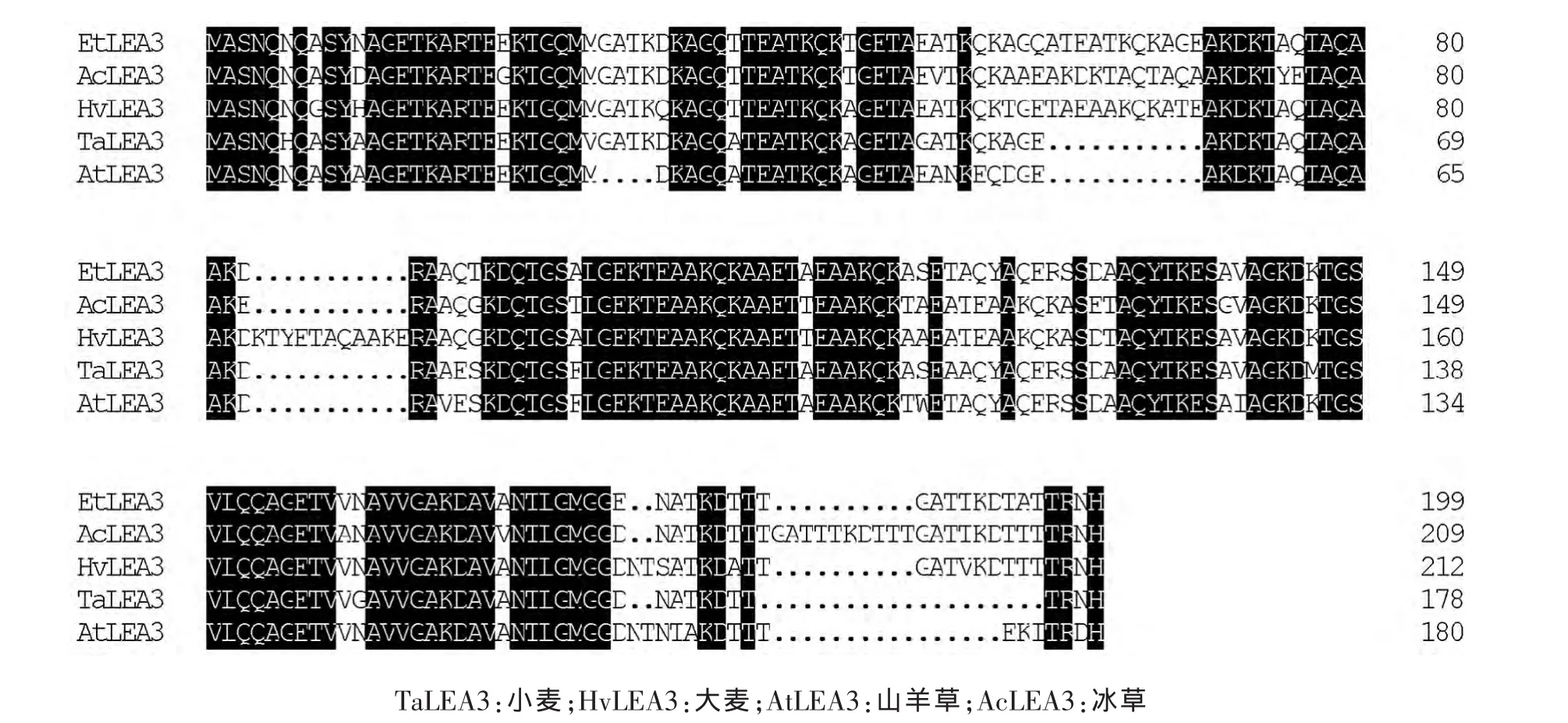
图3 旱麦草EtLEA3蛋白同其他植物的LEA3蛋白氨基酸序列比对分析Fig.3 Multiple alignment of EtLEA3 protein amino acid sequences with that of other plants species
2.3 蛋白理化性质、亲/疏水性及跨膜结构的分析
利用ProtParam分析EtLEA3蛋白的理化性质,结果表明相对分子量为20.38 kDa,等电点为9.08。
蛋白质的亲水性/疏水性的预测是蛋白质二级结构以及功能域的一个重要过程。运用protscale软件对旱麦草EtLEA3氨基酸序列进行亲水性/疏水性分析(正值为疏水,负值为亲水)。结果表明该蛋白有很强的亲水性,并且199个氨基酸在亲水性方面呈现一定的周期性,形成几个亲水性高峰(图4),有利于形成兼性α-螺旋结构,从而达到结合水分子以保护细胞免受水分亏缺时的伤害[12]。
利用TMHMM软件对EtLEA3蛋白进行了跨膜区分析,结果表明该蛋白不含有跨膜区,是非跨膜蛋白(图 5)。
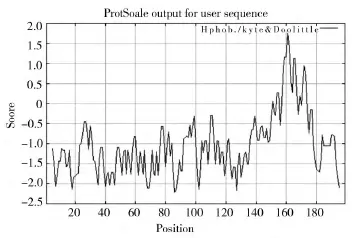
图4 旱麦草EtLEA3蛋白的亲水性/疏水性分析Fig.4 Hydrophilicity/hydrophobicity analysis of EtLEA3 protein fromE.triticeum
2.4 蛋白二级和三级结构预测
通过 http://npsa-pbil.ibcp.fr/网站预测 EtLEA3蛋白的二级结构 (图6)。由图6可知,旱麦草EtLEA3蛋白的二级结构主要以α-螺旋为主,占整个氨基酸序列的71.36%,其次是无规则卷曲,无β-折叠和转角结构,这也与大多数第3组LEA蛋白的结构特点相一致[8-10]。
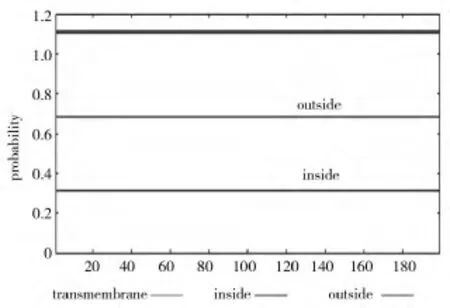
图5 旱麦草EtLEA3蛋白的跨膜结构分析Fig.5 Transmembrane structure of EtLEA3 protein
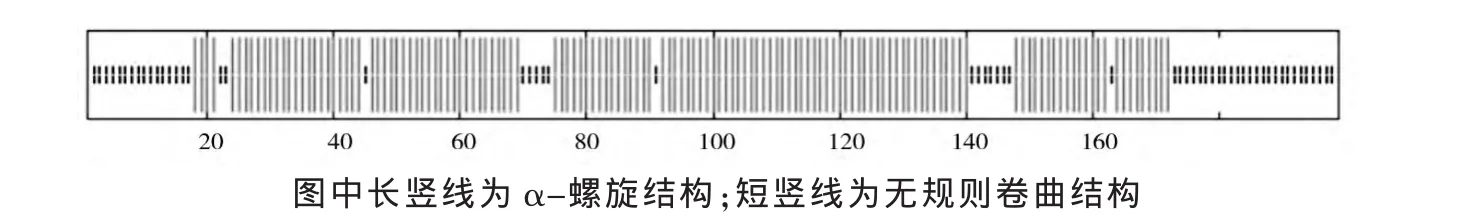
图6 旱麦草EtLEA3蛋白的二级结构预测Fig.6 Secondary structure of EtLEA3 protein
利用SWISS-MODEL中自动建模功能,预测得到EtLEA3蛋白的三维结构(图7),表明该蛋白的三维结构是以α-螺旋为主形成较为紧凑的结构。
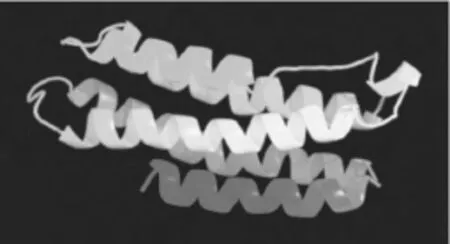
图7 旱麦草EtLEA3蛋白的三级结构Fig.7 Tertiary structure of EtLEA3 protein
2.5 EtLEA3蛋白与已克隆的LEA3蛋白的系统进化树分析
应用MEGA5.0软件将编码的氨基酸序列及从GenBank获取的其它植物的LEA3推导的氨基酸序列进行进化树比对分析,结果见图8。由图8可见:
旱麦草EtLEA3蛋白与禾本科单子叶植物小麦、山羊草、大麦、无芒雀麦和高粱等的LEA3蛋白聚为一类;水稻、玉米和冰草的LEA3蛋白聚为一类;3个双子叶植物拟南芥、菜豆和大豆的LEA3蛋白聚为一类。旱麦草EtLEA3蛋白与小麦和山羊草的LEA3蛋白遗传距离最近。
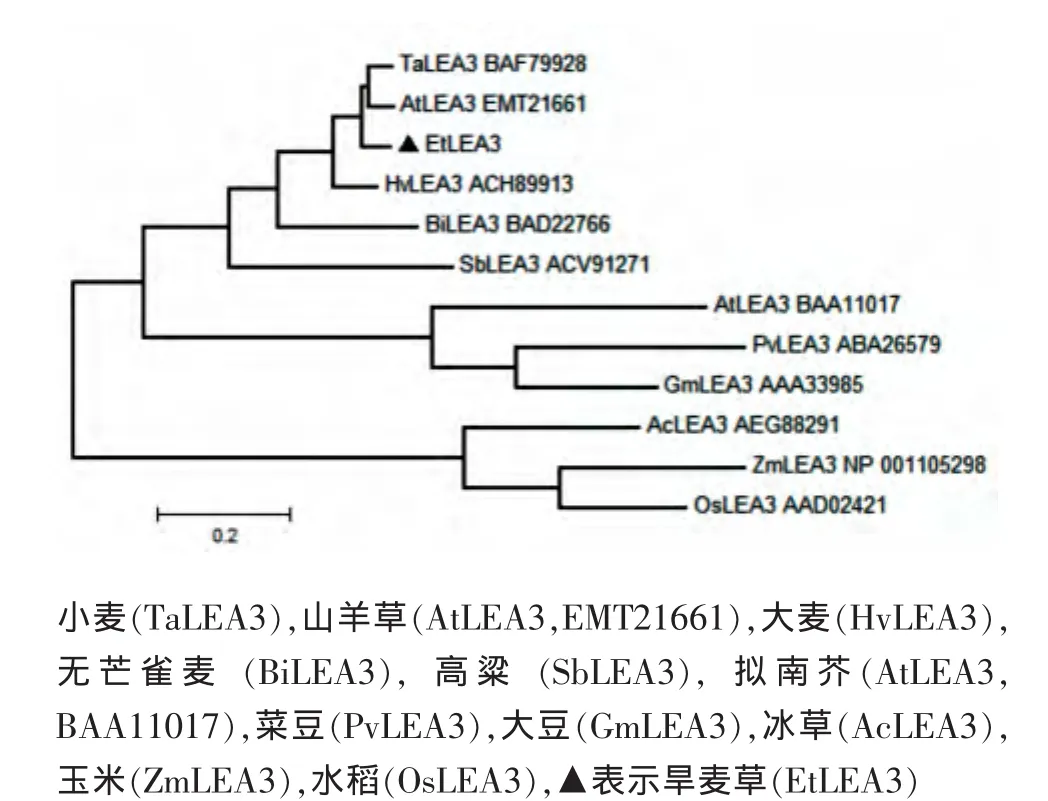
图8 植物LEA3蛋白的系统进化树Fig.8 Phylogentic tree of LEA3 protein from different plant species
3 讨论
通常根据LEA蛋白保守基元序列的差异,可将LEA蛋白分为6种不同的类型[11],类型Ⅰ的基本特征是具有20个保守氨基酸组成的基序,如小麦Em蛋白;类型Ⅱ的LEA蛋白也称作脱水素(dehydrin),基本特征是在羧基末端存在2~11个氨基酸组成的富含 Lys的 K-区(EKKGIMDKIKEKIPG)和在中部含有一连串Ser组成的S-区段;类型Ⅲ含有1段由11个氨基酸组成的基元序列(T/AA/TO/EA/TA/TK/RQ/EDK/RA/TXE/DQ)串联重复排列;类型Ⅳ具有保守的氨基末端形成螺旋和1个多变的羧基末端,具有无规则卷曲结构;类型Ⅴ含有比其他类型的LEA蛋白更多的疏水氨基酸,可能形成球形结构。类型Ⅵ为其他缺乏高度残基专一性的LEA蛋白如棉花的LEA D95等。氨基酸序列分析结果表明EtLEA3的氨基酸构成了5个串联的11个氨基酸基元重复序列 (图2),所以我们认为EtLEA3属于第3组LEA蛋白基因家族的成员。不同植物的第3组LEA蛋白大小差异很大,通常由172~555个氨基酸组成[8,11],该基元序列的拷贝数也有较大的差异,如高粱的LEA蛋白有7个[9],大豆中则超过30个[20],而旱麦草EtLEA3蛋白只有5个串联的保守基元序列,推测LEA3蛋白保守基元序列拷贝数的差异可能与植物的抗旱性机制相关,具体机制有待进一步研究。
对旱麦草EtLEA3蛋白氨基酸序列和蛋白质的结构预测结果表明,EtLEA3蛋白具有较强的亲水性。11个氨基酸残基组成的保守串联重复序列,可形成兼性α-螺旋结构,在植物细胞脱水时能提供疏水区的亲水表面[11]。三级结构预测结果也表明,EtLEA3蛋白是以α-螺旋为主形成较为紧凑的结构。在水分缺乏时,LEA蛋白的亲水α-螺旋结构扩大了与离子结合的表面积,并存在周期性的核电离子空间结合位点,在脱水时核电离子结合于这些位点,从而缓冲了因脱水使离子强度提高造成的细胞伤害[21],这可能是旱麦草EtLEA3蛋白的重要功能之一。
本研究结果表明,LEA基因的表达或蛋白累积与植物的渗透胁迫抗性成正相关。例如,在严重干旱的小麦幼苗中,第3组LEA蛋白累积量的大小与组织的耐旱能力密切相关[3,22]。Xu等[8]将大麦LEA3蛋白(HVA1)基因转入水稻中,结果表明转基因水稻提高了对水分胁迫和高盐胁迫的耐受性。王瑛等[23]将大麦的LEA3基因转入紫花苜蓿中,获得耐盐能力增强的转基因植株。有研究[11]表明,大多数LEA蛋白主要由碱性的亲水氨基酸组成,无半胱氨酸和色氨酸,其疏水面有利于形成同型二聚体,处在外表面带电的基团,可中和因脱水而增加的离子。由LEA蛋白的结构推测其功能是在种子成熟干燥过程中,或渗透胁迫条件下保护细胞免受水势降低的损伤,在植物细胞中起保护作用[3-4,21]。本研究得到的旱麦草EtLEA3抗旱基因的ORF,经过核苷酸及氨基酸序列对比分析确定为第3组LEA蛋白成员。目前对该基因的功能研究工作正在进行中。
[1]Shinozaki K,Y amaguchi Shinozaki K,S eki M.R egulatory network of gene expression in the drought and cold stress responses[J].Current Opinion in Plant Biology,2003,6:410-417.
[2]Farooq M,Wahid A,KobayashiN,etal.Plantdrought stress:effects,mechanisms and management[J].Agronomy for Sustainable Development,2009,29(1):185-212.
[3]Bhagi P,Zhawar V K,Gupta A K.Antioxidant response and Lea genes expression under salt stress and combined salt plus water stress in two wheat cultivars contrasting in drought tolerance[J].Indian J Exp Biol,2013,51(9):746-57.
[4]Espelund M,SaebØe Larssen S,Hughes D W,et al.Late embryogenesis-abundant genes encoding proteins with different numbers of hydrophilic repeats are regulated differentially by abscisic acid and osmotic stress[J].Plant J,1992,2(2):241-252.
[5]ShinozakiK,Yamaguchi ShinozakiK.Gene expression and signal transduction in water stress response[J].Plant Physiol,1997,115:327-334.
[6]刘一鸣,刁丰秋,张雷,等.胡萝卜LEA基因家族新成员DcLEA1的克隆及其结构与表达特性分析[J].自然科学进展,2004,14(8):882-891.
[7]Wang L,Li X,Chen S,et al.Enhanced drought tolerance in transgenic Leymus chinensis plants with constitutively expressed wheat TaLEA3[J].Biotechnol Lett,2009,31(2):313-319.
[8]Xu D,Duan X,Wang B,et al.Expression of a late embryogenesis abundant protein gene,HVA1,from barley confers tolerance to water deficit and salt stress in transgenic rice[J].Plant Physiol,1996,110:249-257.
[9]孙晓娇,汤晓倩,于丽霞,等.高粱LEA3蛋白基因和启动子的克隆及序列分析[J].植物分类与资源学报,2013,35(5):585-593.
[10]Liu Y,Wang L,Xing X,et al.ZmLEA3,a multifunctional group 3 LEA protein from maize(Zea mays L.),is involved in biotic and abiotic stresses[J].Plant Cell Physiol,2013,54(6):944-959.
[11]Ingram J,Barte ls D.The molecular basis of dehydration tolerance in plants[J].Annu Rev Plant Physiol Plant Mol Biol,1996,47:377-403.
[12]Dure L.A repeating 11 mers amino acid motif and plant desiccation[J].Plant J,1993,3:363-369.
[13]钱刚,翟旭光,韩兆雪,等.西藏青稞LEA3蛋白新抗旱基因的克隆与序列分析[J].作物学报,2007,33(2):292-296.
[14]Liu Y,ZhengY,ZhangY,etal.Soybean PM2 protein(LEA3)confers the tolerance of Escherichia coli and stabilization of enzyme activity under diverse stresses[J].Curr Microbiol,2010,60(5):373-378.
[15]HandSC,MenzeM A,TonerM,etal.LEA proteins during water stress:not just for plants anymore[J].Annu Rev Physiol,2011,73:115-134.
[16]张继益,董玉琛.小麦的野生近源植物—旱麦草属[J].生物多样性,1997,5(1):26-30.
[17]徐芳,赵云霞,魏艳玲,等.小拟南芥液泡膜H+-PPase基因OpVP1的克隆、序列分析及表达[J].石河子大学学报:自然科学版,2013,31(1):77-83.
[18]詹杰鹏,崔百明,彭明,等.海岛棉DELLA蛋白基因GbGAI2的克隆及其在胚珠中表达分析[J].石河子大学学报:自然科学版,2012,30(5):529-534.
[19]院海英,顾超,徐芳,等.小拟南芥Na+/H+逆向转运蛋白基因的克隆及生物信息学分析[J].石河子大学学报:自然科学版,2011,29(4):401-407.
[20]李乐,许红亮,杨兴露,等.大豆LEA基因家族全基因组鉴定、分类和表达[J].中国农业科学,2011,44(19):3945-3954.
[21]Shao H B,Liang Z S,Shao M A.LEA proteins in higher plants:structure,function,gene expression and regulation[J].Colloids Surf B Biointerfaces,2005,45(3-4):131-135.
[22]Ried J L,Walker Simmons M K.Group 3 late embryo enesis abundant proteins in desiccation tolerant seedlings of wheat[J].Plant Physiol,1993,102:125-131.
[23]王瑛,朱宝成,孙毅,等.外源lea3基因转化紫花苜蓿的研究[J].核农学报,2007,21(3):249-252.
