高效液相色谱-串联质谱检测基因组DNA甲基化方法的建立
2014-10-27张航胡俊杰汤瑞华叶金波任晓虎胡韬黄培武杨细飞黄海燕刘建军
张航,胡俊杰,汤瑞华,叶金波,任晓虎,胡韬,黄培武,杨细飞,黄海燕,刘建军
1.深圳大学 生命科学学院,广东 深圳 518060;2.深圳市疾病预防控制中心 现代毒理学重点实验室,广东 深圳 518055
DNA甲基化是在特异的DNA甲基转移酶(DNA methyltransferase,DNMT)催化作用下,将S-2腺苷甲硫氨酸(S-2 adenosine methionine,SAM)上的甲基转移到胞嘧啶C-5位点形成甲基化DNA分子的过程[1]。DNA甲基化是表观遗传修饰的主要方式之一,其在维持正常细胞功能、遗传印记、胚胎发育过程中起着极其重要的作用[2]。胞嘧啶甲基化的异常与肿瘤抑癌基因的沉默密切相关,并在很多疾病的发生发展中起决定性作用[3-4]。近年来,不断有研究显示人类肿瘤的发生、发展与DNA甲基化的异常有关,而且早在肿瘤临床确诊之前就可检测出特异基因的甲基化异常现象[5]。因此,甲基化可以作为肿瘤等早期诊断的生物标志物和预后评估指标,对肿瘤筛查、风险评估、早期诊断、分期分型、预后判断及治疗监测都具有重要意义[5]。随着DNA甲基化研究的不断深入,其检测方法也层出不穷。这些方法针对不同的研究目的,运用不同的处理,几乎涵盖了从基因到基因组各个层次水平的研究。在本实验中,我们总结了前人的各种研究方法,建立了在色谱技术检测基础上用串联质谱检测的高效液相色谱-串联质谱(HPLC-ESI-MS/MS)技术。该方法具有高灵敏度、高通量、高分辨率且重复性好等优点,为DNA甲基化的快速精确检测提供了新策略。
1 材料和方法
1.1 材料
LC-20A型全自动高效液相色谱系统(岛津公司);API 4000 Q-Trap型质谱检测器(AB SCIEX公司);186001301型Atlantis dC18色谱柱(2.1 mm×150 mm i.d,5 μm粒径)(Waters公司);色谱柱保护柱(2.1 mm×20 mm i.d,5 μm粒径)(Phenomenon公司);MILLI-QA10超纯水系统(Millipore公司);For⁃ma 725型-80℃低温冰箱(Thermo Forma公司);BP1211S型电子天平(Sartorius公司)。
2-脱氧鸟嘌呤核苷(2-deoxyguanosine,dG)、鸟嘌呤核苷(guanosine,G)、腺嘌呤核苷(adenosine,A)、2-脱氧腺嘌呤核苷(2-deoxyadenosine,dA)、尿嘧啶核苷(uridine,U)、胞嘧啶核苷(cytidine,C)、胸腺嘧啶核苷(thymidine,T)、2-脱氧胞嘧啶核苷(2-deoxycytidine,dC)、5-甲基-脱氧胞苷(5-methyl-de⁃oxycytidine,5-mdC)、甲酸(色谱纯)、乙腈(色谱纯)、甲醇(色谱纯)、碳酸氢铵(色谱纯)、醋酸铵(色谱纯)、碱性磷酸酶购自Sigma-Aldrich公司;实验用水为超纯水。
1.2 溶液配制
1.2.1 储备液和标准工作溶液 用电子天平分别称取A、G、T、C、dA、dG、dC、U和5-mdC各50 mg,溶于50 mL H2O,配制为1 mg/mL的储备液,充分振荡至溶液清澈。将 5-mdC和dG的储备液用H2O稀释配制成浓度梯度为1、10、100、500、1000 ng/mL的混合标准工作溶液。稀释储备液,将不同浓度的5-mdC(5、10、15、20、25、30、35、40、45、50 ng/mL)加入500 ng/mL dG中,所形成的混合标样中5-mdC与dG的质量比值分别为1%、2%、3%、4%、5%、6%、7%、8%、9%和10%,充分振荡混匀。以上溶液均保存于-20℃冰箱,使用时提前室温平衡。
1.2.2 流动相 A相为甲酸-水(1 mL甲酸溶于1000 mL H2O),B相为甲酸-甲醇(1 mL甲酸溶于1000 mL甲醇)。
1.3 色谱和质谱条件
1.3.1 色谱条件 将固定相色谱柱与保护柱连接,并连接流动相样品瓶(A相和B相)。流动相设置洗脱时间为0~24 min,B流动相所占流动相总体积为0~18%,流速为0.22 mL/min,柱温为25℃,进样量为10 μL。
1.3.2 质谱条件 离子源为电喷雾离子源,干燥器温度为450℃,喷雾器为氮气(N2),喷雾压力为4500 V,扫描方式为多反应检测(multiple reaction moni⁃toring,MRM)模式。启动自动优化程序后,分别将5-mdC和dG的标准工作溶液进样检测分析,进行碰撞能和去簇电压等质谱工作参数的优化,同时对检测限、基质效应、标准工作曲线和重现性等参数进行探讨分析。
2 结果
2.1 母/子离子对
采用针泵连续进样,在正离子模式下分别对标准溶液进行母离子全扫描,确定分子离子,优化各分子离子的锥孔电压。分别以上述离子为母离子,对其子离子进行全扫描。优化后的多反应监测母子离子对是:A(m/z 268.1→136.1)、dA(m/z 252.3→135.9)、C(m/z244.1→112.2)、dC(m/z228.0→112.2)、5-mdC(m/z 241.9→126.3)、G(m/z 284.1→152.3)、dG(m/z268.2→152.1)、T(m/z243.3→127.2)和 U(m/z 245.1→112.9)。选取其中信号较强、干扰较小的子离子与母离子组成监测离子对。5-mdC和dG的标准工作溶液(5 μg/mL)经质谱检测后的全扫描质谱图显示了电喷雾形成的离子化5-mdC及dG分别在质荷比(mass-to-charge ratio,m/z)为241.9及268.1处存在最强的响应峰,分别对应于相应的母离子。同时5-mdC和dG对应的子离子分别是m/z 126.3及m/z 152.3(图1)。所以,在进行5-mdC及dG的定量检测时,MRM模式采用的母/子离子对分别是5-mdC(m/z 241.9→126.3)和dG(m/z 268.2→152.1)。
2.2 碰撞能和去簇电压
对5-mdC和dG标准品连续进样监测分析后,得出了1000 ng/mL的5-mdC和dG标准品在ESI阳离子电离模式条件下的响应值与不同的碰撞能和去簇电压间的关系(图2)。经自动优化程序优化后的5-mdC和dG碰撞能均为15 eV,而去簇电压分别是40和45 V。
2.3 MRM总离子流图

图1 在ESI阳离子电离模式下5-mdC(A)和dG(B)的子离子图

图2 在ESI阳离子电离模式下5-mdC和dG(1000 ng/mL)的响应值随碰撞能和碎片电压的变化
图3给出了标准溶液的正常核苷和修饰核苷(A、dA、C、dC、5-mdC、G、dG、T和U)的典型MRM总离子流图,说明以上化合物能实现良好的基线分离,且它们各自的保留时间先后有序,互不干扰,从而有效确保了其他核苷不会干扰5-mdC和dG的定量检测分析。
2.4 标准工作曲线和检测限
以混合标样中5-mdC和dG物质的质量比值和它在质谱中的响应面积的比值构建回归曲线,获得定量基因组DNA甲基化率的标准工作曲线(图4,回归曲线方程为y=0.626x+0.0212,R2=0.9999),具备良好的线性相关性,可用于甲基化率的定量分析。1000 ng/mL的5-mdC和dG不断梯度稀释后连续进样分析,在信噪比≥10的条件下,5-mdC的最低定量限(limit of quantitation,LOQ)为 1.65 fmol,dG 的LOQ为2.47 fmol,相当于0.2 ng的DNA发生了1%的甲基化也能检出。
2.5 基质效应
为了排除酶解缓冲液(包括0.1 mmol/L醋酸铵1 μL、1 mol/L碳酸氢铵1 μL、碱性磷酸酶溶解缓冲液1 μL和H2O 7 μL)和H2O对5-mdC和dG的响应值的影响,我们将不同浓度的5-mdC和dG标准品分别加入酶解缓冲液和H2O混匀,进样检测,比较响应值差异,结果见表1、2,响应值比为90%~110%,说明基质效应不会干扰样品的实际检测分析。
2.6 重现性
以全基因组DNA甲基化率分别为1%、5%、10%的标准样品为质量控制(quality control,QC)样品,在1 d内的3个时间点(天内误差)和连续5 d内(天间误差)分别检测分析QC样品,并计算QC样品中5-mdC含量的相对标准偏差(RSD,%)。结果显示质量控制标准样品5-mdC含量的天内误差和天间误差均小于8%,说明该技术具有很好的稳定性。
3 讨论
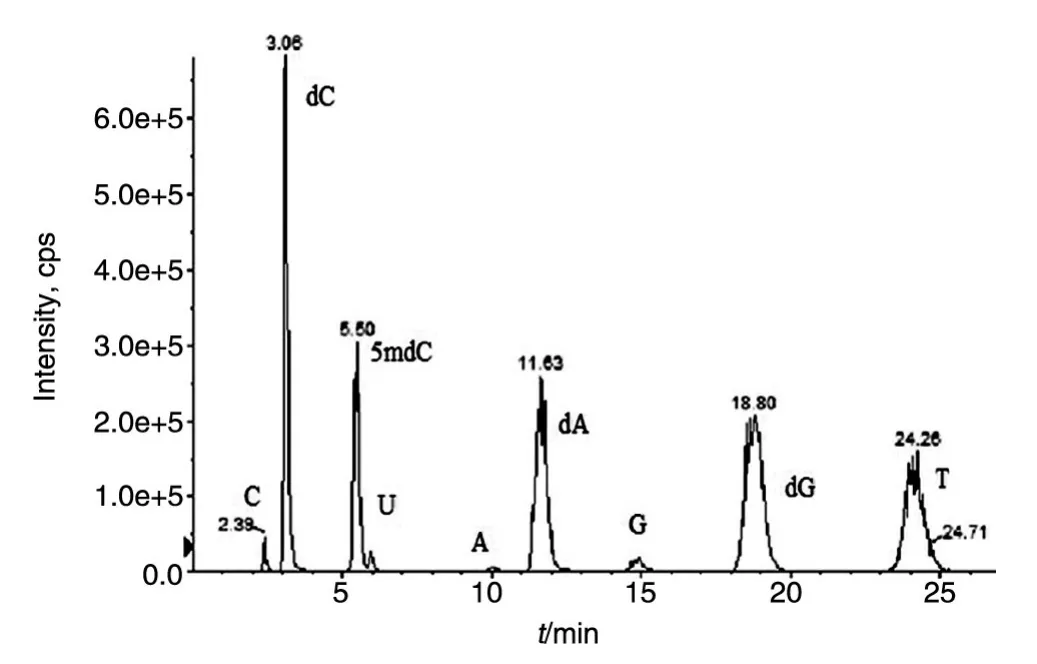
图3 正常和修饰核苷的典型MRM离子流图

图4 基因组DNA甲基化率标准工作曲线
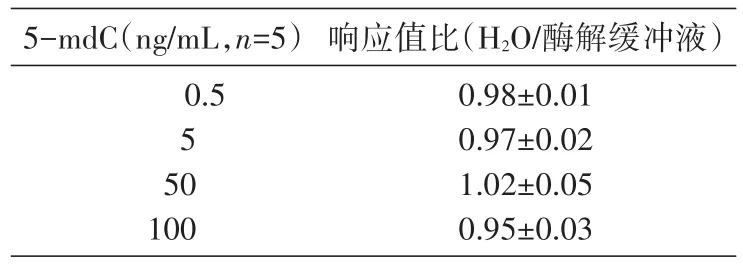
表1 5-mdC的基质效应
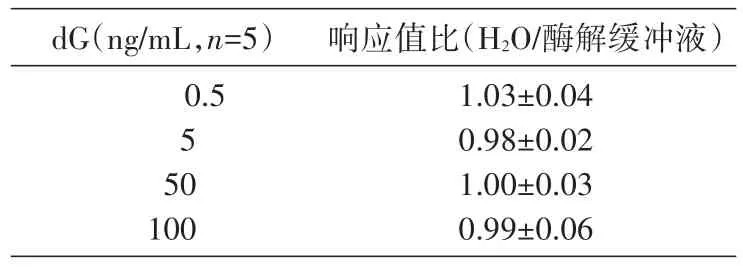
表2 dG的基质效应
近年来,基因组DNA甲基化的研究方法不断发展,主要可分为全基因组甲基化检测和特异基因的甲基化检测。全基因组亚硫酸氢盐和亚硫酸氢盐焦磷酸测序,是以亚硫酸氢盐修饰为基础的检测基因组整体甲基化水平的方法,两者都具有精确度高和能定量的优点[6]。前者还具有较强的可靠性和较高的基础分辨率,但检测过程复杂,既耗时又价格昂贵;后者简单快速,不足之处是只能检测长度小于100 bp的DNA序列[6]。SssⅠ甲基转移酶法是在该酶的作用下使基因组DNA的CpG位点发生甲基化,通过测定剩余的放射性标记的SAM即可得到原基因组整体甲基化水平。这种方法的缺点是所使用的SssⅠ甲基转移酶不稳定,致使结果不够精确,并且常常伴随放射性污染[7]。特异性的抗5-mdC抗体免疫荧光法[8],所需样品量大且精确性较低。高效液相色谱法[9]是一种比较传统的方法,能够定量测定基因组整体DNA甲基化水平,却不能对甲基化的CpG位点进行定位。这些方法都伴随着各种各样的不足与缺陷,给实验者在甲基化检测过程中带来很多困扰。本实验建立的HPLC-ESI-MS/MS技术具有高灵敏度、高通量、高准确率、高分辨率且重复性好的特点,有望成为甲基化研究的关键方法之一,尤其在检测大样本中的微量甲基化时显示了极大优势。
为了消除仪器对化合物响应值的波动所造成的定量误差,HPLC-ESI-MS/MS技术成为DNA甲基化定量方法的核心之一,相关报道采用外标法或利用5-mdC和dC的同位素内标进行定量,包括15N3-dC和15N5-dG[10]、[U-15N]-dC和[U-15N]-5-mdC[11]等,但由于同位素内标价格昂贵和外标操作上的繁琐而不能大量投入使用。我们利用5-mdC和dG标准品构建标准曲线,以dG为内标对全基因组DNA甲基化水平定量分析,利用G只能和C配对的原理,得到了全基因组DNA甲基化率的表达式:甲基化率=100×[5-mdC]/[dG]。虽然DNA序列中的部分dG可发生修饰,但此种修饰的碱基占正常碱基的比例微乎其微,所引起的误差不会对检测结果造成影响,可以忽略不计[10]。并且,以dG为内标,能克服外标法检测分析所存在的信号不稳定和误差较大的问题。
我们建立的HPLC-ESI-MS/MS检测技术具有灵敏度高和特异性好的特点,经过条件优化,母/子离子对能特异地联合鉴定目标化合物,可检出0.2 ng的DNA中发生的1%甲基化,灵敏度可达亿分之一以上。而且,抗干扰能力强,MRM总离子流图显示各核苷能实现良好的基线分离,不会对5-mdC和dG的定量检测分析产生干扰;酶解缓冲液与ddH2O响应值比为90%~110%,基质效应不会影响样品的检测分析。该方法还具备良好的稳定性,质量控制标准样品甲基化率的天内误差和天间误差均小于8%。同时,用该方法所建立的标准工作曲线具备良好的线性相关性,可用于甲基化率的定量分析。
[1]Reik W,Dean W,Walter J,et al.Epigenetic reprogramming in mammalian development[J].Science,2001,293:1089-1093.
[2]Suzuki M M,Bird A.DNA methylation landscapes:provoca⁃tive insights from epigenomics[J].Nat Rev Genet,2008,9(6):465-476.
[3]Issa J P.CpG island methylator phenotype in cancer[J].Nat Rev Cancer,2004,4:988-993.
[4]Curtin K,Slattery M L,Samowitz W S.CpG island methyla⁃tion in colorectal cancer:past,present and future[J].Patholog Res Int,2011,2011:1-8.
[5]Laird P W.The power and the promise of DNA methylation markers[J].Nat Rev Cancer,2003,3(4):253-266.
[6]Frommer J G,McDonald L E,Millar D S,et al.A genomic squencing protocol that yields a positive display of 5-methyl⁃cytosine residues in individualDNA strands[J].Proc Natl Acad Sci USA,1992,89(5):1827-1831.
[7]Wu J,Issa J,Hermen J,et al.Expression of an exogenous eukaryotic DNA methyl transferase gene induces transforma⁃tion of NIN3T3 cells[J].Proc Natl Acad Sci USA,1993,90(19):8891-8895.
[8]Oakeley E J,Podesta A,Jost J P.Developmental changes in DNA methylation of the two tobacco pollen nuclei during mat⁃uration[J].Proc Natl Acad Sci USA,1997,94:11721-11725.
[9]Kuo K C,McCune R A,Gehrke C W,et al.Quantitative re⁃versed-phase high performance liquid chromatographic deter⁃mination of major and modified deoxyribonucleosides in DNA[J].Nucleic Acids Res,1980,8:4763-4776.
[10]Li Xingnan,Franke A A.High-throughput and cost-effective global DNA methylation assay by liquid chromatography-mass spectrometry[J].Anal Chim Acta,2011,703(1):58-63.
[11]Lim U,Flood A,Choi S W,et al.Genomic methylation of leukocyte DNA in relation to colorectal adenoma among as⁃ymptomatic women[J].Gastroenterology,2008,134(1):47-55.
