miR let-7家族在胰腺导管腺癌发生发展中的作用
2014-09-12刘孟刚刘宏鸣杨俊涛
曾 玮 刘孟刚 刘宏鸣 谢 斌 袁 涛 杨俊涛 蓝 翔 陈 平
胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)是临床上常见的消化系统恶性肿瘤,其发病隐匿,预后差,死亡率高,治疗手段有限[1]。而PDAC的发生发展过程中,相关mRNA与ncRNA应该是一个相互调控的动态网络过程[2-3]。据此,本文尝试分析胰腺癌RNA表达数据库内多套mRNA与miRNA的表达谱数据,定义差异表达的分子,并利用已知的“转录因子-miRNA-mRNA”构建调控网络。定义新的PDAC基因与miRNA。分析结果发现一组miRlet-7家族miRNA分子可能在调控网络中其到重要作用,可能参与调控ALDH1A1、ZEB1与HMGA等基因,影响PDAC的发生与发展。
1 材料与方法
1.1 神经胶质瘤mRNA与miRNA表达谱数据
PDAC相关差异表达mRNA与miRNA数据来自于Pancreatic Expression Database(PED)数据库[4]。在数据库内筛选出比较PDAC组织样本与正常组织样本(包括配对样本和非配对样本)的mRNA表达谱数据集共12组,得到差异表达基因(t testP<0.05,|Fold Change|>2)。同时,筛选出比较PDAC组织样本与正常组织样本(包括配对样本和非配对样本)的miRNA表达谱数据集共6组,得到差异表达基因(t testP<0.05,|Fold Change|>2)。
1.2 miRNA与mRNA调控关系数据及miRNA疾病相关数据库
miRNA与mRNA调控关系数据均为文献报道证实数据。其中,转录因子调控miRNA数据来源于transmir数据库[5]。miRNA靶向调控mRNA数据来自miRNAwalk数据库[6]。miRNA与疾病关系数据来源于人类miRNA疾病数据库(HMDD)[7]及人类miRNA疾病与生物学过程数据库(PhenomiR)[8]。已知癌基因数据来自CGC(Cancer Gene Census)数据库[9],共收集488个已知癌基因。
1.3 构建miRNA-mRNA调控网络与核心调控子网络
寻找所有差异RNA分子间存在的调控关系,包括“转录因子-miRNA”以及“miRNA-mRNA”2种调控关系,最后将所有得到的不同分子间的调控关系进行整合,形成miRNA-RNA调控关系网络。
核心调控子网络定义:①所有子网络中节点均为差异表达mRNA或者miRNA;②子网络中转录因子、miRNA及靶基因至少各存在一个;③子网络节点数不得少于5个。
2 结果
2.1 差异表达基因与差异表达miRNA
分析整理PED数据库中的mRNA表达谱数据,共得到465个差异表达的基因。包括392个上调基因,73个下调基因。差异基因中有82个为已知癌基因[9]。所有差异表达基因用biNGO软件[10]进行GOslim富集分析,结果发现差异表达基因大多富集到“细胞增殖”、“细胞分化”、“细胞骨架”及“细胞死亡”等功能(表1)。相关功能都与PDAC发生发展有密切关系[3]。
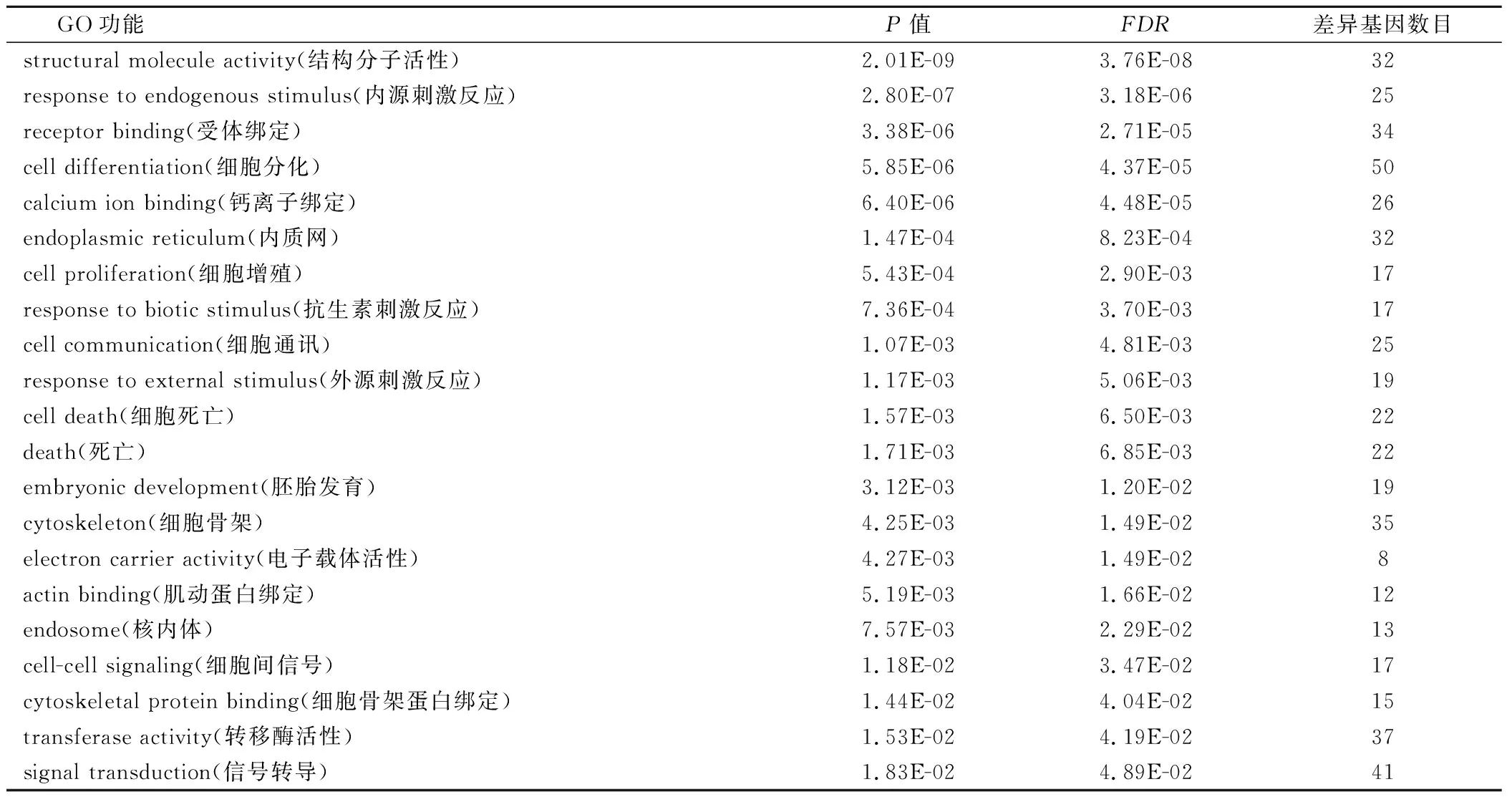
表1 差异表达基因GOslim功能富集结果
分析miRNA表达谱数据,得到97个差异表达的miRNA。包括87个上调的miRNA,10个下调的miRNA。97个miRNA其中有32个文献报道与PDAC疾病相关[7-8]。另外部分miRNA有研究表明与其他肿瘤发生发展密切相关。例如,hsa-mir-125a可以促进非小细胞肺癌细胞的侵袭功能[11],hsa-miR-222可以影响子宫内膜癌的细胞分化[12]。
2.2 构建miRNA-mRNA调控网络
通过transmir数据库[5],发现其中有33个差异表达的miRNA可被11个差异表达的转录因子调控,形成48个“转录因子-miRNA”的调控关系。同时通过miRNAwalk数据库[6],发现其中7个差异miRNA可能调控了47个靶基因,形成了126个“miRNA靶向mRNA”的关系。
将以上2种调控关系整合成一个包含转录因子、miRNA及靶基因的整合网络。该网络包含88个节点,涉及174个调控关系。其中差异表达基因52个,差异表达miRNA 36个。在整合网络中,通过遍历搜索发现6个miR-let-7家族(hsa-let-7i、hsa-let-7d、hsa-let-7b、hsa-let-7g、hsa-let-7c、hsa-let-7e)与周围的分子形成一个涉及到13个节点的子网络(表2)。其中EGR1、TGFB1及ZEB1为对miRNA有调控作用的转录因子,ALDH1A1和HMGA2为靶基因。该5个基因全部属于已知癌基因[9];其中ALDH1A1基因目前已经被证实与PDAC的预后直接相关,可被作为PDAC预后分子标记[13]。HMGA2也被证实与PDAC的恶性程度呈显著正相关,是PDAC非常重要的标记分子[14]。Simone Brabletz等[2]证实ZEB1可与miR-200家族相互调控影响PDAC的发展。这一现象在网络中已有所体现。此外,EGR1与TGFB1很早已被证明与PDAC的发展及转移密切相关[3]。虽然目前已有文献报道miR-let-7家族在胰腺癌组织中呈高表达[15],然而还未有文献报道miR-let-7家族在PDAC发生发展过程中的分子机制,miR-let-7家族与ALDH1A1、ZEB1、HMGA2、TGFB1及EGR1等基因间的调控关系及其与PDAC的发生发展的关系,至今未见文献报道。因此,通过miRNA/mRNA调控网络分析发现,miR let-7与上述分子很可能存在调控关系,进而影响PDAC。

表2 核心调控子网络中基因与miRNA的差异表达情况
3 讨论
miRlet-7家族是生物体内一组进化来源相同的miRNA分子,由于其序列相似,所以在某些情况下,其在生物体内参与的功能有很多类似。目前,已有研究讨论miRlet-7家族与细胞分化以及肿瘤的关系[16-17],曲杰等[16]通过合成hsa-let-7b转染乳腺癌细胞,发现其可以明显降低细胞的迁移。Kong 等[17]发现hsa-let-7可以负向调控EZH2基因的表达,进而影响前列腺癌细胞的结构域增殖。虽然,Kent等[15]已经发现let-7家族在胰腺癌中显著高表达,但未深入研究miR let-7家族在胰腺癌中具体分子机制。本文利用PDAC的全基因组表达数据与miRNA表达数据,寻找定义神经胶质瘤发展过程中的核心mRNA-miRNA调控网络,并由此得到miR-let-7家族相关的“mRNA-miRNA”核心调控网络。其网络中所有其他基因与miRNA目前都已被大量文献证实与PDAC的发生发展有重要关系。此外,部分mRNA-miRNA调控关系也已被研究证实与PDAC相关。所以可能提示,hsa-let-7家族可以与ALDH1A1、ZEB1、HMGA2、TGFB1与EGR1等基因相互调控并影响PDAC的发生发展。因此,深入研究miR-let-7家族与上述分子之间的具体调控机制,并如何影响PDAC的发生发展将是我们下一步工作的重点。
由于肿瘤发病机制的高异质性与复杂性,目前研究积累的成果还远远不能解释PDAC发病的分子机制[18]。定义PDAC相关癌基因与miRNA对研究其发病机理和治疗诊断具有非常重要意义。我们利用大规模胶质瘤样本的RNA分子表达数据,代入“转录因子-miRNA-基因”数据,寻找PDAC发病的调控网络。挖掘批量的PDAC相关分子调控机制与相关RNA分子。此外,我们未使用算法预测的miRNA/mRNA调控关系,只使用目前文献报道过的mRNA/miRNA调控关系,提高了miRNA/mRNA调控网络的准确性与稳定性。我们相信,随着癌症基因与ncRNA的表达数据的进一步积累,同时,伴随着miRNA与基因之间的调控关系数据的进一步增加。通过本文的mRNA/ncRNA调控网络分析方法,将可以挖掘更多的潜在的肿瘤相关RNA分子。
[1] Shi J,Wu C.Meta analyses of risk factors for pancreatic cancer in China 〔J〕.Pancreatic Cancer Study,2004,4(3):154-158.
[2] Simone Brabletz,Karolina Bajdak,Simone Meidhof,et al.The ZEB1/miR-200 feedback loop controls Notch signaling in cancer cells〔J〕.EMBO J,2011,30(4):770-782.
[3] Gong X,Qin XL.Evolve of pancreatic cancer’s medicine cure 〔J〕.Chinese Clinical Oncology,2007,12(1):68-74.
[4] Ma W,Yang D,Gu Y,et al.Finding disease-specific coordinated functions by multi-function genes:insight into the coordination mechanisms in diseases〔J〕.Genomics,2009,94(2):94-100.
[5] Wang J,Lu M,Qiu C,et al.TransmiR:a transcription factor-microRNA regulation database〔J〕.Nucleic Acids Res,2010,38(Database issue):D119-122.
[6] Dweep H,Sticht C,Pandey P,et al.miRWalk--database:p-rediction of possible miRNA binding sites by “walking” the genes of three genomes〔J〕.J Biomed inform,2011,44(5):839-847.
[7] Lu M,Zhang Q,Deng M,et al.An analysis of human microRNA and disease associations〔J〕.Plos One,2008,3(10):3420.
[8] Ruepp A,Kowarsch A,Schmidl D,et al.PhenomiR:a kno-wledgebase for microRNA expression in diseases and biological processes〔J〕.Genome Biology,2010,11(1):R6.
[9] Futreal PA,Coin L,Marshall M,et al.A census of human cancer genes〔J〕.Nature Reviews,2004,4(3):177-183.
[10] Maere S,Heymans K,Kuiper M.BiNGO:a Cytoscape plugin to assess overrepresentation of gene ontology categories in biological networks〔J〕.Bioinformatics,2005,21(16):3448-3449.
[11] 江黎黎,张清富,常继红,等.hsa-miR-125a-5p促进非小细胞肺癌细胞侵袭〔J〕.中国肺癌杂志,2009,12(9):951-955.
[12] Qian K,Hu L,Chen H,et al.Hsa-miR-222 is involved in differentiation of endometrial stromal cells in vitro〔J〕.Endocrinology,2009,150(10):4734-4743.
[13] Kahlert C,Bergmann F,Beck J,et al.Low expression of aldehyde dehydrogenase 1A1(ALDH1A1)is a prognostic marker for poor survival in pancreatic cancer〔J〕.BMC Cancer,2011,11(3):275.
[14] Piscuoglio S,Zlobec I,Pallante P,et al.HMGA1 and HMGA2 protein expression correlates with advanced tumour grade and lymph node metastasis in pancreatic adenocarcinoma〔J〕.Histopathology,2012,60(3):397-404.
[15] Kent OA,Mullendore M,Wentzel EA,et al.A resource for analysis of microRNA expression and function in pancreatic ductal adenocarcinoma cells〔J〕.Cancer Biol Ther,2009,8(21):2013-2024.
[16] 曲 杰,王胜林,吕喜英,等.MiR let-7b 抑制乳腺癌MCF-7 细胞迁移及其机制〔J〕.江苏大学学报(医学版),2012,22(4):328-331.
[17] Kong D,Heath E,Chen W,et al.Loss of let-7 up-regulates EZH2 in prostate cancer consistent with the acquisition of cancer stem cell signatures that are attenuated by BR-DiM〔J〕.PLos One,2012,7(3):33729.
[18] Abramson MA,Jazag A,van der Zee JA,et al.The Molecular Biology of Pancreatic Cancer〔J〕.Gastrointest Cancer Res,2007,1(4Suppl 2):S7-S12.
