春大豆花芽cDNA文库的构建及质量分析
2014-09-10王楠周莹姚丹尹俊琦曲静王丕武
王楠,周莹,姚丹,尹俊琦,曲静,王丕武
(1吉林农业大学农学院,吉林长春 130118;2吉林农业大学生命科学学院,吉林长春 130118)
春大豆花芽cDNA文库的构建及质量分析
王楠1,周莹1,姚丹2,尹俊琦1,曲静1,王丕武1
(1吉林农业大学农学院,吉林长春 130118;2吉林农业大学生命科学学院,吉林长春 130118)
【目的】为了解大豆多荚、多粒相关基因的调控机制,构建了大豆花芽cDNA文库,根据要求初步鉴定了所构建文库的质量.【方法】以大豆吉农18突变体的幼嫩花芽为材料提取总RNA,采用SMART技术合成双链cDNA.经蛋白酶K的消化及SfiⅠ酶切后,将所得cDNA克隆到λTriplEx质粒载体中,成功构建了大豆花芽全长cDNA文库.【结果和结论】将初始文库经扩增后保存,检测扩增文库滴度为2.13×108pfu/mL,重组率接近95.3%,菌落PCR鉴定插入片段主要分布在0.5~2.0 kb.插入片段平均大小在1.0 kb左右.表明本研究所构建的文库既满足了目的基因的分离筛选,又可保证全长cDNA文库的获得,该文库的构建为进一步开展相关基因的克隆及分子生物学研究奠定基础.
大豆;花芽;cDNA文库构建;质量分析
cDNA文库是高效、大规模获得基因序列信息的有效方法,为新基因的发现和功能基因的研究提供便捷、高效的途径.与其他基因组文库相比较,cDNA文库中获得的基因组是已经经过剪切将内含子去除了的cDNA,因此可以在cDNA文库中较便捷地获得基因序列的信息[1],并且可以直接用于该基因完整的mRNA信息的表达[2].通过构建cDNA表达文库不仅可保护濒危珍惜生物资源,而且可以提供构建分子标记连锁图谱所用的探针,更重要的是可以用于分离全长基因进而开展基因功能研究.
我国北方春大豆生长一般分为出苗期、开花期、结荚期、鼓粒期和成熟期5个生育阶段.根据以往的研究,春大豆花芽分化过程中干物质积累量会随着花芽分化迅速上升,单位面积干物质量的增加可达到提高产量的目的[3-5].春大豆花芽分化时主茎伸展4~5片复叶,此时是营养生长与生殖生长交错期,大豆植株生活力旺盛、生长发育快、营养器官与生殖器官同时建造,此时花芽部位所含基因丰富,为构建全长cDNA文库奠定基础.目前,对春大豆花芽的研究多限于生理及表观形态学的研究,如陈传梅等[6]研究的大豆花芽分化和物候期的机理模型,吕薇等[7]研究的大豆花芽分化和发育的扫描电子显微镜观察;另有对大豆成花诱导、花发育及开花逆转过程中的相关基因的研究,如马启斌[8]关于GmNMH7基因在大豆成花诱导、花发育及开花逆转过程中的表达研究.但从分子水平揭示其与产量相关性的研究甚少,只有在柑橘[9]、擎天凤梨[10]等方面有相关报道.本试验选取大豆吉农18突变体为材料,构建突变体幼嫩花芽全长cDNA文库,以期从分子生物学角度研究调控大豆多荚、多粒相关机制,从而为大豆相关基因的克隆及增产的分子机理研究提供依据.
1 材料与方法
1.1 材料
1.1.1 植物材料本研究选取大豆吉农18突变体为试验材料,该材料单株4粒荚结荚率为30%左右,与对照材料比,提高近5倍;突变体小区产量较对照材料提高15%左右.2012年7月在大棚内试验田中取大豆吉农18突变体6~7叶期的幼嫩花芽,用锡箔纸包裹迅速置于液氮中速冻后,储存于-80℃超低温冰箱备用.
1.1.2 引物序列合成cDNA第1条链引物CDSⅢ/3'PCR Primer(12 μmol/L):5'-ATTCTAGAGGCCGAGGC GGCCGACATG-d(T)30N-1N-3'(N=A,G,C或T;N-1=A,G或C),SMART IVTM Oligo-nucleotide(12μmol/L):5'-AAGCAGTGGTATCAACGCAGAGTGGCCATTACGGCCGGG-3';合成cDNA第2条链引物5'PCR Primer(12 μmol/L):5'-AAGCAGTGGTATCAACGCAGAGT-3'.
1.1.3 试剂RNAiso Plus总RNA提取试剂盒,购自TaKaRa公司;文库构建试剂盒SMART cDNA Library construction kit,购自Clontech公司,其中包含宿主菌大肠埃希菌XL1-Blue菌株、BM25.8菌株及TripIEx2载体;双链cDNA合成试剂盒Advantage®2 PCR Kit,购自Clontech公司;包装蛋白MaxPlaxTMLambda packaging extract,购自EPICENTRE公司.
1.2 试验方法
1.2.1 总RNA的提取及mRNA的纯化取1 g大豆叶片,按照RNAiso Plus试剂盒使用说明书的步骤提取大豆叶片总RNA.取3 μL上样量,电压为8~10 V/cm,经φ为1%甲醛变性凝胶电泳,检测提取RNA的质量及完整性.利用DNaseI消化,消除样品中可能含有的微量基因组DNA,用微量分光光度计检测总RNA的浓度和纯度.
1.2.2 双链cDNA的合成取3.0 μL纯化后的mRNA,按照SMART cDNA Library construction kit试剂盒说明书中步骤,首先合成单链cDNA,孵育结束后放在冰上终止反应.取2.0 μL单链cDNA,按照SMART cDNA Library construction kit试剂盒说明书中步骤,进行以下反应:95℃5 min;95℃30 s,68℃6 min,30个循环;最后68℃6 min结束反应.取5μL产物进行11 g/L的琼脂糖凝胶电泳,检测产物质量.
1.2.3 蛋白酶K的消化及SfiⅠ酶切取50.0 μL双链cDNA(2~3 μg)于高温灭菌的0.5 mL离心管中,按照SMART cDNA Library construction kit试剂盒说明书步骤操作,所得沉淀干燥后加入79 μL去离子水回溶.向回溶的cDNA中加入下列试剂:10×SfiⅠ缓冲液10 μL;SfiⅠ酶10 μL;100×BSA 1 μL;混用后50℃孵育2 h,孵育后加入2 μL φ为1%二甲苯氰混匀.
1.2.4 cDNA的分级分离按试剂盒说明书要求准备好16个1.5 mL离心管及CHROMA SPIN-400分级分离柱.将柱内基质摇匀,消除柱内气泡,移除底盖使柱内缓冲液流尽.加入过柱缓冲液700 μL使其自然流尽.将混有二甲苯氰的cDNA加入到柱中,待cDNA渗入基质后加入过柱缓冲液100 μL.待自然流尽后加入过柱缓冲液600 μL,将制备好的16个离心管迅速放在柱下方,每管1滴,直到缓冲液流尽为止.每管取3 μL进行11 g/L琼脂糖凝胶电泳,150 V电泳10 min.选择符合试验要求的3~4管,收集到新的离心管中.加入醋酸钠(3 mol/L;pH 4.8)1.6 μL,糖原(20 mg/mL)1.3 μL,φ为95%乙醇溶液(-20℃) 408 μL,-20℃冰浴过夜.14 000 r/min离心20 min,小心移除上清后用去离子水7 μL重悬沉淀.
1.2.5 cDNA克隆入TriplEx2载体为保证目的片段与载体更好地连接,本试验设立3个连接反应梯度(表1),以建立最优的连接效率.
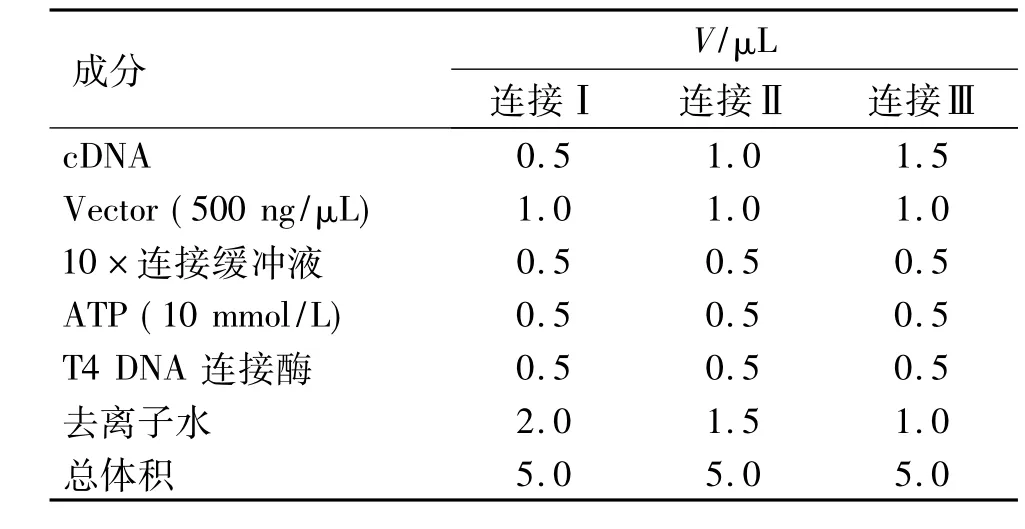
表1 梯度连接反应Tab.1 Ligations using three different ratios of cDNA to phage vector
1.2.6 包装反应3个连接产物分别用EPICENTR公司的MaxPlaxTMLambda packaging extracts包装,体外包装方法按试剂盒说明书执行,所得混合液即为原始cDNA文库.
1.2.7 文库质量的鉴定1)文库滴度的检测:取1 μL包装好的连接产物,用1×Lambda dilution buffer按1∶100稀释.取稀释产物10 μL,加入过夜培养的XL1-Blue 200 μL,37℃条件培养15 min.之后加入3 mL熔化的LB/MgSO4顶层琼脂培养基,快速混匀并倒在37℃预热的90 mm LB/MgSO4平板上,快速旋转平板使顶层琼脂培养基均匀分布在平板上.37℃条件培养6~18 h,每2 h观察1次噬菌斑生长情况.

2)文库重组率的检测:应用蓝白斑筛选鉴定文库重组率,在3 mL熔化的LB/MgSO4顶层琼脂培养基中加入IPTG(0.1 mol/L)50 μL,X-Gal(0.1 mol/L)50 μL,根据噬菌斑数量计算文库滴度及重组率.

3)文库插入片段大小的检测:随机挑取培养皿中的噬菌斑进行菌液PCR.PCR反应条件:94℃预变性5 min;94℃1 min,55℃30 s,72℃2 min,35个循环;72℃延伸7 min.反应结束后取5 μL进行11 g/L琼脂糖凝胶电泳检测.通过电泳筛选阳性克隆,送北京三博远志公司测序.
1.2.8 序列分析利用NCBI的VecScreen程序对所测序列进行识别,去除载体,利用DNAStar软件SeqMan程序做多序列聚类比对,将获得的EST序列在GenBank上进行BLAST比对分析.
2 结果与分析
2.1 总RNA的提取及mRNA的纯化
本研究利用RNAiso Plus试剂盒提取大豆花芽总RNA,经φ为1%甲醛变性凝胶电泳检测,结果(图1)表明:本研究提取的吉农18突变体材料总RNA中28.0 s、18.0 s和5.8 s条带清晰,28.0 s条带亮度基本上是18.0 s条带亮度的2倍,并且条带清晰,说明所提取的RNA完整性较好,质量较高.经NanoDrop(ND-1000)Spectrophotometer测定其在230、260及280 nm处的吸收值(D),结果显示其D260nm/D280nm均在1.8~2.0之间,D260nm/D230nm均在2.2~2.6之间,RNA质量浓度为219~324 ng/μL,说明蛋白质及盐类等污染较小,所提RNA纯度较高,达到反转录所需RNA浓度,符合构建cDNA文库的要求.

图1 吉农18大豆花芽突变体总RNA电泳图Fig.1 Electrophoresis of total RNA from flower bud mutants of soybean Jinong 18
2.2 双链cDNA的合成
取5 μL双链cDNA经11 g/L琼脂糖凝胶电泳检测,从图2中可以看出,cDNA条带基本在0.5~2.0 kb间弥散,且集中在0.75~1.00 kb之间,中间有几条与组织特异性高丰度mRNA相对应的亮带,结果表明双链cDNA合成符合文库构建标准.
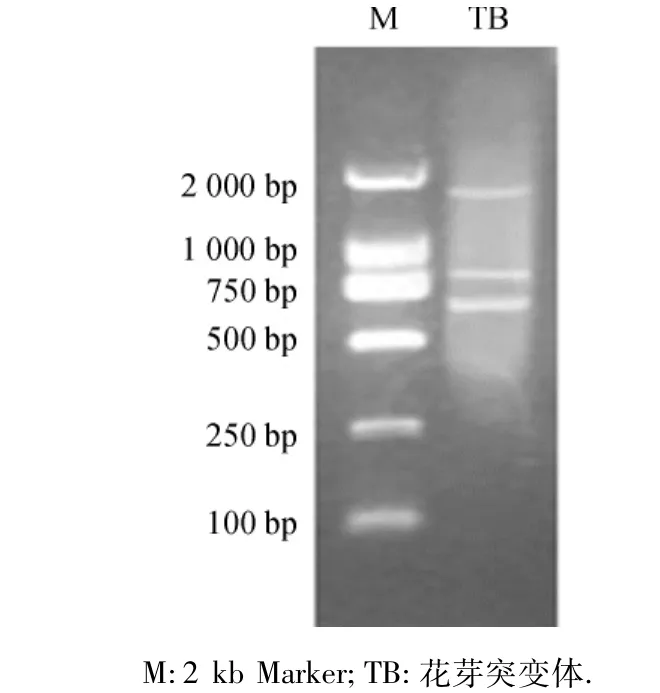
图2 吉农18大豆花芽突变体双链cDNA电泳图Fig.2 Electrophoresis of ds cDNA from flower bud mutants of soybean Jinong 18
2.3 cDNA的分级分离
双链cDNA合成后通过蛋白酶K的消化和SfiⅠ酶切后,用CHROMA SPIN-400柱分级分离,将引物及小片段cDNA(<0.5 kb)过滤掉.经11 g/L琼脂糖凝胶电泳检测,结果(图3)表明:16管中1~4号管、9~16号管没有条带,5~8号管有清晰的、大小为500~2 000 bp的条带,根据文库构建的标准,收集大小在500~2 000 bp之间的条带,因此,本研究收集5~8号的离心管用于后续文库构建.
2.4 cDNA文库质量的检测
2.4.1 cDNA克隆入λTriplEx2载体和库容量的检测按照不同比例,设置3个cDNA与载体的连接反应,包装后侵染E.coliXL1-Blue菌株,观察噬菌斑的数量,并计算噬菌体滴度,选择最佳的连接效率.未扩增文库滴度测试结果见表2.

图3 吉农18大豆花芽突变体cDNA片段分级分离电泳图Fig.3 Electrophoresis of cDNA size of fractionation from flower bud mutants of soybean Jinong 18
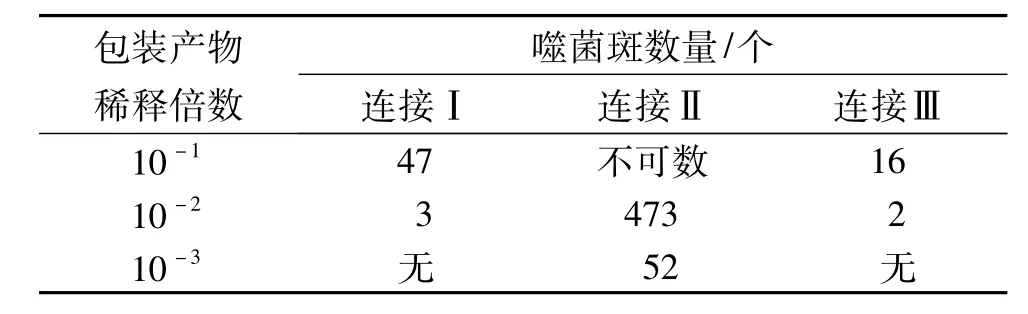
表2 未扩增文库滴度测试结果Tab.2 Results of an unamplified library titer test
经检测,原始文库滴度为1.3×106pfu/mL,重组率为93.6%.扩增文库的滴度为2.13×108pfu/mL,重组率为95.3%.重组率大于80%,文库滴度大于1.0×106pfu/mL的文库即为合格文库,因此本研究所构建的文库为合格的cDNA文库.
2.4.2 文库插入片段大小的检测从文库中随机挑取30个单克隆,进行菌落PCR检测.检测结果(图4)表明,挑取的30个克隆均出现清晰条带,条带的分布主要集中在0.75~2.00 kb之间.其中,0.20~0.50 kb间有1个克隆,占总克隆数的3.3%;0.50~0.75 kb间有7个克隆,占总克隆数的23.3%,0.75~1.00 kb间有12个克隆,占总克隆数的40.0%;1.00~2.00 kb间有10个克隆,占总克隆数的33.3%;平均长度为1.0 kb左右.进一步检测结果证明,本研究构建的大豆花芽cDNA文库完全满足cDNA文库构建的库容要求.

图4 吉农18大豆花芽突变体cDNA文库中插入片段大小凝胶电泳图Fig.4 Electrophoresis of inserts in the library from flower bud mutants of soybean Jinong 18 by PCR
2.5 测序及EST分析
从cDNA原始文库中随机挑取53个克隆进行测序,利用NCBI的VecScreen程序对所测序列进行识别,去除载体序列、短序列和低相对分子质量序列后共获得有效序列42条,有效序列大小在117~523 bp之间,其中序列大小为100~200 bp的有9条,占总数的21.4%;200~300 bp的有16条,占总数的38.1%; 300~400 bp的有13条,占总数的31.0%;400~500 bp的有3条,占总数的7.1%;500~600 bp的有1条,占总数的2.4%.利用Phrap程序进行多序列聚类拼接,获得23个非重复序列,将获得的EST序列利用NCBI的BlastX进行相似性的比对分析,结果如表3.
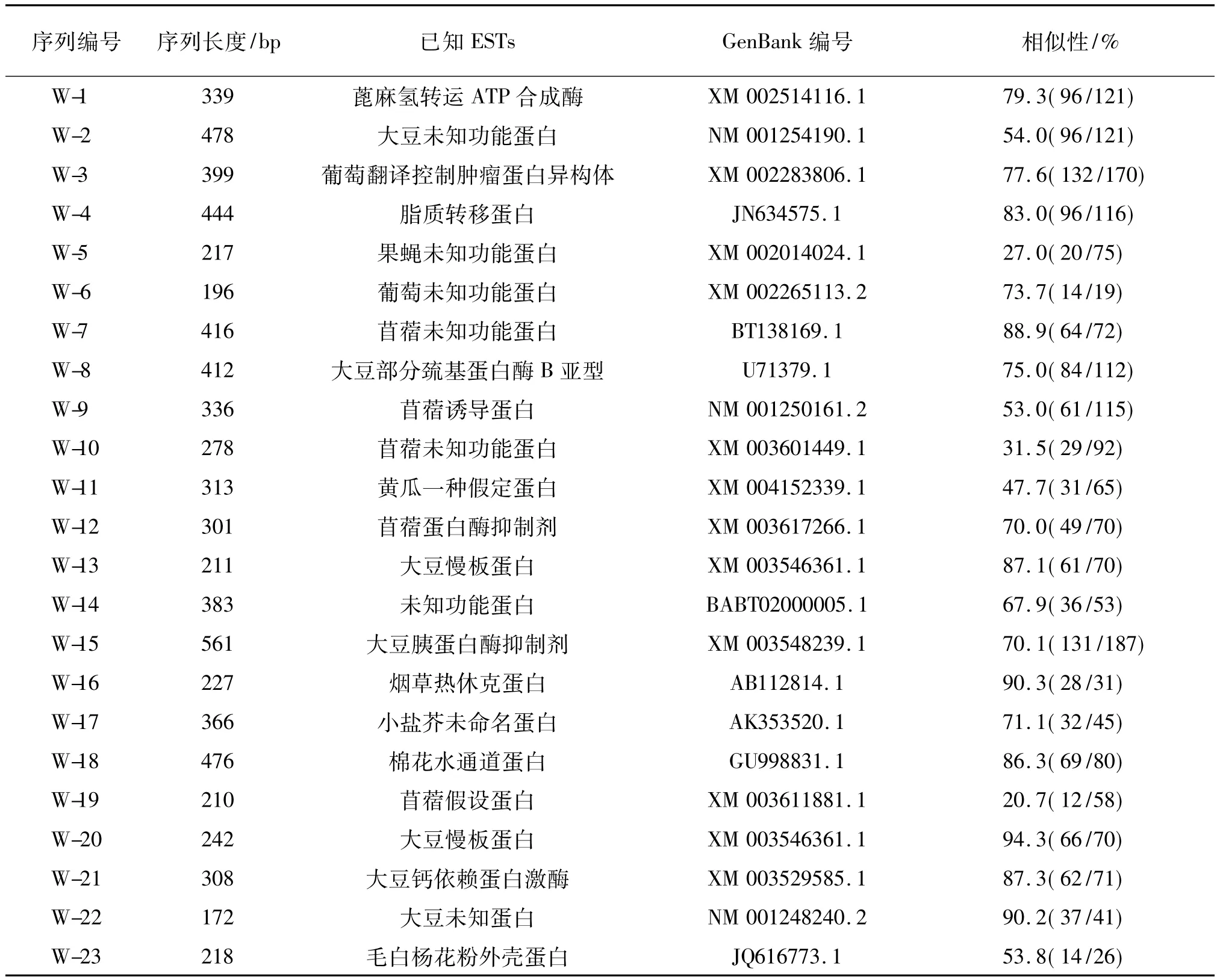
表3 功能已知ESTs的比对结果(BlastX)Tab.3 The results(BlastX)for a comparison between homology and function genes deposited in GenBank
3 讨论
3.1 大豆花芽cDNA文库质量评价
评价cDNA文库的质量主要包括2个方面:一是cDNA文库的代表性,即mRNA的种类,它可以用一个量化的标准来衡量,就是cDNA文库的库容量[11].本试验构建的原始cDNA文库的滴度为1.3×106pfu/mL,一般文库滴度要求不小于1.0×106pfu/mL.与目前已经构建的全长cDNA文库相比较,董志敏等[12]构建的大豆叶片全长cDNA文库原始滴度为2.13×108pfu/mL;房学爽等[13]构建的珙桐叶片cDNA文库原始滴度为6.3×106pfu/mL.由此可证明本试验所构建的cDNA文库可以满足大规模测序所需.二是重组cDNA片段序列的完整性,即重组的cDNA片段足够长,可以反映出基因的天然结构.本试验所构建的cDNA文库的插入片段在0.5 kb以上的占96.6%,平均长度在1.0 kb左右,表明本试验获得了较高比率的全长cDNA.
3.2 SMART法全长cDNA文库的构建
cDNA文库的建立,可以获得更多拥有优良性状的基因,cDNA文库的质量决定了是否能获得全长的cDNA克隆[14-15].构建高质量的cDNA文库的先决条件是高质量、高纯度的RNA.以往常用的cDNA合成方法中,都要有足够的试验材料来提取高纯度的mRNA,而SMART技术弥补了这一缺点,可以直接利用总RNA来合成cDNA[16-17],并且起始材料用量较少,使用0.05~1.00 μg的总RNA就可以利用LDPCR技术构建双链cDNA,获得一个大于106 pfu的全长cDNA文库,对于稀有材料而言具有较高的应用价值[12,18].通过LD-PCR的方法,避免了当mRNA内部存在二级结构,或是mRNA长度过长时不能反转录完全的现象发生.LD-PCR扩增中使用的聚合酶混合物包含N-末端缺失突变的Taq酶、具有3'、5'端矫正功能的聚合酶以及特有的用于自然热启动的抗体,使合成的cDNA提高了精确度和高效性.cDNA通过分级分离后,过滤掉了较小的cDNA片段和酶切反应后DNA片段的残留物,不仅保证了大片段cDNA的富集,还减少了后期筛选的工作量[19].SMART技术中提供的SfiⅠ酶含有2个不同的酶切位点,可保证cDNA以正确的方向连接到载体中,提高了表达的机率.在进行SfiⅠ酶切反应时,要注意控制酶的用量,酶的浓度过大可能使酶产生信号活性,酶的体积过低会明显抑制酶的活性[20].本试验构建库试剂盒中所提供的载体为噬菌体,利用该载体构建的cDNA文库库容量大,可以筛选出低丰度的mRNA,而通过质粒载体构建的cDNA文库包含的克隆数较少,只能筛选出高丰度的mRNA,因此考虑到大豆中可能含有低丰度的mRNA的情况,因此选择λ噬菌体为载体进行连接反应可保证构建文库的质量.将制备好的cDNA克隆到载体中,通过平行试验决定插入片段和载体的最佳比例,一般推荐的二者之比在2~5之间,如果比值较大会直接降低连接效率,比值过小容易使DNA造成自连.此外,SMART技术提供的噬菌体臂中包含LacZ和Ampr2个筛选基因,以不同的读码框架通过插入区,从而增加了插入区基因的表达概率,并且可以进行蓝白斑筛选,提高了产生高滴度文库的机率[21].
3.3 EST分析
目前,大豆花芽的遗传规律研究较少,本研究经初步测序获得了一些关于初级代谢、蛋白质合成的相关基因.后期研究将通过双向测序、Primer walking测序等手段,完成部分基因的全长cDNA的获得,在后续的研究中,如果通过上述方法还是不能获得全长cDNA的少数基因,可通过文库杂交等方法获得.
致谢:感谢吉林农业大学生物技术中心对本研究的支持和帮助!
[1]CARNINCI P,SHIBATA Y,HAYATSU N,et al.Normalization and subtraction of cap-trapper-selected cDNAs to prepare full-length cDNA libraries for rapid discovery of new genes[J].Genome Res,2000,10(10):1617-1630.
[2]晏慧君,黄兴奇,程在全.cDNA文库构建策略及其分析研究进展[J].云南农业大学学报,2006,21(1):1-6.
[3]王四清,高聚林,刘克礼,等.大豆花荚形成与花荚脱落的研究[J].内蒙古农业科技,2006(2):8-10.
[4]郑丽娜,周明兵.毛竹快速拔节过程节间组织cDNA文库的构建与分析[J].福建林业科技,2012,39(3):19-23.
[5]何德,李翠新.重金属胁迫后旱柳全长cDNA文库的构建[J].广西林业科学,2012,41(3):221-224
[6]陈传梅,赵达,李志刚,等.大豆花芽分化和物候期的机理模型[J].中国油料作物学报,2012,34(5):502-507.
[7]吕薇,崔琳,王学东.大豆花芽分化和发育的扫描电子显微镜观察[J].电子显微学报,2009,28(6):585-590.
[8]马启彬.GmNMH7基因在大豆成花诱导、花发育及开花逆转过程中的表达[D].北京:中国农业科学院,2003.
[9]郑铁庚.椪柑花器官和幼果的cDNA文库构建及EST分析[D].武汉:华中农业大学,2008.
[10]刘建新,沈福泉,田丹青,等.擎天凤梨花器官全长cDNA文库的构建及EST分析[J].分子植物育种,2009,7(6):1137-1143.
[11]龚达平,解敏敏,孙玉合.烟草叶片全长cDNA文库构建及EST序列分析[J].中国农业科学,2012,45(9):1696-1702
[12]董志敏,李英慧,张宝石,等.大豆叶片全长cDNA文库的构建与鉴定[J].作物杂志,2006(5):1-4.
[13]房学爽.珙桐叶片cDNA文库构建[D].长沙:中南林业科技大学,2008.
[14]钟肖芬,卫剑文,赵贵军,等.平颏海蛇毒腺cDNA表达文库的构建[J].中山大学学报:自然科学版,2001,40 (3):66-69.
[15]刘文华,王义良,陈慧萍,等.玫瑰红绿海葵触手cDNA表达文库的构建和初步分析[J].生物工程学报,2002,18(6):749-753.
[16]涂艳阳,徐如祥,杨志林,等,人脑瘤组织全长cDNA文库的构建[J].第一军医大学学报,2003,23(9):916-920.
[17]CHENCHIK A,ZHU Y Y,DIATCHENKD L,et al.Generation and use of high-quality cDNA from small amout of total RNA by SMART PCR[M]∥SIEBER P D,LARRICK J W.RT-PCR methods for gene cloning and analysis.Natick,MA:Eaton Publishing,1998:305-319.
[18]毛新国,景蕊莲,孔秀英,等.几种全长cDNA文库构建方法比较[J].遗传,2006,28(7):865-873.
[19]WELLENREU T R,SCHUPP I,POUSTKA A,et al,The Germ an cDNA Consortum1 SMART Amplification combined with cDNA size fractionation in order to obtain large full-length clones1BMC[J].Genomics,2004,5(36):1-8.
[20]王敏,刘萍,石明旺,等.野生大豆种子cDNA文库的构建与分析[J].生物技术通讯,2005,16(5):509-511.
[21]朱利军,长孙东亭,罗素兰.全长cDNA文库构建方法及应用研究[J].海南大学学报:自然科学版,2009,27 (2):185-190.
【责任编辑周志红】
Construction and quality analysis of cDNA library from flower buds of spring soybean cultivars
WANG Nan1,ZHOU Ying1,YAO Dan2,YIN Junqi1,QU Jing1,WANG Piwu1
(1 College of Agronomy,Jilin Agricultural University,Changchun 130118,China; 2 College of Life Science,Jilin Agricultural University,Changchun 130118,China)
【Objective】To understand soybean pods and multigrain gene regulation mechanisms of improving soybean production,a soybean flower bud cDNA library was constructed.The quality of library construction was initially identified according to the requirements.【Method】In order to study the novel genes from flower bud mutants of soybean,a full-length cDNA library from flower bud mutants of soybean Jinong 18 was constructed.Total RNA from young flower bud mutants of soybean was extracted.Double strand cDNA was synthesized by SMART method.After proteinase K digestion andSfiⅠdigestion,the ds cDNA fragments were ligated to the λTriplEx vector.A cDNA library of soybean was successfully constructed.【Result and conclusion】With the unamplified library stored after amplification,the titer of the amplified library was estimated as 2.13×108pfu/mL and the recombination rate was approximately 95.3%.PCR results showed that the inserts varied from 0.5 to 2.0 kb with an average size of 1.0 kb or so.It indicated that this library could be used for full-length genes screening and cloning of low abundance genes.The library will lay a foundation for the genes clon and molecular biological research.
soybean;flower bud;cDNA library construction;quality analysis
Q 785;S336
A
1001-411X(2014)01-0073-06
王楠,周莹,姚丹,等.春大豆花芽cDNA文库的构建及质量分析[J].华南农业大学学报,2014,35(1):73-78.
2013-01-24优先出版时间:2013-11-07
优先出版网址:http:∥www.cnki.net/kcms/detail/44.1110.S.20131107.1610.014.html
王楠(1987—),女,硕士,E-mail:759774740qq.com;通信作者:王丕武(1958—),男,教授,博士,E-mail: peiwuw@yahoo.com.cn
吉林省科技支撑重点项目(20090203);吉林省发改委科学技术研究项目(20090001)
