小片段干扰RNA抑制人肝癌细胞株中YAP基因表达的研究
2014-09-07张素青陈橼张一心刘继斌朱烨菁钱俊李德春
张素青 陈橼 张一心 刘继斌 朱烨菁 钱俊 李德春
(1.苏州大学附属第一医院普外科,江苏苏州 215006;2.江苏省南通市肿瘤医院普外科,江苏南通 226361;3.南通大学临床医学系,江苏南通 226019)
目前认为,癌基因的激活与肿瘤发病密切相关,阻断癌基因的高表达可以防止正常细胞癌变,甚至可使恶性肿瘤细胞转为正常细胞[1]。YAP(yes-associated protein)是一种癌基因,其通过Hippo通路促进细胞凋亡,进而导致细胞接触性抑制的丧失,促进细胞癌变[2]。之前,我们的研究认为,抑制YAP的表达可望提高化疗药物治疗肿瘤的疗效。本研究则利用小片段干扰RNA(small interfering RNA,siRNA)来抑制YAP基因表达以观察肝癌细胞株对阿霉素(adriamycin,ADM)的敏感性变化。
1 资料与方法
1.1 细胞株和试剂 肝癌细胞株Huh-7、PLC/PRF/5、MHCC 97H、MHCC 97L购自中国科学院上海细胞生物学研究所。脂质体LipofectamineTM 2000购自美国Invitrogen公司。胎牛血清、DMEM培养液、ADM、四甲基偶氮唑蓝(methyl thialzolyl tetrazolium,MTT)及二甲基亚砜、蛋白免疫印迹(Western blotting)试剂盒、标准蛋白均由上海睿智化学研究有限公司提供。兔抗人YAP一抗购自美国ImmunoWay公司。羊抗兔二抗购自北京中衫金桥生物技术有限公司。siRNA序列由上海吉玛制药技术有限公司合成。
1.2 方法
1.2.1 siRNA序列的设计 参照文献[3]采用在线设计软件设计并筛选出了针对YAP基因启动子区域的siRNA序列。上游引物:5′-GACGACCAAUAGCUCAGAUTT-3′,下游引物:5′-UCUGAGCUAUUGGUC GUCTT-3′。
1.2.2 细胞培养及瞬时转染 肝癌细胞株Huh-7、PLC/PRF/5、MHCC 97H、MHCC 97L用10%胎牛血清的DMEM培养液,置于37℃、CO2体积分数为5%的条件下中培养3 d,定期观察细胞的生长情况,培养3 d时用胰酶消化并传代。消化Huh-7、PLC/PRF/5、MHCC 97H、MHCC 97L细胞后接种于24孔培养板中继续培养,待细胞融合度为70%时,换为无血清培养液,并按脂质体Lipofecta-mineTM 2000的说明书将siRNA-1、siRNA-2、siRNA-3、siRNA-4分别转染肝癌细胞株,同时设立不转染siRNA的空白组、荧光标记组、对照组(NC组)。转染4 h后,换为DMEM完全培养液培养,24 h后用于荧光显微镜下观察转染效果。48 h后收集转染效率最高的细胞,提取蛋白;采用蛋白免疫印迹(Western blotting)法检测YAP的表达。
1.2.3 MTT比色法检测细胞的存活率 按每孔5000个细胞的密度接种于96孔板中,置于37 ℃、CO2体积分数为5%的培养箱中培养24 h,然后按脂质体Lipofectamine TM 2000的说明书以siRNA瞬时转染,转染4 h后加入不同浓度(0、0.2、0.4、0.6、0.8、1.0、1.5、2.0 μg/mL)的ADM,继续培养48 h后每孔加入MTT液10 μL继续培养4 h,然后每孔加入三联溶解液(10%SDS,5%异丁醇,0.01 mol/L HCl)100 μL过夜,测定各孔在波长560 nm/700 nm处的光密度(A)值。空白对照组的孔中加入等量DMEM培养液代替ADM/siRNA,每组培养的细胞均重复3个孔。根据公式计算细胞的存活率:细胞存活率=(实验组A值/对照组A值)×100%。
1.3 统计学处理 采用SPSS 11.0软件进行统计学处理,组间比较采用t检验,免疫组织化学染色结果的比较采用χ2检验,以P<0.05为差异有统计学意义。
2 结 果
2.1 siRNA转染对肝癌细胞株生物学特性的影响 siRNA转染24 h后,4种肝癌细胞均缩小,大小不等,胞质凝缩,细胞增殖受到抑制;而对照组细胞无明显变化。见图1。

(见转续)
(续图1)
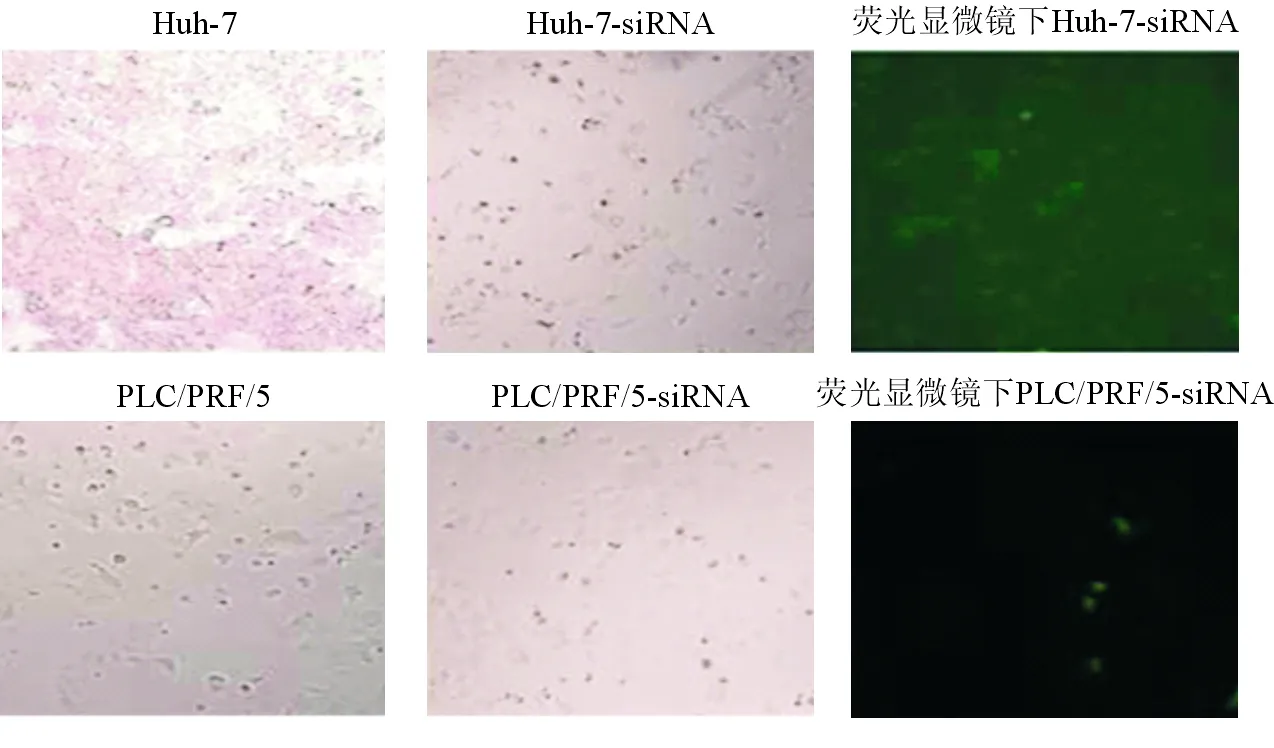
左侧为空白对照组,中间为siRNA转染组,右侧为转染后荧光显微镜下表现
2.2 Western blotting检测siRNA转染48 h后的YAP表达水平 用siRNA转染Huh-7、PLC/PRF/5、MHCC 97H、MHCC 97L后与相应的对照组(NC组)进行比较,YAP的表达减少。见图2。
2.3 MTT比色法检测肝癌细胞的存活率 各肝癌细胞株均以siRNA转染,转染4 h后给予ADM。结果显示,siRNA转染组存活率明显低于对照组(P<0.05)。见图3。
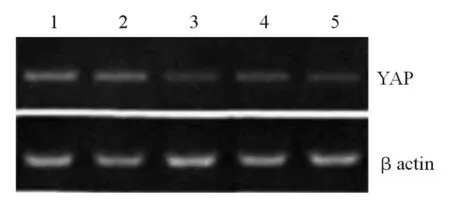
1:NC组;2:Huh-7-siRNA组;3:PLC/PRF/5-siRNA组;4:MHCC 97H-siRNA组;5:MHCC 97L-siRNA组
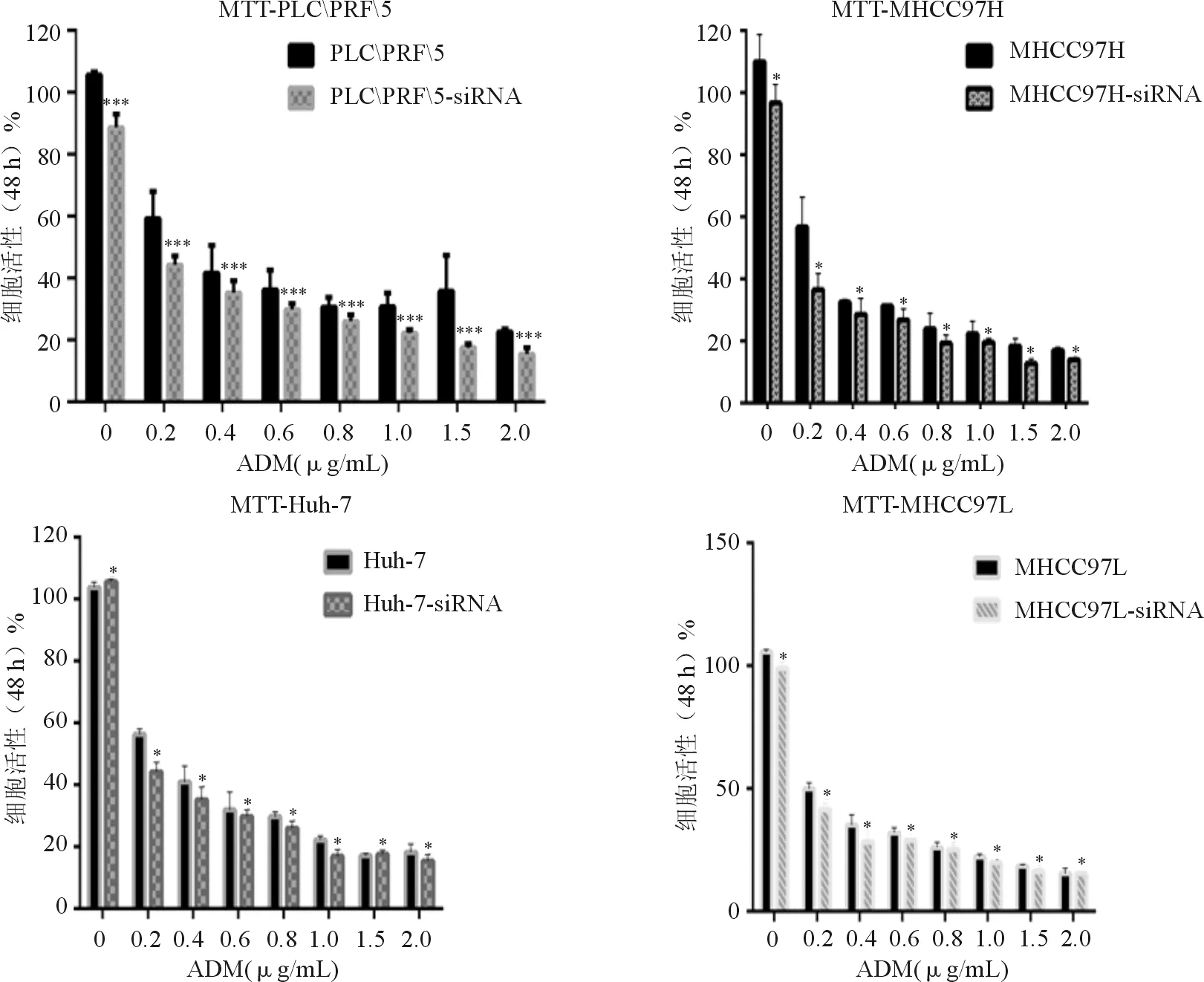
转染siRNA-3的肝癌细胞株即转染组与空白对照组比较,*P<0.05,***P<0.01
3 讨 论
一般认为,YAP在肿瘤的发生发展中起重要作用。YAP除了通过Hippo通路促进细胞癌变外[4],还有研究[5]认为,YAP具有转录激活因子的功能,通过结合转录因子(p73、TEAD等)调节下游基因转录,从而影响细胞的凋亡、增殖和侵袭等生物学行为。Overholtzer等[6]研究发现,正常的乳腺上皮细胞可因YAP高表达而过度增殖,发生上皮-间质转化,体外培养时呈现锚定非依赖性生长
Da等[7]对98例胃癌患者的研究发现,YAP表达阳性率为48%,且YAP的表达与凋亡抑制蛋白Survivin表达呈正相关。Bai等[8]也得出类似结论。Sudol等[2]的研究发现,YAP与肝癌患者的甲胎蛋白水平密切相关。
化疗是中晚期无手术指征的肝癌患者的主要治疗手段。ADM是常用的肝癌化疗药物,可抑制RNA和DNA的合成,对RNA合成的抑制作用尤为显著。术后的肝癌患者通常对ADM的敏感度低,另外,肝癌对ADM等化疗药物的耐药性一直是个未解决的问题。
本研究针对YAP基因的启动子区域设计并合成siRNA序列,筛选抑制效果较好的siRNA进行实验。用荧光显微镜观察和Western blotting法检测发现,转染siRNA后给予ADM处理的肝癌细胞存活率明显低于未转染siRNA组,表明siRNA抑制YAP表达可增强肝癌细胞株对ADM的敏感性。
综上所述,本研究证实,siRNA干扰YAP表达后,肝癌细胞株对ADM敏感性增强,这为应用YAP抑制剂逆转肝癌的耐药性提供了依据。
[1]Carneiro F,Oliveira C,Leite M,et al.Molecular targets and biological modifiers in gastric cancer[J].Semin Diagn Pathol,2008,25(4):274-287.
[2]Sudol M.Yes-associated protein(YAP65) is a proline-rich phosphoprotein that binds to the SH3 domain of the Yes proto-oncogene product[J].Oncogene,1994,9(8):2145-2152.
[3]Xu MZ,Yao TJ,Lee NP,et al.Yes-associated protein is an independent prognostic marker in hepatocellular carcinoma[J].Cancer,2009,115(19):4576-4585.
[4]Zhao B,Wei X,Li W,et al.Inactivation of YAP oncoprotein by the Hippo pathway is involved in cell contact inhibition and tissue growth control[J].Genes Dev,2007,21(21):2747-2761.
[5]Liu AM,Xu MZ,Chen J,et al.Targeting YAP and Hippo signaling pathway in liver cancer[J].Expert Opin Ther Targets,2010,14(8):855-868.
[6]Overholtzer M,Zhang J,Smolen GA,et al.Transforming properties of YAP,a candidate oncogene on the chromosome 11q22 amplicon[J].Proc Natl Acad Sci U S A,2006,103(33):12405-12410.
[7]Da CL,Xin Y,Zhao J,et al.Significance and relationship between Yes-associated protein and survivin expression in gastric carcinoma and precancerous lesions[J].World J Gastroenterol,2009,15(32):4055-4061.
[8]Bai HB,Gayyed MF,Lam-Himlin DM,et al.Expression of Yes-associated protein modulates Survivin expression in primary liver malignancies[J].Hum Pathol,2012,43(9):1376-1385.
