转GsbZIP33基因苜蓿的耐盐性分析
2014-08-25陈明明成舒飞王家佳赵超越朱延明
陈明明,成舒飞,王家佳,赵超越,才 华,朱延明
(东北农业大学生命学院 农业生物功能基因重点实验室,黑龙江 哈尔滨 150030)
植物生产层
转GsbZIP33基因苜蓿的耐盐性分析
陈明明,成舒飞,王家佳,赵超越,才 华,朱延明
(东北农业大学生命学院 农业生物功能基因重点实验室,黑龙江 哈尔滨 150030)
本研究以前期获得的耐盐转基因苜蓿(Medicagosativa)的两个株系b32和b77及其受体对照野生型苜蓿品种为材料,经200 mmol·L-1NaCl处理15 d 后,系统地测定了与耐盐性相关的生理生化指标,包括叶片净光合速率(Pn),抗氧化酶(SOD,CAT,POD)活性、可溶性糖含量,Na+、K+含量等。结果表明,与野生型相比,转基因苜蓿新株系b32、b77的可溶性糖含量分别增加了48.8%和39.6%;SOD活性分别升高了71%和89%;CAT活性分别升高了21%和13%;POD活性分别升高了32.9%和34.1%,Na+含量分别降低了57%和44%,K+含量分别增长了21%和13%;两个转基因株系(b32、b77)的光合速率是对照的2.4倍和2.5倍,以上指标差异均极显著(P<0.01),表明转GsbZIP33的两个株系b32和b77提高了耐盐性。
净光合速率;抗氧化酶;可溶性糖;离子含量;耐盐性
碱性亮氨酸拉链(Basic Region/Leucine Zipper motif,bZIP)转录因子是真核生物转录因子中分布最广泛、最保守的一类转录因子,植物bZIP转录因子包含DNA序列相结合的富含碱性氨基酸的结构域和与其相邻的亮氨酸拉链区[1]。根据DNA结合的特异性和序列相似性,将bZIP转录因子分成Ⅰ~Ⅺ共11个亚族[2]或13个亚族(A~S)[3],bZIP转录因子家族可以与ABA响应元件结合因子(AREB) 或ABRE结合蛋白(ABF)结合,启动下游基因的表达。植物界中bZIP转录因子广泛存在并参与各种生物学过程,包括病菌防御、光及各种胁迫信号、种子萌发和花器官发育[4]。研究表明,在烟草(Nicotianatabacum)里超表达转ThbZIP基因后,增加了过氧化物酶(POD)和超氧化物歧化酶(SOD)的活性并且调节了可溶性糖和可溶性蛋白的合成,从而改善了耐盐性[5];组成性表达ABP9的转基因拟南芥(Arabidopsisthaliana)改善了胁迫下植物的光合能力[6],还可以降低细胞活性氧水平,增强活性氧清除相关基因的表达,减轻活性氧的伤害[7]; Yang等[8]发现,转AtbZIP24基因拟南芥的耐盐能力与叶中钠离子的积累减少有关;超表达AtbZIP60通过增强抗氧化酶的活性减弱了盐诱导的氧化伤害[9];在拟南芥中超表达PtrABF基因,可以通过清除活性氧和调节压力响应基因的表达从而增强耐胁迫性[10];超表达GmbZIP1基因的拟南芥在盐胁迫条件下会诱导气孔关闭,减少失水从而增强耐盐性[11]。以上研究表明,bZIP转录因子在盐胁迫反应中起着重要的积极作用。
迄今,国内外学者对苜蓿的耐盐碱转基因做了大量的研究,将AlfinI基因导入苜蓿中,增强了植物生长且耐盐性得到提高[12],另外,BADH、GmDREB1、H+-PPase、SsNHX1、NTHK1等基因也被导入苜蓿,转基因株系均表现出不同程度的耐盐性。将GsGST14[13]、GsZFP1[14]、GsCBRLK[15]等基因转化肇东苜蓿获得大量的转基因株系,也具有较强的耐盐性。如何从大量的转基因材料中筛选出耐盐碱新品(种)系,对其进行耐盐碱相关的生理生化指标检测是一项很重要的工作。本研究以前期获得的转GsbZIP33苜蓿两个新株系b32和b77为材料,系统测定与耐盐相关的生理生化指标,以期鉴定其耐盐能力。
1 材料与方法
1.1试验材料
耐盐转GsbZIP33苜蓿株系b32和b77及其受体对照品种非转基因野生型肇东苜蓿(M.sativacv.Zhaodong)。
1.2试验处理
本研究采用无性扦插繁殖的方式获得扦插苗,选取生长状态良好且长势一致的转基因和非转基因株系的扦插苗进行耐盐性分析。根据魏正巍[16]前期研究中对肇东苜蓿耐盐性的分析,选择两个盐浓度处理:0和200 mmol·L-1NaCl,每个处理设3次重复,每个重复有8株,每两天更换一次1/2Hoagland营养液配制的NaCl溶液,以防止因水分蒸发造成的NaCl浓度变大,处理15 d后取样,测定各项生理生化指标。
1.3测定项目及方法
可溶性糖含量测定采用蒽酮比色法[17];SOD活性测定采用氮蓝四唑法[16]; 过氧化氢酶活性(CAT)的测定采用紫外分光光度法[18];POD活性测定采用愈创木酚显色法[17];光合指标测定采用Li-6400便携式光合测定系统分析仪(USA,LI-COR),测定时间为09:30-11:30;K+、Na+含量的测定采用火焰光度法[19]。
1.4统计分析
数据利用统计软件SPSS 13.0进行显著性检验,采用Excel 2010制图。
2 结果与分析
2.1转基因株系可溶性糖含量测定
在未胁迫处理时,转基因株系b32和b77与野生型(对照)相比,可溶性糖含量无明显差异(图1)。在200 mmol·L-1NaCl胁迫下,转基因和对照株系的可溶性糖含量均升高,但两个转基因株系(b32和b77)的可溶性糖含量都显著高于野生型(P<0.01)。其中,对照为27.35 μg·g-1,转基因株系b32和b77的可溶性糖含量分别为40.70和38.19 μg·g-1,分别较对照高出48.8%、39.6%。
2.2转基因株系抗氧化酶(SOD,CAT,POD)活性测定
在未胁迫处理时,转基因株系b32和b77的SOD活性均较低,在200 mmol·L-1NaCl胁迫后,转基因和对照株系的SOD活性迅速增强。其中,转基因株系的SOD活性分别为128.66和142.37 U·g-1,对照为75.20 U·g-1,两个转基因株系的SOD活性都显著高于对照(P<0.01),分别较对照高出71%、89%(图1)。
在未胁迫处理时,两个转基因株系的CAT活性显著高于对照(P<0.01),在200 mmol·L-1NaCl胁迫后,转基因和对照的CAT活性迅速增强。其中,对照为38.21 U·mg-1·min-1,b32和b77的CAT活性分别为46.50和43.23 U·mg-1·min-1,均显著高于对照,分别较对照高出21%和13%(图1)。
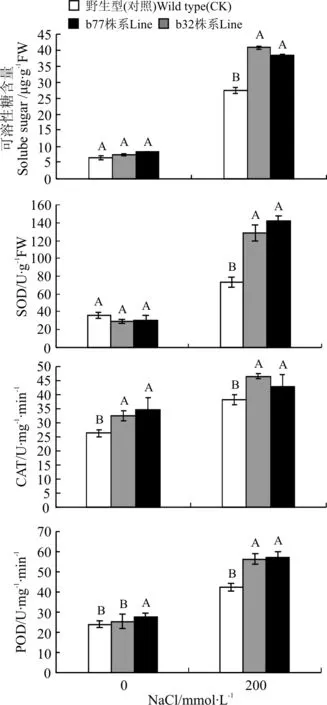
图1 转基因株系可溶性糖含量,SOD,CAT和POD活性的测定Fig.1 Measurement of soluble sugar content,SOD,CAT and POD in transgenic lines
注:不同大写字母表示同一处理不同株系之间差异极显著(P<0.01)。下同。
Note: Different capital letters for the same treatment show significant difference among three lines at 0.01 level.The same below.
在未胁迫处理时,转基因株系b77的POD活性显著高于对照(P<0.01),b32与对照无明显差异,在200 mmol·L-1NaCl胁迫后,转基因和对照的POD活性迅速增强。其中对照为42.5 U·mg-1·min-1,b32和b77的POD活性分别为56.5和57.0 U·mg-1·min-1,两个转基因株系的POD活性都极显著高于对照,分别高出32.9%、34.1%(图1)。
2.3转基因苜蓿K+、Na+、K+/Na+含量测定
在未胁迫处理时,转基因株系b32和b77与对照相比,K+、Na+含量无明显差异(图2)。在200 mmol·L-1NaCl处理下,转基因和对照株系K+、Na+离子的积累量较未胁迫处理均表现为K+含量降低,Na+含量提高,K+/Na+比值降低(图2)。胁迫处理后,转基因株系b32和b77的Na+含量分别为4.46和5.78 mg·g-1,对照为10.36 mg·g-1;转基因株系b32和b77的K+含量分别为5.716和5.336 mg·g-1,对照为4.706 mg·g-1,两个转基因株系(b32和b77)的K+显著高于对照(P<0.01),分别增长了21%和13%,Na+含量都显著低于对照,分别降低了57%和44%。

图2 转基因株系Na+,K+,K+/Na+和光合速率测定Fig.2 Measurement of Na+,K+, K+/Na+ and photosyntheticrate content in transgenic lines
2.4转基因苜蓿光合速率测定
在未胁迫处理时,转基因株系b32和b77与对照相比,光合速率无明显差异(图2)。在200 mmol·L-1NaCl胁迫下,转基因株系的光合速率分别为2.76和2.83 μmol·m-2·s-1,对照为1.14 μmol·m-2·s-1,转基因和对照株系的光合速率较未胁迫均减弱,但两个转基因株系(b32、b77)的光合速率都显著高于野生型(P<0.01),两个转基因株系的光合速率分别是对照的2.4倍和2.5倍。
3 讨论与结论
植物耐盐性受多重因素的调节和控制,包括无机渗透调节、有机渗透调节、调控抗氧化防御系统和调节光合作用等。细胞中可溶性糖的含量是反映细胞渗透调节的生理指标之一[20]。渗透调节是植物适应盐胁迫的一种重要调节机制,保护植物细胞在盐胁迫下的渗透失衡,稳定亚细胞组织如细胞膜和蛋白质。可溶性糖含量提高,可降低细胞渗透势,使细胞具有较强的保水能力,维持细胞的正常膨压,以维持正常的生理活动。本研究中,在200 mmol·L-1NaCl胁迫下,可溶性糖的含量明显高于对照,由此表明GsbZIP33基因作为转录因子,能提高转基因苜蓿的渗透调节能力,增强了耐盐性。
植物抗氧化防御机制是植物抵抗盐胁迫的重要途径。盐分能增加细胞膜的透性,加强脂质的过氧化,破坏膜结构[21],而光合电子传递链仍保持较高的活性,这会导致电子传递链PSI端的过还原,从而产生大量超氧阴离子(·O2-)、H2O2和·OH等多种活性氧自由基[21]。SOD能催化·O2-发生歧化反应,降低超氧离子对膜系统的伤害,CAT、POD是清除过氧化氢的关键酶,H2O2可作为底物直接激活CAT酶的活力,也可以通过基因表达的方式激活过氧化氢酶基因的表达[22]。三者协同作用,共同清除细胞内过多的自由基,避免细胞的结构受到损伤。有研究报道,bZIP基因是组织游离氧损害的关键基因,对植物抗逆胁迫起关键作用[23],本研究中,在200 mmol·L-1NaCl胁迫下,转基因苜蓿苗的3种抗氧化酶活性均显著高于野生型。综合已有结果,说明转基因株系清除盐胁迫产生的过氧化氢的能力明显高于对照,有利于保护细胞内的蛋白和膜系统免受氧化胁迫的伤害,增强其在高盐环境下的耐受能力。
高等植物耐盐还通过调节无机离子的种类、数量和比例来维持细胞内微环境的稳定。细胞内钠、钾离子浓度及比例的变化与植物的耐盐性存在一定的关系,并影响细胞膜的透性、盐离子毒害程度及离子的区隔化等逆境生理过程。转基因番茄(Lycopersiconesculentum)的耐盐机理是在于根系对K+、Na+的选择吸收和K+、Na+在根、茎、叶等整株水平上的区域化分配[24]。本研究中,在200 mmol·L-1NaCl处理下,转基因苜蓿苗地上部的钠离子含量比对照低,推测转基因苜蓿通过根系对土壤中盐分离子的选择吸收,限制钠离子向地上部的运输,降低对叶器官的伤害,使植物耐盐。
盐往往造成植物生物量的下降,而生物量的改变与光合作用的变化密切相关[25],盐害可以使叶绿素含量降低,影响光合电子传递,降低RUBP羧化酶的羧化效率,从而降低光合效率,因此,盐胁迫下植物能否保持较高的光合效率十分关键。bZIP基因是叶绿体内光合损伤的适应性反应基因,可以保护和修复光合元件[23]。在前期研究中发现,经盐胁迫后,转基因苜蓿叶绿素含量也明显高于未转基因株系[26]。本研究中,转基因和对照相比,光合速率下降的幅度较小,说明转基因光合元件受迫害的程度较小,转基因苗耐盐能力增强,推测GsbZIP33基因直接或间接调节了光合相关基因的表达调控。
综合各项指标的测定结果,转GsbZIP33基因的苜蓿可以通过提高盐胁迫下苜蓿苗的可溶性糖的含量和抗氧化酶活性,升高K+/Na+,增强光合速率,从而减轻植株的盐伤害的程度, 增强了耐盐性。
[1]Liu C T,Mao B G,Ou S J,Wang W,Liu L C,Wu Y B,Chu C C,Wang X P.OsbZIP71,a bZIP transcription factor,confers salinity and drought tolerance in rice[J].Plant Molecular Biology,2014,84(1-2):19-36.
[2]Nijhawan A,Jain M,Tyagi A K.Genome survey and gene expression analysis of the basic leucine zipper transcription factor family in rice[J].Plant Physiology,2008,146:333-350.
[3]Correa L G,Riano-Pachon D M,Schrago C G,dos Santos R V,Mueller-Roeber B.Vincentz M.The role of bZIP transcription factors in green plant evolution:Adaptive features emerging from four founder genes[J].PloS ONE,2008,3(8):1-16.
[4]Jakoby M,Weisshaar B,Dröge-Laser W,Vicente-Carbajosa J,Tiedemann J,Kroj T,Parcy F.bZIP transcription factors inArabidopsis[J].Trends in Plant Science,2002,7(3):106-111.
[5]Wang Y C,Gao C Q,Liang Y N,Wang C,Yang C P,Liu G F.A novelbZIPgene fromTamarixhispidamediates physiological responses to salt stress into tobacco plants[J].Plant Physiology,2010,167:222-230.
[6]Zhang X,Wollenweber B.Water deficits and heat shock effects on photosynthesis of a transgenicArabidopsisthalianaconstitutively expressingABP9,a bZIP transcription factor[J].The Journal of Experimental Botany,2008,59:839-848.
[7]Zhang X,Wang L,Meng H,Wen H T,Fan Y L,Zhao J.MaizeABP9 enhances tolerance to multiple stresses in transgenicArabidopsisby modulating ABA signaling and cellular levels[J].Plant Molecular Biology,2011,75(4-5):365-378.
[8]Yang O S,Popova V O,Suthoff U,Luking I,Dietz K J,Golldaci D.TheArabidopsisbasic leucine zipper transcription factorAtbZIP24 regulates complextranscriptional networks involved in abiotic stress resistance[J].Gene,2009,436:45-55.
[9]Tang W,Michael P,Fei Y J,Liu L C,Xu F,Cai X D,Yuan L Y,Wu Q S,Zhou M Q.Overexpression ofAtbZIP60deltaCgene aleviates induced oxidative damage in transgenic cell cultures[J].Plant Molecular Biology Reporter,2012,30:1183-1195.
[10]Hang X S,Liu J H,Chen X J.AbZIPtranscription factor isolated fromPoncirustrifoliata,enhances dehydration and drought tolerance in tobacco via scavenging ROS and modulating expression of stress-responsive genes[J].BMC Plant Biology,2010,10:1-18.
[11]Gao S Q,Chen M,Xu Z S,Zhao C P,Li L C,Xu H J,Tang Y M,Zhao X,Ma Y Z.The soybeanGmbZIP1 transcription factor enhances multipleabiotic stress tolerances in transgenic plants[J].Plant Molecular Biology,2011,75:537-553.
[12]Winicov I.Alfin1 transcription factor overexpression enhances plant root growth under normal and saline conditions and improves salt tolerance in alfalfa[J].Planta,2000,210:416-422.
[13]Wang Z Y,Song F B,Cai H.Zhu Y M,Bai X,Ji W,Li Y,Hua Y.Over-expressingGsGST14 fromGlycinesojaenhances alkaline tolerance of transgenicMedicagosativa[J].Biologia Plantarum,2012,56(3):516-520.
[14]Tang L L,Cai H,Ji W,Luo X,Wang Z Y,Wu J,Wang X D,Cui L,Wang Y,Zhu Y M,Bai X.Overeixpression ofGsZFP1 enhances salt and drought tolerance in transgenic alfalfa (MedicagosativaL.)[J].Plant Physiology and Biochemistry,2013,71:22-30.
[15]Liu J,Cai H,Zhu Y M,Ji W,Liu Y,Bai X.Over-expressingGsCBRLKfromGlycinesojaenhances tolerance to salt stress in transgenic alfalfa (Medicagosativa)[J].Functional Plant Biology,2013,40(10):1048-1056.
[16]魏正巍.耐盐碱GsGST13/SCMRP和GsCBRLK/SCMRP双价基因对苜蓿的遗传转化及转基因植株的培育[D].哈尔滨:东北农业大学,2012.
[17]李合生.植物生理生化实验原理和技术[M].北京:高等教育出版社,1985:67-70.
[18]王学奎.植物生理生化实验原理和技术[M].北京:高等教育出版社,2006:167-173.
[19]景艳霞,袁庆华.NaCl胁迫对苜蓿幼苗生长及不同器官中盐离子分布的影响[J].草业学报,2011,20(2):134-139.
[20]刘建新,王鑫,王凤琴.水分胁迫对苜蓿幼苗渗透调节物质积累和保护酶活性的影响[J].草业科学,2005,22(3):18-21.
[21]冯昌军,罗新义,沙伟,王凤国.低温胁迫对苜蓿品种幼苗SOD、POD活性和脯氨酸含量的影响[J].草业科学,2005,22(6):29-32.
[22]Michelet B,Boutry M.The plasma membrane H+-ATPase(A highly regulated enzyme with multiple physiological functions)[J].Plant Physiology,1995,108(1):1-6.
[23]吴英杰,王超.柽柳bZIP基因调控耐盐相关基因的表达[J].植物生理学通讯,2010,46(12):1237-1242.
[24]田吉林,杨玉爱,何玉科.转HAL1基因番茄的耐盐性[J].植物生理与分子生物学学报,2003,29(5):409-414.
[25]包爱科,杜宝强,王锁民.紫花苜蓿耐盐、抗旱生理机制研究进展[J].草业科学,2011,28(9):1700-1705.
[26]刘晶.GsbZIP33和GsCBRLK基因转化肇东苜蓿及其耐盐性分析[D].哈尔滨:东北农业大学,2012.
(责任编辑 武艳培)
SaltstresstoleranceoftransgenicalfalfawiththeGsbZIP33gene
CHEN Ming-ming, CHENG Shu-fei, WANG Jia-jia, ZHAO Chao-yue, CAI Hua, ZHU Yan-ming
(Key Laboratory of Agricultural Biological Functional Genes, Northeast Agricultural University, Harbin 150030, China)
In the present study,the physiological and biochemical indicators of two salt-tolerant transgenic lines (b32 and b77) and the wild type alfalfa were compared after 15 days treated with 200 mmol·L-1NaCl, including soluble sugar content, the activities of catalase (CAT),peroxidase (POD) and superoxide dismutase (SOD), photosynthetic rate (Pn) and the content of K+and Na+.Compared with the wild type plants, the soluble sugar content of the two transgenic lines increased by 48.8% and 39.6%, respectively, the SOD activities increased by 71% and 89 %, CAT activities increased by 21% in b32 and 13% in b77, POD activities increased by 32.9% and 34.1%, Na+contents decreased by 57% and 44% and K+contents increased by 23% and 13%, respectively.The photosynthetic rate of transgenic plants increased by 2.4 times compared with the wild type plants.This study demonstrated that the transgenic alfalfa (b32 and b77) had a greater tolerance of saline stress compared with non-transgenic plants.
net photosynthetic rate; antioxidant enzyme; soluble sugar; ion content; salt tolerance
ZHU Yan-ming E-mail:ymzhu2001@neau.edu.cn
2014-01-24 接受日期:2014-05-20
转基因生物新品种培育重大专项(2011ZX08004-002);国家自然科学基金项目(31171578);黑龙江省高校科技创新团队建设计划(2011TD005)
陈明明(1988-),女,山东莱芜人,在读硕士生,主要从事植物基因工程研究。E-mail:chenming100210@163.com
朱延明(1955-),男,黑龙江哈尔滨人,教授,博士,主要从事植物基因工程与分子生物学研究。 E-mail:ymzhu2001@neau.edu.cn
S816;S551+.703.4;Q943.2
:A
:1001-0629(2014)11-2077-05
10.11829j.issn.1001-0629.2014-0050
