基于锚定PCR技术对10种重要农业害虫微卫星DNA位点的筛选及其特征分析
2014-08-15郎坤玲沈长朋陶云荔
李 慧, 郎坤玲, 沈长朋, 李 洁, 陶云荔, 褚 栋
青岛农业大学农学与植物保护学院,山东省植物病虫害综合防控重点实验室,山东 青岛 266109
微卫星DNA(microsatellite DNA)一般指基因组中由短的重复单元(一般为1~6个碱基)组成的DNA串联重复序列,被称为短串联重复序列(short tandem repeats,STRs)或简单重复序列(simple sequence repeats,SSRs)。微卫星DNA广泛分布于真核生物的基因组中,在原核生物的基因组中也有少量分布(罗文永等,2003)。微卫星DNA符合孟德尔遗传模式,共显性遗传,通常具有丰富的多态性,易于检测,是一类很好的分子标记(代金霞,2005;何平,1998;刘佳妮等,2008)。微卫星DNA分子标记有广泛的应用价值,现已被应用于入侵害虫的起源、入侵途径及模式和种群扩散等研究中(褚栋等,2007、2012;高长生等,2011;Chuetal.,2011)。
微卫星DNA的获得方式主要有几种途径:(1)筛选基因文库法,用含有微卫星的探针在基因文库中筛选含有微卫星序列的阳性克隆并测序,再根据微卫星DNA两端的侧翼序列设计引物,最后用该引物对应的微卫星DNA位点做定性分析(Rassmannetal.,1991)。此方法工作量大,效率低。(2)微卫星富集法,用含微卫星序列的探针进行杂交富集,构建其富集文库(Kandpaletal.,1994; Karagyozovetal.,1993)。这种方法操作较复杂,大量扩增目的片段的同时,产生了大量的冗余序列(张增翠和侯喜林,2004)。(3)搜索GenBank、EMBL 和 DDBJ 等公共数据库以获得含SSR序列的方法,开发的微卫星位点的多态性一般不是很高(段惠生等,2012)。近年来,由于锚定PCR技术简便、高效、多态性好,在微卫星筛选中得到了广泛应用(盛良明等,2007;张俊鹏等,2012)。该方法是由Fisheretal.(1996)提出,用锚定简并微卫星引物对基因组DNA进行扩增。此方法是一种分离微卫星的简单、快捷的方法,避免了SSR富集的随机性,具有明确的目的性和较强的富集能力,提高了微卫星的分离效率,且通常每个序列都含有2个完整的微卫星位点,开发效率极高(张增翠和侯喜林,2004)。
目前,桃小食心虫CarposinasasakiiMatsumura、桃蛀螟Conogethespunctiferalis(Guenée)、玉米螟Ostrinianubilalis(Hübner)、二点委夜蛾Proxenuslepigone(Moschler)、花蓟马Frankliniellaintonsa(Trybom)、黄胸蓟马Thripshamaiiensis(Morgan)、棕榈蓟马ThripspalmiKarny、斑翅果蝇Drosophilasuzukii(Matsurmura)、稻水象甲LissorhoptrusoryzophilusKuschel、扶桑绵粉蚧PhenacoccussolenopsisTinsley等这些重要农业害虫的微卫星位点尚未见报道。本试验用锚定PCR技术对上述10种重要农业害虫进行微卫星DNA位点筛选,并对微卫星DNA序列进行分析比较,旨在探索该方法在农业害虫微卫星DNA筛选中的有效性,为进一步挖掘农业害虫的微卫星位点及研究其种群迁移扩散等奠定基础。
1 材料与方法
1. 1 试验材料及DNA提取
本研究中所用的物种均放于95%乙醇中,-20 ℃下保存。基因组DNA的提取方法参照Chuetal.(2005),供试虫体样本数量均为1头,提取的DNA放于-20 ℃下保存。
1. 2 锚定PCR扩增
以提取的基因组DNA为PCR反应的模板,使用根据需要设计的5′锚定简并引物NNNNNNNNKKVRVRV(CA)6进行扩增。建立50 μL反应体系,体系中含有DNA模板3 μL、buffer 5 μL、dNTPs 1 μL、简并引物1 μL(10 μmol·L-1)、ddH2O 39. 5 μL、Taq酶0. 5 μL。PCR反应程序:94 ℃预变性4 min,35个循环(94 ℃变性30 s,55 ℃复性45 s,72 ℃延伸2 min),72 ℃再延伸7 min。
1. 3 克隆与测序
将扩增产物经2%琼脂糖凝胶电泳检测,使用试剂盒(北京全式金生物技术有限公司)切胶回收长度500~750 bp的DNA,与PMD18-T连接并转入大肠杆菌Trans1-T1感受态细胞,涂抹于含有氨苄西林(0. 1 mg·mL-1)的LB琼脂平板上。挑取单菌落,放于含有氨苄西林(0. 1 mg·mL-1)的LB液体培养基中振荡培养。取2 μL菌液,用通用引物M13对插入片段进行PCR扩增。13 μL反应体系中含有菌液2 μL、buffer 1. 3 μL、dNTPs 0. 26 μL、M13通用引物0. 26 μL(10 μmol·L-1)、ddH2O 9. 3 μL、Taq酶0. 13 μL。PCR程序:94 ℃预变性4 min,35个循环(94 ℃变性30 s,55 ℃复性45 s,72 ℃延伸2 min),72 ℃再延伸7 min。PCR产物经1%琼脂糖凝胶电泳检测,挑选阳性克隆。
1. 4 序列测定与分析
随机选取片段大小为500~750 bp的50个阳性克隆进行测序。利用Sequencher 5. 0 Demo软件(http:∥www.genecodes.com/)去除冗余序列,再用SSR Hunter1. 3软件对DNA序列进行SSR位点的搜索,设置重复数至少为5,构成重复元件的核苷酸数最多为6个。
2 结果与分析
2. 1 简并引物的扩增及PCR产物的克隆与测序
用含有CA重复单元的5′锚定简并引物扩增10个物种的DNA,扩增片段较弥散,多集中于200~2000 bp。经PCR筛选,各个物种的克隆片段在500~750 bp之间。阳性克隆率在10个物种中有较大的差异,最高为棕榈蓟马(95. 8%),最低为黄胸蓟马(20. 9%),平均阳性克隆率为57. 8%。10个物种的微卫星比率在70. 8%~100%之间,最高为扶桑绵粉蚧(100%),最低的是二点委夜蛾(70. 8%),微卫星平均比率为91. 3%。克隆效率差异较大,最高为稻水象甲(88. 2%),最低为黄胸蓟马(19. 9%),克隆平均效率为52. 8%(表1)。

表1 微卫星文库的筛选结果Table 1 The screening result of microsatellite library
*阳性克隆率指阳性克隆占所有克隆的百分比;**微卫星比率指含有微卫星的克隆占所有阳性克隆的百分比;***克隆效率指含有微卫星的克隆占所有克隆的百分比。*Positive clone rate refers to the percentage of positive clones in all clones;**Microsatellite rate refers to the percentage of microsatellites identified in all positive clones;***Cloning efficiency refers to the percentage of microsatellites identified in all clones.
2. 2 微卫星类型
去除重复序列后进行微卫星搜索和分类统计,结果表明,冗余率(重复序列的数量/总序列的数量)在不同物种间差异较大,最低为棕榈蓟马(2. 3%),最高为扶桑绵粉蚧(71. 7%)(图1)。各个物种平均每条序列含微卫星位点数为1. 9~2. 4,桃小食心虫、扶桑绵粉蚧和稻水象甲的平均位点数小于2,其他物种均大于2。
从微卫星位点(核苷酸碱基组成)分布类型的角度分析,除简并引物中所含有的CA/TG重复序列外,还发现其他类型的重复序列,如:二核苷酸AT、GC、CT/AG等3种重复单元;三核苷酸AAC、GGT、GCA、GCC、TCC等10种重复单元;四核苷酸TCGC、GACA、GTGC、ATCT、TGTC等5种重复单元。不同物种以二核苷酸CA/TG重复序列数目最多,在89. 2%~100%之间,二核苷酸(CT/AG、AT、GC)、三核苷酸和四核苷酸所占比例较低。其中,花蓟马、扶桑绵粉蚧和稻水象甲二核苷酸的比例为100%,扶桑绵粉蚧的CA/TG重复为100%(表2)。从微卫星位点(重复次数)分布类型的角度分析,10个物种中,具有最高重复次数的是玉米螟,达39次,其最低重复次数为5(SSR hunter的最低重复次数设置为5次)。大部分物种的SSR序列较短,如桃小食心虫的SSR片段长度为10~36 bp,玉米螟的SSR片段长度为10~78 bp。各个物种的平均重复次数为6. 7~8. 9次(图2)。
根据Weber(1990)的微卫星分类标准,本研究中涉及的10个物种的微卫星类型90%以上都是完全型,不完全型和复合型的SSR所占比例较少。不完全型的微卫星比率最高的物种为玉米螟(5. 7%),复合型的微卫星比率最高的物种为桃小食心虫(5. 0%)。其中,扶桑绵粉蚧和桃蛀螟的微卫星类型均为完全型(图3)。

图1 序列冗余率分布

表2 微卫星位点类型分布(基于核苷酸碱基数目)Table 2 Distribution of microsatellite types (based on nucleotide numbers)
*包括重复单位CTG、GCA、GCC、TCC、GAA、AGA、TTA、TCC、AAC、CCA;**包括重复单位TGTC、GTGC、ATCT、TCTG、GACA。*including the repeat unit as follows: CTG, GCA, GCC, TCC, GAA, AGA, TTA, TCC, AAC, CCA;**including the repeat unit as follows: TGTC, GTGC, ATCT, TCTG, GACA.
3 讨论
本研究表明,10个物种中阳性克隆率最高为95. 8%,平均阳性克隆率为57. 8%,高于通过探针杂交法获得的阳性克隆率(安建东等,2011;郝卓然等,2012;吉亚杰和张德兴,2004)。本研究中,微卫星比率最高为100%,微卫星平均比率为91. 3%,高于通过探针杂交法获得的微卫星比率(程月琴等,2013;张国彦和翟保平,2008)。盛良明等(2007)利用锚定PCR技术测定结果表明,阳性克隆率和微卫星比率均达到了100%;张俊鹏等(2012)也证明利用该技术筛选的微卫星比率极高,达97. 7%。徐波等(2012)对使用磁珠富集法与锚定PCR法开发背瘤丽蚌Lamprotulaleai(Gray)微卫星标记进行比较,发现锚定PCR技术获得的阳性克隆比率高。此外,本试验也发现10个物种的冗余率(2. 3%~71. 7%)、假阳性率(0~29. 2%)之间差异较大。
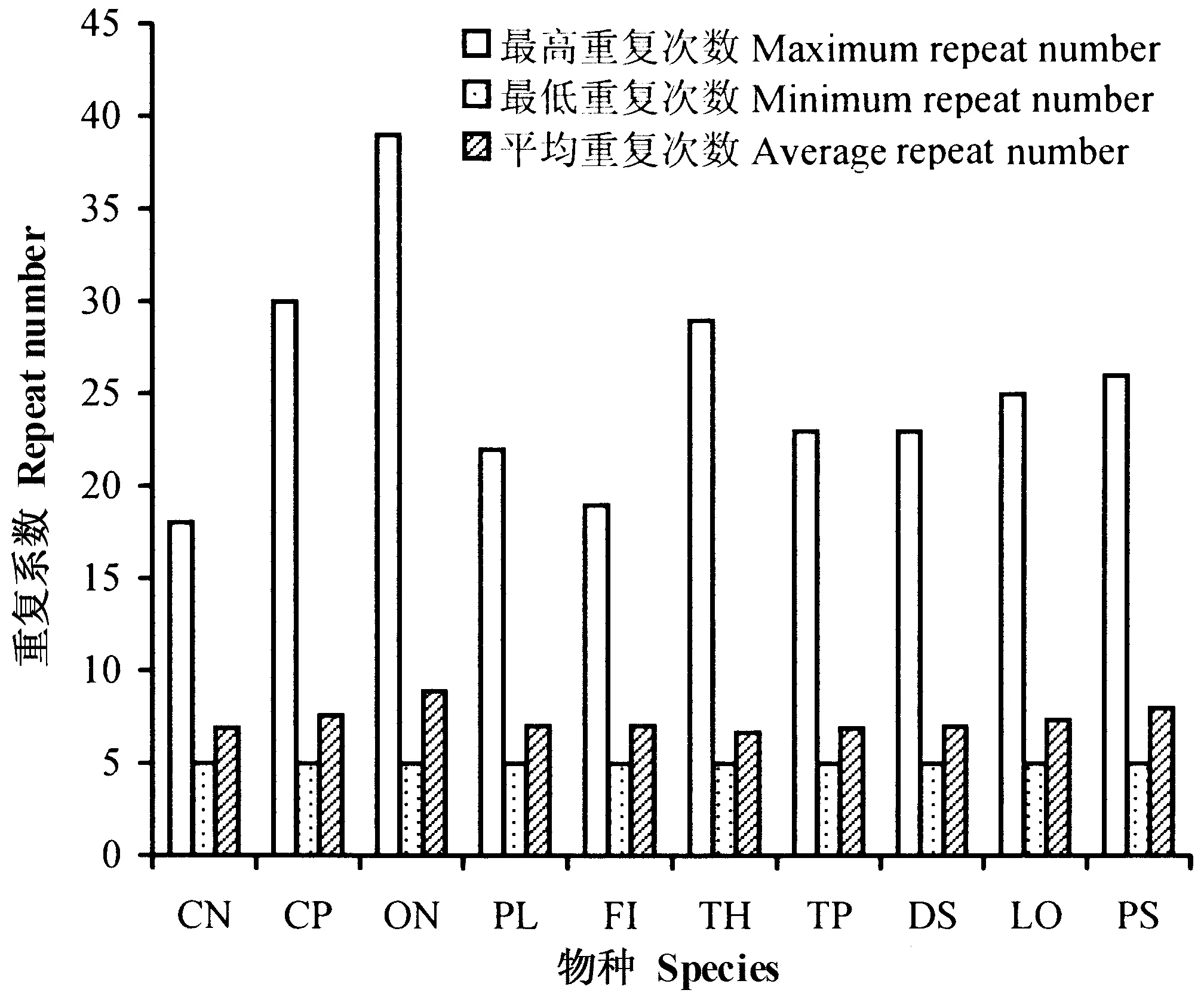
图2 微卫星重复次数分布图Fig.2 Distribution of repeat number of microsatellite in the species analysed CN=Carposina nipponensis, CP=Conogethes punctiferalis, ON=Ostrinianubilalis, PL=Proxenus lepigone, FI=Frankliniella intonsa, TH=Thrips hawaiiensis, TP=Thrips palmi, DS=Drosophila suzukii, LO=Lissorhoptrus oryzophilus, PS=Phenacoccus solenopsis.

图3 微卫星类型分布[基于Weber(1990)标准]
在所有动物基因组中,(CA)n微卫星的含量最为丰富(Brenneretal.,1993)。因此,本研究使用(CA)n简并引物来研究10个物种,以获得更多的微卫星位点。本试验中,二核苷酸、三核苷酸、四核苷酸重复单元在鳞翅目、缨翅目、双翅目、同翅目和鞘翅目中差异不明显,二核苷酸在不同目间的比例为95. 1%~100%,三核苷酸的比例为0~4. 9%,四核甘酸为0~1. 73%,较高的二核苷酸比例可能与锚定引物筛选方法有关。
本试验所设计的锚定简并引物中含有6个微卫星重复,测序结果表明,获得的克隆两端大部分至少含有6个二核苷酸重复,并且每条序列大部分都含有2个以上的微卫星位点,说明锚定简并引物扩增可以有效获得微卫星DNA位点。本试验中分离的微卫星的平均重复数为6. 35~7. 50次,最高重复次数为34次。马丽华(2008)通过该方法获得的微卫星的平均重复数为7. 19次。而通过磁珠富集法获得的微卫星的重复次数大部分都在10次以上(安建东等,2011;连灏等,2012)。这些结果表明,锚定PCR技术获得的微卫星重复数低于磁珠富集法(马丽华,2008;徐波等,2012)。
Weber(1990)按照微卫星核心序列的不同将微卫星分为3种类型:完全型(perfect)、不完全型(imperfect)和复合型(compound)。本试验中,10个物种的微卫星类型90%以上都是完全型,不完全型和复合型的微卫星DNA所占比例较小。完全型微卫星DNA是动物基因组中比例最大的结构类型,而不完全型和复合型微卫星比例的高低在不同物种中存在差异(连灏等,2012;马雅军等,2008;王蕾等,2009;张丽等,2009)。
分子遗传标记是近来现代遗传标记学发展较快的领域之一,微卫星则被认为是各类分子遗传标记中最有价值的一种。随着生物统计学研究的不断深入,微卫星在度量品种遗传多样性、估测品种间遗传距离及构建系统发生树等研究中显示出巨大优势,其应用前景非常广阔(徐兴莉和杨虎,2011)。本研究采用5′锚定PCR技术大大降低了研究成本,提高了筛选效率,是从物种基因组中分离微卫星位点的一个较好策略。
安建东, 黄家兴, 董捷, 周冰峰. 2011. 火红熊蜂微卫星标记的筛选及种特异性分析. 昆虫学报, 54(12): 1423-1432.
程月琴, 焦振彬, 张佩, 叶永忠, 王红卫. 2013. 地黄微卫星富集文库构建及特性分析. 种子, 32(5): 12-16.
褚栋, 李显春, 张友军. 2012. 基于微卫星标记对中国Q型烟粉虱早期入侵种群与B型烟粉虱种群的遗传结构分析. 昆虫学报, 55(12): 1376-1385.
褚栋, 张友军, 万方浩. 2007. 分子标记技术在入侵生态学研究中的应用. 应用生态学报, 18(6): 1383-1387.
代金霞. 2005. 微卫星DNA标记技术及其应用. 农业科学研究, 26(1): 67-70, 79.
段惠生, 张安盛, 赵传志, 于毅, 褚栋. 2012. 西花蓟马EST-SSR信息分析标记筛选及其与Genomic-SSR的多态性比较. 昆虫学报, 55(6): 634-640.
高长生, 国栋, 刘国霞, 陶云荔, 张友军, 褚栋. 2011. Q型烟粉虱东地中海种群遗传多样性的mtCOI与SSR分析. 昆虫学报, 54(12): 1416-1422.
郝卓然, 梁利群, 常玉梅, 任波. 2012. 扁吻鱼微卫星分子标记的筛选及特征分析. 华北农学报, 27(增刊): 40-45.
何平. 1998. 真核生物中的微卫星及其应用遗传. 遗传, 20(4): 42-47.
吉亚杰, 张德兴. 2004. 鳞翅目昆虫基因组中微卫星DNA的特征以及对其分离的影响. 动物学报, 50(4): 608-614.
连灏, 石米娟, 杜富宽, 江遥, 黄容, 廖兰杰, 汪亚平, 朱作言. 2012. 草鱼(AG)微卫星标记克隆及特征分析. 水生生物学, 36(1): 29-34.
刘佳妮, 桂富荣, 李正跃. 2008. SSR分子标记技术在入侵昆虫学研究中的运用. 植物保护, 34(3): 7-11.
罗文永, 胡骏, 李晓方. 2003. 微卫星序列及其应用. 遗传, 25(5): 615-619.
马丽华. 2008. 应用5′锚定PCR开发谷子微卫星标记. 石家庄: 河北师范大学.
马雅军, 樊勇, 吴静. 2008. 雷氏按蚊多态微卫星DNA位点的筛选和特征. 寄生虫与医学昆虫学报, 15(3): 150-153.
盛良明, 薛华柏, 王化坤, 徐春明, 王三红, 章镇. 2007. 利用5′锚定PCR技术分离枇杷微卫星标记. 江苏农业学报, 23(1): 50-53.
王蕾, 刘继红, 张立冬, 孙效文. 2009. 牙鲆基因组(CAG)n微卫星DNA特征分析. 中国水产科学, 16(6): 807-815.
徐波, 汪桂玲, 李家乐. 2012. 磁珠富集法与5′锚定PCR法开发背瘤丽蚌微卫星标记的比较. 生态学杂志, 31(4): 923-930.
徐兴莉, 杨虎. 2011. 微卫星DNA标记技术的特点及其在动物研究中的应用. 畜禽业, (12): 34-35.
张国彦, 翟保平. 2008. 东方粘虫(Pseudaletiaseparate(Walker))微卫星富集文库的构建与分析. 生态学报, 28(8): 3860-3867.
张俊鹏, 孙典巧, 王日昕, 徐田军. 2012. 刺参CT/AG微卫星标记的筛选. 浙江海洋学院学报: 自然科学版, 31(2): 97-102.
张丽, 樊勇, 马雅军. 2009. 中华白蛉微卫星DNA序列的分离和多态位点筛选的初步研究. 中国寄生虫学与寄生虫病杂志, 27(6): 503-507.
张增翠, 侯喜林. 2004. SSR分子标记开发策略及评价. 遗传, 26(5): 763-768.
Brenner S, Elgar G, Sandford R, Macrae A, Venkatesh B A and Paricio S. 1993. Characterization of the pufferfish (Fugu) genome as a compact model vertebrate genome.Nature, 366: 265-268.
Chu D, Gao C S, De Barro P, Wan F H and Zhang Y J. 2011. Investigation of the genetic diversity of an invasive whitefly (Bemisiatabaci) in China using both mitochondrial and nuclear DNA markers.BulletinofEntomologicalResearch, 101: 467-475.
Chu D, Zhang Y J, Cong B, Xu B Y, Wu Q J and Zhu G R. 2005. Sequences analysis of mtDNA COI gene and molecular phylogeny of different geographical populations ofBemisiatabaci(Gennadius).AgriculturalSciencesinChina, 4: 533-541.
Fisher P J, Gardner R C and Richardson T E. 1996. Single locus microsatellite isolated using 5′-anchored PCR.NucleicAcidsResearch, 24: 4369-4371.
Kandpal R P, Kandpal G and Weissman S M. 1994. Construction of libraries enriched for sequence repeats and jumping clones, and hybridization selection for region-specific markers.ProceedingsoftheNationalAcademyoftheSciencesoftheUnitedStatesofAmerica, 91(1): 88-92.
Karagyozov L, Kalcheva I D and Chapman V M. 1993. Construction of random small-insert genomic libraries highly enriched for simple sequence repeats.NucleicAcidsResearch, 21: 3911-3912.
Rassmann K, Schlotterer C and Tautz D. 1991. Isolation of simple sequence loci for use in polymerase chain reaction-based DNA fingerprinting.Electrophoresis, 12(2-3): 113-118.
Weber J L. 1990. Informativeness of human (dC-dA)n. (dG-dT)npolymorphisms.Genomics, 7: 524-554.
