氧化石墨烯保护的核酸分子探针及其在生物分析中的应用
2014-08-07张薇婷杨朝勇
崔 亮,张薇婷,林 晖,朱 志,杨朝勇
(厦门大学 化学化工学院,固体表面物理化学国家重点实验室,谱学分析与仪器教育部重点实验室,福建 厦门 361005)
石墨烯(graphene)是由碳原子以sp2杂化轨道形成六角型蜂巢晶格的平面薄膜[1-2],得益于石墨烯独特的物理、化学及结构性能,石墨烯及其衍生物已被广泛应用于各种领域,包括电子学、材料科学、能源技术、催化科学等[3-5].作为石墨烯的一种衍生物,氧化石墨烯(graphene oxide,GO)是通过对石墨烯氧化加工而成,具有水溶性好、表面易于修饰等特性,因而在生物分析领域得到了广泛的应用[6],其中一个代表性应用就是利用核酸分子探针与GO的相互作用实现一系列生物分析.自2009年,第一篇报道关于DNA分子探针结合GO用于高灵敏检测DNA和蛋白质[7]以来,GO已经发展为一个通用分析平台用于核酸、蛋白质、核酸酶、小分子及金属离子的检测[8-10].除此之外,核酸与GO相互作用带来的新的效应,如吸附于GO表面的核酸能够抵抗核酸酶的降解,也被证明并得到了新的应用[11].在本文里,我们综述了科学家们对GO保护核酸抵抗核酸酶降解的研究及在此研究基础上展开的一系列应用[12].
1 GO对核酸的吸附、保护性能及机理探讨
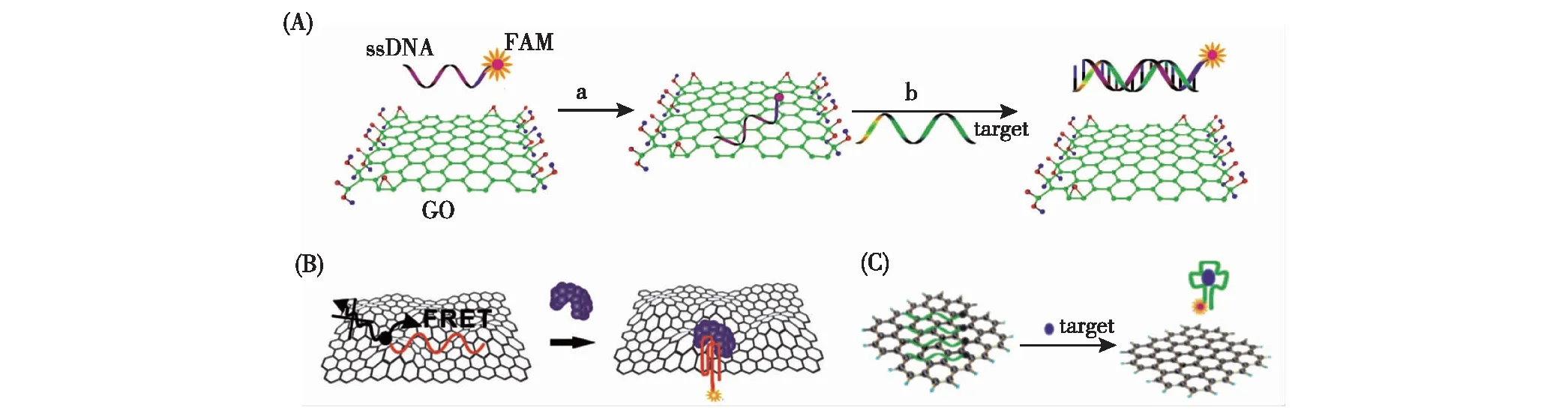
图1 荧光标记的ssDNA用于核酸(A)[7]、蛋白质(B)[13]及小分子(C)[14]的检测原理Fig.1 Fluorophore-labeled ssDNA probes for detection of nucleic acid (A)[7],protein (B)[13] and small molecule (C)[14]
2009年,Yang等[7]报道了荧光标记的单链DNA(single strand DNA,ssDNA)探针能够吸附于GO表面,同时荧光信号被淬灭;当与ssDNA完全互补的目标DNA(complementary DNA,cDNA)出现以后,2条链互补结合形成刚性的双链结构,能够脱离GO表面,被淬灭的荧光信号得到恢复,从而实现对目标DNA的检测(图1(A)).除此以外,当核酸适体(aptamer)与相对应的目标物如蛋白质(图1(B))[13]、小分子(图1(C))[14]结合以后,单链的核酸适体形成了稳定的二级或三级结构,同样能够脱离GO表面,从而利用单荧光标记的核酸适体探针对相对应的目标物进行检测.Fan等[15]研究了GO对ssDNA的吸附作用机理,他们使用分子动力学(molecular dynamics,MD)模拟研究了ssDNA及双链DNA(double strand DNA,dsDNA)在GO表面的作用图谱.通过π-π共价键堆积反应,ssDNA上的核苷酸碱基环能够与GO表面的六边形晶胞产生紧密结合,介导ssDNA稳定地吸附于GO表面.而cDNA的出现使其形成双链,碱基之间氢键相互作用使dsDNA无法再与GO作用,造成dsDNA脱离.
基于ssDNA能够稳定地吸附于GO表面,而dsDNA或者形成了稳定二级/三级结构的ssDNA能够脱离GO的性质,人们开发了各种基于GO平台的化学/生物学传感器用于分析检测,这在许多综述性文章中已有报道[16-22].
ssDNA吸附于GO也带来了其他的生物学效应.2010年,Tang等[11]使用电泳、荧光偏振等方式研究了当ssDNA吸附于GO表面以后,核酸酶对DNA的水解能力.如图2(A)所示,自由的ssDNA与DNase I孵育20 min后,部分被水解(lane 2),60 min后,全部被降解(lane 3).然而,当ssDNA吸附于GO表面以后,即使经过了60 min的DNase I消化,DNA仍然没有发生任何变化.荧光偏振实验同样证明了该现象,荧光标记的ssDNA在与DNase I作用以后,偏振信号逐渐降低,表明由于ssDNA被水解,荧光分子的偏振信号发生了改变.而当荧光标记的ssDNA吸附于GO表面以后,加入DNase I,偏振信号没有发生任何改变,表明ssDNA分子仍然完整地存在.以上结果均表明,ssDNA吸附于GO表面后,能够被GO保护抵抗核酸酶的降解.
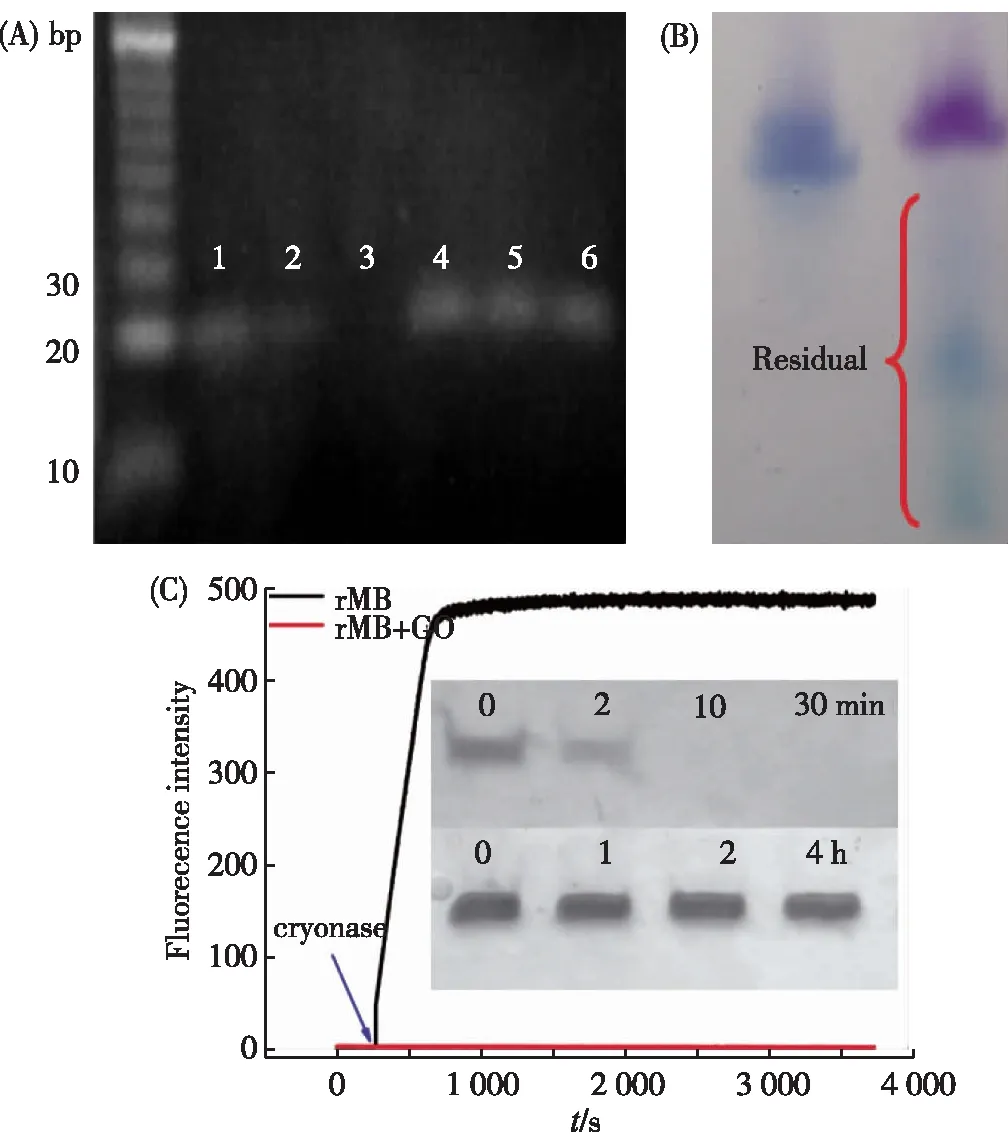
(A)ssDNA以及ssDNA/GO复合物与DNase I的凝胶 电泳结果[11].1.ssDNA,2.ssDNA与DNase I反应20 min, 3.ssDNA与DNase I反应60 min,4.ssDNA/GO复合物, 5.ssDNA/GO与DNase I反应20 min,6.ssDNA/GO与 DNase I反应60 min;(B)凝胶电泳分析probe-GO复合物 (左)以及probe/RNA-GO复合物(右)与DNase I 的作用结果[23];(C) rMB和rMB-GO在cryonase存在下的 实时荧光扫描及电泳结果图(插图)[25].图2 ssDNA、RNA及二者的GO 复合物与核酸酶的相互作用Fig.2 Interation between nuclease and ssDNA, RNA,ssDNA/GO and RNA/GO complexes
在此基础上,我们实验室研究了当吸附于GO表面的ssDNA形成双链脱离GO以后与DNase I的作用效果[23].如图2(B)所示,ssDNA吸附于GO表面后,加入DNase I孵育60 min,ssDNA未被降解,这与前面的研究结果是一致的[11].当加入与其互补的RNA以后,再与DNase I孵育60 min,由于DNase I只能作用于DNA而对RNA没有降解效果,互补的RNA保持完整状态,而ssDNA完全被降解(其中蓝色条带为DNA,紫色条带为RNA).该结果表明,当吸附于GO表面的ssDNA脱离GO表面以后,由于不再受GO的保护,因此能够被核酸酶降解.
以上的结果同样表明,GO保护DNA不受核酸酶降解的性质仅与DNA是否吸附于GO表面相关.当存在足够的盐离子,dsDNA同样能够吸附于GO表面,因此能够抵抗核酸酶的降解[24].
GO除了能够保护DNA不受核酸酶降解以外,同样能够对RNA产生保护能力.我们实验室系统地研究了吸附于GO表面的RNA的抗酶切效果[25].我们使用RNA 分子信标(RNA molecular beacon,rMB)作为研究对象,使用cryonase核酸酶(该酶能够降解一切类型的核酸)作为工具酶.如图2(C)所示,无论荧光还是电泳实验(图2(C)插图),自由的rMB在与cryonase核酸酶作用后短短的时间里即被水解,而当吸附于GO表面后,与cryonase核酸酶长时间作用仍然无任何变化,表明GO能够保护吸附于其表面的RNA不被降解.
到目前为止,愈来愈多的研究报道了金纳米颗粒、硅纳米颗粒、碳纳米管以及GO等纳米材料对核酸的保护能力,并对其保护机理进行了探讨[26-30].我们在此处总结了几种具有说服力的保护机理[31].
首先,在解释金纳米颗粒和硅纳米颗粒对DNA的保护能力时,有人认为这些纳米材料能够诱导其表面产生一个不利于核酸酶发挥活性的盐离子环境,从而导致核酸酶无法对吸附于纳米材料表面/周围的核酸发挥作用[26,28,30].其次,另一个观点则认为纳米材料改变了吸附于其表面的核酸的构象,从而导致核酸酶无法识别[29,32].目前广为接受的一个理论是“位阻效应(steric hindrance)”[11,24,30,33].该理论认为,纳米材料和核酸酶同是纳米尺寸,因此,核酸酶无法攻击纳米材料表面的“小分子”核酸[25].
这些观点在解释一些特定的现象时看似完美,但新的实验现象往往不断挑战着这些理论.比如,“位阻效应”理论认为核酸酶无法攻击吸附于纳米材料表面的DNA,但在2011年,Mirkin等[34]报道了DNA修饰的金纳米颗粒显著地增加了RNase H对RNA的降解能力,该现象与“位阻效应”背道而驰.2012年,Li等[32]报道在一定条件下,dsDNA也能够吸附于GO表面,并且仍然能够被DNase I和EcoR I攻击.但是根据位阻效应,吸附于GO表面的dsDNA能够抵抗核酸酶的攻击,这也有悖于位阻效应.因此,纳米材料保护核酸的机理,仍然存在许多未知,有待于科学家们进一步发掘.
尽管GO对核酸的保护机理仍然有待研究确认,其对核酸的保护能力却是毋庸置疑的.与其他纳米材料无异,GO对核酸的良好保护效果一经发现,便被广泛应用于体内生物成像、生物医学等领域.与此同时,在体外分析中GO-核酸体系一方面可用于构建稳定的RNA探针用于生物分析,另一方面,建立起全新的分析平台——循环酶切信号放大方法(cyclic enzymatic amplification method,CEAM)检测分析物,能够显著提高待测物的检测灵敏度.在接下来的章节中,我们将分别讨论GO保护的核酸分子探针在生物分析领域的各种应用.
2 GO提高RNA分子探针的稳定性
核酸分子探针,包括DNA和RNA分子探针.目前,DNA分子探针已被深入研究和广泛报道,而RNA分子探针的研究工作却相对较少.造成这种现象的主要原因是:RNA具有相对活跃的化学反应基团[35],同时,环境中无处不在的核糖核酸酶,致使RNA容易被降解,最终易导致高背景以及假阳性信号[36].因此,为使RNA分子探针得到广泛应用,我们首要发展稳定RNA的方法[31].

图3 GO保护RNA探针的原理(A)及rMB、GO-rMB的荧光光谱图(B)和信背比(C)[25]Fig.3 Principle of protection of RNA probe with GO(A), fluorescence spectra (B) and signal-background ratio (C) of rMB and GO-rMB[25]
基于GO对核酸分子的保护效果,我们实验室提出了使用GO稳定RNA探针的方法[25].其原理如图3(A)所示,没有GO保护的RNA分子探针,如rMB,在实验室环境下易被降解,因此容易产生假阳性信号.在GO的保护下,rMB只能够对特异的目标物响应,不会产生高背景和假阳性信号,从而有效地检测目标物.我们的方案得到了实验数据的验证,如图3(B)所示,对于自由的rMB,放置于实验室环境下,荧光信号逐日上升,表明rMB在实验室环境极易被降解,到达15 d后,信号趋于饱和,表明rMB已经完全被降解.相对应的,对于GO保护的rMB,经过15 d的实验室环境放置,信号没有发生变化.在此基础上通过考察与cDNA杂交后的信背比(S/B)比较了2种rMB的工作能力,如图3(C)所示,对于未被保护的rMB,第5天信背比已经下降至1.68,而GO保护的rMB到达第15天后,其信背比仍能保持在7.5左右,充分说明RNA分子探针在GO的保护下能够稳定工作.
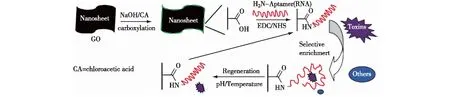
图4 RNA-GO纳米片用于分离和富集肽毒素的方案[38]Fig.4 Schematic fabrication process of RNA-graphene oxide nanosheets[38]
借助RNase H能够特异性水解与DNA杂交的RNA,而对单独的DNA或RNA无作用效果,Corn等基于RNA微阵列发展了用于高灵敏的RNase H辅助的信号放大方法、用于检测基因组DNA的SPR成像技术,然而,由于RNA探针极不稳定,他们最终抛弃了这种方法[37].借助于GO的保护能力,我们重新发展了RNase H辅助的信号放大方法用于检测DNA,该方法实现了0.1 pmol/L DNA检测,并具有区分单碱基错配识别的能力,更重要的是,由于GO的保护,我们的方法能够使RNA探针稳定存在,避免了复杂的操作,从而发展了操作简单、灵敏度高的DNA检测方法[25].
我们同时发展了GO保护的RNA核酸分子探针用于生物分析.设计了GO保护的血管内皮生长因子(VEGF)和茶碱的RNA核酸适体分子探针,分别用于VEGF和茶碱的高灵敏检测,分别实现了2 nmol/L VEGF和2 μmol/L茶碱的灵敏检测.这些方法避免了对RNA繁琐而复杂的操作[25].
GO保护的RNA分子探针除了用于分析物的检测,Zhou等[38]也将其用于富集并捕获饮用水中痕量存在的肽毒素(peptide toxins).如图4所示,他们将肽毒素的RNA核酸适体通过化学交联的方式固定于GO表面,在遇到肽毒素以后,RNA核酸适体识别并与其结合,导致大量的肽毒素富集于GO表面,起到富集捕获的效果.通过调节pH及温度,捕获的肽毒素被释放,使得该GO保护的RNA分子探针能够循环使用.该方法设计简单、制作方便、特异性高,有望在水污染纯化及复杂体系中的样品制备等领域得到广泛应用.
3 基于GO的CEAM
GO对核酸的保护能力除了应用于在实验室环境下稳定RNA探针的存在,还能够与特异的核酸酶结合,发展高灵敏高选择性的放大化分析方法用于提高检测物的灵敏度.
如前所述,荧光标记的ssDNA探针能够吸附于GO表面,目标DNA或其他目标分子与其结合形成双链或具有稳定二级/三级结构的DNA能够脱离GO,荧光信号恢复,从而实现对目标物的检测.然而,在这一类检测方式中,一个目标物只能触发一个分子探针信号的打开,这种1∶1的检测方案限制灵敏度的进一步提高.当研究者们发现GO能够有效地保护ssDNA不受核酸酶降解以后,便发展了CEAM[39-41],实现了一个目标物分子触发多个核酸分子探针信号打开,将对目标物的检测灵敏度提高了2~3个数量级.
我们实验室将该CEAM方法用于MicroRNA(miRNA)的检测[23].miRNA是在真核生物中发现的一类内源性的具有调控功能的非编码RNA,其表达水平与人类许多疾病密切相关,这使miRNA成为新的生物标志物,在癌症等重大疾病的早期诊断中意义重大[42-43].因此,miRNA的定量检测和表达分析是十分重要的[44].基于DNase I对DNA的特异性水解性能,我们实现了放大化高灵敏的miRNA检测[23].其原理如图5(A)所示,将与miRNA互补的DNA荧光探针与GO孵育,此时探针的荧光被淬灭并能够抵抗DNase I的降解.目标miRNA出现后与DNA探针形成了miRNA/DNA双链复合物脱离GO表面,发出荧光信号.同时,脱离了GO表面的DNA探针不再被GO保护,成为DNase I的水解底物.由于DNase I无法水解miRNA,miRNA释放并与下一条DNA探针杂交,再形成双链脱离GO表面,构成水解—杂交—脱附—水解的CEAM过程.同时,针对不同的miRNA设计不同荧光标记的荧光探针能够实现对miRNA的多目标检测[31].
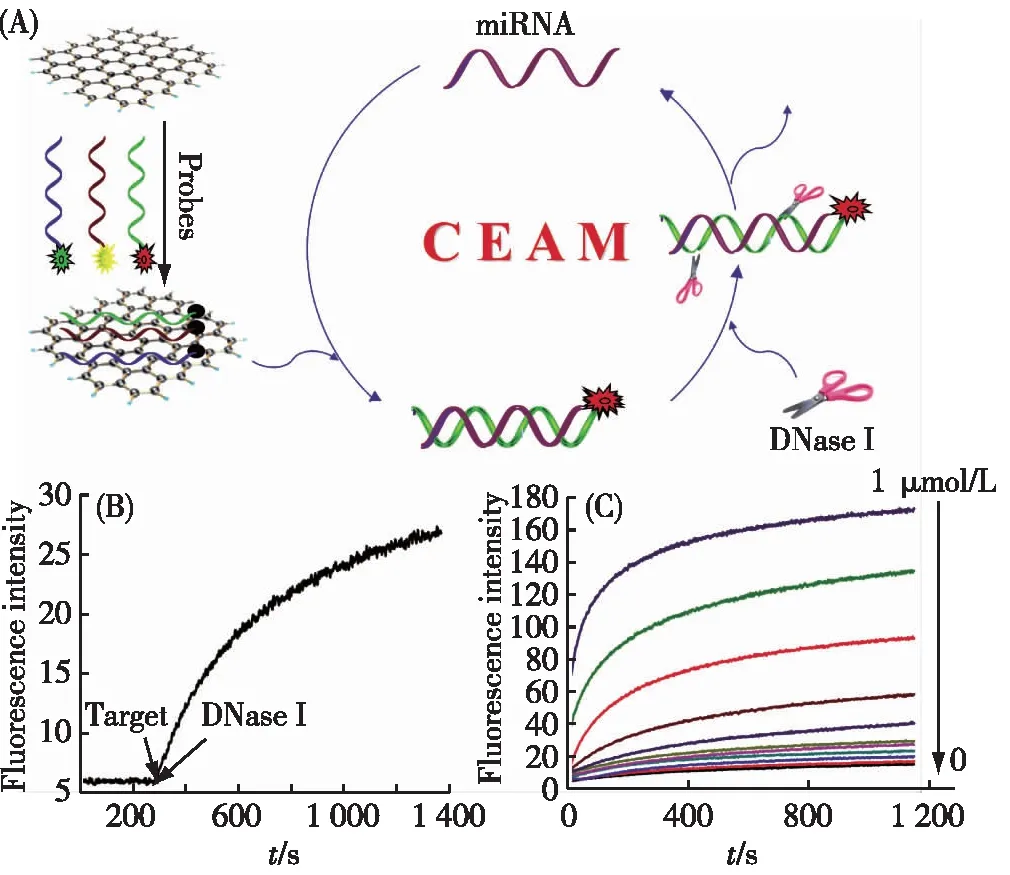
图5 GO保护的ssDNA探针用于miRNA的高灵敏分析[23]Fig.5 Analysis of miRNA based on GO-protected ssDNA probes[23]
图5(B)证明了该方法的信号放大效果.对于GO/探针体系,加入0.1 nmol/L的miRNA,由于在1∶1的检测方案下,此浓度的miRNA触发的荧光信号无法达到仪器能够识别的级别,因此从仪器上观察到的荧光信号没有任何变化.此时加入DNase I,荧光信号在20 min内上升了4倍,说明DNase I辅助的循环酶切放大成功发生,证明了该方法的可行性.使用该方法,我们实现了9 pmol/L的miRNA检测灵敏度(图5(C)),与常规的方法相比,灵敏度提高了3个数量级.同时,该方法能够较好地区分序列高度相似的let 7家族的几种miRNA(他们之间只有一个或几个碱基之间的差别).另外,借助于GO对核酸探针的保护效果,我们的方法能够直接用于复杂的生物体系如细胞裂解液中miRNA检测,并能够区分不同细胞系之间miRNA的表达水平[12].
Yang等设计了用于三磷酸腺苷(ATP)、可卡因等小分子的高灵敏检测CEAM[14].以检测ATP为例,羧基荧光素(FAM)标记的ATP核酸适体探针吸附于GO表面,荧光信号被淬灭,同时DNase I无法将其降解.ATP出现后,与其核酸适体结合形成复合物,脱离GO表面,发出荧光信号.同时,脱离了GO表面的ATP核酸适体分子探针不再受GO保护,成为DNase I的水解底物,从而导致探针被水解,并将ATP释放,释放的ATP重新与另一条核酸适体探针结合,实现解吸附-被水解-释放ATP的循环过程,最终实现了一个ATP分子持续触发多个核酸分子探针释放出荧光信号的过程,从而能够提高对ATP的检测灵敏度.通过这种方式,实现40 nmol/L的ATP检测,相对于1∶1的检测方案(检测限:10 μmol/L),灵敏度提高了200倍.该作者同时将该方法用于可卡因的检测,实现了50 nmol/L的检测灵敏度.
基于同样的原理,Tan等[45]发展了胰岛素的高灵敏检测方法,他们实现了5 nmol/L的胰岛素检测,该方法的建立将有利于发展糖尿病早期诊断的方法.
除建立荧光检测的CEAM方法,CEAM也被拓展到电化学领域.Chen等[46]建立了磁控的GO传感平台用于放大化、多目标、电化学检测ATP和可卡因,分别实现了0.1和1.5 pmol/L的检测灵敏度.
4 以GO为载体的细胞内检测与成像方法
近年来,功能化核酸(functional nucleic acids,FNAs)凭借其独特的优势,在生物分析及生物医学领域得到广泛的应用,包括靶向药物运输、基因调控、体内检测与成像等[47-51].然而,作为一种带负电的生物大分子,FNAs需要通过载体将其带入细胞或体内[28,52];另外,由于细胞内存在各种各样的核酸酶,外源FNAs极易受到降解,因此需要能够稳定/保护FNAs存在的载体或方法[53-54].因此,高效、无毒并能够稳定FNAs的运输载体是必不可少的[28].GO作为一种独特的碳纳米材料,细胞毒性低、能够有效地进入细胞[55-56],更重要的是,能够吸附核酸并保护其不受核酸酶的降解,从而被作为运输载体而广泛应用于细胞及活体内分析.
Lin等[57]将单荧光标记的ATP核酸适体探针与GO共同孵育,探针吸附于GO表面导致荧光淬灭,将此探针/GO复合物用于检测老鼠上皮细胞内的ATP.借助于GO对探针的保护效果,以及GO能够自由进入细胞等优点,该方法成功实现了细胞内ATP的成像检测.在此基础上,为了实现对细胞内不同核苷酸的同时/多目标检测,他们分别设计了FAM标记的ATP核酸适体,Cy5标记的GTP核酸适体,以及Alex546N标记的随机序列,并将他们与GO共同孵育,利用GO对荧光光谱的广谱淬灭效果,所有的荧光探针荧光均被淬灭.当与细胞共同孵育以后,2种核酸适体与其对应的目标物结合,能够发出不同的荧光信号,从而实现对细胞内核苷酸的多目标检测(图6)[58].
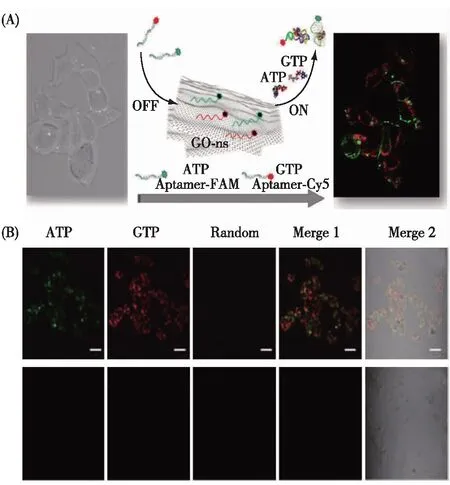
图6 GO作为载体用于细胞内核苷酸的 多目标检测方案(A)和效果(B)[58]Fig.6 Scheme (A) and performance (B) of GO delivery of multiplex aptasensors for intracellular nucleotides detection[58]
与此类似,Yang等[59]使用GO作为载体将分子信标载入HeLa细胞内用于生存素(survivin)mRNA的检测.他们分别设计了生存素mRNA的分子信标及随机序列分子信标,并将其分别与GO孵育,考察了GO存在和不存在的情况下,生存素mRNA的分子信标及随机序列的分子信标与HeLa细胞相互作用的效果.结果表明,吸附于GO表面的生存素mRNA的分子信标能够在GO的载体作用下进入细胞,并在遇到生存素mRNA后打开,在荧光显微镜下观察到荧光信号.作为对照,自由的生存素mRNA分子信标以及吸附于GO表面的随机序列分子信标在与HeLa作用以后,没有可观察到的荧光信号.该结果表明,GO能够作为载体将分子信标带入到细胞内用于检测目标mRNA.
随着GO被广泛应用的同时,其不足之处也逐渐暴露,其中之一是其尺寸的不均一性.单原子排列的GO在其二维平面上的随机扩展,长度能够从纳米级别延伸到微米级别.这种尺寸的不均一性造成其氧化程度不均一、实验重复性差以及进入细胞困难等诸多问题[60].为此,科学家们发展了一系列纳米尺寸的GO(nanoscale graphene oxide,nGO;graphene oxide quantum dots),并将其应用于体内/体外的生物传感、生物医学等领域.
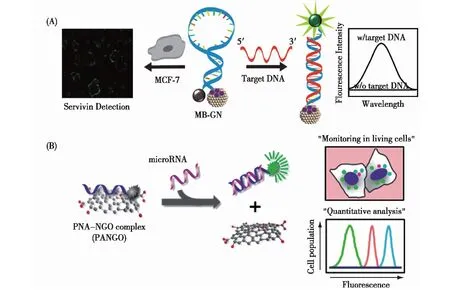
图7 纳米尺寸的GO用于细胞内mRNA(A)[61]及miRNA(B)[62]的成像分析Fig.7 Scheme of strategy for mRNA(A)[61]/miRNA(B)[62] sensor based on nGO
Seo等[61]设计了球形的graphite nanoparticle (GN) 交联的分子信标用于细胞内基因检测.这种GN尺寸均一(4~5 nm),当分子信标与其交联以后,能够保护MB不受核酸酶降解,同时,由于其具有较小的尺寸和较好的进入细胞的能力,而被设计为用于细胞内特定基因检测的工具(图7(A)).
肽核酸是一种以多肽骨架取代糖磷酸主链的DNA类似物,与天然核酸相比,肽核酸呈电中性;同时,肽核酸与天然核酸杂交形成的双链稳定性要高于天然的核酸双链.基于这些特性,科学家们研究了荧光标记的肽核酸探针与nGO结合用于体外及体内分析物的传感.一方面,电中性的肽核酸与nGO具有更强的结合力,因此降低了背景信号;另一方面,肽核酸探针与cDNA/cRNA的结合具有更强的稳定性,从而能够摆脱nGO的吸附,提高了检测信号.他们将肽核酸探针/nGO用于miRNA检测,在1∶1的检测方案下实现了1 pmol/L的检测灵敏度.在此基础上,实现了细胞内miRNA的定量检测(图7(B))[62].
上述研究,以GO/nGO为载体,有效地实现了细胞内mRNA、miRNA、ATP、GTP等重要生物分子的成像检测.同时,也证明了作为一种新材料,GO:1) 能够作为载体有效地运输FNAs进入细胞;2) 能够保护FNAs不受细胞内核酸酶的降解,因此能够使其稳定存在;3) 能够与FNAs形成复合物构建传感平台,用于细胞成像.
5 结论和展望
自2004年第一次报道以来,石墨烯和GO就以其突出的物理/化学性质而被应用于各种领域[1,63].近年来,GO对核酸的结合能力以及由此带来的抗酶切效果,吸引了科学家们的广泛关注并展开了新的应用.一方面,GO能够保护RNA探针分子不受环境中核酸酶的攻击,从而发展了稳定RNA存在的方法用于特定目标物的检测、富集及分离;另一方面,GO对ssDNA的吸附及dsDNA的脱吸附,导致相应的保护/去保护效果,激发了科学家们发展CEAM用于目标分析物的高灵敏检测.相对于常规的1∶1检测方案,CEAM实现了2~3个数量级灵敏度的提高.在体内,GO对核酸的保护效果,以及GO本身自由进入细胞的能力、体内无毒性等优点使其能够成为优秀的“共靶向”分子,用于细胞内检测和成像等[12].
目前,对GO保护核酸的研究及应用仍然有待深入.一方面,GO能够保护核酸抵抗核酸酶降解的机理仍然有待探讨,新的理论和假说需要提出[31].另一方面,GO保护核酸的应用仍然处于概念论证(proof-of-concept)阶段.为了能够使该研究真正应用于实际检测或临床体系,需要验证更多的目标物以及更复杂的生物体系.最后,除GO相关的研究之外,本文也提到了其他纳米材料与功能核酸之间的相互作用及由此带来的应用.众所周知,近年来新的纳米材料层出不穷,如碳纳米颗粒[64]、C60[65]、上转换纳米颗粒及其他纳米材料[66-67],这些新的纳米材料与核酸又具有怎样的相互作用关系、这些纳米材料本身又具有什么样的独特的性能,这部分研究工作将是引人入胜又充满挑战,而纳米材料与功能化核酸的结合将对分子生物学、生物化学、分子医学等领域产生新的进步性影响.
[1] Novoselov K S,Geim A K,Morozov S V,et al.Electric field effect in atomically thin carbon films[J].Science,2004,306 (5696):666-669.
[2] Geim A K,Novoselov K S,The rise of graphene[J].Nature Materials,2007,6(3):183-191.
[3] Rao C N,Sood A K,Subrahmanyam K S,et al.Graphene:the new two-dimensional nanomaterial[J].Angew Chem Int Ed,2009,48(42):7752-7777.
[4] Yang W,Ratinac K R,Ringer S P,et al.Carbon nanomaterials in biosensors:should you use nanotubes or graphene?[J].Angew Chem Int Ed,2010,49 (12):2114-2138.
[5] Shen H,Zhang L,Liu M,et al.Biomedical applications of graphene[J].Theranostics,2012,2(3):283-294.
[6] Guo S,Dong S,Graphene nanosheet:synthesis,molecular engineering,thin film,hybrids,and energy and analytical applications[J].Chemical Society Reviews,2011,40(5):2644-2672.
[7] Lu C H,Yang H H,Zhu C L,et al.A graphene platform for sensing biomolecules[J].Angew Chem Int Ed,2009,48(26):4785-4787.
[8] Loh K P,Bao Q L,Eda G,et al.Graphene oxide as a chemically tunable platform for optical applications[J].Nature Chemistry,2010,2(12):1015-1024.
[9] Liu Y,Dong X,Chen P.Biological and chemical sensors based on graphene materials[J].Chemical Society Reviews,2012,41(6):2283-2307.
[10] Morales-Narvaez E,Merkoci A.Graphene oxide as an optical biosensing platform[J].Advanced Materials,2012,24(25):3298-3308.
[11] Tang Z,Wu H,Cort J R,et al.Constraint of DNA on functionalized graphene improves its biostability and specificity[J].Small,2010,6 (11):1205-1209.
[12] Cui L,Song Y,Ke G.et al.Graphene oxide protected nucleic acid probes for bioanalysis and biomedicine[J].Chemistry-A European Journal,2013,19(32):10442-10451.
[13] Chang H X,Tang L H,Wang Y,et al.Graphene fluorescence resonance energy transfer aptasensor for the thrombin detection[J].Analytical Chemistry,2010,82(6):2341-2346.
[14] Lu C H,Li J,Lin M H,et al.Amplified aptamer-based assay through catalytic recycling of the analyte[J].Angew Chem Int Ed,2010,49 (45):8454-8457.
[15] He S J,Song B,Li D,et al.A graphene nanoprobe for rapid,sensitive,and multicolor fluorescent DNA analysis[J].Advanced Functional Materials,2010,20(3):453-459.
[16] Stankovich S,Dikin D A,Dommett G H,et al.Graphene-based composite materials[J].Nature,2006,442 (7100):282-286.
[17] Li F,Huang Y,Yang Q,et al.A graphene-enhanced molecular beacon for homogeneous DNA detection[J].Nanoscale,2010,2 (6):1021-1026.
[18] Lu C H,Li J,Liu J J,et al.Increasing the sensitivity and single-base mismatch selectivity of the molecular beacon using graphene oxide as the "nanoquencher"[J].Chemistry-A European Journal,2010,16(16):4889-4894.
[19] Somers R C,Bawendi M G,Nocera D G.CdSe nanocrystal based chem-/bio-sensors[J].Chemical Society Reviews,2007,36(4):579-591.
[20] Chung C,Kim Y K,Shin D,et al.Biomedical applications of graphene and graphene oxide[J].Accounts of Chemical Research,2013,46(10):2211-2224.
[21] Chen D,Feng H,Li J.Graphene oxide:preparation,functionalization,and electrochemical applications[J].Chemical Reviews,2012,112(11):6027-6053.
[22] Wang Y,Li Z,Wang J,et al.Graphene and graphene oxide:biofunctionalization and applications in biotechnology[J].Trends in Biotechnology,2011,29(5):205-212.
[23] Cui L,Lin X,Lin N,et al.Graphene oxide-protected DNA probes for multiplex microRNA analysis in complex biological samples based on a cyclic enzymatic amplification method[J].Chemical Communications,2012,48(2):194-196.
[24] Lei H,Mi L,Zhou X,et al.Adsorption of double-stranded DNA to graphene oxide preventing enzymatic digestion[J].Nanoscale,2011,3(9):3888-3892.
[25] Cui L,Chen Z,Zhu Z,et al.Stabilization of ssRNA on graphene oxide surface:an effective way to design highly robust RNA probes[J].Analytical Chemistry,2013,85(4):2269-2275.
[26] He X X,Wang K,Tan W,et al.Bioconjugated nanoparticles for DNA protection from cleavage[J].J Am Chem Soc,2003,125(24):7168-7169.
[27] Chen C C,Liu Y C,Wu C H,et al.Preparation of fluorescent silica nanotubes and their application in gene delivery[J].Advanced Materials,2005,17(4):404-407.
[28] Giljohann D A,Seferos D S,Daniel W L,et al.Gold nanoparticles for biology and medicine[J].Angew Chem Int Ed,2010,49(19):3280-3294.
[29] Wu Y,Phillips J A,Liu H,et al.Carbon nanotubes protect DNA strands during cellular delivery[J].ACS Nano,2008,2(10):2023-2028.
[30] Seferos D S,Prigodich A E,Giljohann D A,et al.Polyvalent DNA nanoparticle conjugates stabilize nucleic acids[J].Nano Letters,2009,9 (1):308-311.
[31] 崔亮.核酸分子探针信号放大新策略及其在生物分析中的应用[D].厦门:厦门大学,2013.
[32] Tang L,Chang H,Liu Y,et al.Duplex DNA/graphene oxide biointerface:from fundamental understanding to specific enzymatic effects[J].Advanced Functional Materials,2012,22(14):3083-3088.
[33] Nielsen L J,Olsen L F,Ozalp V C.Aptamers embedded in polyacrylamide nanoparticles:a tool for in vivo metabolite sensing[J].ACS Nano,2010,4 (8):4361-4370.
[34] Prigodich A E,Alhasan A H,Mirkin C A.Selective enhancement of nucleases by polyvalent DNA-functionalized gold nanoparticles[J].J Am Chem Soc,2011,133 (7):2120-2123.
[35] Oivanen M,Kuusela S,Lonnberg H.Kinetics and mechanisms for the cleavage and isomerization of the phosphodiester bonds of RNA by bronsted acids and bases[J].Chemical Reviews,1998,98(3):961-990.
[36] Carrasquilla C,Lau P S,Li Y,et al.Stabilizing structure-switching signaling RNA aptamers by entrapment in sol-gel derived materials for solid-phase assays[J].J Am Chem Soc,2012,134 (26):10998-11005.
[37] Goodrich T T,Lee H J,Corn R M.Direct detection of genomic DNA by enzymatically amplified SPR imaging measurements of RNA microarrays[J].J Am Chem Soc,2004,126 (13):4086-4087.
[38] Hu X,Mu L,Wen J,et al.Immobilized smart RNA on graphene oxide nanosheets to specifically recognize and adsorb trace peptide toxins in drinking water[J].Journal of Hazardous Materials,2012,213/214:387-392.
[39] Cui L,Ke G,Wang C,et al.A cyclic enzymatic amplification method for sensitive and selective detection of nucleic acids[J].Analyst,2010,135 (8):2069-2073.
[40] Cui L,Ke G,Zhang W Y,et al.A universal platform for sensitive and selective colorimetric DNA detection based on Exo III assisted signal amplification[J].Biosensors and Bioelectronics,2011,26 (5):2796-2800.
[41] Yang C J,Cui L,Huang J,et al.Linear molecular beacons for highly sensitive bioanalysis based on cyclic Exo III enzymatic amplification[J].Biosensors and Bioelectronics,2011,27 (1):119-124.
[42] Farh K K H,Grimson A,Jan C,et al.The widespread impact of mammalian microRNAs on mRNA repression and evolution[J].Science,2005,310 (5755):1817-1821.
[43] Grimson A,Farh K K,Johnston W K,et al.MicroRNA targeting specificity in mammals:determinants beyond seed pairing[J].Molecular Cell,2007,27 (1):91-105.
[44] Cui L,Zhu Z,Lin N,et al.A T7 exonuclease-assisted cyclic enzymatic amplification method coupled with rolling circle amplification:a dual-amplification strategy for sensitive and selective microRNA detection[J].Chemical Communications,2014,50:1576-1578.
[45] Pu Y,Zhu Z,Han D,et al.Insulin-binding aptamer-conjugated graphene oxide for insulin detection[J].Analyst,2011,136 (20):4138-4140.
[46] Tang D,Tang J,Li Q,et al.Ultrasensitive aptamer-based multiplexed electrochemical detection by coupling distinguishable signal tags with catalytic recycling of DNase I[J].Analytical Chemistry,2011,83 (19):7255-7259.
[47] Stein C A,Cheng Y C.Antisense oligonucleotides as therapeutic agents--is the bullet really magical?[J].Science,1993,261(5124):1004-1012.
[48] Wang K M,Tang Z W,Yang C Y J,et al.Molecular engineering of DNA:molecular beacons[J].Angew Chem Int Ed,2009,48 (5):856-870.
[49] Kaur H,Babu B R,Maiti S.Perspectives on chemistry and therapeutic applications of locked nucleic acid (LNA)[J].Chemical Reviews,2007,107 (11):4672-4697.
[50] Seferos D S,Giljohann D A,Hill H D,et al.Nano-flares:probes for transfection and mRNA detection in living cells[J].J Am Chem Soc,2007,129(50):15477-15479.
[51] Cutler J I,Auyeung E,Mirkin C A.Spherical nucleic acids[J].J Am Chem Soc,2012,134(3):1376-1391.
[52] Lebedeva I,Stein C A.Antisense oligonucleotides:promise and reality[J].Annual Review of Pharmacology and Toxicology,2001,41:403-419.
[53] Uchiyama H,Hirano K,Kashiwasake-Jibu M,et al.Detection of undegraded oligonucleotides in vivo by fluorescence resonance energy transfer.Nuclease activities in living sea urchin eggs[J].Journal of Biological Chemistry,1996,271 (1):380-384.
[54] Yang C J,Wang L,Wu Y,et al.Synthesis and investigation of deoxyribonucleic acid/locked nucleic acid chimeric molecular beacons[J].Nucleic Acids Research,2007,35 (12):4030-4041.
[55] Jeong H K,Jin M,Ra E J,et al.Enhanced electric double layer capacitance of graphite oxide intercalated by poly(sodium 4-styrensulfonate) with high cycle stability[J].ACS Nano,2010,4 (2):1162-1166.
[56] Liu Z,Robinson J T,Sun X,et al.PEGylated nanographene oxide for delivery of water-insoluble cancer drugs[J].J Am Chem Soc,2008,130 (33):10876-10877.
[57] Wang Y,Li Z,Hu D,et al.Aptamer/graphene oxide nanocomplex for in situ molecular probing in living cells[J].J Am Chem Soc,2010,132 (27):9274-9276.
[58] Wang Y,Li Z,Weber T J,et al.In situ live cell sensing of multiple nucleotides exploiting DNA/RNA aptamers and graphene oxide nanosheets[J].Analytical Chemistry,2013,85 (14):6775-6782.
[59] Lu C H,Zhu C L,Li J,et al.Using graphene to protect DNA from cleavage during cellular delivery[J].Chemical Communications,2010,46 (18):3116-3118.
[60] Chou S S,De M,Luo J Y,et al.Nanoscale graphene oxide (nGO) as artificial receptors:implications for biomolecular interactions and sensing[J].J Am Chem Soc,2012,134(40):16725-16733.
[61] Piao Y X,Liu F,Seo T S.A novel molecular beacon bearing a graphite nanoparticle as a nanoquencher for in situ mRNA detection in cancer cells[J].Acs Applied Materials & Interfaces,2012,4(12):6784-6788.
[62] Ryoo S R,Lee J,Yeo J,et al.Quantitative and multiplexed microRNA sensing in living cells based on peptide nucleic acid and nano graphene oxide (PANGO)[J].ACS Nano,2013,7(7):5882-5891.
[63] Allen M J,Tung V C,Kaner R B.Honeycomb carbon:a review of graphene[J].Chemical Reviews,2010,110 (1):132-145.
[64] Liu H,Ye T,Mao C.Fluorescent carbon nanoparticles derived from candle soot[J].Angew Chem Int Ed,2007,46(34):6473-6475.
[65] Zheng X,Liu Q,Jing C,et al.Catalytic gold nanoparticles for nanoplasmonic detection of DNA hybridization[J].Angew Chem Int Ed,2011,50(50):11994-11998.
[66] Wang F,Banerjee D,Liu Y S,et al.Upconversion nanoparticles in biological labeling,imaging,and therapy[J].Analyst,2010,135(8):1839-1854.
[67] Haase M,Schafer H.Upconverting nanoparticles[J].Angew Chem Int Ed,2011,50(26):5808-5829.
