海洋细菌NTa发酵产琼胶酶条件的初步优化
2014-07-02马芮萍朱艳冰肖安风蔡慧农
马芮萍,朱艳冰,倪 辉,肖安风,蔡慧农
(集美大学生物工程学院,福建 厦门 361021)
0 引言
琼脂主要是从海洋红藻中提取出来的一种细胞间多糖,由琼脂糖和硫琼胶组成.琼脂糖是不含有硫酸酯盐的非离子型多糖,由D-半乳糖和3,6-内醚-α-L-半乳糖以α-1,3糖苷键和β-1,4糖苷键交替连接形成的线性分子,是形成凝胶的组分;硫琼胶结构较复杂,具有和琼脂糖相似的基本二糖重复单位,但带有硫酸酯、甲氧基、葡萄糖醛酸和丙酮醛酸等基团,是非凝胶组分[1].琼胶酶可催化水解琼脂糖分子内的糖苷键,根据其作用于琼脂糖的方式分为两大类:α-琼胶酶(E.C.3.2.1.158)和β-琼胶酶(E.C.3.2.1.81),前者可切断琼脂糖的α-1,3糖苷键生成琼寡糖,如琼二糖、琼四糖及琼六糖,后者切断琼脂糖的β-1,4糖苷键,作用产物为新琼寡糖[1].琼胶酶水解琼脂形成的琼脂低聚糖具有减缓淀粉水解、抗氧化、抑制脂质过氧化及保湿等功能,广泛用于食品、化妆品及制药工业等领域[1].除可用于琼脂低聚糖的制备,琼胶酶也是生物研究领域的重要工具酶,用于分离制备海藻原生质体,从海藻中提取不饱和脂肪酸、类胡萝卜素及甜菜碱等生物活性物质[2].因此,琼胶酶及其产酶菌株是近年来海洋生物资源开发利用的热点,具有重要的应用价值.
微生物来源的琼胶酶产生菌主要是从海水、海洋沉积物、海洋软体动物、淡水、土壤及海藻中发现的,包括假单孢菌属 (Pseudomonas sp.)[3]、纤维菌属 (Cellvibrio sp.)[4]、噬琼胶菌属(Agarivorans sp.)[5]、不动杆菌属 (Acinetobacter sp.)[6]、红球菌属 (Rhodococcus sp.)[7]、噬细胞菌属 (Cytophaga sp.)[8]、弧菌属 (Vibrio sp.)[9]、假交替单孢菌属 (Alteromonas sp.)[10]、微球菌属 (Micrococcus sp.)[11]、桃色杆菌属 (Persicobacter sp.)[12]、盐杆菌属 (Salegentibacter sp.)[13]和紫色杆菌属(Janthinobacterium sp.)[14]等.目前,还未见寡养单胞菌属(Stenotrophomonas sp.)细菌产琼胶酶的研究报道.本文尝试从厦门红树林泥土样品中分离出寡养单胞菌属(Stenotrophomonas sp.)海洋细菌,探讨了该菌株产琼胶酶的最适培养基组成和最佳产酶温度.
1 材料和方法
1.1 材料
泥土样品于2012年10月采自厦门集美红树林.
1)选择培养基:琼脂20 g,NaNO35 g,豆饼粉0.5 g,MgCl·6H2O 0.02 g,K2HPO40.1 g,FeSO4·7H2O 0.02 g,过滤海水1000 mL,pH=7.5.
2)液体种子培养基:细菌学蛋白胨1 g,酵母浸膏0.5 g,KNO35 g,NaCl 30 g,MgSO4·7H2O 5 g,无水CaCl20.2 g,K2HPO40.1 g,FeSO4·7H2O 0.02 g,蒸馏水1000 mL,pH=7.5.
3)摇瓶基础发酵培养基:琼脂2 g,KNO35 g,NaCl 30 g,MgSO4·7H2O 5 g,无水CaCl20.2 g,K2HPO40.1 g,FeSO4·7H2O 0.02 g,蒸馏水 1000 mL,pH=7.5.
1.2 主要试剂和仪器
细菌基因组DNA提取试剂盒、DNA凝胶回收试剂盒、Taq酶均购于广州东盛生物科技有限公司,其他试剂均为市售分析纯.
WFJ型可见分光光度计 (尤尼柯仪器有限公司);YXQ-LS-30SH立式压力蒸汽灭菌器 (上海博迅实业有限公司医疗设备厂);数显恒温水浴锅 (常州国华电器有限公司);PCR仪 (美国BIORAD公司);5417R高速冷冻离心机 (德国Eppendorf公司).
1.3 方法
1)产琼胶酶细菌的分离 称取适量泥土样品,用无菌过滤海水进行10倍梯度稀释.取稀释后的泥土样品200 μL,分别涂布于选择培养基,22℃培养5 d.选取菌落周围具有明显水解圈或凹陷的菌株进行3次以上划线纯化培养,根据菌落的凹陷程度和菌落周围的透明圈,选取产琼胶酶能力最强的菌株 (命名为NTa)进行下一步研究.
2)16S rRNA基因的PCR扩增、序列测定以及同源性分析 利用细菌基因组DNA提取试剂盒提取菌株的基因组DNA,并以此为模板采用16S rRNA基因的通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-GGCTA CCTTGTTACGACTT-3')进行PCR扩增.反应条件为:95℃5 min;94℃ 45 s,55℃ 45 s,72℃ 90 s,30个循环;72℃ 10 min.PCR产物利用琼脂糖凝胶电泳检测后,由北京六合华大基因科技股份有限公司测序.所得16S rRNA基因序列通过NCBI(http://www.ncbi.nlm.nih.gov/)Blast与GenBank数据库中的已知序列进行相似性比对,运用MEGA 4.0的Neighbor-Joining(NJ)方法构建系统发育树.
3)生理生化鉴定 菌株的生长温度、革兰氏染色、芽孢染色、明胶液化实验、硝酸盐还原实验和卵磷脂酶实验参照《常见细菌系统鉴定手册》[15].
4)琼胶酶粗酶样品制备 将菌株NTa种子液接种于摇瓶基础发酵培养基,25℃下180 r/min培养36 h后,发酵液于4℃、12000 r/min离心10 min,取上清液即为发酵粗酶液.
5)生物量的测定 取发酵液1 mL,12000 r/min离心10 min,倾去上清液,补充蒸馏水至3 mL,混合均匀后以蒸馏水作空白对照,在600 nm处测定吸光度值,检测微生物的生物量.
6)琼胶酶活力的测定 取50 μL粗酶液加入450 μL含0.5%琼脂 (质量分数)的50 mmol/L NaH2PO4-Na2HPO4缓冲液 (pH=7.0)中,40℃水浴反应10 min,加入500 μL DNS溶液,置于沸水浴中5 min,冷却后,用蒸馏水定容至5 mL,充分混匀后,于540 nm处测定吸光度值.以灭活的酶液作对照.在此条件下,每分钟释放1 μmoL还原糖所需的酶量定义为一个酶活力单位 (U).
7)总糖的测定 发酵液稀释10倍后,吸取1 mL于试管中,加入2 mL蒽酮试剂,沸水浴反应10 min,冷却后,室温放置10 min,在620 nm处测定吸光度值.
8)不同碳源对菌株产酶的影响 发酵基础培养基其他成分不变,分别以0.2%(m/V)琼脂、江蓠、海藻酸钠、岩藻多糖、葡萄糖和麦芽糖为唯一碳源配制培养基,培养后测定生物量及发酵液琼胶酶活力,试验不同碳源对菌株产酶的影响.
9)混合碳源对菌株产酶的影响 发酵基础培养基其他成分不变,添加0.2%(m/V)的琼脂后,再分别添加0.2%(m/V)葡萄糖、麦芽糖、海藻酸钠和岩藻多糖作为额外碳源配制培养基,培养后测定生物量及发酵液琼胶酶活力,试验额外添加的不同碳源对菌株产酶的影响.
10)不同氮源对菌株产酶的影响 发酵基础培养基其他成分不变,保持氮元素含量为0.07%(m/V),分别以硫酸铵、硝酸钾、硝酸铵、蛋白胨和酵母浸膏为唯一氮源配制培养基,培养后测定生物量及发酵液琼胶酶活力,试验不同氮源对菌株产酶的影响.
11)NaCl质量分数对菌株产酶的影响 发酵培养基中NaCl质量分数(m/V)分别为1.0%,2.0%,3.0%,4.0%,5.0%和6.0%,培养后测定生物量、发酵液琼胶酶活力及总糖含量,试验NaCl的质量分数对菌株产酶的影响.
12)培养温度对菌株产酶的影响 培养温度分别为19℃、22℃、25℃、28℃和31℃,培养后测定生物量、发酵液琼胶酶活力及总糖含量,试验温度对菌株产酶的影响.
13)Stenotrophomonas sp.NTa发酵产琼胶酶的动态规律分析 将菌种接入优化后的发酵培养基,并按照优化后的条件进行培养,每隔8 h取发酵液测定生物量、琼胶酶活力及总糖含量,试验菌株发酵产琼胶酶的动态规律.
14)统计分析 试验数据为3次平行试验的平均值,应用Excel软件计算平均值和标准偏差,SPSS17.0软件 (SPSS Inc.H,Chicago,IL)对结果进行差异显著性分析(P〈0.05).
2 结果
2.1 产琼胶酶菌株的分离
NTa在琼脂平板上可观察到菌落周围有非常明显的凹陷,该菌株在平板上形成规则的圆形菌落,表面光滑湿润,边缘整齐,呈乳白色.
2.2 菌株的鉴定
以提取的菌株NTa的基因组DNA为模板、27F和1492R为引物进行PCR扩增,在大约1500 bp处得到单一、明亮的条带 (见图1).目的基因进行测序,得到1451个碱基序列,将序列提交到GenBank获得登录号为KF516076.利用BLAST程序将所得16S rRNA基因序列与GenBank数据库的序列进行比对,发现菌株NTa的16S rRNA基因序列与寡养单胞菌属其他菌株的序列相似度在96%以上.一般认为,16S rRNA基因序列相似度大于95%时,可初步判定属于同一个属[16].选取一些相似性较高的序列,通过MEGA 4.0软件构建系统发育树,结果(见图2)显示,菌株NTa位于寡养单胞菌属的分枝下,与 Stenotrophomonas sp.2325(JX174202.1)和Stenotrophomonas malltophilia G46(JF783988)的系统发育关系最为密切,其序列相似度达到99.86%和99.72%.

图1 16S rRNA基因的PCR产物Fig.1 PCR product of 16S rRNA gene
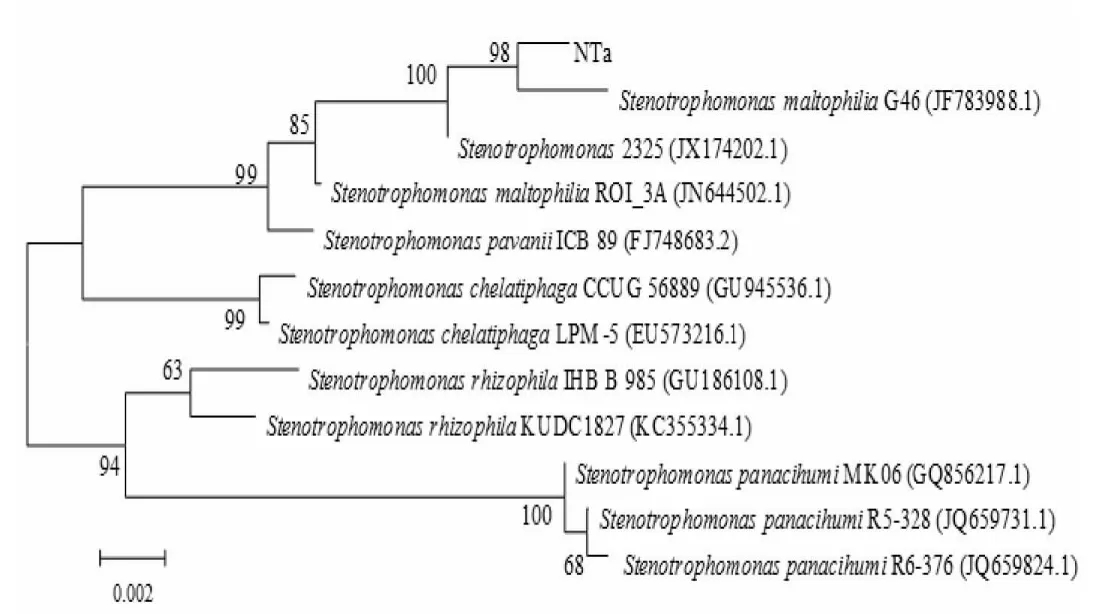
图2 菌株NTa 16S rRNA基因序列系统发育树Fig.2 Phylogenetic tree derived from 16S rRNA gene sequence of strain NTa
菌株NTa为革兰氏阴性,无芽孢,菌体呈直杆状.菌株的部分生理生化鉴定结果显示,明胶液化阴性,硝酸盐还原阳性,卵磷脂酶阴性,利用葡萄糖和麦芽糖产酸不产气,可在19~40℃生长,最适生长温度为25℃.根据菌株的典型生理生化特征及16S rRNA基因系统进化分析,将菌株NTa鉴定为寡养单胞菌(Stenotrophomonas sp.).
2.3 不同碳源对菌株产酶的影响
在无碳源的基础发酵培养基中按0.2%(m/V)的比例分别加入6种碳源,发酵后测定生物量及琼胶酶活力,结果如图3a所示.由图3a可见,Stenotrophomonas sp.NTa在以琼脂和江蓠作为唯一碳源时生长良好,酶活力分别为1.44 U/mL和1.25 U/mL,产酶量前者是后者的1.15倍.当以海藻酸钠、岩藻多糖、葡萄糖和麦芽糖为唯一碳源时菌株可以生长,但是产酶能力弱.这些结果表明,琼脂和江蓠都可以诱导菌株产琼胶酶,琼胶降解菌Stenotrophomonas sp.NTa发酵产琼胶酶的最佳碳源是琼脂.此外,其他碳源也可微弱诱导菌株产琼胶酶.
2.4 混合碳源对菌株产酶的影响
从图3b中可以看出,添加质量分数0.2%琼脂时,同时分别添加质量分数为0.2%的葡萄糖、麦芽糖、海藻酸钠和岩藻多糖,琼胶酶活力降低.额外添加葡萄糖和麦芽糖时,菌株生长旺盛,是以琼脂为唯一碳源时的1.29倍,但是酶活力降低了10.80%.添加海藻酸钠时菌株生长良好,但酶活力降低了12.89%.添加岩藻多糖后菌株基本不产琼胶酶.综上所述,菌株Stenotrophomonas sp.NTa发酵产琼胶酶时不需要额外添加碳源,在后续研究中以琼脂为唯一碳源进行培养.
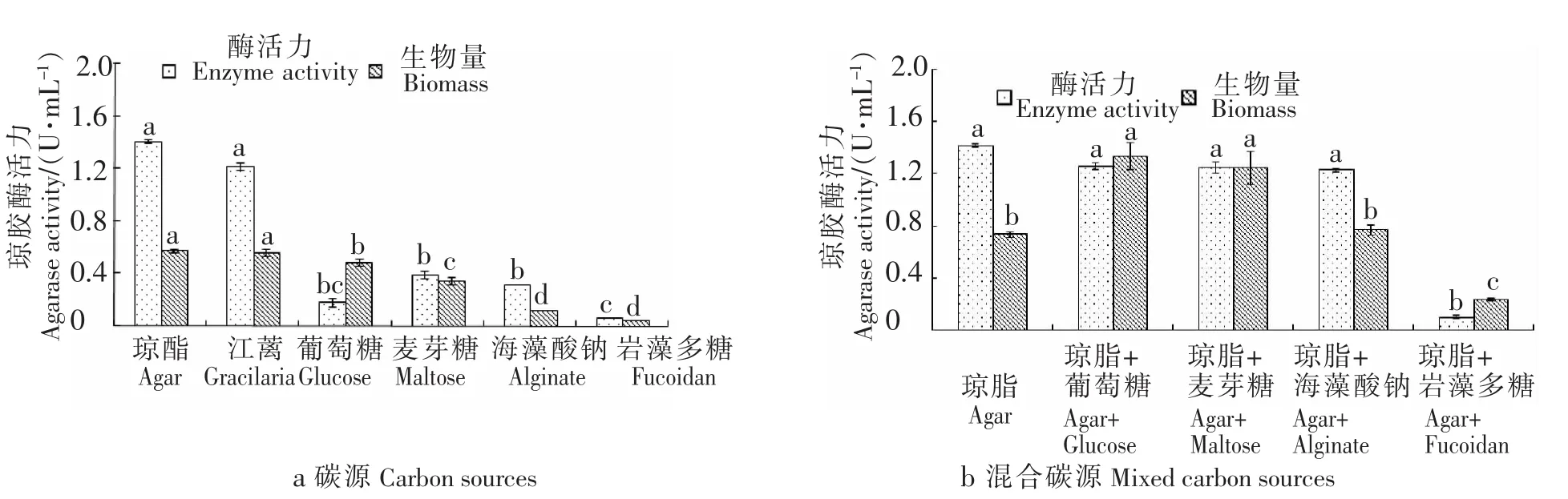
图3 不同碳源对菌株NTa产琼胶酶及菌体生长的影响Fig.3 Effects of different carbon sources on agarase production and microorganism growth for the fermentation of Stenotrophomonas sp.NTa
2.5 不同氮源对菌株产酶的影响
本文比较了常规氮源对Stenotrophomonas sp.NTa产琼胶酶的影响,结果如图4所示.研究发现,在以酵母浸膏作为氮源时,菌株的生物量和产酶能力均达到最大,琼胶酶活力达到1.45 U/mL,其次是KNO3,琼胶酶活力达到1.44 U/mL,但是生物量明显低于以酵母浸膏作为氮源时.当以 (NH4)2SO4和NH4NO3作为氮源时,菌株仅可微弱生长,但是基本不产琼胶酶.因此,选取酵母浸膏为菌株Stenotrophomonas sp.NTa发酵产琼胶酶的最佳氮源.
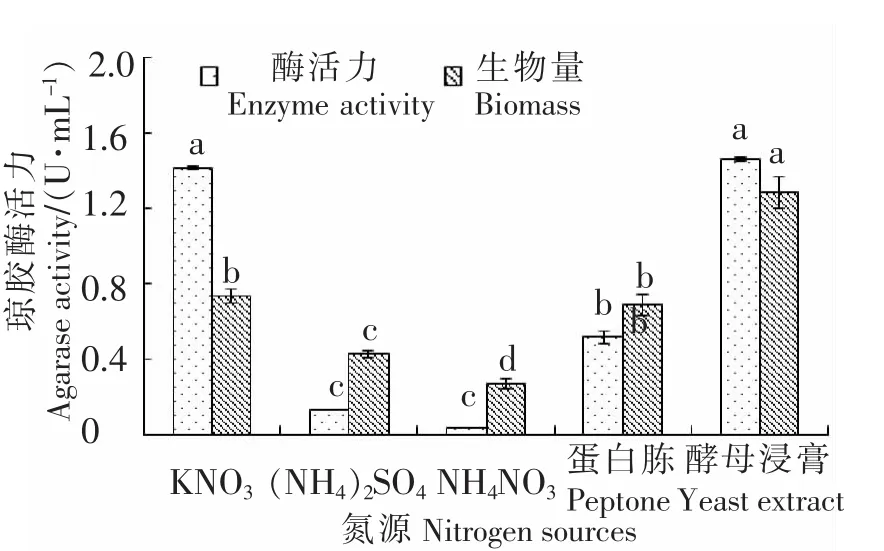
图4 氮源对菌株NTa产琼胶酶及菌体生长的影响Fig.4 Effects of nitrogen sources on agarase production and the growth of Stenotrophomonas sp.NTa
2.6 NaCl质量分数对菌株产酶的影响
菌株 Stenotrophomonas sp.NTa来源于海洋,受NaCl影响较大.NaCl质量分数对菌株产琼胶酶的影响结果 (见表1)表明,在低质量分数NaCl条件下,不利于菌株产酶.该菌株在NaCl质量分数为3% ~6%时,表现出较高的产琼胶酶能力.随着NaCl质量分数的增加,菌株的生物量和产酶能力均呈现先上升后下降的趋势,而总糖含量呈现出先下降后上升的趋势.当NaCl质量分数为5%时,生物量和酶活力达到最大,分别为1.45 U/mL和1.74 U/mL.因此选用质量分数为5%的NaCl作为最佳添加浓度.
2.7 温度对菌株产酶的影响
温度对Stenotrophomonas sp.NTa发酵产琼胶酶的影响结果 (见表2)显示,在考察温度范围内,菌株的生物量和产酶能力随着温度的升高均呈现先上升后下降的趋势,而总糖含量呈现出先下降后上升的趋势.在28℃进行培养时,生物量和酶活力都达到最大值,分别为1.69 U/mL和1.86 U/mL,同时总糖含量降低了87.66%.
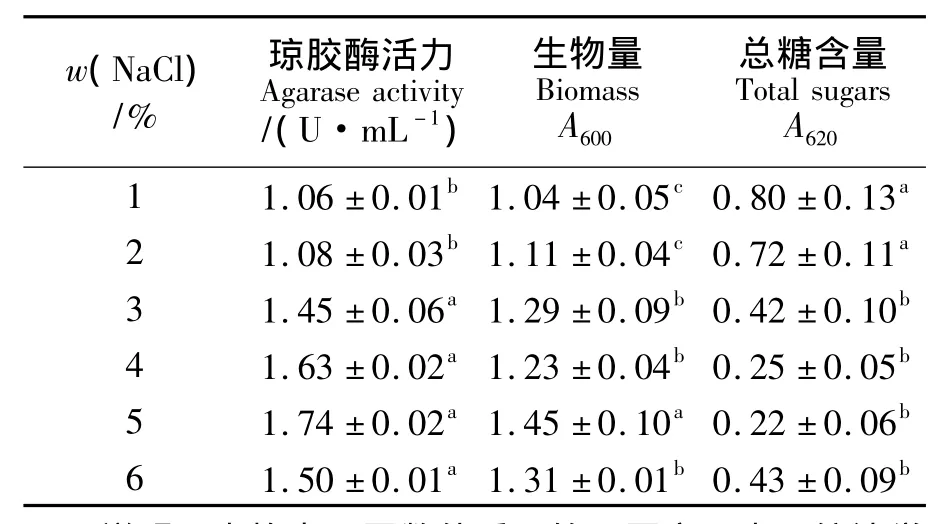
表1 NaCl质量分数对菌株NTa产琼胶酶及菌体生长的影响Tab.1 Effect of NaCl concentration on agarase production and the growth of Stenotrophomonas sp.NTa
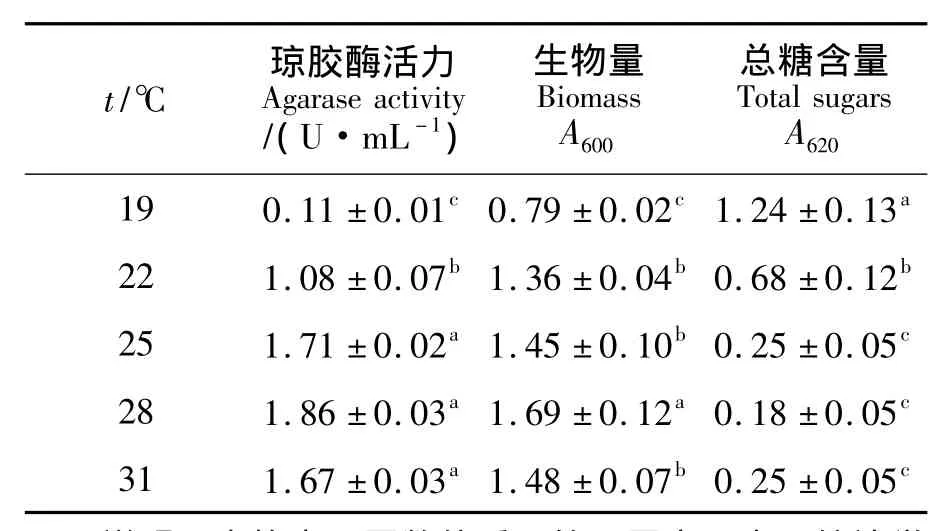
表2 温度对菌株NTa产琼胶酶及菌体生长的影响Tab.2 Effect of temperature on agarase production and microorganism growth fermented by Stenotrophomonas sp.NTa
2.8 Stenotrophomonas sp.NTa发酵产琼胶酶的动态规律分析
对液态发酵琼胶酶的动态规律进行分析,结果如图5所示.在0~8 h内,菌株生长处于延滞期,产酶能力微弱.在8~16 h内,菌株迅速生长,产酶能力依旧微弱.在16~32 h内,菌株的生物量继续增大,琼胶酶活力迅速升高.到40 h时,菌株的生物量达到最大,产酶能力最强,琼胶酶活力达到1.98 U/mL.培养基的总糖含量在0~32 h内持续下降,在32 h后趋于稳定.
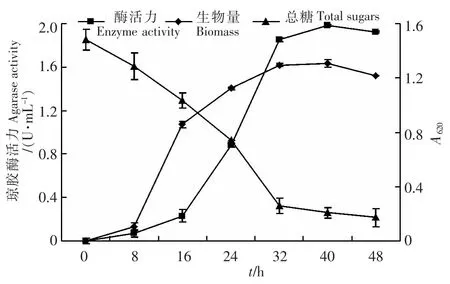
图5 Stenotrophomonas sp.NTa发酵产琼胶酶的动态规律分析Fig.5 Dynamic process of the agarase production by Stenotrophomonas sp.NTa during fermentation
3 讨论
本文利用选择性培养基从厦门红树林泥土样品中分离得到能降解琼脂的海洋细菌NTa,根据菌株的16S rRNA基因序列分析及典型生理生化特性分析,将NTa归属于寡养单胞菌属(Stenotrophomonas sp.).该属是1993年分离出来的一个新属,属于黄单胞菌目 (Xanthomonadales),黄单胞菌纲 (Xanthomonadaceae),目前尚未见该菌属相关琼胶酶活力及产酶条件的报道.因此,寡养单胞菌属(Stenotrophomonas sp.)是开发琼胶酶的另一类菌种来源.
在选择碳源的实验中发现,Stenotrophomonas sp.NTa发酵产琼胶酶的最佳碳源是琼脂,以其为唯一碳源时产酶活力最高.同时,其他碳源也可微弱诱导菌株产琼胶酶.此研究结果与Lakshmikanth等[3]的研究结果不同,Pseudomonas aeruginosa AG LSL-11只有以琼胶为碳源时才可诱导其分泌琼胶酶.发酵培养基中同时存在琼脂和葡萄糖时,菌株NTa生长旺盛,但产酶能力降低.Vander Meulen等[8]的研究发现,当将琼胶与葡萄糖混合作为碳源时,琼胶酶的产量显著下降为琼胶单独诱导产酶量的20%,表明,当培养基中额外添加葡萄糖时,琼胶酶受到分解代谢物阻遏.琼脂与其他多糖混合作为碳源可完全抑制Pseudomonas aeruginosa AG LSL-11分泌琼胶酶[3],但可以增强Acinetobacter sp.AG LSL-1分泌琼胶酶的能力[6].
氮源是影响微生物生长代谢的重要因素,本研究中采用酵母浸膏为氮源发酵菌株NTa时菌体的生物量及琼胶酶的产量明显高于其他氮源,酵母浸膏富含完全蛋白质、均衡的必需氨基酸及核苷酸,有利于Stenotrophomonas sp.NTa的吸收利用,促进其生长及分泌琼胶酶.蛋白胨含有大量的氨基酸及小肽,有利于微生物的吸收利用.Alexeeva研究表明,必须添加蛋白胨以保证微生物产糖酶[17].但对Stenotrophomonas sp.NTa的生长和产琼胶酶,蛋白胨不是合适的氮源,添加蛋白胨不利于微生物产琼胶酶.本研究中采用无机氮源(NH4)2SO4和NH4NO3时菌株仅可微弱生长,但是基本不产琼胶酶,可能是由于铵根离子存在时不利于菌株生长和产琼胶酶.微生物的种类不同,利于产酶的最适氮源也有所不同.Vander Meulen等的研究发现,Cytophaga flevvensis生长优先利用铵态氮,其分泌琼胶酶的最佳氮源为NH4NO3[8].王静雪等报道,柠檬酸三铵是海洋弧菌QJH-12产琼胶酶的最佳氮源[9].
海洋微生物是琼胶酶重要来源,因此NaCl对菌体产酶具有重要作用.发酵培养基中NaCl的质量分数为5%时,Stenotrophomonas sp.NTa生长茂盛,产酶能力最高.海洋弧菌QJH-12发酵产酶时的最适NaCl质量分数为1%[9],Tamlana sp.MA-B22发酵产酶的最适NaCl质量分数为2.5%[18].
培养温度对微生物产酶具有重要影响,本研究中28℃是Stenotrophomonas sp.NTa生长和产酶的最佳温度,而Cytophaga flevvensis产琼胶酶的最适培养温度为20℃[8],Pseudomonas aeruginosa AG LSL-11的为30 ℃[3],Tamlana sp.MA-B22的为25 ℃[18],Acinetobacter sp.AG LSL-1分泌琼胶酶的最适温度为37 ℃[6].
在优化后的最适条件下发酵40 h后,Stenotrophomonas sp.NTa发酵液的酶活力达到1.98 U/mL.Pseudomonas aeruginosa AG LSL-11的琼胶酶最大产量为0.32 U/mL[3],Acinetobacter sp.AG LSL -1的琼胶酶最大产量为 0.45 U/mL[6],Agarivorans albus YKW -34的琼胶酶最大产量为 0.87 U/mL[5],Vibrio sp.QJH-12的琼胶酶最大产量为1.84 U/mL[9].菌株Stenotrophomonas sp.NTa的产酶能力具有一定的优势,为进一步开发海洋琼胶酶资源提供了基础材料.
[1]CHI W J,CHANG Y K,HONG S K.Agar degradation by microorganisms and agar-degrading enzymes[J].Appl Microbiol Biotechnol,2012,94(4):917-930.
[2]FU X T,KIM S M.Agarase:review of major sources,categories,purification method,enzyme characteristics and applications[J].Mar Drugs,2010,8(1):200-218.
[3]LAKSHMIKANTH M,MANOHAR S,PATNAKAR J,et al.Optimization of culture conditions for the production of extracellular agarase from newly isolated Pseudomonas aeruginosa AG LSL-11 [J].World J Microb Biot,2006,22(5):531-537.
[4]RHEE Y J,HAN C R,KIM W C,et al.Isolation of a novel freshwater agarolytic Cellvibrio sp.KY-YJ-3 and characterization of its extracellular bata-agarase[J].J Microbiol Biotechn,2010,20(10):1378-1385.
[5]FU X T,LIN H,KIM S M.Optimization of medium composition and culture conditions for agarase production by Agarivorans albus YKW-34[J].Process Biochem,2009,44(10):1158-1163.
[6]LAKSHMIKANTH M,MANOHAR S,SOUCHE Y,et al.Extracellular β-agarase LSL-1 producing neoagarobiose from a newly isolated agar-liquefying soil bacterium Acinetobacter sp.AG LSL-1[J].World J Microb Biot,2006,22(10):1087-1094.
[7]FENG Z H,LI M.Purification and characterization of agarase from Rhodococcus sp.Q5,a novel agarolytic bacterium isolated from printing and dyeing wastewater[J].Aquaculture,2013,372:74-79.
[8]VANDER MEULEN H J,HARDER W.Production and characterization of the agarase of Cytophaga flevensis[J].Antonie Van Leeuwenhoek,1975,41(4):431-447.
[9]王静雪,江晓路,牟海津,等.海洋弧菌QJH-12发酵产琼胶酶条件的优化 [J].海洋科学,2007,31(7):8-14.
[10]KIM JONGHEE,HONG SOON-KWANG.Isolation and characterization of an agarase-producing bacterial strain,Alteromonas sp.GNUM-1,from the West Sea,Korea[J].J Microbiol Biotechn,2012,22(12):1621-1628.
[11]CHOI H J,HONG B J,PARK J J,et al.Production of agarase from a novel Micrococcus sp.GNUM-08124 strain isolated from the East Sea of Korea[J].Biotechnol Bioproc E,2011,16(1):81-88.
[12]韩文君,赵帅,刘会会,等.一株多糖降解菌的分离、鉴定与琼脂糖降解能力[J].微生物学报,2012,52(6):776-783.
[13]VIJAYARAGHAVAN R,RAJENDRAN S.Studies on agar degrading Salegentibacter sp.and characterization of its agarase[J].Int J Biosci,2011,1(5):56-64.
[14]SHI Y L,LU X Z,YU W G.A new β-agarase from marine bacterium Janthinobacterium sp.SY12[J].World J Microb Biot,2008,24(11):2659-2664.
[15]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
[16]FRY N K,WARWICK S,SAUNDERS N A,et al.The use of 16S ribosomal RNA analyses to investigate the phylogeny of the family Legionellaceae[J].J Gen Microbiol,1991,137:1215-1222.
[17]ALEXEEVA Y V,IVANOVA E P,BAKUNINA I Y,et al.Optimization of glycosidases production by Pseudoaleromonas issachenkonii KMM 3549T[J].Lett Appl Microbiol,2002,35:343-346.
[18]卢斌,柯才焕,杨明,等.一株高产琼胶酶菌株MA-B22的分子鉴定及产酶条件优化 [J].水产学报,2009,33(6):1037-1043.
