大鼠PER2基因RNAi慢病毒载体的构建及 干扰效率的测定
2014-06-27王翔凌孙宁玲马丽萍郭晓夏王鲁雁何湘君
王翔凌,孙宁玲,马丽萍,郭晓夏,姜 娟,王鲁雁,何湘君
(1. 北京大学人民医院心内科;2. 北京大学人民医院中心实验室,北京 100044; 3.卫生部北京医院心内科,北京 100730)
随着近年来人们生活方式的改变,我国高血压的发病率持续升高,其所导致的心脑血管疾病的危害日趋严重。既往研究发现,除血压水平外,血压昼夜节律异常也可导致心、脑、肾等靶器官损害,危害人类的生存与健康[1]。已有大量研究证实,血压的昼夜节律变化与时钟基因所产生的体内生物钟周期振荡密切相关[2]。
正常生理情况下生物钟通过时钟基因的“转录-翻译”反馈回路自主产生约24 h的节律性振荡,整合时钟输入信号与外界环境保持同步化,控制下游钟控基因的表达。目前,已发现的哺乳动物时钟基因包括PER、CLOCK、BMAL1、CRY基因以及时钟输出基因DBP、HLF、TEF等[3]。哺乳动物的生物钟由中枢生物钟和外周生物钟构成。中枢生物钟位于下丘脑视交叉上核(SCN),外周生物钟存在于除SCN以外的多数组织,如心脏、血管、肝脏等,属组织或细胞的内在特性之一[4]。研究发现,自发性高血压大鼠的肥厚心肌和主动脉组织中PER2蛋白表达高于正常大鼠,但昼夜时相相似[5]。体外培养血管平滑肌细胞也证实,PER2、DBP、BMAL1可在72 h内显示昼夜节律,血管紧张素Ⅱ(angiotensin Ⅱ, AngⅡ)短期刺激后,PER2基因的表达可显著增强[6]。以上均提示,钟基因PER2可能参与中枢生物钟节律调控及外周生物钟与中枢生物钟同步化,影响血压节律和导致靶器官损害。
RNA干扰(RNA interference, RNAi)指在进化过程中由双链RNA(double-stranded RNA,dsRNA)诱发的、高度保守的、同源mRNA高效特异降解的现象[7,9]。RNA干扰技术可特异性剔除或关闭特定基因的表达,现已被广泛用于探索基因功能、传染性疾病及恶性肿瘤的基因治疗等领域。慢病毒载体感染效率高, 是携带干扰RNA的理想载体[8]。本实验旨在寻找合适的RNA干扰位点,构建针对大鼠PER2基因的有效的RNAi慢病毒载体,为进一步研究PER2基因与异常血压节律及靶器官损害的相关性奠定基础。
1 材料与方法
1.1材料大鼠胸主动脉血管平滑肌细胞系A7r5细胞购自博慧斯生物(ATCC,CRL-1444);含100 U/mL青霉素、100 μg/mL链霉素的高糖DMEM培养基、100 mL/L胎牛血清、D-Hank’s溶液、Opti-MEM I无血清介质、无抗生素的DMEM培养基均购自美国Gibco公司;0.5 g/L胰酶EDTA溶液购自Hyclone公司;慢病毒-NC病毒液(1×108TU/mL)由上海吉玛制药技术有限公司提供;96孔板和6孔板购自美国Corning公司;dNTPs购自Sangon公司;MMLV逆转录酶(200 U/μL)购自Promega公司;RNA聚合酶抑制剂(40 U/μL)购自TYOBO公司;TaqDNA多聚酶(5 U/μL)购自TaKaRa公司;Polybrene、Real-time PCR Universal试剂盒(试剂盒号Cat号GMRS-001)购自上海吉玛制药技术有限公司。
1.2方法
1.2.1细胞培养 大鼠胸主动脉血管平滑肌A7r5细胞(以下简称A7r5细胞)常规使用含100 mL/L胎牛血清、100 IU/mL青霉素、100 μg/mL链霉素的高糖DMEM培养基在37 ℃、50 mL/L CO2饱和湿度培养箱中培养,定时换液,传代。
1.2.2大鼠PER2基因慢病毒shRNA干扰载体的构建 以大鼠PER2基因(GenBank登录号:AB 016532.1,GI:3845582)的cDNA序列为模板,遵循siRNA设计原则[9],选择RNAi靶序列,并进行BLAST基因组同源性分析,确定4个作用干扰位点为本实验的PER2基因靶序列,确保选择的siRNA序列与基因组中其他基因至少存在3个以上的碱基不同。由上海吉玛制药技术有限公司合成相应的4组shRNA表达质粒,并进行慢病毒载体的包装及测序鉴定。
1.2.3大鼠PER2基因慢病毒shRNA干扰载体转染A7r5细胞 当A7r5细胞在10 cm培养皿中培养至80%~90%融合时,进行胰酶消化后形成单细胞悬液。血球计数板计数,按5×105细胞/孔的密度接种6孔板,混匀后37 ℃、50 mL/L CO2培养24 h。将慢病毒原液200 μL(慢病毒滴度检测为1×108TU/mL),用含100 mL/L胎牛血清的DMEM培养液5倍稀释,根据细胞状态及类型,加入终质量浓度为5 μg/mL的Polybrene。吸去6孔板中的培养液,每孔加入上述1 mL稀释的病毒液,同时设立空白对照组,于37 ℃、50 mL/L CO2培养24 h。吸弃6孔板中的稀释病毒液,每孔加入1.5 mL含100 mL/L胎牛血清的DMEM培液,根据细胞状态,如有必要可分出1/3~1/5,于37 ℃、50 mL/L CO2继续培养48、72 h,通过荧光显微镜观察和计数转染荧光细胞评估转染效率。
1.2.4转染不同慢病毒shRNA干扰载体后A7r5细胞PER2基因mRNA表达的检测 ①总RNA提取:荧光趋于稳定时采用Trizol方法提取细胞总RNA,所得RNA溶解于DEPC处理的ddH2O中,采用琼脂糖凝胶电泳分析RNA的完整度,采用分光光度法测定RNA的浓度和纯度;②反转录为cDNA:在无菌EP管中配制如下体系,即2 μL的总RNA、1.2 μL的反转录酶、11.5 μL DEPC-H2O,70 ℃温育混合物5 min,冰上骤冷。再加入4 μL 5×Reaction Buffer、0.8 μL 10 mmol/L dNTPs mix、0.5 μL(100 U)MMLV RTase RNaseH,在42 ℃温育45 min,85 ℃加热10 min终止反应,冰上骤冷,反应液用作PCR模板;③Real-time PCR:采用实时荧光定量PCR检测PER2基因mRNA的表达水平。PER2上游引物5′-GAACTCCACAAACTTGGCATCACT-3′,下游引物5′-AGAACTCGGACATGTTTGCTGTG-3′,PCR扩
增产物大小128 bp,由MX3000实时定量PCR仪完成(美国Stratagen公司生产)。总反应体积为20 μL,按照10 μL的2×Real-time PCR Master Mix、2 μL的cDNA逆转录产物、0.1 μL的0.1 μmol/L上、下游引物、0.4 μL的TaqDNA聚合酶(2.5 U/μL)、7.4 μL的灭菌去离子水。PCR循环参数为:95 ℃ 3 min变性,1个循环,95 ℃ 30 s,62 ℃ 40 s,72 ℃ 30 s,共40个循环。PCR扩增产物以10 g/L琼脂糖电泳,EB染色,用Bio-Rad公司的GS-700凝胶成像仪对电泳带进行紫外吸光度扫描,以PER2/GAPDH的相对灰度表示该基因mRNA的相对表达量。

2 结 果
2.1大鼠PER2基因慢病毒shRNA载体的鉴定根据RNAi的设计原则,以PER2基因的第417、859、1057、1 363位点为干扰点,将siRNA序列与慢病毒载体质粒pGLV-U6-EGFP(图1)连接,构成4种PER2基因shRNA表达重组体pLentivirus-per2-rat-417、pLentivirus-per2-rat-859、pLentivirus-per2-rat-1057和pLentivirus-per2-rat-1363,依序标记为C229、C230、C231和C232,基因测序结果证实插入序列正确(表1)。

图1慢病毒载体质粒LV1-pGLV-U6-EGFP的结构图
Fig.1 cDNA structure of LV1-pGLV-U6-EGFP lentivector
表1针对大鼠PER2基因的4个siRNA靶点序列
Tab.1 Four target sequences of siRNA for rat PER2

标号靶位点基因序列(5'~3')基因测序NCTTCTCCGAACGTGTCACGTC229per2-rat-417GCACACAAAGAGCTGATAAGGC230per2-rat-859GCAGTGGCTTAGATTCTTTCAC231per2-rat-1057GCTATGAAGCTCCTAGAATTCC232per2-rat-1363GCAGGAAGATATCCTTCATCA
2.2大鼠PER2基因慢病毒shRNA载体转染效率的评价转染不同浓度荧光标记的pLenti-virus-per2-rat干扰载体72 h后,荧光倒置显微镜下观察转染效率。结果显示,转染pLentivirus-per2-rat干扰载体后血管平滑肌细胞形态与母系细胞(图2A)相比呈圆形者增多,细胞排列较前稍显紊乱(图2B、2C);各组细胞均可见荧光表达,高MOI(multiplicity of infection)组表达绿色荧光细胞较多,但死亡细胞也不断增加,故选择MOI值约50的比例转染,发绿荧光的细胞数约占70%,死亡的细胞较少(图2B、2C)。由于该细胞系较难转染,选择进行2次侵染的方式提高转染效率。
2.3大鼠PER2基因慢病毒shRNA载体对A7r5细胞表达PER2基因mRNA的抑制作用琼脂糖凝胶电泳结果显示,28S rRNA与18S rRNA的亮度比值约为2∶1,提示总RNA质量较好,无降解(图3)。图4可见NTC仅有引物二聚体,显示整个扩增体系正常,无模板污染;各条带单一,显示扩增产物特异性良好。对各组细胞的PER2基因与内参基因GAPDH的Ct差值(ΔCt)、ΔCt与阴性对照的Ct差值(ΔΔCt)数据进行正态性检验,结果符合正态分布,选择单因素方差分析对各组细胞PER2基因被抑制的效果进行评价,结果显示,各组间存在统计学差异(P<0.05)。进一步行两两比较显示,C231组和C232组显著抑制大鼠PER2基因表达,两组分别与阴性对照组、C229组和C230组比较具有统计学差异(P<0.05),抑制效率可达84%和91%,C231与C232两组间无统计学差异(表2)。

图2自然光下正常A7r5细胞(A)及侵染72h后的转染效率(B、C)
Fig.2 Normal A7r5 VSMCs in natural light (A) and the efficiency of pLentivirus-per2-rat transfection after 72 h (B, C)
B:亮视野;C:暗视野。
图3总RNA的电泳图
Fig.3 Electrophoretogram of total RNA extraction

图4PCR扩增产物的电泳图
Fig.4 Electrophoretogram of PCR amplification products
表2大鼠PER2基因慢病毒shRNA载体对A7r5细胞PER2基因mRNA表达的抑制作用

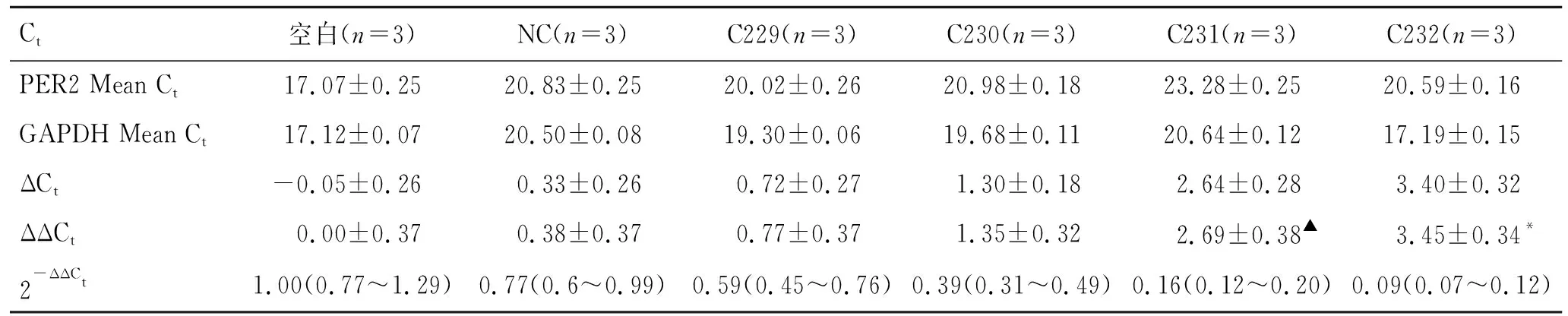
Ct空白(n=3)NC(n=3)C229(n=3)C230(n=3)C231(n=3)C232(n=3)PER2 Mean Ct17.07±0.2520.83±0.2520.02±0.2620.98±0.1823.28±0.2520.59±0.16GAPDH Mean Ct17.12±0.0720.50±0.0819.30±0.0619.68±0.1120.64±0.1217.19±0.15ΔCt-0.05±0.260.33±0.260.72±0.271.30±0.182.64±0.283.40±0.32ΔΔCt0.00±0.370.38±0.370.77±0.371.35±0.322.69±0.38▲3.45±0.34*2-ΔΔCt1.00(0.77~1.29)0.77(0.6~0.99)0.59(0.45~0.76)0.39(0.31~0.49)0.16(0.12~0.20)0.09(0.07~0.12)
Mean Ct是3个重复孔Ct 值的平均值,该项“±”后的数值为3次重复的标准差。ΔCt指同一样品中,待检基因与内参基因平均Ct值的差值。ΔΔCt指其余样品的ΔCt值和对照样品相应基因的ΔCt值之差。2-ΔΔCt是按照ΔΔCt所计算,后面置信区间也是根据ΔΔCt的范围所计算。单因素方差分析显示,C231与阴性对照(NC)及C229、C230比较,▲P<0.05;C232与阴性对照(NC)及C229、C230比较,*P<0.05。
3 讨 论
PER2基因是时钟基因的重要组分之一,其与PER1、PER3、CRY1、CRY2等共同组成调节蛋白的负向分支,和转录因子的正向分支(BMAL1、CLOCK和NPAS2)通过转录、翻译和后翻译等过程,形成一系列相互作用自发调节的负反馈环路,产生24 h周期振荡,形成了昼夜节律(circadian rhythm)[3]。哺乳动物包括人类和小鼠的心血管系统同样存在时钟基因的节律性表达[10-12]。研究证实,动物体内的PER2基因常在夜间高表达,而BMAL1常在白昼表达增加,PER基因作用于BMAL1的E-box,刺激BMAL1基因的表达。已证实BMAL1靶基因敲除小鼠也出现血压昼夜节律的消失[13]。持续黑暗状态下,CRY1/CRY2缺陷小鼠(双基因敲除/DKO)与野生型小鼠(WT)比较白天为高血压(啮齿类此时间血压下降)[14]。故推测若抑制PER2,有可能CLOCK/ BMAL1异源二聚体形成将减少,可能导致时钟基因 PER、BMAL1和输出基因包括Dbp等蛋白翻译下降,影响神经体液的昼夜分泌,影响血压昼夜节律,参与高血压的发生发展。
动脉粥样硬化是高血压患者常见的靶器官损害,血管平滑肌细胞(vascular smooth muscle cells, VSMCs)增殖是动脉粥样硬化的基础。研究表明,Ang Ⅱ可诱导培养的乳大鼠心肌细胞核内时钟基因PER2、BMAL1、DBP表达增强[15],提示心脏局部的RAS在促进心肌肥厚的过程中与时钟体系的基因表达变化之间有某种联系,而作为参与高血压发病机制的重要成分,VSMCs在调节血压节律及细胞增殖方面的作用尚未明确。本实验选取血管平滑肌细胞作为RNA干扰抑制的靶细胞,将为我们探索时钟基因组分在血压节律调节、细胞增殖方面的作用提供一种途径。
RNA干扰技术已广泛用于基因研究及应用,该技术简单快速、高效经济、效果特异,可进行基因功能分析[16]、信号转导通路研究[17]、新药开发[18]及基因治疗等多个领域[19-21]。合成siRNA的方法有两大类:体外合成和体内转录。体外合成包括化学合成、体外转录及酶消化法,其中化学合成法为应用已找到最有效的siRNA序列进行单核苷酸直接化学合成,简便、有效、产物纯度高,但合成周期长、作用时间短、转染效率差别大而且价格高。体内转录的原理是利用携带siRNA的质粒、病毒表达载体或带有siRNA表达盒的PCR产物,转染入目的细胞并整合至宿主细胞基因组而表达产生靶基因的siRNA起到RNA干扰作用。表达载体常用能在哺乳动物细胞中表达shRNA的含有RNA聚合酶Ⅲ(pol Ⅲ) 启动子的载体[22]。该方法可提高对原代细胞、干细胞、不分化细胞等较难转染细胞的转染率,插入较大的片段(-7.5 kb)并将免疫反应降到最低,使得目的基因整合至宿主细胞基因组的几率明显增加,可获得目的基因siRNA的高水平的表达,提高沉默基因效力。本实验在预实验过程中曾选用siRNA化学合成法及shRNA体外转录方法,效果均欠佳,分析原因主要为细胞转染效率低,即使提高siRNA或shRNA的浓度及选择不同细胞浓度和脂质体2000的比例,转染效率也维持在30%~50%左右,远不能达到实验所需要求。这与siRNA或shRNA本身的转染率相对较低及A7r5细胞系本身难于被转染有关。结果证实,调整siRNA的制备方式,以慢病毒为载体将shRNA导入慢病毒pGLV-U6-EGFP质粒后转染细胞改善转染效果,使血管平滑肌细胞内表达靶基因Per2的siRNA从而达到沉默PER2基因成为可能。
由于本实验初期时siRNA及shRNA转染效率低及浓度过大可导致细胞死亡,故在选择病毒载体时充分考虑其感染效率、对细胞活力的影响及安全性等因素。而慢病毒载体系统为VSV-G膜蛋白包裹,可增加病毒感染谱;其LTR的转录激活能力低于逆转录病毒,激活原癌基因能力也相对较低;重组慢病毒颗粒以同时采用“自我灭活”修饰,阻止子代病毒自我复制和转移,从而保证产生的慢病毒具备最低的细胞毒性和良好的生物安全性。本实验结果也证实,通过更换导入方式、2次侵染及调整合适的感染复数(MOI值)等方法,慢病毒载体的转染效率较siRNA和shRNA明显增加,未出现明显靶细胞A7r5死亡。
本实验共构建4组siRNA干扰序列,通过Real-time PCR检测转染后A7r5细胞PER2基因的mRNA表达,初步证实C231组(pLentivirus-per2-rat-1057)和C232组(pLentivirus-per2-rat-1363)干扰序列可有效沉默大鼠PER2基因的表达,可进一步通过了解PER2基因在蛋白水平的表达变化证实其沉默效果。其他序列不能有效沉默基因表达,可能与选择的干扰序列位点是否符合siRNA的设计原则、所选序列作用位点的二级结构和碱基配对程度及细胞转染后的活力有关。
本研究针对大鼠PER2基因设计选择不同的siRNA序列,连接到LV1-pGLV-U6-EGFP质粒中构建大鼠PER2基因shRNA慢病毒载体,通过检测慢病毒载体转染的大鼠血管平滑肌A7r5细胞的PER2基因mRNA表达情况,筛选出有效干扰PER2基因表达的siRNA序列,为进一步研究时钟基因各组分的表达、调控等基因功能,及其在血压昼夜节律调节、异常血压节律形成和靶器官损害等疾病发生机制中的作用奠定了基础。
参考文献:
[1] 中国高血压防治指南修订委员会.中国高血压防治指南2010 [J]. 中华心血管病杂志, 2011, 39(7):579-616.
[2] KOHSAKA A, WAKI H, CUI H, et al. Integration of metabolic and cardiovascular diurnal rhythms by circadian clock[J]. Endocr J, 2012, 59(6):447-456.
[3] RICHARDS J, GUMZ ML. Advances in understanding the peripheral circadian clocks[J]. FASEB J, 2012, 26(9):3602-3613.
[4] OKAMURA H, YAMAGUCHI S, YAGITA K. Molecular machinery of the circadian clock in mammals[J]. Cell Tissue Res, 2002, 309:47-48.
[5] 丁延魁,黄建寨,张莉,等. 高血压大鼠肥厚心肌和主动脉中PER2蛋白表达的变化[J]. 中国临床药理学与治疗, 2006, 11(6): 625-628.
[6] NONAKA H, EMOTO N, IKEDA K, et al. Angiotensin II induces circadian gene expression of clock genes in cultured vascular smooth muscle cells[J]. Circulation, 2001,104:1746-1748.
[7] XIA H, MAO Q, PAULSON HL, et al. siRNA mediated gene silencinginvitroandinvivo[J]. Nat Biotechnol, 2002, 20:1006-1010.
[8] LI M, HUSIC N, LIN Y, et al. Optimal promoter usage for lentiviral vector-mediated transduction of cultured central nervous system cells[J]. J Neurosci Methods, 2010, 189:56-64.
[9] WILSON RC, DOUDNA JA. Molecular mechanisms of RNA interference[J]. Annu Rev Biophys, 2013, 42:217-239.
[10] TAKEDA N, MAEMURA K. Circadian clock and vascular disease[J]. Hypertens Res, 2010, 33(7):645-651.
[11] RUDIC RD, FULTON DJ. Pressed for time: the circadian clock and hypertension[J]. J Appl Physiol, 2009, 107(4):1328-1338.
[12] ANEA CB, ZHANG M, STEPP DW, et al. Vascular disease in mice with a dysfunctional circadian clock[J]. Circulation, 2009, 119:1510-1517.
[13] LI P, SUR SH, MISTLBERGER RE, et al. Circadian blood pressure and heart rate rhythms in mice[J]. Am J Physiol Regul Integr Comp Physiol, 1999, 276:R500-R504.
[14] MASUKI S, TODO T, NAKANO Y, et al. Reduced alpha-adrenoceptor responsiveness and enhanced baroreflex sensitivity in Cry-deficient mice lacking a biological clock[J]. J Physiol, 2005, 566:213-224.
[15] 黄新艳,魏立,李庆平,等. 血管紧张素Ⅱ对乳大鼠心肌细胞时钟基因表达的影响[J]. 中国药理学与毒理学, 2005, 19(3):194-198.
[16] ASHRAFI K, CHANG FY, WATT’S JL, et al. A genome-wide RNAi screen for modifiers of the circadian clock in human cells [J]. Cell, 2009, 139(1):199-210.
[17] MULLENDERS J, FABIUS AWM, MADIREDJO M, et al. A large scale shRNA barcode screen identifies the circadian clock component ARNTL as putative regulator of the p53 tumor suppressor pathway[J]. PLoS One, 2009, 4(3):e4798:1-10.
[18] NOLTEA, WALKER T, SCHNEIDER M, et al. Small interfering RNA eluting surfaces as a novel concept for intravascular local gene silencing[J]. Mol Med, 2011, 17(11-12):1213-1222.
[19] KAUR IP, SHARMA G.siRNA: a new approach to target neuropathic pain[J]. Bio Drugs, 2012, 26(6):401-412.
[20] LIU Y, SHEN Y, QIN C, et al.Growth inhibition and apoptosis of human B-cell lymphomainvitroandinvivoby Bcl-2 short hairpin RNA[J]. Oncol Rep, 2013, 29(1):244-252.
[21] LARES MR, ROSSI JJ, OUELLET DL. RNAi and small interfering RNAs in human disease therapeutic applications[J]. Trends Biotechnol, 2010, 28(11):570-579.
[22] MIYAGISHI M, TAIRA K. U6 promoter-driven siRNAs with four uridine 3′overhangs efficiently suppress targeted gene expression in mammalian cells[J]. Nat Biotechnol, 2002, 20(5):497-500.
