属级芯片筛查技术在马铃薯纺锤块茎类病毒属检测中的应用
2014-06-19张永江辛言言朱水芳邓丛良
张永江,辛言言,朱水芳,邓丛良
1 中国检验检疫科学研究院,北京 100029 2 北京出入境检验检疫局检验检疫技术中心,北京 101312
马铃薯纺锤块茎类病毒属 (Pospiviroid) 属于马铃薯纺锤块茎类病毒科 (Pospiviroidae),根据国际病毒分类委员会 (International Committee on Taxonomy of Viruses, ICTV) 第九次分类报告,该属包括菊花矮化类病毒(Chrysanthemum stunt viroid, CSVd)、柑橘裂皮类病毒 (Citrus exocortis viroid, CEVd)、金鱼花潜隐类病毒 (Columnea latent viroid, CLVd)、血苋类病毒1 (Iresine viroid 1, IrVd-1)、墨西哥心叶茄类病毒 (Mexican papita viroid, MPVd)、马铃薯纺锤块茎类病毒 (Potato spindle tuber viroid, PSTVd)、辣椒小果类病毒 (Pepper chat fruit viroid, PCFVd)、番茄顶缩类病毒 (Tomato apical stunt viroid, TASVd)、番茄褪绿矮缩类病毒 (Tomato chlorotic dwarf viroid, TCDVd) 及番茄雄性株类病毒 (Tomato planta macho viroid,TPMVd) 等10种类病毒[1]。该属类病毒可侵染马铃薯、番茄、菊花及柑橘等多种重要的经济作物,并可通过种子及苗木迅速扩散传播,进而导致严重的经济损失[2-6]。近年来,该属新的类病毒种不断被鉴定,从2007年的8种 (CSVd、CEVd、CLVd、IrVd-1、MPVd、PSTVd、TASVd和 TCDVd) 增加到 2011年的 10种 (新增PCFVd和 TPMVd);新的寄主也不断被报道,如TASVd在2010–2012年间报道的新增寄主就有蓝花茄Lycianthes rantonnetii、扭管花Streptosolen jamesonii、木曼陀罗属Brugmansiasp.、素馨叶白英Solanum jasminoides及夜香树属Cestrumsp.等[7-9];新发生的国家和地区也不断增加,如TASVd的分布已从2003年首次报道的突尼斯迅速扩散到了目前的奥地利、芬兰、印度尼西亚、以色列、意大利、科特迪瓦、塞内加尔、荷兰、比利时、德国、法国及斯洛文尼亚[10-12]等国家。上述 3种“新”情况表明,该属类病毒发现及传播扩散速度较以往大大增加,因而马铃薯等相关产业所面临的危险也将大大增加。为了尽早检测到植物中的类病毒,防止其传播扩散,需要有效的技术来作为支撑。
现有的检测方法如指示植物法、聚丙烯酰胺凝胶电泳法、核酸斑点杂交法及RT-PCR法等都是针对该属特定类病毒种的特异性检测技术,在上述3种“新”情况下,这些特异性检测技术将会延缓发现新类病毒的速度,也会增大检测的强度;同时,很多寄主受侵染后会无症状表现,增加了特异性检测的难度,漏检的可能性非常大。为了解决这个问题,可以采用广谱性筛查技术。广谱性筛查技术 (Broad spectrum screening techniques, BSST) 是目前流行的一类技术,如用于免疫学检测的属级抗体技术、用于分子生物学检测的科或属级引物及属级兼容性探针芯片技术等。这类技术往往具备某类病原物较高分类单元上的鉴定特征,因而具有较高的兼容性,可以对科或属中变异大且变异位点多的病原物进行鉴定;特别是对于科或属内可能出现的新种,具有较强的发现能力;同时,这类技术具有高通量的特点,一次样品反应可检测到科或属内已知和未知的病原物;这些特点使得该类技术可以大大提高新发病原物预报式筛查的效率,从而提前发现可能的危险性病原物,如 Zhang等[13]建立的植物病毒属级寡核苷酸芯片可以有效筛查到样品中的新病毒,表明了这类筛查技术的实用性。
为了弥补目前特异性检测技术的不足,提高 3种“新”情况下筛查马铃薯纺锤块茎类病毒属类病毒的能力,本文开展了该属筛查芯片技术的研究,着重解决具备属级鉴定特征芯片探针的获得及其有效性验证的问题,初步建立了该类病毒属属级水平上的筛查芯片体系。
1 材料与方法
1.1 材料与试剂
CSVd及TPMVd阳性材料来自美国ATCC(ATCC-PV-105,ATCC-45052)。
9N随机引物为Invitrogen公司产品;纳米磁珠检测试剂盒来源由北京出入境检验检疫局植物实验室提供;ExTaqDNA聚合酶、M-MLV反转录酶、dNTPs、RNasin为大连TaKaRa公司产品;CbcScript酶为AMBION公司产品;RNA及DNA纯化试剂盒购自MN公司。
1.2 RT-PCR反应
根据NCBI中发表的序列,分别设计CSVd及TPMVd的种简并性全长扩增引物如表1。
总RNA提取:称取100 mg样品置于研钵中,加入适量的研磨液,用研磨棒充分研磨,使样品均匀分散。将研磨后的样品转入离心管,5 000 r/min离心5 min。取100 μL离心上清于1.5 mL离心管中,再加入适量纳米磁珠和1倍体积的结合液,上下颠倒充分混匀,室温放置5 min。磁分离后弃上清,用200 μL清洗液清洗2−5次。加入50 μL洗脱液,缓慢抽吸均匀,室温放置2−5 min后,磁分离,小心吸取上清液至新的离心管中进行下游实验。
反转录:体系 20 µL,DEPC-H2O 10 µL、dNTPs 1 µL、下游引物 2 µL (20 µmol/L)、RNA模板 1 µL (200 ng/μL);70 ℃反应 5 min,冰上放置5 min;加入M-MLV酶5×缓冲液 4 µL、M-MLV酶 1 µL、RNA 酶抑制剂 1 µL,42 ℃反应 1 h。
PCR扩增:即在 0.2 mL的反应管中加入cDNA 产物 2 μL、上游引物 0.5 μL (20 µmol/L)、下游引物 0.5 μL (20 µmol/L)、dNTPs 0.5 μL、Taq酶0.5 μL、PCR缓冲液2 μL及DEPC-H2O 14 μL,然后按照PCR反应程序进行扩增,产物进行琼脂糖凝胶电泳检测。

表1 CSVd和TPMVd RT-PCR引物Table 1 Primers used for RT-PCR of CSVd and TPMVd
1.3 芯片体系
探针设计步骤如下:
1) 从美国国立生物技术信息中心 (NCBI)及国际病毒学分类委员会(ICTV)数据库下载类病毒属全基因组及核酸序列数据。
2) 分别使用类病毒全基因组序列和全部核酸序列设计探针。使用全部核酸序列时需进行筛选,去除小于250 bp长度的核酸序列。并使用NCBI的BLASTN对核酸序列去冗余,即如果某核酸序列 90%以上长度与其他序列有 95%相似度,则删除此序列。
3) 从 NCBI RefSeq数据库下载全部植物mRNA序列数据、全部植物病毒基因组序列数据,作为探针设计的特异性数据库。
4) 使用BLASTN联配类病毒属内序列,以5个碱基作为间隔,连续提取所有40 bp的核酸序列,对选取的核酸序列进行筛选,标准为GC含量在 40%与 60%之间、单个核苷酸含量不大于 50%、没有多于 4个核苷酸的连续重复及不形成多于6个核苷酸的发卡结构。
5) 使用BLASTN将通过步骤4) 筛选的序列与步骤 3) 得到的特异性数据库以及非此类病毒属的其他所有植物类病毒核酸序列进行特异性比对,筛选标准为联配序列长度乘以相似性小于25 bp,并且无20 bp以上的完全吻合序列匹配。
6) 使用BLASTN将通过步骤5) 筛选的探针序列与本类病毒属的所有类病毒序列进行比对,比对标准为联配序列长度乘以相似性大于34 bp,且有连续15 bp的序列匹配。满足此标准的序列,即为可以检测到相应类病毒的探针。
7) 使用维也纳大学理论化学研究所(Institute for Theoretical Chemistry, University of Vienna)开发的RNAcofold程序计算探针与相应类病毒序列结合的自由能及自身形成RNA二级结构的自由能。
8) 将步骤5) 得到的探针序列进行排序,排序标准依次为探针对本类病毒属所有类病毒的覆盖率、探针与本类病毒属序列结合总自由能、探针与本类病毒属病毒序列结合总相似度、探针与特异性数据库非特异结合相似度及探针形成自身RNA二级结构自由能。
9) 将步骤5) 得到的探针去冗余,去掉重复探针,使得探针之间最大重复长度不超过25 bp。重复的探针依步骤8) 的标准排序,只保留最佳的探针。此步骤得到的探针即为非重复的特异性探针。
10) 按步骤 8) 的标准对步骤 9) 得到的探针进行排序,依排序结果依次选择探针,使本类病毒属每个类病毒序列至少有3个探针对应。选择时,如果某些类病毒序列已经达到 3个探针对应,则对其他未被选择的探针的排序,优先考虑对其他未达到 3个探针对应的类病毒序列的覆盖率,之后再按照步骤8) 的标准进行排序。如果经过步骤9) 无法达到本类病毒属每个类病毒序列至少 3个探针对应的标准,则在步骤 3) 降低序列保守性标准,重新设计,依此循环直到所有类病毒序列都有3个探针对应。如果无法从保守区域设计探针,或类病毒属内序列无保守区域,则使用所有类病毒序列全长序列设计探针。探针设计标准与步骤4) 到步骤9) 相同。
样品标记、芯片杂交与洗涤方法均按照文献[14]的方法进行。芯片数据分析时,探针的信号值为探针前景值的中位值减去背景值的中位值。信噪比为图像对应点内所有信号值的中位值与背景值中位值的比值。样品中若至少一条探针的信号值≥600且信噪比≥3,判为阳性 (为Pospiviroid类病毒侵染);探针的信号值<600且信噪比<2,判为阴性 (不为Pospiviroid类病毒侵染);其余情况判为可疑,需重复验证。
1.4 芯片探针有效性验证
分别取CSVd及TPMVd的PCR扩增产物按照1.3的方法进行标记、杂交及检测,分析芯片探针的有效性。
1.5 芯片与RT-PCR灵敏度比较实验
将 CSVd总 RNA (200 ng/μL) 进行 10倍梯度稀释后用作模板,分别按照1.2和1.3的方法进行芯片与RT-PCR检测,比较两者的灵敏度。

表2 寡核苷酸探针Table 2 Nucleotide probes
2 结果与分析
2.1 探针设计与芯片制备
采用生物信息学方法分析 Pospiviroid类病毒的核苷酸序列,根据设定的探针设计标准,获得了19条40单体单元 (Monomerie unit, mer)的属级鉴定特征的寡核苷酸探针 (表 2)。将寡核苷酸探针通过芯片点样仪点制到醛基基片上制备芯片。每条探针重复5次,矩阵为10×12。芯片包括4个部分:芯片固定阳性质控Hex,杂交阳性质控 PC,杂交阴性质控 NC,其他位点为检测探针 (图1)。

图1 芯片矩阵图 (10×12)Fig. 1 Microarray construction (10×12).
2.2 探针有效性验证
应用CSVd及TPMVd的样品对芯片探针进行验证,杂交结果如图2及图3所示。图2中1#、3#、9#探针及图 3中 1#、3#、4#、18#探针的信号值均大于600,信噪比均大于3,达到阳性结果判定的标准,说明本研究建立的芯片杂交体系能够在样品检测中获得有效的探针信号点,可以对样品中是否含有该属类病毒进行判断。图2中的8#探针虽然具有肉眼可见的信号,且其信噪比大于3,但由于其信号值小于600,因而判为可疑点,在实际样品检测时需进行重复检测 (表3)。
2.3 芯片灵敏度检测结果
CSVd总 RNA按 101、102及 103梯度稀释后进行cDNA合成、Klenow酶标记和杂交,反应结束后对芯片进行洗涤扫描。结果如图 4所示,其中对照Hex、PC和NC杂交符合预期效果;而样品杂交结果则随着稀释梯度的增大,探针信号强度越来越小;103稀释梯度仅有9号探针信号达到阳性探针判断的标准,因此所建立的芯片的灵敏度阈值大约是103。具体阳性探针信号值及信噪比见表4。
2.4 RT-PCR灵敏度检测结果
CSVd总 RNA 按 100、101、102及 103梯度稀释后进行RT-PCR检测,电泳结果表明可从阳性材料中扩增出约354 bp的特异性目标条带。随着总RNA稀释倍数的增大,可以观察到明显的浓度梯度谱带,稀释梯度限点为103(图5)。

图2 菊花矮化类病毒样品杂交Fig. 2 Hybridization using CSVd.
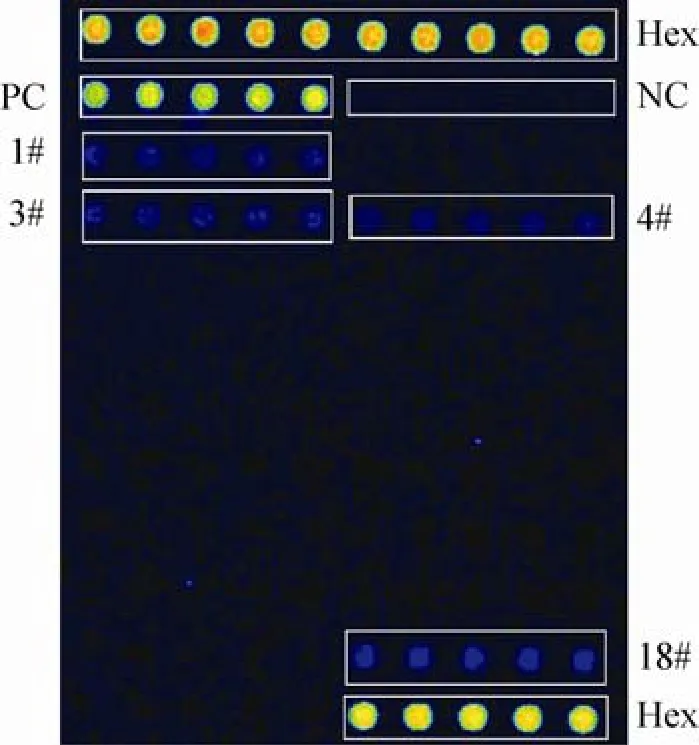
图3 番茄雄性株类病毒样品杂交Fig. 3 Hybridization using TPMVd.
表3 芯片探针有效性验证结果Table 3 Results of microarray probes efficency validation

图4 芯片灵敏度结果Fig. 4 Sensitivity test of microarray.
表4 芯片灵敏度结果Table 4 Results of microarray sensitivity
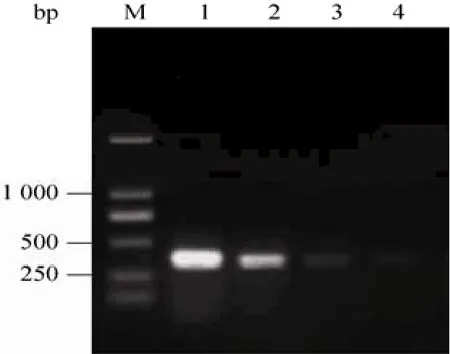
图5 RT-PCR灵敏度检测结果Fig. 5 Sensitivity test of RT-PCR. 1: 100 dilution; 2:101 dilution; 3: 102 dilution; 4: 103 dilution; M: marker DL2000.
3 讨论
足够量的标记产物是芯片杂交时获得足够强度杂交信号的重要环节。选择何种标记策略,需要根据样品中病原物含量的多寡及病原物基因组二级结构的复杂程度等因素来确定,目前主要有 3种标记方式:随机引物反转录获得标记的 cDNA作为标记产物;随机引物反转录后特异性引物PCR获得标记的PCR产物作为标记产物;特异性引物RT-PCR获得标记的PCR产物作为标记产物[14]。其中以第一种标记方式为最好,可以使芯片具有更强的实用性。为此,本文最初采用第一种标记策略来获取标记产物,但得到的产物与芯片杂交后没有获得杂交信号 (结果未显示),这种状况与的研究结果相同[15],可能是类病毒基因组复杂的二级结构所致。由于本研究的重点在于验证所获得的属级探针的有效性,故在后续的实验中采用了特异性引物扩增来获得标记产物的策略;为了增加芯片的实用性,需要进一步研究更简便有效的标记方式。
Supported by:National Department Public Benefit Research Foundation (No. 201110035).
Corresponding author:Shuifang Zhu. Tel/Fax: +86-10-64896608; E-mail: zhushf020420@gmail.com
受质检公益性行业科研专项 (No. 201110035) 资助。
芯片灵敏度的高低决定了芯片在实际应用中的效果。一般情况下,对于同一种样品,RT-PCR的检测灵敏度要高于芯片检测灵敏度;也有两者持平的情况;甚至后者高于前者的情况[16-17],这种情况的影响因素较多,比如琼脂糖凝胶电泳检测中凝胶的厚度、电泳缓冲液的新鲜程度或电泳电压的高低等都可能会影响到PCR的检测灵敏度,使其检测的灵敏度要比理论上的灵敏度低。在本文中,因为使用的是类病毒种的简并引物,获得的PCR产物的特异性要比随机引物或科属简并引物获得的PCR产物特异性强,产物量也大,与芯片杂交时获得的杂交信号的质量就高,信号就强,两者的检测灵敏度持平,均可检测到 10–3稀释梯度的样品总RNA。对于特异性检测芯片来说,其特异性与灵敏度一样,也是建立有效芯片检测技术的重要一面。不过,因为本文建立的芯片是广谱性的筛查芯片,而非特异性的检测芯片,因此,信号点的探针对应的类病毒种类不一定绝对是探针序列来源的类病毒种类;是何种类病毒最终要由序列测定来确定。所以,本文没有应用其他属的类病毒进行芯片的特异性研究,这并不影响该技术的建立和使用。
目前对于 Pospiviroid类病毒的检测方法有生物学测定[18]、聚丙烯酰胺凝胶电泳[19]、Northern 杂交[20-21]、RT-PCR[22-24]及 RT-PCRELISA[25]等,这些方法适用于样品中已知类病毒的特异性检测,对于潜在的未知类病毒则无法进行有效检测;且一次反应最多检测几种类病毒。下一代测序技术 (Next generation sequencing technologies, NGST) 的发展为潜在未知类病毒的有效检测提供了一个选择。通过小RNA库的构建及深度测序,NGST可以鉴定样品中已知和未知的类病毒种类。Li等应用小RNA深度测序技术分析了受感染的番茄样品,发现其中感染了马铃薯纺锤块茎类病毒、柏平缕瓜花叶病毒和一种新的马铃薯Y病毒属病毒[26]。不过,NGST费用昂贵、数据量庞大且分析复杂。为了弥补上述技术的缺陷,本研究根据Pospiviroid类病毒的序列设计了19条40 mer的具有该属鉴定特征的探针,制备相应芯片后以CSVd及TPMVd样品进行验证,结果表明探针有效性良好,具有较高的灵敏度,初步建立了Pospiviroid芯片筛查技术,为该属类病毒的筛查探索了一种有效和有前景的技术体系;对今后在植物类病毒的检测鉴定中应用广谱筛查技术作为快速和高通量筛查方法具有借鉴意义。
[1]King AMQ, Lefkowitz E, Adams MJ, et al. Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses. Amsterdam:Elsevier/Academic Press, 2011.
[2]Pfannenstiel MA, Slack SA. Response of potato cultivars to infection by the potato spindle tuber viroid. Phytopathology, 1980, 70(9): 922–926.
[3]Cho WK, Jo Y, Jo KM, et al. A current overview of two viroids that infect chrysanthemums:Chrysanthemum stunt viroid and Chrysanthemum chlorotic mottle viroid. Viruses-Basel, 2013, 5(4):1099–1113.
[4]Batuman O, Gilbertson RL. First report of Columnea latent viroid (CLVd) in tomato in mali.Plant Dis, 2013, 97(5): 692.
[5]Verhoeven JT, Roenhorst JW, Owens RA. Mexican papita viroid and tomato planta macho viroid belong to a single species in the genus Pospiviroid.Arch Virol, 2011, 156(8): 1433–1437.
[6]Chung BN, Pak HS. Seed Transmission of Chrysanthemum stunt viroid inChrysanthemum.Plant Pathol J, 2008, 24(1): 1–35.
[7]Verhoeven JThJ, Botermans M, Jansen CCC, et al.First report of tomato apical stunt viroid in the symptomless hostsLycianthes rantonnetiiandStreptosolen jamesoniiin the Netherlands. Plant Dis, 2010, 94(6): 791.
[8]Olivier E, Demonty J, Govers K, et al. First report of aBrugmansiasp. infected by tomato apical stunt viroid in Belgium. Plant Dis, 2011, 95(4): 495.
[9]北美植物保护组织植物检疫预警系统番茄顶缩类病毒[EB/OL]. [2013-08-06]. http://www.pestalert.org/viewNewsAlert.cfm?naid=60&keyword=Tomato%20apical%20stunt%20viroid (in Chinese).
[10]欧洲植物保护组织植物检疫番茄顶缩类病毒[EB/OL]. [2013-08-06]. http://www.eppo.int/QUARANTINE/Alert_List/viruses/TASVD0.htm(in Chinese).
[11]Grausgruber-Gröger S, Gottsberger RA. First report of tomato apical stunt viroid and Chrysanthemum stunt viroid inSolanum jasminoidesin Austria.New Disease Reports, 2011, 24: 4.
[12]Viršček Marn M, Mavrič Pleško I. First report of tomato apical stunt viroid inSolanum jasminoidesin Slovenia. New Disease Reports, 2012, 26: 7.
[13]Zhang YJ, Yin J, Li GF, et al. Oligonucleotide microarray with a minimal number of probes for the detection and identification of thirteen genera of plant viruses. J Virol Methods, 2010, 167:53–60.
[14]Zhang YJ, Xin YY, Zhu SF, et al. Establishment of the screening oligonucleotide microarray of Apscaviroid in genus level. Biotechnol Bull, 2013,2: 151–156 (in Chinese).
张永江, 辛言言, 朱水芳, 等. 苹果锈果类病毒属属级寡核苷酸芯片筛查方法的建立. 生物技术通报, 2013, 2: 151–156.
[15]类病毒芯片检测 [EB/OL]. [2013-07-24].http://cost853.acw-online.ch/pdf-files/spain%20me eting/Posters/Tomlinson%20mircoarray%20v2.pdf(in Chinese).
[16]Shi FY, Miao JK, Zhang LP, et al. Detection of bovine, goat, pig and chicken derived ingredients in animal products with universal PCR-microarray method. Chin J Biotech, 2010, 26(6): 823–829 (in Chinese).
石丰运, 缪建锟, 张利平, 等. 运用基因芯片技术检测牛、山羊、猪和鸡源性成分. 生物工程学报, 2010, 26(6): 823–829.
[17]Zhai JH, Song YJ, Du ZM, et al. Study on universal microarray system for detection of common clinical infectious bacteria. Chin J Public Health, 2003,19(4): 430–431 (in Chinese).
翟俊辉, 宋亚军, 杜宗敏, 等. 通用基因芯片检测感染性细菌方法的研究. 中国公共卫生, 2003,19(4): 430–431.
[18]Lü DQ. Detection and control of potato spindle tuber disease. Chin Potato J, 2005, 19(6): 361–365(in Chinese).
吕典秋. 马铃薯纺锤块茎类病毒(PSTVd)的检测与防治研究进展. 中国马铃薯, 2005, 19(6):361–365.
[19]Lü DQ, Li XZ, Bai YJ, et al. Application of NASH and R-PAGE techniques to the detection of potato viroid. J Northeast Agricul Univ, 2003, 34(1):15–18 (in Chinese).
吕典秋, 李学湛, 白艳菊, 等. NASH 技术和R-PAGE技术在马铃薯类病毒检测上的应用. 东北农业大学学报, 2003, 34(1): 15–18.
[20]Antignus Y, Lachman O, Pearlsman M. The spread of Tomato apical stunt viroid (TASVd) in greenhouse tomato crops is associated with seed transmission and bumble bee activity. Plant Dis,2007, 91(1): 47–50.
[21]Cohen O, Batuman O, Stanbekova G, et al.Construction of a multiprobe for the simultaneous detection of viroids infecting citrus trees. Virus Genes, 2006, 33(3): 287–292.
[22]Ragozzino E, Faggioli F, Barba M. Development of a one tube-one step RT-PCR protocol for the detection of seven viroids in four genera:Apscairoid, Hostuviroid, Pelamoviroid and Pospiviroid. J Virol Methods, 2004, 121: 25–29.
[23]Wang XF, Zhou CY, Tang KZ, et al. A rapid one-step multiplex RT-PCR assay for the simultaneous detection of five citrus viroids in China. Eur J Plant Pathol, 2009, 124: 175–180.
[24]Verhoeven JThJ, Botermans M, Meekes ETM, et al. Tomato apical stunt viroid in the Netherlands:most prevalent pospiviroid in ornamentals and first outbreak in tomatoes. Eur J Plant Pathol, 2012,133: 803–810.
[25]Shamloul AM, Faggioli F, Keith JM, et al. A novel multiplex RT-PCR probe capture hybridization(RT-PCR-ELISA) for simultaneous detection of six viroids in four genera: Apscavroid, Hosuviroid,Pelamoviroid, and Pospiviroid. J Virol Methods,2002, 105: 115–121.
[26]Li R, Gao S, Hernandez AG, et al. Deep sequencing of small RNAs in tomato for virus and viroid identification and strain differentiation. PLoS ONE,2012, 7: e37127.
