基于分子信标的DNA自组装立体结构
2014-06-05殷志祥
刘 静, 殷志祥
(安徽理工大学 理学院,安徽 淮南 232001)
文献[1]首先建立了分子信标技术,很快该技术被广泛应用于医学、生物学及分子生物学等诸多领域。分子信标技术具有极高的特异性,操作简单、灵敏度高,特别是在进行实时定量检测、临床诊断及基因检测方面具有极强的优势。文献[2-3]利用分子信标构造的可满足性问题的DNA计算模型和文献[4]基于分子信标的逻辑门的计算模型对于分子信标的研究具有重大意义;文献[5-6]将分子信标应用于DNA计算中。
1 分子信标技术的原理
分子信标是一种荧光标记的寡核苷酸链,一般有25~35个核苷酸。在结构上,它是由环状区、茎干区、荧光基团和淬灭基团组成。环状区一般由15~30个核苷酸组成,并且可以与靶分子特异性结合。茎干区一般由5~8个碱基对组成,在分子信标与靶分子结合过程中可发生可逆性解离,而荧光基团一般是连接在5′端的,淬灭基团一般连在3′端。在自由状态时,分子信标呈发夹结构,荧光基团和淬灭基团相距较近,然后发生荧光共振能量转移,使荧光基团发出的荧光被淬灭基团吸收并以热的形式散发,荧光几乎完全被淬灭;当分子信标与靶分子结合时,荧光基团与淬灭基团的距离变大时,荧光就会恢复,如图1所示。
就目前的研究来看,如何找到更好的方法能够最大限度地挖掘分子信标表达和处理信息的能力、如何能找到分子信标环部识别区长度与荧光颜色轻度或跟茎干的长度等之间的关系,从而寻找到信息容量更大、灵敏性更高、结构更复杂、更加通用的分子信标模型是进一步研究的重点。
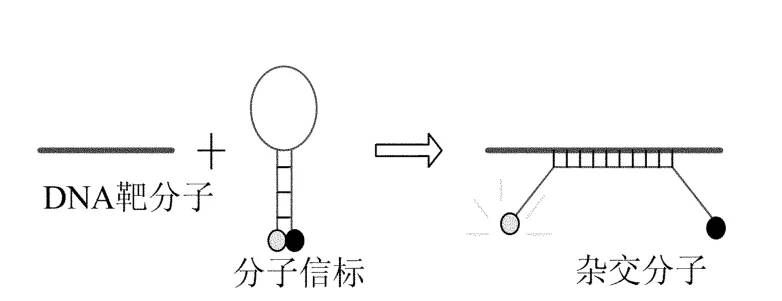
图1 分子信标的原理
2 DNA自组装
随着DNA计算的逐步深入,DNA计算正逐步朝着自组装方向发展,因为它极有可能成为下一代计算机的“图灵机”。文献[7]首次提出利用DNA分子构成自组装瓦片结构;文献[8]给出了求解累计异或运算的方法;文献[9]将自组装思想用于求解布尔逻辑运算;文献[10]实现一维元胞自动机,后来又对DNA进行编码,成功构造出各种纳米结构[11];文献[12-14]提出了二维自组装求解可满足性问题的方法、非确定性算法、子集合问题等。以上研究主要是针对一维和二维的结构展开的,对于三维的自组装模型研究还处于起步阶段[15-16]。
二维结构和三维结构的自组装在概念上是没有区别的,二维结构自组装中运用的原理可以运用到三维结构中。例如,文献[17]合成了立方体和去角的八面体等纳米结构;文献[18]构建了DNA四面体、十二面体等,证明了DNA作为支架可以用来构建三维结构。
3 三维DNA自组装立体结构
分子信标技术本身具有简单便捷、实时监测、灵敏度高等特点,伴随着该技术的不断发展,不少研究人员也在考虑将该技术与其他相关的模型进行结合。诸如与链置换技术结合,进而可以有效地构建DNA逻辑门[19];与DNA变构相结合,使得通过DNA分子的不同形态构建可控的荧光纳米装置,随着DNA分子的变构,通过检测荧光强度就能准确了解DNA分子的构象变化[20];与某些酶切技术相结合,可以有效地进行检测,有时比PCR检测更具灵敏性[21]。这些技术的结合能够让两者的优势都得以发挥,进而优势互补,可取得令人兴奋的结果。
从自组装的结构来说,三维立体结构相比于一维的线性结构和二维的平面结构在发展纳米技术、药物载体及DNA计算等方面都具有巨大的优势。文献[22]将DNA自组装和荧光技术相结合,利用DNA分子碱基互补配对,杂交后再用连接酶连接,得到了环链结构。文献[23]将DNA荧光技术应用在检测纳米装置的开关。这些三维结构的尝试对于DNA自组装立体结构的发展都具有重大的意义。
在这些研究的基础上,本文试图探究一种稳定性更高、产量更高的DNA自组装立体结构。文献[24]提出的关于纳米DNA结构中,DNA四面体的三维结构是天然的“积木”,而且可以迅速地合成一个单一的立体异构体,且具有产量高、三角架结构的传送稳定性等特点。现构想DNA四面体的设计过程如图2所示。首先组装了3条短的DNA链和1个分子信标,6对互补域(图中相同标号部分)杂交成6条边。每条链包含3个区域并且运行1周。在该DNA的骨架中,5′和3′端都有一个缺口,给予适宜的条件,分子信标会找到其靶分子,最后会形成图2b和图2c所示的发夹结构。在该结构中5条边是20个碱基互补配对的双螺旋结构,并且其长度为6.8nm,第6条边的长度为3.4nm,它包含10个碱基,与分子信标结合后形成4个碱基的发夹环和12个核苷酸的环域。
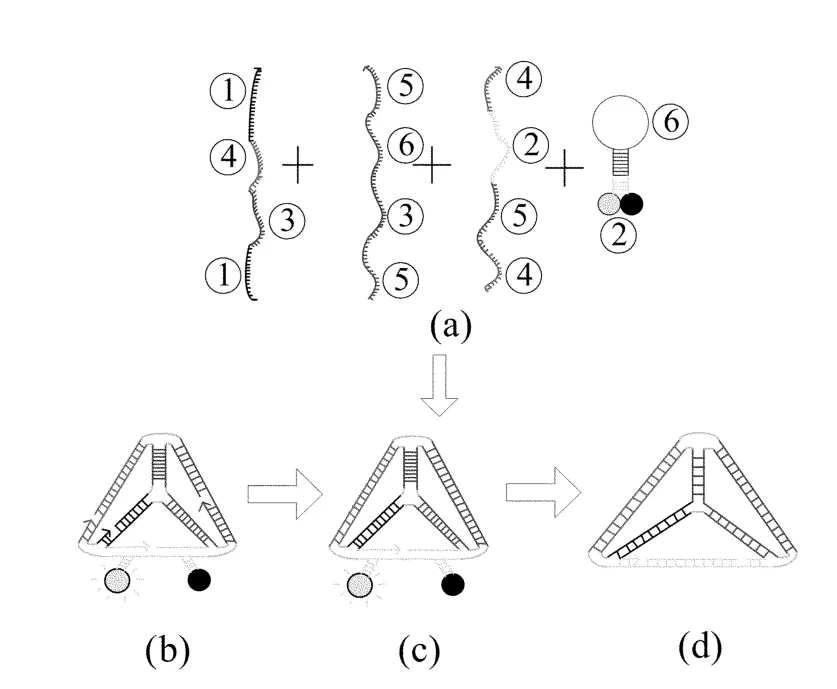
图2 DNA自组装立体结构
组装四面体的过程是将其放在含盐的缓冲液中,加热至95℃,再冷却至室温,此时要特别注意退火的温度,保证分子信标与DNA链杂交,要形成发夹结构的其他3条链都进行磷酸酸化,再用T4DNA连接酶处理后,就会发生自组装反应。首先让分子信标与其他3条链进行反应,形成发夹结构,这种发夹结构是可以重构的,当发夹形成闭合状态时,含有5个碱基配对的链和分子信标的发夹结构的茎就会形成1个缺口,并进一步形成发夹循环,该过程的能量是由12个核酸杂交所释放的。在图2d中表示的是由2个缺口的2条连续的30个碱基互补配对的双链。作为释放能量的分子信标的核酸是没有与四面体形成的复合物杂交的。图2b、图2c和图2d演示了四面体打开和闭合的发夹模型。还可以采用聚丙烯酰胺凝胶电泳(PAGE)来观察并检验其重构性,确定发夹循环是正确形成的,如图3所示。
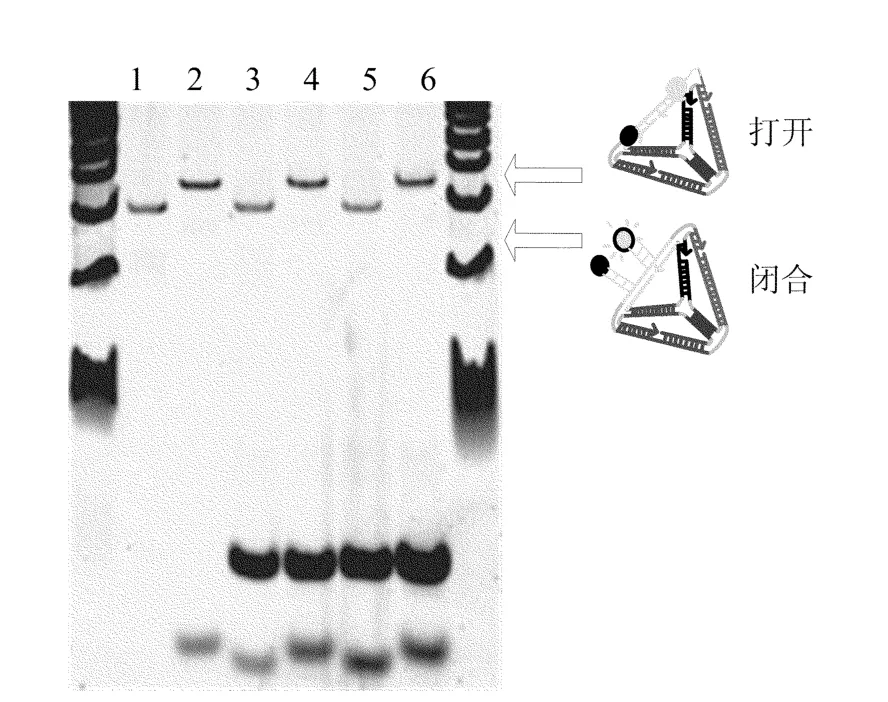
图3 PAGE演示分析移动性闭合状态和打开状态
4 三维结构的应用
上述结合分子信标的自组装三维结构不仅有图像结构信息,而且最重要的是可以随着结构的变化应用于纳米技术、药物载体和DNA计算等方面。在DNA计算方面,关于立体结构应用方面的研究相对较少,但是可以从其独特的优势,看出它适合解决NP难的问题,因为其复杂的三维结构可以缩短自组装的时间、空间复杂度、生物酶的使用量,从而提高实验成功率和精确度。将它运用于逻辑运算[25-27],并将其整合制作成 DNA芯片,利用其高效并行的计算能力,推动DNA计算机的发展。
布尔逻辑运算得名于英国数学家乔治·布尔,现广泛应用于电子学、计算机软件和硬件中,在计算机中用布尔逻辑运算定义一些布尔逻辑函数,即操作符。每个函数可以根据1个或多个输入,用逻辑算法来得到输出值(真值为1,假值为0)。因此,逻辑运算具有重要的意义。
本文拟在分子维度方面利用上述立体结构解决简单布尔逻辑表达式的求值问题,现利用该立体结构设计构建“and”门和“or”门,主要包括的工作有:①DNA自组装模型结构的设计、信号传入和识别、输出信息;② 总体上的设计,包括在该过程中温度的控制、溶液浓度的控制、酶的使用量以及防止错配等问题。
利用立体结构自组装and逻辑门,如图4所示。它的5条边是20个碱基互补配对的双螺旋结构,长度为6.8nm,第6条边的长度是3.4nm,包含10个碱基。在运行过程中,自DNA链放入盐酸溶液中后,需要加热至95℃,冷却,加入分子信标,再开始加入需要识别的信号溶液1和2,几分钟后即可检测到荧光,检测得到荧光后加热,3条链和分子信标会分离,回到最初的状态。
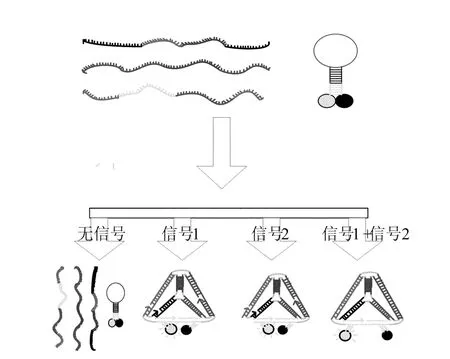
图4 自组装and逻辑门
类似地,自组装“or”逻辑门也可以采用相同的方式进行。在选择的过程中,只要有1个值为“真”,即可成立。示意图如图5所示。
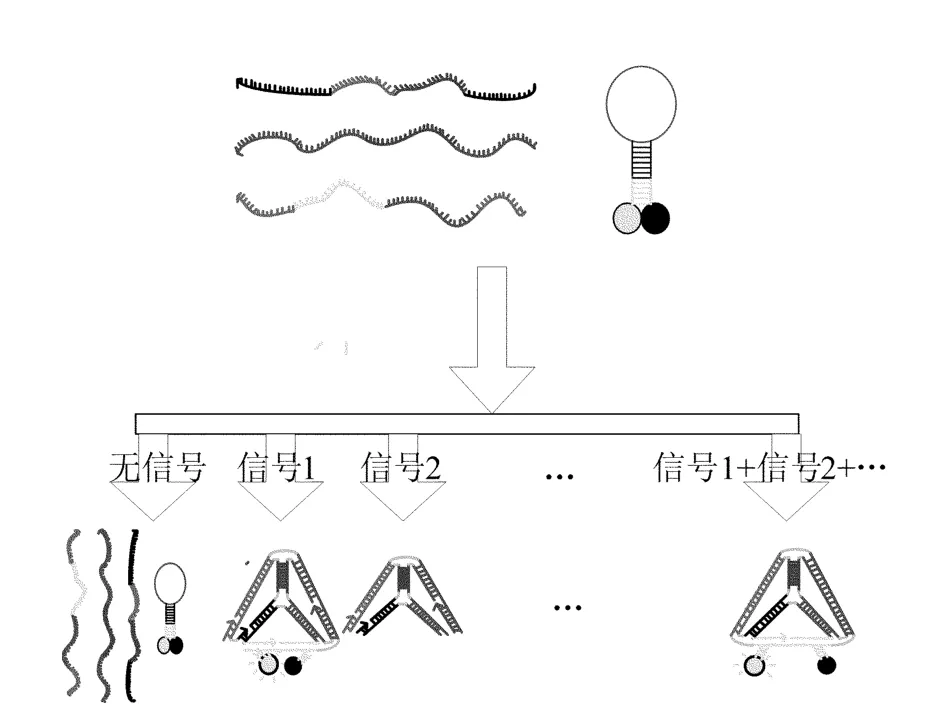
图5 自组装or逻辑门
在上述and和or逻辑门的基础上,本文可以做复杂的逻辑运算,为不失一般性,选取的合取范式如下:
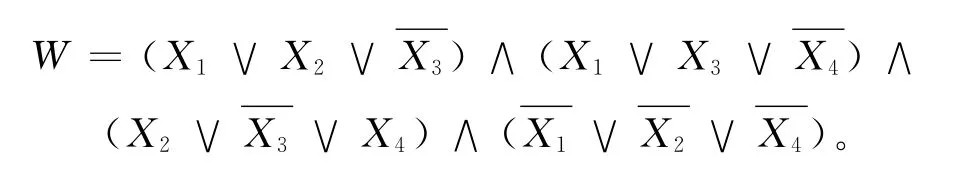
具体运算过程如图6所示,其实验方法如下:分别计算每个子句的值;利用自组装自底向上的特点,再分别计算次子句的值;计算出整个逻辑运算W的值。应用上述立体结构,本文首先构造DNA链,让其进行第1层的子句运算,因为三维结构本身就具有多个变量同时计算的特点,在本例中只需3步即可得到结果。
将DNA链放在含盐的缓冲液中,加热至95℃,冷却至室温,再加入分子信标,然后进行磷酸酸化,保证发夹结构的形成,再用T4DNA连接酶进行处理。可以在不同的区域检测到3处荧光,还有一部分并未形成发夹结构。再进行一次上述处理后进行检测,没有形成发夹结构。综上所述,可以得到W=0。
应用该结构,不仅可以简化二维结构中的多次运算,而且由于它可以重构而反复使用,具有一定的实际价值。
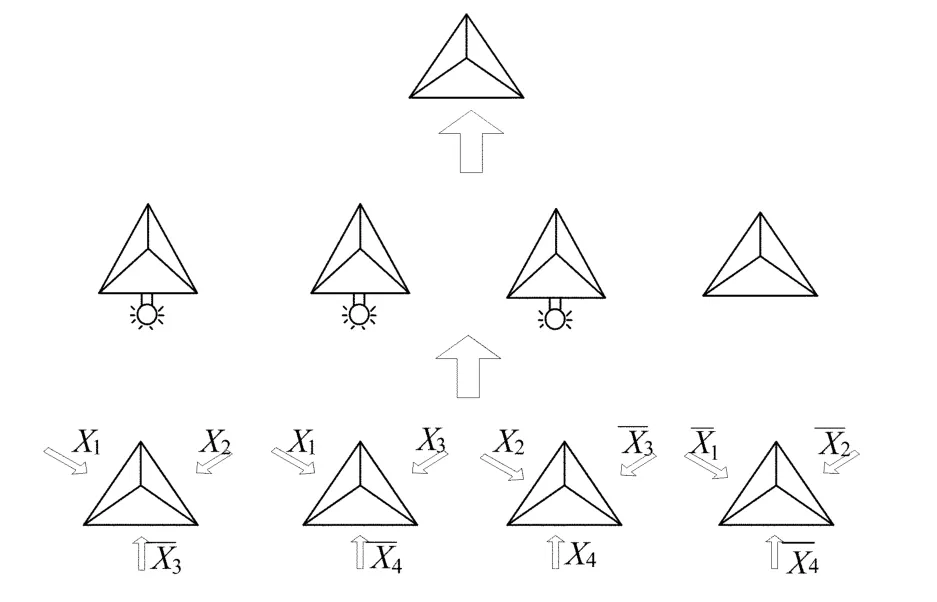
图6 DNA自组装自下而上的运算过程
5 结束语
本文利用已有的分子信标技术和DNA自组装模型的研究成果,主要研究了一种DNA自组装立体结构——带有分子信标的DNA四面体,它能够完成自行装配,且实验操作简单。另外,该模型也为相关领域(如计算机科学、密码学及纳米智能等)提供了新的思路和结合点。随着三维DNA自组装模型的深入研究,DNA计算在未来的实际应用中必将发挥重大作用。
[1]Tyagi S,Kramer F R.Molecular beacon:probes that fluoresce upon hybridization[J].Nat Biotech,1996,14(3):303-308.
[2]殷志祥,张凤月,许 进.基于分子信标的DNA计算[J].生物数学学报,2003,18(4):497-501.
[3]殷志祥.图与组合优化中的DNA计算[M].北京:科学出版社,2004:37-40.
[4]刘文斌,朱翔鸥,殷志祥.基于分子信标的逻辑门的计算模型[J].安徽理工大学学报:自然科学版,2008,28(1):65-69.
[5]Wang Y K,Li J S,Tang H X.Strategy for molecular beacon binding readout:separating molecular recongnition element and signal reporter[J].Analytical Chemistry,2009,81(23):9703-9708.
[6]LaBean T H,Reif J H,Seeman N C,et al.Construction,analysis,ligation,and self assembly of DNA triple crossover complexes[J].Journal of the American Chemical Society,2000,122:1848-1860.
[7]Seeman N C.DNA nanotechnology:novel DNA constructions[J].Annual Review of Biophysics and Bimolecular Structure,1998,27:225-248.
[8]Mao C,LaBean T H,Reif J H,et al.Logical computation using algorithmic self-assembly of DNA triple-crossover molecules[J].Nature,2000,407(6803):493-496.
[9]Carbone A,Seeman N C.Circuits and programmable self-assembling DNA structures[J].Proceedings of the National Academy of Sciences of the United States of America,2001,99(20):12577-12582.
[10]Rothemund P W K,Papadakis N,Winfree E.Algorithmic self-assembly of DNA Sierpinski triangles[J].PLOS Biology,2004,2(12):2041-2053.
[11]Rothemund P W K.Folding DNA to create nanoscale shapes and patterns [J].Nature,2006,440(7082):297-302.
[12]Michail G L,LaBean T H.2DDNA self-assembly for satisfiability[J].DIMACS Series in Discrete Mathematics and Theoretical Computer Science,1999,44:139-152.
[13]Brun Y.Solving satisfiability in the tile assembly model with a constant-size tileset [J].Journal of Algorithms,2008,63(4):151-166.
[14]Brun Y.Solving NP-complete problems in the tile assembly model[J].Theoretical Computer Science,2008,395(1):31-36.
[15]周炎涛,李肯立,罗 兴,等.一种基于DNA自组装模型求解最大独立集问题的算法[J].湖南大学学报:自然科学版,2012,39(9):39-44.
[16]Zang Wenke,Liu Xiyu,Liu Wenju.DNA computing approach on surface to solve minimal set covering problem[J].Application Research of Computers,2012,29(4):1220-1222.
[17]Goodman R P,Schaap I A T,Tardin C F,et al.Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication[J].Science,2005,310:1661-1665.
[18]Shih W M,Quispe J D,Joyce G F.A 1.7-kilobase singlestranded DNA that folds into a nanoscale octahedron[J].Nature,2004,427(2):618-621.
[19]Georg S,David S,Yu Z D,et al.Enzyme-free nucleic acid logic circuits[J].Science,2006,314:1585-1588.
[20]Mao Chengde,Sun Weiqiong,Shen Zhiyong.A nanomechanical device based on the B-Z transition of DNA[J].Nature,1999,397(14):144-146.
[21]Wang Liaman,Hall Jeff G,Lu Manchun.A DNA computing readout operation based on structure-specific cleavage[J].Nature Biotechnology,2001,19:1053-1059.
[22]Weizmann Y,Braunschweig A B,Wilner O I.A polycatenated DNA scaffold for the one-step assembly of hierarchical nanostructures[J].Proceedings of the National A-cademy of Sciences of the Uniteal States of America,2008,105(14):5289-5294.
[23]Andersen E S,Dong Mingdong,Nielsen M M,et al.Selfassembly of a nanoscale DNA box with a controllable lid[J].Nature,2009,459:73-77.
[24]Goodman R P,Schaap I A T.Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication [J].Science,2005,310:1661-1665.
[25]Zhang C,Ma L N,Dong Y F,et al.Molecular logic computing model based on DNA self-assembly strand branch migration[J].Chinese Sci Bull,2012,57(31):2909-2915.
[26]黄玉芳,程 珍,周 康,等.基于DNA Tile自组装的布尔逻辑运算[J].计算机学报,2009,32(12):2347-2354.
[27]Zhang C,Yang J,Xu J.Molecular logic computing model based on self-assembly of DNA nanoparticles[J].Chinese Sci Bull,2011,56(27):2276-2282.
