大豆含硫氨基酸相关酶基因发掘
2014-05-25邱红梅郝文媛高淑芹马晓萍郑宇宏孟凡凡范旭红王洋王跃强王曙明
邱红梅,郝文媛,高淑芹,马晓萍,郑宇宏,孟凡凡,范旭红,王洋,王跃强,王曙明
大豆国家工程研究中心,吉林省农业科学院大豆研究所,长春 130033
大豆含硫氨基酸相关酶基因发掘
邱红梅,郝文媛,高淑芹,马晓萍,郑宇宏,孟凡凡,范旭红,王洋,王跃强,王曙明
大豆国家工程研究中心,吉林省农业科学院大豆研究所,长春 130033
大豆(Glycine max)含硫氨基酸合成途径中的酶基因是含硫氨基酸组分的重要调控基因,发掘相关酶基因对高含硫氨基酸分子育种具有重要意义。文章采用大豆物理与遗传整合图谱,通过BioMercator2.1将113个含硫氨基酸合成途径酶基因及33个控制含硫氨基酸含量的QTL整合到遗传图谱Consensus Map 4.0上,依据酶基因位点与QTL的一致性以及QTL的效应值,初步筛选到16个与含硫氨基酸合成相关的候选基因。通过生物信息学方法对候选基因进行拷贝数、SNP、表达谱等分析,鉴定到 12个相关酶基因,分别位于 D1a、M、A2、K和G等8个连锁群上。生物信息学分析显示这些基因所在QTL可解释含硫氨基酸遗传变异的6.0%~38.5%,其中9个基因的间接效应值超过10%。12个相关酶基因参与含硫氨基酸代谢的重要途径,且多在子叶、花中高丰度表达,存在丰富的SNP。这些基因可作为候选基因进行功能标记开发,将为大豆分子设计育种奠定基础。
大豆;含硫氨基酸;合成途径;酶基因;生物信息学
大豆(Glycine max)在世界上被广泛种植,常用来制做各种豆制品。据统计,全球 70%的植物蛋白来自大豆[1]。但其含硫氨基酸组分较低,在豆制品中需添加含硫氨基酸。然而,在处理过程中会形成挥发性硫化物并增加成本[2,3]。发掘或创制高含硫氨基酸种质是提高大豆含硫氨基酸组分最有效的途径之一[4]。大豆含硫氨基酸包括甲硫氨酸和半胱氨酸,合成途径受遗传及环境等因素的调控和影响[5]。遗传因素包括氨基酸代谢途径酶基因及控制含硫氨基酸含量的QTL等[6]。解析遗传调控基础,挖掘含硫氨基酸合成途径相关的酶基因,是进行高含硫氨基酸种质分子辅助选择与分子设计育种的基础。
目前,已知丝氨酸和乙酰CoA在丝氨酸乙酰转移酶(SAT)催化下形成 O-乙酰丝氨酸;O-乙酰丝氨酸与硫化物在半胱氨酸合成酶(CS)的催化下形成半胱氨酸[7~9]。半胱氨酸与 O-磷酰高丝氨酸在胱硫醚-γ-合酶(CGS)的催化下形成胱硫醚。后者又在 β-胱硫醚酶(CBL)作用下,形成高半胱氨酸,最后在甲硫氨酸合酶(MS)的催化下生成甲硫氨酸[10,11]。在大豆中半胱氨酸与 O-乙酰高丝氨酸在 CGS的催化下可直接形成高半胱氨酸,其在半胱氨酸甲基转移酶(CMT)的催化下生成甲硫氨酸。大豆基因组测序完成后,在 KEGG数据库上公布了大豆含硫氨基酸合成途径(http://www.kegg.jp/pathway/gmx00270),注明了特有的相关酶。这些酶由 113个基因编码,NCBI数据库收录了这些酶基因的相关信息(http://www.ncbi.nlm.nih.gov/ gene?LinkName= biosystems_gene_all)[12]。于妍等[13]利用大豆基因组物理和遗传整合图谱,将17个大豆天冬氨酸代谢途径酶基因分别定位在F、G、J及I等11个连锁群上。Panthee等[14]利用RIL群体,发现10个控制含硫氨基酸含量的QTL,分别定位在F、G、M、D1a、D2等连锁群上,效应值在 7.6%~22.9%之间。Carlson等[15]利用两个RIL群体得到21个控制含硫氨基酸含量的QTL,分别定位在K、O、C1、C2、A2、K、G和F等连锁群上,效应值在3.0%~26.0%之间。Fallen等[16]利用RIL群体,定位了2个控制含硫氨基酸含量的QTL,分别定位在 F、I连锁群上,效应值分别为 5.2%和6.0%。
上述研究只是从代谢途径或遗传调控基础单方面解析大豆含硫氨基酸的合成,缺乏两者间的关联性分析。本研究利用大豆物理与遗传整合图谱及BioMercator2.1,将113个含硫氨基酸合成酶基因与33个控制含硫氨基酸含量的 QTL共定位到整合遗传图谱Consensus Map 4.0上[17],通过酶基因与QTL一致性位点,筛选与含硫氨基酸合成相关的酶基因。
利用生物信息学方法对候选基因进行基因组拷贝数、SNP、表达谱等分析,进一步鉴定相关酶基因,为含硫氨基酸功能标记开发提供参考。
1 材料和方法
1.1 含硫氨基酸合成酶基因和QTL信息的收集
在NCBI数据库(http://www.ncbi.nlm.nih.gov)下载113个含硫氨基酸合成途径酶基因序列,包括基因名称、所在染色体、物理位置、基因注释等。
收集 33个多年多点重复鉴定的控制含硫氨基酸含量的QTL信息,包括QTL名称、所在染色体位置、临近标记、染色体位置、LOD值、贡献率和置信区间(C.I.)等[14~16]。
1.2 大豆含硫氨基酸合成途径酶基因的遗传定位
利用 Soybase(http://www.soybase.org/dlpages/ind-ex.php)物理(Glyma1.1)与遗传(Consensus Map 4.0)整合图谱,将酶基因的物理位置转换为遗传位置,找到临近的左右标记,并通过 BioMercator2.1软件定位到Consensus Map 4.0图谱上。
1.3 大豆含硫氨基酸相关基因的初步鉴定
根据BioMercator2.1软件要求的QTL文件格式整理数据,利用 BioMercator2.1软件将控制含硫氨基酸含量的QTL映射到Consensus Map 4.0图谱上,构建控制含硫氨基酸含量的QTL“一致性图谱”。通过酶基因位点与控制含硫氨基酸含量的QTL的一致性,筛选相关酶基因。
1.4 相关酶基因的生物信息学分析
利 用 Soybase(http://soybase.org/GlycineBlast-Pages/)的Blast分析软件对基因进行拷贝数分析。利用NCBI/ UniGene(http://www.ncbi.nlm.nih.gov/unigene)对基因的表达谱进行分析。利用 NCBI/dbSNP (http://www.ncbi.nlm. nih.gov/snp/)对基因的SNP进行分析。利用 PSORT(http: //psort.hgc.jp/form.html)预测蛋白的亚细胞定位。
2 结果与分析
2.1 含硫氨基酸合成途径酶基因的遗传定位
利用大豆物理与遗传整合图谱及BioMercator2.1,将 113个大豆含硫氨基酸合成途径酶基因分别定位到Consensus Map 4.0的20个连锁群上(表1)。N、H、E连锁群上的基因最少为3个,A2连锁群上的基因最多为10个。酶基因在连锁群的中部、末端均有分布,无位置偏好性。113个酶基因共编码30个含硫氨基酸合成途径酶,平均 3.8个基因同源。O、F、I连锁群上存在3对串联重复基因,分别为编码甲硫基戊烯加双氧酶(MTD)的100812492和100500632、编码酪氨酸转氨酶(TAT)的100800297和100800830、编码 MTD 的 100500592和 100306042。其中100812492、100500632、100500592和100306042为同源基因。D1a连锁群上的100799480与100798061为同源基因,编码胞嘧啶甲基转移酶(CMT)。N连锁群上的100819100与OAS-TL1为同源基因,编码半胱氨酸合成酶(CS)。A2连锁群上的100793294与100808025为同源基因,编码羧酸氧化酶(ACO)。编码亚精胺合成酶(SS)的同源基因有 9个,在 D1a、 D1b、C2、A2、O、B2和 G连锁群上各 1个,在D2连锁群上有2个。表明大豆染色体加倍时,重要功能基因也加倍,但随着进化基因序列发生变化,在不同连锁群形成同源基因[18]。
2.2 含硫氨基酸代谢相关酶基因的初步鉴定
通过酶基因位点与控制含硫氨基酸含量的QTL的一致性,筛选到17个含硫氨基酸合成途径酶基因,分别位于D1a、C1、M、A2和K等10个连锁群上(图1)。D1a连锁群上的100790506,所在半胱氨酸QTL的效应值为8.5%。C1连锁群上的100776541,所在半胱氨酸QTL的效应值合计23.6%。M连锁群上的100815091,所在甲硫氨酸QTL的效应值为22.9%。A2连锁群上的100778925、100793294,所在甲硫氨酸QTL的效应值为9.0%; 100798053所在半胱氨酸QTL的效应值为3.0%,因效应值太小,舍去该基因。K连锁群上的100799311,所在半胱氨酸和甲硫氨酸QTL的效应值合计18.0%;CYS所在半胱氨酸和甲硫氨酸QTL内,效应值合计11.0%。
O 连锁群上的 100812480、100775420和100808736,所在半胱氨酸QTL的效应值为19.0%。F连锁群上的100805448,所在半胱氨酸+甲硫氨酸和半胱氨酸 QTL的效应值合计 27.0%;100779947所在半胱氨酸+甲硫氨酸、甲硫氨酸和半胱氨酸QTL的效应值合计38.5%。D2连锁群上的100791928,所在半胱氨酸+甲硫氨酸QTL的效应值7.6%。G连锁群上的100776863,所在半胱氨酸QTL的效应值为18.8%;100800410所在半胱氨酸和甲硫氨酸 QTL的效应值合计30.9%。I连锁群上的100792700,所在半胱氨酸QTL的效应值为6.0%。虽不排除QTL内含有其他调控基因,但相关效应值可作为本研究的筛选依据。分析结果表明,初步鉴定的16个候选基因,可解释含硫氨基酸含量的部分遗传变异。
2.3 候选基因的生物信息学分析
通过上述分析在控制含硫氨基酸含量的QTL位点内,得到16个重要酶基因, 这些基因可能与储藏含硫氨基酸组分有关,但并未包括全部氨基酸合成途径关键酶基因。对这些基因进行生物信息学分析,分别得到候选基因的拷贝数、SNP、高表达器官等信息(表2)。这些基因多为2~3个拷贝,含有多个 SNP,多在子叶和花中表达,编码的酶多定位于
细胞质。100790506有2个拷贝,9个同源基因,62个SNP,在子叶与花中高丰度表达,编码的SS定位于细胞质。100776863是 100790506的同源基因,在下胚轴高丰度表达,编码的SS定位于细胞核。氨基酸的合成多在细胞质及叶绿体中进行,因此100776863与含硫氨基酸的积累无关。100776541在基因组中有 2个拷贝,在叶芽与茎中高丰度表达,与储藏含硫氨基酸相关的基因多在子叶或花中表达,因此该基因与含硫氨基酸的积累无关。100779947是100776541基因的拷贝,在子叶与根中高丰度表达。100815091在根中高丰度表达,表明在含硫氨基酸前期积累过程中,可能存在氨基酸从根到子叶的运输。100778925有2个拷贝,4个同源基因,10个SNP,编码的氨基环丙烷羧酸合酶(ACS)定位于细胞质。100791928是 100778925的同源基因,编码的ACS定位于细胞核,因此判定该基因与含硫氨基酸的积累无关。
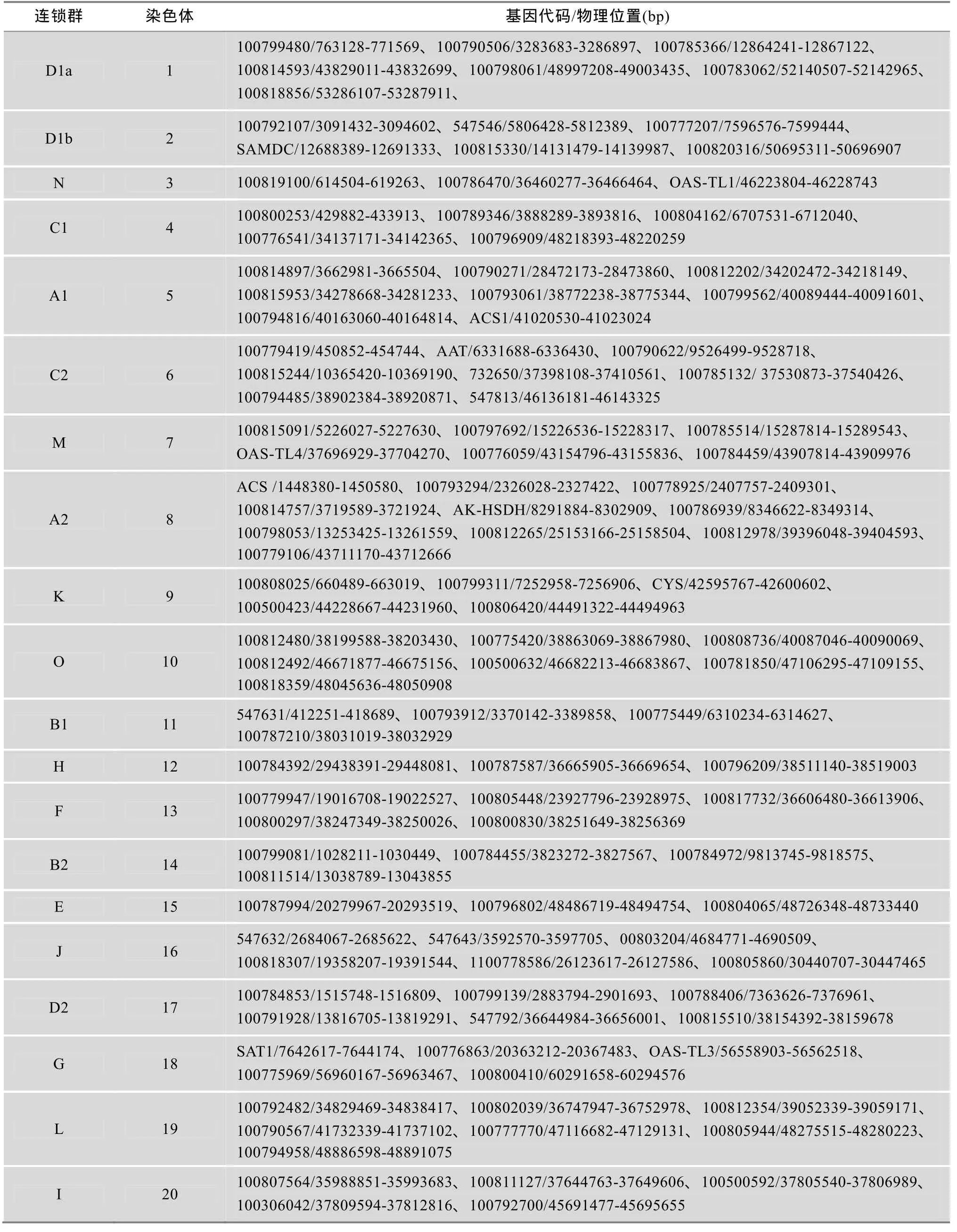
表1 大豆含硫氨基酸合成途径酶基因的遗传定位

图 1 大豆含硫氨基酸合成途径酶基因与QTL一致性位点
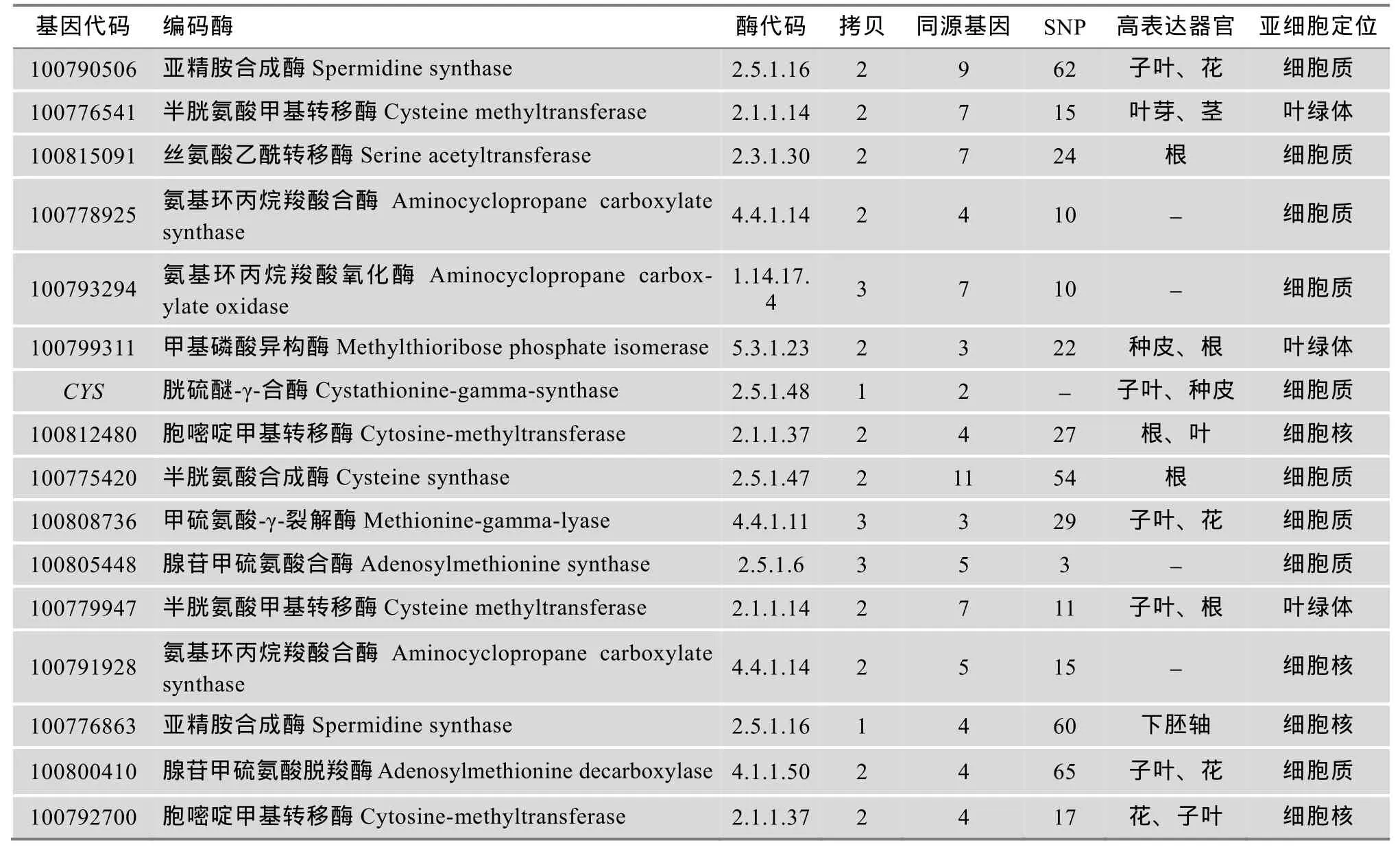
表2 相关酶基因的生物信息学分析
100793294有 3个拷贝,7个同源基因,10个 SNP,未检索到其表达谱信息,但在大豆EST数据得到多条与其高度比配的序列。CYS在NCBI数据库中未检索到SNP,但通过本地Blast与Clustal-1.81得到了SNP。100812480是100792700的拷贝,编码的CMT均定位于细胞核。但100792700在花和子叶高丰度表达,亚细胞定位结果为软件预测,还需进一步验证,因此,该基因可能与含硫氨基酸的积累有关,可作为候选基因进一步研究。
2.4 相关酶基因遗传调控的代谢途径
通过生物信息学分析,初步鉴定到12个与大豆含硫氨基酸合成相关的酶基因,分别调控半胱氨酸与甲硫氨酸合成、甲硫醇的生成及甲硫氨酸循环途径(图2)。图2结果表明,在甲硫氨酸合成途径上,半胱氨酸与O-乙酰高丝氨酸在CGL基因的调控下可直接形成高半胱氨酸,无需CBL基因参与。100815091和 100775420调控半胱氨酸的合成。100815091编码的 SAT(2.3.1.30)催化丝氨酸与乙酰 CoA生成 O-乙酰丝氨酸,此反应是可逆反应,受底物浓度调控[19]。

图2 相关酶基因调控的代谢途径
100775420编码的CS(2.5.1.47)催化O-乙酰丝氨酸与硫化物生成半胱氨酸,该过程是硫元素进入植物新陈代谢的唯一途径,受底物浓度、酶活等因素影响[20]。CYS和100779947调控甲硫氨酸的合成。CYS编码的CGS(2.5.1.48)催化半胱氨酸与O-乙酰高丝氨酸生成高半胱氨酸。CGS在大豆中能催化5个化学反应,是重要的多功能酶。100779947编码的CMT(2.1.1.14)催化高半胱氨酸生成甲硫氨酸。100808736编码的甲硫氨酸-γ-裂解酶(MGL,4.4.1.11)催化甲硫氨酸生成甲硫醇,该反应受甲硫氨酸浓度反馈抑制,研究表明,MGL的活性也影响半胱氨酸的含量[21,22]。
100805448、100792700、100778925、100793294、100800410、100790506和100799311调控甲硫氨酸循环途径。100805448编码的腺苷甲硫氨酸合酶(AMS,2.5.1.6)催化甲硫氨酸生成腺苷甲硫氨酸,细胞中大约80%的甲硫氨酸都经过2.5.1.6形成腺苷甲硫氨酸。在植物中腺苷甲硫氨酸是主要的甲基供体,通常作为底物参与多种生化途径,包括多胺和乙烯的合成。100778925编码的氨基环丙烷羧酸合酶(ACS,4.4.1.14)催化腺苷甲硫氨酸生成氨基酸环丙烷羧酸盐,100793294编码的氨基环丙烷羧酸氧化酶(ACO,1.14.17.4)催化氨基酸环丙烷羧酸盐生成乙烯。ACO是乙烯生物合成的限速酶,ACO基因的表达具有组织和环境特异性,随发育、环境的不同而变化[23]。在逆境下乙烯含量增加,甲硫氨酸相对含量降低,推测可导致储藏蛋白及含硫氨基酸含量的减少。
3 讨 论
大豆基因组测序后,在 NCBI(http://www.ncbi.nlm. nih.gov)上公布了大豆基因组序列及预测基因的功能注释。NCBI是一个开放的数据库,研究者可以提交基因的EST、SNP信息,这些都是实验性的研究结果。利用EST建立了基因的UniGene数据库,这里的表达谱信息是可靠的。通过基因组注释还构建了许多代谢途径模型。基因组注释可通过各种电子注释工具,如KEGG 的自动注释功能,实现粗模型的数据提取[24]。KEGG数据库里收录了大豆含硫氨基酸合成途径。本文将KEGG中含硫氨基酸合成途径与控制含硫氨基酸含量的QTL进行关联分析,初步鉴定相关的候选酶基因;再依据基因的表达谱(来自UniGene数据库)及亚细胞定位信息进一步鉴定候选基因。此方法数据来源可靠,候选基因的筛选更准确。
大豆基因组发生过两次大规模复制,形成了一个高度重复的基因组,约 75%的基因存在多个同源基因[12]。本文初步鉴定的12个相关酶基因,均有同源基因。其中编码CGS(2.5.1.48)的CYS基因有2个同源基因,编码CS(2.5.1.47)的100775420有11个同源基因。同源基因数在 3~10个的为非家族(Nonfamily,NF)基因,研究表明 NF基因调控植物基本生命代谢过程[25]。基因的拷贝可以降低进化的选择压,但重复基因之间可能存在功能的冗余[26]。本文通过目标性状遗传调控位点,在同源基因中筛选到含硫氨基酸相关的酶基因,表明并非所有的同源基因都参与某一目标性状的遗传调控,有效排除了基因功能的冗余。
本文初步鉴定到的与大豆含硫氨基酸合成相关酶基因,所在的QTL均可解释大豆含硫氨基酸的部分遗传变异。Laxman等[27]发现合成储藏蛋白时,游离含硫氨基酸是重要的影响因子,当含硫氨基酸缺乏时,tRNA硫醇化受阻,减少储藏蛋白的翻译,进而减少储藏含硫氨基酸组分。SAT是含硫氨基酸合成途径的第一个关键酶。Tabe等[28]在羽扇豆(Lupinus angustifolius L.)中过表达拟南芥的SAT基因,结果表明O-乙酰丝氨酸的含量增加了5倍,游离含硫氨基酸增加了26倍,但储藏含硫氨基酸含量无变化。Nguyen等[29]在水稻中异位表达大肠杆菌的SAT基因,研究表明游离甲硫氨酸在叶片中增加2.7倍,在籽粒中增加 1.4倍,但储藏甲硫氨酸含量无变化。Matityahu等利用种子特异启动子在烟草中高丰度表达拟南芥CGS基因,结果烟草种子中游离甲硫氨酸、脯氨酸、丝氨酸含量增加[30]。Song等[31]将拟南芥的 CGS基因(AtD-CGS)转化到大豆品种自贡冬豆中,3个转基因品系的游离含硫氨基酸增加了 3.8~7倍,其中有 2个品系储藏含硫氨基酸分别增加1.8和2.3倍。上述研究表明储藏含硫氨基酸由多基因调控,通过表达个别基因通常对提高含硫氨基酸组分的效果不显著。Tabe等[32]在种子成熟前期,检测到多个含硫氨基酸合成途径酶基因高水平表达。Liao等[33]在2个菜豆(Phaseolus vulgaris L.)品系中,对 10个含硫氨基酸代谢途径酶基因进行了表达差异分析,明晰了10个酶基因的转录模式,初步构建了多基因遗传调控网络。本研究得到的 100775420与100808736定位于同一半胱氨酸QTL内,前者编码CS,后者编码MGL。MGL基因转录水平与CS基因转录水平呈负相关,进而影响半胱氨酸的含量[34]。本研究得到的12个与大豆含硫氨基酸合成相关的酶基因均存在 SNP,表明这些基因可能存在等位基因,因此可以利用这些基因进行候选基因关联分析,开发功能标记,为大豆分子辅助及设计育种奠定基础。
[1] Friedman M, Brandon DL. Nutritional and health benefits of soy proteins. J Agric Food Chem, 2001, 49(3): 1069-1086.
[2] George AA, Lumen BO. A novel methionine-rich protein in soybean seed: Identification, amino acid composition, and N- terminal sequence. J Agric Food Chem, 1991, 39(1): 224-227.
[3] Clarke EJ, Wiseman J. Developments in plant breeding for improved nutritional quality of soya beans I. protein and amino acid content. J Agric Sci, 2000, 134(2): 111-124.
[4] Ufaz S, Galili G. Improving the content of essential amino acids in crop plants: goals and opportunities. Plant Physiol, 2008, 147(3): 954-961.
[5] Krishnan HB. Engineering soybean for enhanced sulfur amino acid content. Crop Sci, 2005, 45(2): 454-461.
[6] Yi H, Ravilious GE, Galant A, Krishnan HB, Jez JM. From sulfur to homoglutathione: thiol metabolism in soybean. Amino Acids, 2010, 39(4): 963-978.
[7] Bick JA, Leustek T. Plant sulfur metabolism-the reduction of sulfate to sulfite. Curr Opin Plant Biol, 1998, 1(3): 240-244.
[8] Ravina CG, Chang CI, Tsakraklides GP, Mcdermott JP, Vega JM, Leustek T, Gotor C, Davies JP. The sac mutants of chlamydomonas reinhardtii reveal transcriptional and posttranscriptional control of cysteine biosynthesis. Plant Physiol, 2002, 130(4): 2076-2084.
[9] Kopriva S. Regulation of sulfate assimilation in Arabidopsis and beyond. Ann Bot, 2006, 97(4): 479-495.
[10] Hirel B, Bertin P, Quilleré I, Bourdoncle W, Attagnant C, Dellay C, Gouy A, Cadiou S, Retailliau C, Falque M, Gallais A. Towards a better understanding of the genetic and physiological basis for nitrogen use efficiency in maize. Plant Physiol, 2001, 125(3): 1258-1270.
[11] Gallais A, Hirel B. An approach to the genetics of nitrogen use efficiency in maize. J Exp Bot, 2004, 55(396): 295-306.
[12] Schmutz J, Cannon SB, Schlueter J, Ma J X, Mitros T, Nelson W, Hyten DL, Song QJ, Thelen JJ, Cheng JL, Xu D, Hellsten U, May GD, Yu Y, Sakurai T, Umezawa T, Bhattacharyya MK, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu SQ, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du JC, Tian ZX, Zhu LC, Gill N, Joshi T, Libault M, Sethuraman A, Zhang XC, Shinozaki K, Nguyen HT, Wing RA, Cregan P, Specht J, Grimwood J, Rokhsar D, Stacey G, Shoemaker RC, Jackson SA. Genome sequence of the palaeopolyploid soybean. Nature, 2010, 463(7278): 178-183.
[13] 于妍, 姜威, 唐敬仙, 刘春燕, 陈庆山, 胡国华. 大豆天冬氨酸代谢途径关键酶基因电子定位与结构分析. 大豆科学, 2010, 29(1): 22-27.
[14] Panthee DR, Pantalone VR, Sams CE, Saxton AM, West DR, Orf JH, Killam AS. Quantitative trait loci controlling sulfur containing amino acids, methionine and cysteine, in soybean seeds. Theor Appl Genet, 2006, 112(3): 546-553.
[15] Carlson CM. Genetic control of protein and amino acid content in soybean determined in two genetically connected populations[dissertation]. Raleigh, N.C: North Carolina State University, 2011: 1-177.
[16] Fallen BD, Hatcher CN, Allen FL, Kopsell DA, Saxton AM, Chen P, Kantartzi SK, Cregan PB, Hyten DL, Pantalone VR. Soybean seed amino acid content QTL detected using the universal Soy Linkage Panel 1. 0 with 1, 536 SNPs. J Plant Geno Sci, 2013, 1(3): 68-79.
[17] Hyten, DL, Choi IY, Song QJ, Specht JE, Carter TE, Shoemaker RC, Hwang EY, Matukumalli L K, Cregan PB. A high density integrated genetic linkage map of soybean and the development of a 1536 universal Soy linkage panel for quantitative trait locus mapping. Crop Sci, 2010, 50(3): 960-968.
[18] He LL, Zhao M, Wang Y, Gai JY, He CY. Phylogeny, structural evolution and functional diversification of the plant PHOSPHATE1 gene family: a focus on Glycine max. BMC Evol Biol, 2013, 13: 103.
[19] Wirtz M, Hell R. Functional analysis of the cysteine synthase protein complex from plants: structural, biochemical and regulatory properties. J Plant Physiol, 2006, 163(3): 273-286.
[20] Kopriva S, Mugford SG, Matthewman C, Koprivova A. Plant sulfate as similation genes: redundancy versus specialization. Plant Cell Rep, 2009, 28(12): 1769-1780.
[21] Goyer A, Collakova E, Shachar-Hill Y, Hanson AD. Functional characterization of a methionine gamma-lyase in Arabidopsis and its implication in an alternative to the reverse trans-sulfuration pathway. Plant Cell Physiol, 2007,48(2), 232-242.
[22] Joshi V, Jander G. Arabidopsis methionine γ-lyase is regulated according to isoleucine biosynthesis needs but plays a subordinate role to threonine deaminase. Plant Physiol, 2009, 151(1): 367-378.
[23] Yamagami T, Tsuehisaka A, Yamada K, Haddon WF, Harden LA, Theologis A. Bioehemical diversity among the 1-amino-cyclopropane-1-carboxylate synthase isozymes eneoded by the Arabidopsis gene family. J Biol Chem, 2003, 278(49): 49102-49112.
[24] Moriya Y, Itoh M, Okuda S, Yoshizawa AC, Kanehisa M. KAAS: an automatic genome annotation and pathway reconstruction server. Nucleic Acids Res, 2007, 35(Web Server issue): W182-W185.
[25] Ye CY, Li T, Yin HF, Weston DJ, Tuskan GA, Tschaplinski TJ, Yang XH. Evolutionary analyses of non-family genes in plants. Plant J, 2013, 73(5): 788-797.
[26] 王晓波, 滕婉, 何雪, 童依平. 大豆谷氨酰胺合成酶基因的分类及根瘤特异表达GmGS1β2基因功能的初步分析. 作物学报, 2013, 39(12): 2145-2153.
[27] Laxman S, Sutter BM, Wu X, Kumar S, Guo XF, Trudgian DC, Mirzaei H, Tu BP. Sulfur amino acids regulate translational capacity and metabolic homeostasis through modulation of tRNA thiolation. Cell, 2013, 154(2): 416-429.
[28] Tabe L, Wirtz M, Molvig L, Droux M, Hell R. Overexpression of serine acetlytransferase produced large increases in Ο-acetylserine and free cysteine in developing seeds of a grain legume. J Exp Bot, 2010, 61(3): 721-733.
[29] Nguyen HC, Hoefgen R, Hesse H. Improving the nutritive value of rice seeds: elevation of cysteine and methionine contents in rice plants by ectopic expression of a bacterial serine acetyltransferase. J Exp Bot, 2012, 63(16): 5991-6001.
[30] Matityahu I, Godo I, Hacham Y, Amir R. Tobacco seeds expressing feedback-insensitive cystathionine gamma-synthase exhibit elevated content of methionine and altered primary metabolic profile. BMC Plant Biol, 2013, 13: 206.
[31] Song SK, Hou WS, Godo I, Wu CX, Yu Y, Matityahu I, Hacham Y, Sun S, Han TF, Amir R. Soybean seeds expressing feedback-insensitive cystathionine γ-synthase exhibit a higher content of methionine. J Exp Bot, 2013, 64(7): 1917-1926.
[32] Tabe L, Popelka C, Chiaiese P, Higgins TJV. Manipulating the sulfur composition of seeds. In: Sulfur Metabolism in Plants. Proceedings of the International Plant Sulfur Workshop. Netherlands: Springer, Vol. 1. 2012: 35-45.
[33] Liao D, Pajak A, Karcz SR, Chapman P, Sharpe AG, Austin RS, Datla R, Dhaubhadel S, Marsolais F. Transcripts of sulphur metabolic genes are co-ordinately regulated in developing seeds of common bean lacking phaseolin and major lectins. J Exp Bot, 2012, 63(17): 6283-6295.
[34] Liao D, Cram D, Sharpe AG, Marsolais F. Transcriptome profiling identifies candidate genes associated with the accumulation of distinct sulfur γ-glutamyl dipeptides in Phaseolus vulgaris and Vigna mungo seeds. Front Plant Sci, 2013, 4: 60.
(责任编委: 张红生)
Gene mining of sulfur-containing amino acid metabolic enzymes in soybean
Hongmei Qiu, Wenyuan Hao, Shuqin Gao, Xiaoping Ma, Yuhong Zheng, Fanfan Meng, Xuhong Fan, Yang Wang, Yueqiang Wang, Shuming Wang
National Engineering Research Center for Soybean, Soybean Research Institute, Jilin Academy of Agricultural Sciences, Changchun 130033, China
The genes of sulfur-containing amino acid synthetases in soybean are essential for the synthesis of sulfur-containing amino acids. Gene mining of these enzymes is the basis for the molecular assistant breeding of high sulfur-containing amino acids in soybean. In this study, using software BioMercator2.1, 113 genes of sulfur-containing amino acid enzymes and 33 QTLs controlling the sulfur-containing amino acids content were mapped onto Consensus Map 4.0, whichwas integrated by genetic and physical maps of soybean. Sixteen candidate genes associated to the synthesis of sulfur-containing amino acids were screened based on the synteny between gene loci and QTLs, and the effect values of QTLs. Through a bioinformatic analysis of the copy number, SNP information, and expression profile of candidate genes, 12 related enzyme genes were identified and mapped on 8 linkage groups, such as D1a, M, A2, K, and G. The genes corresponding to QTL regions can explain 6%-38.5% genetic variation of sulfur-containing amino acids, and among them, the indirect effect values of 9 genes were more than 10%. These 12 genes were involved in sulfur-containing amino acid metabolism and were highly expressed in the cotyledons and flowers, showing an abundance of SNPs. These genes can be used as candidate genes for the development of functional markers, and it will lay a foundation for molecular design breeding in soybean.
soybean(Glycine max); sulfur-containing amino acids; synthetic pathways; enzyme genes; bioinformatics
2014-01-14;
2014-05-06
国家高技术研究发展计划(863计划)项目(编号:2012AA101106),现代农业产业技术体系项目(编号:CARS-04-PS11),吉林省创新工程项目(编号:C32120402),吉林省自然科学基金项目(编号:20140101142JC)和东北农业大学大豆生物学教育部重点实验室开放基金项目(编号:SB12A02)资助
邱红梅,硕士研究生,专业方向:大豆种质资源保存与创新。Tel: 0431-87063237;E-mail: qhm2001-2005@163.com;
郝文媛,博士研究生,专业方向:大豆基因资源发掘与利用。E-mail: wenyuan_h@163.com
邱红梅和郝文媛同为第一作者。
王曙明,博士,研究员,研究方向:大豆分子遗传育种。E-mail: shumingw@263.net
10.3724/SP.J.1005.2014.0934
时间: 2014-8-12 14:17:59
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140812.1417.002.html
