植物NBS类抗病基因的进化
2014-05-25罗莎
罗莎
江西农业大学农学院园艺系,南昌 330045
植物NBS类抗病基因的进化
罗莎
江西农业大学农学院园艺系,南昌 330045
NBS(Nucleotide-binding site)类抗病基因是植物中最重要的一类抗病基因,其进化模式、结构特点和功能调控一直是抗病基因研究领域的热点。这类基因具有保守的结构域,广泛存在于植物基因组中,在不同植物基因组中数目差异较大且具有较低的表达量。此外,同源 NBS类抗病基因之间通过频繁的序列交换产生广泛的序列多样性,且抗病基因位点具有较差的线性。依据基因之间序列交换的频率,抗病基因可分为TypeⅠ和TypeⅡ两类。文章从抗病基因的结构、数量、分布、序列多样性、进化模式以及表达调控等方面进行了综述,旨在为后续NBS类抗病基因的相关研究提供参考。
NBS类抗病基因;进化模式;序列多样性;线性关系
植物在抵御病原物侵染的过程中进化出一系列防卫机制。其中,最关键的是由抗病基因介导的抗病性[1]。庄军等[2]对已克隆的抗病基因结构进行了较为详细的介绍,其中最重要的抗病基因是具有核苷酸结合位点(Nucleotide-binding site, NBS)结构域的一类基因,称为 NBS类抗病基因(以下简称抗病基因)。它们通过编码抗病蛋白识别病原物中的无毒蛋白来抵抗病原物,这种识别即基因对基因假说[3]。抗病基因在不同植物基因组中数量差异较大,且分布不均匀。随着多种植物全基因组测序的完成,抗病基因比较研究从以往相关物种的少数位点转变为全基因组的比较。通过全基因组抗病基因位点比较研究,发现抗病基因位点相对基因组内其他基因位点具有较差的线性,造成这一现象的原因可能是抗病基因之间频繁的序列交换以及抗病基因位点附近转座元件造成。抗病基因的复制、重组和点突变,使抗病基因产生丰富的序列多样性。同一家族中不同的抗病基因可能具有完全不同的进化方式和进化速度[4]。迄今为止,抗病基因进化差异的机制尚不清楚。
1 NBS类抗病基因的结构、数量及分布
大部分已克隆的NBS类抗病基因属于NBS-LRR类,这类基因在NBS结构域的C端包含一个富亮氨酸重复(Leucine rich repeat,LRR)的结构域。依据NBS-LRR类基因所编码蛋白的 N端结构,又分为TIR(Toll interleukin receptor)类和CC(Coiled-coil)两类。在单子叶植物中仅发现CC-NBS类基因,而大部分双子叶植物中这两类NBS类基因均存在[5,6]。研究显示,TIR-NBS类基因最早出现在苔藓类植物中,而非TIR-NBS类基因最早发现于晚于苔藓植物五百万年的石松类植物中,所以TIR-NBS类基因的出现早于非TIR-NBS类基因[7]。
随着多种植物全基因组序列的公布,抗病基因被相继鉴定。双子叶植物拟南芥(Arabidopsis thaliana)、葡萄(Vitis vinifera)、杨树(Populus trichocarpa)、苹果(Malus pumila)、甜橙(Citrus sinensis)、苜蓿(Medicago truncatula)、马铃薯(Solanum tuberosum)、黄瓜(Cucumis sativus L.)、甜瓜(Cucumis melo L.)和西瓜(Citrullus lanatus)基因组中分别鉴定到150、459、330、58、333、408、70、75和55个NBS类基因[8~14]。单子叶植物中也有大量NBS类基因存在,水稻两个栽培种(日本晴(Oryza sativa L. japonica)和 93-11 (Oryza sativa L. indica))、小麦(Triticum aestivum)、玉米(Zea mays)、高粱(Sorghum vulgare)和短柄草(Brachypodium distachyon)中分别鉴定到623、725、1219、129、345和239个NBS类基因[15~17]。由此可见,NBS类抗病基因广泛存在于植物基因组,且属于一个大的基因家族。但是,不同物种中NBS类基因数量差异较大,基因数量的多少与基因组大小并没有必然的联系。如:葡萄(475 Mb)、杨树(480 Mb)和水稻(389 Mb)中NBS类基因数量分别达到459、 330和大于600个[2,10,16,18,19],而番木瓜(Carica papaya linnaeus,372 Mb)、黄瓜(367 Mb)、西瓜(425 Mb)和甜瓜(450 Mb)基因组中仅有55、61、44和81个NBS类基因[20~23]。研究表明,葫芦科基因组中NBS类抗病基因数量少的原因可能是某些抗病基因世系(Lineages)的丢失以及现存的抗病基因世系缺少复制[11]。此外,NBS类基因在上述基因组中数量减少,以及出现大量假基因的现象,都可能与抗病基因大量存在的适合度代价(Fitness costs)有关,如大量抗病基因的存在会降低植物活力以及种子产量等[24,25]。不同基因组抗病基因数量差异现象出现的具体机制仍有待进一步深入研究。
NBS类抗病基因大都以多个基因串联的方式在染色体上成簇分布,这种簇的形式有利于抗病基因之间进行序列交换,从而产生更多新的嵌合体基因[26~29]。拟南芥和水稻中分别有66%和76%的NBS类抗病基因位于基因簇中,这种结构大部分是由基因复制和重组产生[9,16]。此外,NBS类抗病基因在基因组中并不是均匀地分布,而是集中分布在某几条染色体上,这种现象普遍存在于拟南芥、水稻、杨树和甘蓝(Brassica oleracea)中[10,30,31]。
2 NBS类抗病基因位点的线性关系
在各类植物全基因组信息缺乏时,抗病基因的进化研究都是基于相关物种的少数位点。随着新的植物基因组序列的公布,基于全基因组的NBS类抗病基因比较研究相继开展。尽管水稻、大麦(Hordeum vulgare L.)和粟(Pennisetum glaucum )等禾本科作物之间在全基因组染色体水平上呈现较好的线性关系,但是这些物种间抗病基因位点的线性较差[16,32]。花生(Arachis hypogaea)、大豆(Giycine max)、苜蓿、拟南芥和葡萄的抗病基因位点同样具有较低的线性[33]。Yang等[34]对两个已测序的水稻亚种日本晴和 93-11基因组中NBS类抗病基因进行了比较研究,发现虽然NBS类抗病基因在这两个基因组中数量不同,但大部分抗病基因在两个基因型中仍然比较保守。其中341对基因在日本晴和93-11中具有等位关系,基因之间的平均多样性(Average diversity, π)为1.52%,包括 171对具有明显等位关系的单基因位点,以及170对具有多个基因的基因对。本课题组研究发现,水稻中420对抗病基因在日本晴和93-11中存在一一对应的关系,基因之间平均核苷酸一致性(Nucleotide identity)高达 99.45%;同时,部分抗病基因仅存在于一个水稻亚种中,在另一个水稻亚种中找不到对应的等位基因,表现出存在/丢失(Presence/absence)多态性。日本晴和93-11中共有124个位点具有存在/丢失多态性,造成这种多态性出现的原因可能是抗病基因位点的非等位交换、丢失位点同源修复或非同源修复等事件以及大量基因转换[16]。这类存在/丢失多样性同样广泛存在于拟南芥和葫芦科的抗病基因位点[11,35]。抗病基因位点的较低线性以及广泛的存在/丢失多样性在一定程度上阻碍了in silico克隆方法在抗病基因上的应用,单一的参考基因组不能完全代表基因组中抗病基因位点的信息,所以整合多物种的抗病基因位点的抗病基因图谱显得非常重要。本课题组整合了水稻、玉米、高粱以及短柄草中NBS类基因位点,构建了禾本科中较完整的抗病基因图谱,为禾本科抗病基因研究提供了丰富的资源[16]。
3 NBS类抗病基因的进化模式
多基因家族的进化模式一般分为两种:协同进化(Concerted evolution)和 Birth-and-death的进化模式。协同进化的特点是家族内所有基因之间会发生序列的交换,作为一个整体共同进化。而birth-anddeath的进化模式指家族内各成员之间不会有序列的交换,相对独立地进化[36]。抗病基因的进化也具有这两种进化模式。Kuang等[4]对生菜中抗霜霉病Dm3基因家族的研究显示,抗病基因家族内成员之间具有不同频率的序列交换,依据交换频率的不同,首次将抗病基因分为TypeⅠ和TypeⅡ两类。TypeⅠ基因同源体之间发生频繁的序列交换,通常以嵌合体的形式存在。在自然界中某一特定的Type Ⅰ基因嵌合体出现的频率很低,同源基因之间没有明显的等位关系,这一类基因的进化模式与协同进化相似(图1)。TypeⅠ基因的进化特点可能与同源基因之间大量的序列交换有关,研究表明抗病基因的序列交换大都通过同源基因之间的非等位交换(Unequal-crossover)和基因转换(Gene conversion)发生,变化的序列被选择保留,从而产生新的抗病基因[1,16,37]。玉米和水稻(日本晴)中的 Rp1位点均是通过非等位交换形成,我们发现水稻中克隆的抗稻瘟病基因Pi37也是由这个位点两个同源基因的非等位交换形成[37]。同源基因之间频繁的序列交换会使同源基因序列逐渐趋于同化,而研究显示抗病基因编码序列并未被同化,仅它们的内含子部分被同化,可能是多样性选择导致的结果[4]。与 TypeⅠ基因不同,TypeⅡ基因同源体之间很少发现序列交换,它们之间只有点突变和缺失突变的差异,同源基因之间有明显等位关系。这类基因的进化类似于birth-and-death的进化模式(图1)。TypeⅠ和 TypeⅡ这两类基因的分化同样出现在其他物种中,马铃薯抗晚疫病R3a基因家族、拟南芥Rpp8基因家族以及玉米抗锈病Rp1基因家族都存在这两类基因的分化[37~39]。迄今为止,这两类基因分化的具体机制仍不清楚。目前存在两种假说:一是结构假说,认为TypeⅡ基因不与其他基因发生序列交换是受其侧翼序列的影响;另一种是功能假说,认为TypeⅡ基因的保守是由于其具有重要的功能[4]。随着研究的深入,已有的证据均与功能假说相悖。首先,之前的研究中没有发现TypeⅡ基因在保证自己不变的前提下作为供体改变其他同源基因的序列;其次,某一个TypeⅡ基因一般在自然群体中出现的频率很高,但是它也可以在一个基因组中完全丢失;最后,许多TypeⅡ基因都是假基因,我们证明造成假基因的突变位点在某一物种的不同基因型甚至野生型中仍然非常保守[4,16]。此外,研究显示基因之间的距离也不是决定序列交换频率的因素,拟南芥Rpp8基因家族中相隔 2.2 Mb的同源体之间能检测到序列交换,但是位于同一个基因簇内相隔只有 4 kb的同源基因之间却没有序列交换[38]。我们对日本晴全基因组中TypeⅠ和TypeⅡ两类NBS类抗病基因侧翼序列的结构进行比较研究,结果显示TypeⅡ基因侧翼序列的 GC含量显著高于 TypeⅠ基因,但侧翼序列的 GC含量的差异是否是影响序列交换频率的关键因素还有待进一步研究[16]。
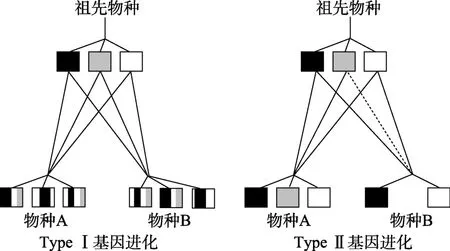
图1 NBS类抗病基因进化模式
4 NBS类抗病基因的多样性
抗病基因通过复制、不等交换和基因间序列交换产生基因多样性,棉花NBS类基因多样性便是由抗病基因的复制产生[40]。通过复制抗病基因拷贝数发生变化,生菜RGC2同源基因拷贝数在不同的生菜群体中具有较大差异,12~22个不等[41]。玉米Rp1基因在不同基因型中拷贝数也变化很大(1~50),这种拷贝数的变化可能是不等交换的结果[42]。玉米 Rp3位点在大部分基因型中具有至少 9个同源基因,而在B73中只有一个拷贝[43]。此外,通过RGC2位点的单倍型筛选,709个生菜个体中具有 366个不同的单倍型[41]。部分 TypeⅡ类 RGC2基因家族(K和L)的多样性与群体多样性不相符,这一类基因在一个群体或物种的不同基因组中如果存在则序列高度保守,同源基因间差异主要表现于拷贝数的变化以及单核苷酸突变,可能是由于TypeⅡ类基因承受着纯化选择的原因(Purifying selection);此外,这类基因也可能在群体某一个基因组中完全丢失,表现为存在/缺失多样性。我们发现转座元件的非等位交换还会导致抗病基因位点在不同基因型中出现的存在/丢失的多态性[16]。拟南芥生态型Ler-0中抗病基因RPS5的丢失是由于Ac-like转座子造成该位点序列发生断裂、修复所导致的[44]。另一部分 TypeⅠ类RGC2基因的多态性主要表现在嵌合体结构的广泛存在以及等位关系的模糊[41]。TypeⅠ类基因这种嵌合体结构是由同源基因之间频繁的序列交换造成的,这种频繁的序列交换使得这类基因内含子序列高度同化,但外显子并没有被同化,可能是由于序列交换之后多样性选择的结果[4]。综上所述,抗病基因具有更丰富的多态性以及大量的缺失,所以抗病基因的定位和注释也相应复杂。
5 NBS类抗病基因的调控
抗病基因的表达虽能诱导植物抗性,但大量抗病基因的高表达会导致植物细胞死亡,对植物细胞是致命的。为了减少植物的负担,抗病基因表达量通常较其他基因低[45,46]。最新研究显示,抗病基因的转录可能通过小RNA调控。两个miRNA(nta-miR6019和nta-miR6020)通过切割N基因转录本可降低其表达量[45]。豆科和茄科中 NBS-LRR基因的表达通过miRNA和siRNA调控,其中苜蓿基因组60%以上的NBS类基因能够与21个核苷酸的siRNA匹配[46,47]。这类小RNA对NBS类基因的转录调控是一种缓冲的作用,防止NBS类基因转录本的剧烈变化,同时NBS类基因转录本对小 RNA的形成也具有反向调控作用[48]。另一种原因可能是小 RNA通过对NBS类基因的抑制,有利于有益菌与植物之间的互作[48]。除对抗病基因的表达调控,还要另一种减少抗病基因对植物基因组负担的方式,许多抗病基因在长期没有对应病原的环境中逐渐进化为假基因甚至在基因组中丢失,从而减少能量的消耗[49]。因此,抗病基因通过各种调控既能保证植物基因组中保存大量具有多样性的抗病基因用以对抗不断变化的病原菌,同时又能减少大量基因表达对植物基因组的负担。
6 结 语
抗病基因与病原物之间存在协同进化的关系,为了适应病原物的快速变化,抗病基因也在不断进化。抗病基因多样性是由基因的复制、重组、突变以及突变后的自然选择产生。附近转座元件以及siRNA的存在也会影响抗病基因的数量、多样性以及基因表达。随着多种植物基因组的获得,越来越多NBS类基因被鉴定,抗病基因的进化研究也不仅仅局限于相关物种的少数位点。通过物种内、物种间抗病基因比较分析,抗病基因进化模式以及进化特点有了进一步的了解。抗病基因结构特征、序列多样性以及进化模式等特殊性,导致抗病基因的克隆相对于其他功能基因的克隆更为复杂和困难。传统的正向遗传学方法在克隆抗病基因的过程中存在一定的局限性。因此,对抗病基因的结构特征、序列多样性以及进化模式等方面的研究,不仅为开发适合抗病基因的克隆策略提供理论依据,而且为作物生产提供更好指导。但尚有一系列的问题亟待进一步明确,如:单子叶植物中为什么没有发现TIRNBS类基因,NBS类抗病基因数量在不同基因组间巨大差异的机制,TypeⅠ和TypeⅡ两类抗病基因分化的具体机制都是未来抗病基因进化研究的方向。此外,目前抗病基因进化研究仅限于单一或某一类抗病基因内进行,但抗病基因的进化并不是独立进行。因此,对整个抗病基因调控网络进化研究以及小RNA对抗病基因进化影响的研究也十分必要。
[1] Hulbert SH, Webb CA, Smith SM, Sun Q. Resistance gene complexes: Evolution and utilization. Annu Rev Phytopathol, 2001, 39: 285-312.
[2] 庄军, 刘志昕. 植物抗病基因的进化. 遗传, 2004, 26(6): 962-968.
[3] Flor HH. Current status of the gene-for-gene concept. Annu Rev Phytopathol, 1971, 9: 275-296.
[4] Kuang H, Woo SS, Meyers BC, Nevo E, Michelmore RW. Multiple genetic processes result in heterogeneous rates of evolution within the major cluster disease resistance genes in lettuce. Plant Cell, 2004, 16(11): 2870-2894.
[5] Hammond-Kosack KE, Jones JDG. Plant disease resistance genes. Annu Rev Plant Physiol Plant Mol Biol, 1997, 48: 575-607.
[6] Bai JF, Pennill LA, Ning JC, Lee SW, Ramalingam J, Webb CA, Zhao BY, Sun Q, Nelson JC, Leach JE, Hulbert SH. Diversity in nucleotide binding site-leucine-rich repeat genes in cereals. Genome Res, 2002, 12(12): 1871-1884.
[7] Yue JX, Meyers BC, Chen JQ, Tian DC, Yang SH. Tracing the origin and evolutionary history of plant nucleotide-binding site-leucine-rich repeat (nbs-lrr) genes. New Phytol, 2012, 193(4): 1049-1063.
[8] Ameline-Torregrosa C, Wang BB, O'Bleness MS, Deshpande S, Zhu H, Roe B, Young ND, Cannon SB. Identification and characterization of nucleotide-binding site-leucinerich repeat genes in the model plant Medicago truncatula. Plant Physiol, 2008, 146(1): 5-21.
[9] Meyers BC, Kozik A, Griego A, Kuang H, Michelmore RW. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis. Plant Cell, 2003, 15(4): 809-834.
[10] Yang SH, Zhang XH, Yue JX, Tian DC, Chen JQ. Recent duplications dominate NBS-encoding gene expansion in two woody species. Mol Genet Genomics, 2008, 280(3): 187-198.
[11] Lin X, Zhang Y, Kuang H, Chen JJ. Frequent loss of lineages and deficient duplications accounted for low copy number of disease resistance genes in Cucurbitaceae. BMC Genomics, 2013, 14: 335.
[12] Xu X, Pan SK, Cheng SF, Zhang B, Mu DS, Ni PX, Zhang GU, Yang S, Li RQ, Wang J, Orjeda G, Guzman F, Torres M, Lozano R, Ponce O, Martinez D, De la Cruz G, Chakrabarti SK, Patil VU, Skryabin KG, Kuznetsov BB, Ravin NV, Kolganova TV, Beletsky AV, Mardanov AV, Di Genova A, Bolser DM, Martin DMA, Li GC, Yang Y, Kuang H, Hu Q, Xiong XY, Bishop GJ, Sagredo B, Mejía N, Zagorski W, Gromadka R, Gawor J, Szczesny P, Huang S, Zhang ZH, Liang CB, He J, Li Y, He Y, Xu JF, Zhang YJ, Xie BY, Du YC, Qu DY, Bonierbale M, Ghislain M, Herrera Mdel R, Giuliano G, Pietrella M, Perrotta G, Facella P, O'Brien K, Feingold SE, Barreiro LE, Massa GA, Diambra L, Whitty BR, Vaillancourt B, Lin H, Massa AN, Geoffroy M, Lundback S, DellaPenna D, Buell CR, Sharma SK, Marshall DF, Waugh R, Bryan GJ, Destefanis M, Nagy I, Milbourne D, Thomson SJ, Fiers M, Jacobs JM, Nielsen KL, Sønderkaer M, Iovene M, Torres GA, Jiang JM, Veilleux RE, Bachem CWB, de Boer J, Borm T, Kloosterman B, van Eck H, Datema E, Hekkert BL, Goverse A, van Ham RCHJ, Visser RGF. Genome sequence and analysis of the tuber crop potato. Nature, 2011, 475(7355): 189-195.
[13] Xu Q, Chen LL, Ruan XA, Chen DJ, Zhu DA, Chen CL, Bertrand D, Jiao WB, Hao BH, Lyon MP, Chen JJ, Gao S, Xing F, Lan H, Chang JW, Ge XH, Lei Y, Hu Q, Miao Y, Wang L, Xiao SX, Biswas MK, Zeng WF, Guo F, Cao HB, Yang XM, Xu XW, Cheng YJ, Xu J, Liu JH, Luo OJ, Tang ZH, Guo WW, Kuang H, Zhang HY, Roose ML, Nagarajan N, Deng XX, Ruan YJ. The draft genome of sweet orange (citrus sinensis). Nat Genet, 2013, 45(1): 59-66.
[14] Kohler A, Rinaldi C, Duplessis S, Baucher M, Geelen D, Duchaussoy F, Meyers BC, Boerjan W, Martin F. Genome-wide identification of nbs resistance genes in Populus trichocarpa. Plant Mol Biol, 2008, 66(6): 619-636.
[15] Li J, Ding J, Zhang W, Zhang YL, Tang P, Chen JQ, Tian DC, Yang SH. Unique evolutionary pattern of numbers of gramineous NBS-LRR genes. Mol Genet Genomics, 2010, 283(5): 427-438.
[16] Luo S, Zhang Y, Hu Q, Chen JJ, Li KP, Lu C, Liu H, Wang W, Kuang H. Dynamic nucleotide-binding site and leucinerich repeat-encoding genes in the grass family. Plant Physiol, 2012, 159(1): 197-210.
[17] Jia JZ, Zhao SC, Kong XY, Li YR, Zhao GY, He WM, Appels R, Pfeifer M, Tao Y, Zhang XY, Jing RL, Zhang C, Ma YZ, Gao LF, Gao C, Spannagl M, Mayer KF, Li D, Pan SK, Zheng FY, Hu Q, Xia XC, Li JW, Liang QS, Chen J, Wicker T, Gou CY, Kuang H, He GJ, Luo YD, Keller B, Xia QJ, Lu P, Wang JY, Zou HF, Zhang R, Xu J, Gao J, Middleton C, Quan ZW, Liu GM, Yang HM, Liu X, He ZH, Mao L, Wang J. Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation. Nature, 2013, 496(7443): 91-95.
[18] Tuskan GA, Difazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A, Schein J, Sterck L, Aerts A, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W, Brun A, Brunner A, Busov V, Campbell M, Carlson J, Chalot M, Chapman J, Chen GL, Cooper D, Coutinho PM, Couturier J, Covert S, Cronk Q,Cunningham R, Davis J, Degroeve S, Déjardin A, Depamphilis C, Detter J, Dirks B, Dubchak I, Duplessis S, Ehlting J, Ellis B, Gendler K, Goodstein D, Gribskov M, Grimwood J, Groover A, Gunter L, Hamberger B, Heinze B, Helariutta Y, Henrissat B, Holligan D, Holt R, Huang W, Islam-Faridi N, Jones S, Jones-Rhoades M, Jorgensen R, Joshi C, Kangasjärvi J, Karlsson J, Kelleher C, Kirkpatrick R, Kirst M, Kohler A, Kalluri U, Larimer F, Leebens-Mack J, Leplé JC, Locascio P, Lou Y, Lucas S, Martin F, Montanini B, Napoli C, Nelson DR, Nelson C, Nieminen K, Nilsson O, Pereda V, Peter G, Philippe R, Pilate G, Poliakov A, Razumovskaya J, Richardson P, Rinaldi C, Ritland K, Rouzé P, Ryaboy D, Schmutz J, Schrader J, Segerman B, Shin H, Siddiqui A, Sterky F, Terry A, Tsai CJ, Uberbacher E, Unneberg P, Vahala J, Wall K, Wessler S, Yang G, Yin T, Douglas C, Marra M, Sandberg G, Van de Peer Y, Rokhsar D. The genome of black cottonwood, Populus trichocarpa (torr. & gray). Science, 2006, 313(5793): 1596-1604.
[19] Jaillon O, Aury JM, Noel B, Policriti A, Clepet C, Casagrande A, Choisne N, Aubourg S, Vitulo N, Jubin C, Vezzi A, Legeai F, Hugueney P, Dasilva C, Horner D, Mica E, Jublot D, Poulain J, Bruyère C, Billault A, Segurens B, Gouyvenoux M, Ugarte E, Cattonaro F, Anthouard V, Vico V, Del Fabbro C, Alaux M, Di Gaspero G, Dumas V, Felice N, Paillard S, Juman I, Moroldo M, Scalabrin S, Canaguier A, Le Clainche I, Malacrida G, Durand E, Pesole G, Laucou V, Chatelet P, Merdinoglu D, Delledonne M, Pezzotti M, Lecharny A, Scarpelli C, Artiguenave F, Pe ME, Valle G, Morgante M, Caboche M, Adam-Blondon AF, Weissenbach J, Quétier F, Wincker P. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature, 2007, 449(7161): 463-467.
[20] Ming R, Hou SB, Feng Y, Yu QY, Dionne-Laporte A, Saw JH, Senin P, Wang W, Ly BV, Lewis KL, Salzberg SL, Feng L, Jones MR, Skelton RL, Murray JE, Chen C, Qian W, Shen J, Du P, Eustice M, Tong E, Tang H, Lyons E, Paull RE, Michael TP, Wall K, Rice DW, Albert H, Wang ML, Zhu YJ, Schatz M, Nagarajan N, Acob RA, Guan P, Blas A, Wai CM, Ackerman CM, Ren Y, Liu C, Wang J, Na JK, Shakirov EV, Haas B, Thimmapuram J, Nelson D, Wang XY, Bowers JE, Gschwend AR, Delcher AL, Singh R, Suzuki JY, Tripathi S, Neupane K, Wei HR, Irikura B, Paidi M, Jiang N, Zhang WL, Presting G, Windsor A, Navajas-Perez R, Torres MJ, Feltus FA, Porter B, Li YJ, Burroughs AM, Luo MC, Liu L, Christopher DA, Mount SM, Moore PH, Sugimura T, Jiang JM, Schuler MA, Friedman V, Mitchell-Olds T, Shippen DE, dePamphilis CW, Palmer JD, Freeling M, Paterson AH, Gonsalves D, Wang L, Alam M. The draft genome of the transgenic tropical fruit tree papaya (Carica papaya linnaeus). Nature, 2008, 452(7190): 991-996.
[21] Huang SW, Li RQ, Zhang ZH, Li L, Gu XF, Fan W, Lucas WJ, Wang XW, Xie BY, Ni PX, Ren YY, Zhu HM, Li J, Lin K, Jin WW, Fei ZJ, Li GC, Staub J, Kilian A, van der Vossen EA, Wu Y, Guo J, He J, Jia ZQ, Ren Y, Tian G, Lu Y, Ruan J, Qian WB, Wang MW, Huang QF, Li B, Xuan ZL, Cao JJ, Asan, Wu ZG, Zhang JB, Cai QL, Bai YQ, Zhao BW, Han YH, Li Y, Li XF, Wang SH, Shi QX, Liu SQ, Cho WK, Kim JY, Xu Y, Heller-Uszynska K, Miao H, Cheng ZC, Zhang SP, Wu J, Yang YY, Kang HX, Li M, Liang HQ, Ren XL, Shi ZB, Wen M, Jian M, Yang HL, Zhang GJ, Yang ZT, Chen R, Ma LJ, Liu H, Zhou Y, Zhao J, Fang XD, Fang L, Liu DY, Zheng HK, Zhang Y, Qin N, Li Z, Yang GH, Yang S, Bolund L, Kristiansen K, Li SC, Zhang XQ, Wang J, Sun RF, Zhang BX, Jiang SZ, Du YC. The genome of the cucumber, Cucumis sativus L. Nat Genet, 2009, 41(12): 1275-1281.
[22] Garcia-Mas J, Benjak A, Sanseverino W, Bourgeois M, Mir G, Gonzalez VM, Hénaff E, Câmara F, Cozzuto L, Lowy E, Alioto T, Capella-Gutiérrez S, Blanca J, Cañizares J, Ziarsolo P, Gonzalez-Ibeas D, Rodríguez-Moreno L, Droege M, Du L, Alvarez-Tejado M, Lorente-Galdos B, Melé M, Yang LM, Weng YQ, Navarro A, Marques-Bonet T, Aranda MA, Nuez F, Picó B, Gabaldón T, Roma G, Guigó R, Casacuberta JM, Arús P, Puigdomènech P. The genome of melon (Cucumis melo L.). Proc Natl Acad Sci USA, 2012, 109(29): 11872-11877.
[23] Guo SG, Zhang JG, Sun HH, Salse J, Lucas WJ, Zhang HY, Zheng Y, Mao LY, Ren Y, Wang ZW, Min JM, Guo XS, Murat F, Ham BK, Zhang ZL, Gao S, Huang MY, Xu YM, Zhong SL, Bombarely A, Mueller LA, Zhao H, He HJ, Zhang Y, Huang SW, Tan T, Pang E, Lin K, Hu Q, Kuang H, Ni PX, Wang B, Liu JG, Kou QH, Hou WJ, Zou XH, Jiang J, Gong GY, Klee K, Schoof H, Huang Y, Hu XS, Dong SS, Liang DQ, Wang J, Wu K, Xia Y, Zhao X, Zheng ZQ, Xing M, Liang XM, Huang BQ, Lv T, Yin Y, Yi HP, Li RQ, Wu MZ, Levi A, Zhang XP, Giovannoni JJ, Li YF, Fei ZJ, Xu Y. The draft genome of watermelon (Citrullus lanatus) and resequencing of 20 diverse accessions. Nat Genet, 2013, 45(1): 51-58.
[24] Brenchley R, Spannagl M, Pfeifer M, Barker GL, D'Amore R, Allen AM, McKenzie N, Kramer M, Kerhornou A, Bolser D, Kay S, Waite D, Trick M, Bancroft I, Gu Y, Huo NX, Luo MC, Sehgal S, Gill B, Kianian S, Anderson O, Kersey P, Dvorak J, McCombie WR, Hall A, Mayer KF, Edwards KJ, Bevan MW, Hall N. Analysis of the bread wheat genome using whole-genome shotgun sequencing. Nature, 2012, 491(7426): 705-710.
[25] Tian DC, Araki H, Stahl E, Bergelson J, Kreitman M. Signature of balancing selection in Arabidopsis. Proc NatlAcad Sci USA, 2002, 99(17): 11525-11530.
[26] Mondragón-Palomino M, Meyers BC, Michelmore RW, Gaut BS. Patterns of positive selection in the complete NBS-LRR gene family of Arabidopsis thaliana. Genome Res, 2002, 12(9): 1305-1315.
[27] Dodds PN, Lawrence GJ, Ellis JG. Contrasting modes of evolution acting on the complex N locus for rust resistance in flax. Plant J, 2001, 27(5): 439-453.
[28] Chen Q, Han Z, Jiang H, Tian D, Yang S. Strong positive selection drives rapid diversification of R-genes in Arabidopsis relatives. J Mol Evol, 2010, 70(2): 137-148.
[29] Ashfield T, Egan AN, Pfeil BE, Chen NW, Podicheti R, Ratnaparkhe MB, Ameline-Torregrosa C, Denny R, Cannon S, Doyle JJ, Geffroy V, Roe BA, Saghai Maroof MA, Young ND, Innes RW. Evolution of a complex disease resistance gene cluster in diploid Phaseolus and tetraploid Glycine. Plant Physiol, 2012, 159(1): 336-354.
[30] Zhou T, Wang Y, Chen JQ, Araki H, Jing Z, Jiang K, Shen J, Tian D. Genome-wide identification of NBS genes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes. Mol Genet Genomics, 2004, 271(4): 402-415.
[31] Yu JY, Tehrim S, Zhang FQ, Tong CB, Huang JY, Cheng XH, Dong CH, Zhou Y,Q Qin R, Hua W, Liu SY. Genome-wide comparative analysis of NBS-encoding genes between Brassica species and Arabidopsis Thaliana. BMC Genomics, 2014, 15: 3.
[32] Gale MD, Devos KM. Plant comparative genetics after 10 years. Science, 1998, 282(5389): 656-659.
[33] Ratnaparkhe MB, Wang XY, Li JP, Compton RO, Rainville LK, Lemke C, Kim C, Tang HB, Paterson AH. Comparative analysis of peanut NBS-LRR gene clusters suggests evolutionary innovation among duplicated domains and erosion of gene microsynteny. New Phytol, 2011, 192(1): 164-178.
[34] Yang S, Feng Z, Zhang X, Jiang K, Jin X, Hang Y, Chen JQ, Tian D. Genome-wide investigation on the genetic variations of rice disease resistance genes. Plant Mol Biol, 2006, 62(1-2): 181-193.
[35] Shen JD, Araki H, Chen LL, Chen JQ, Tian DC. Unique evolutionary mechanism in R-genes under the presence/absence polymorphism in Arabidopsis thaliana. Genetics, 2006, 172(2): 1243-1250.
[36] Nei M, Rooney AP. Concerted and birth-and-death evolution of multigene families. Annu Rev Genet, 2005, 39: 121-152.
[37] Luo S, Peng J, Li K, Wang M, Kuang H. Contrasting evolutionary patterns of the Rp1 resistance gene family in different species of poaceae. Mol Biol Evol, 2011, 28(1): 313-325.
[38] Kuang H, Caldwell KS, Meyers BC, Michelmore RW. Frequent sequence exchanges between homologs of Rpp8 in Arabidopsis are not necessarily associated with genomic proximity. Plant J, 2008, 54(1): 69-80.
[39] Huang SW, van der Vossen EA, Kuang H, Vleeshouwers VGAA, Zhang NW, Borm TJA, van Eck HJ, Baker B, Jacobsen E, Visser RGF. Comparative genomics enabled the isolation of the R3a late blight resistance gene in potato. Plant J, 2005, 42(2): 251-261.
[40] Wei HL, Li W, Sun XW, Zhu SJ, Zhu J. Systematic analysis and comparison of nucleotide-binding site disease resistance genes in a diploid cotton Gossypium raimondii. PLoS ONE, 2013, 8(8): e68435.
[41] Kuang H, van Eck HJ, Sicard D, Michelmore R, Nevo E. Evolution and genetic population structure of prickly lettuce (Lactuca serriola) and its RGC2 resistance gene cluster. Genetics, 2008, 178(3): 1547-1558.
[42] Smith SM, Pryor AJ, Hulbert SH. Allelic and haplotypic diversity at the Rp1 rust resistance locus of maize. Genetics, 2004, 167(4): 1939-1947.
[43] Webb CA, Richter TE, Collins NC, Nicolas M, Trick HN, Pryor T, Hulbert SH. Genetic and molecular characterization of the maize Rp3 rust resistance locus. Genetics, 2002, 162(1): 381-394.
[44] Henk AD, Warren RF, Innes RW. A new Ac-like transposon of Arabidopsis is associated with a deletion of the Rps5 disease resistance gene. Genetics, 1999, 151(4): 1581-1589.
[45] Li F, Pignatta D, Bendix C, Brunkard JO, Cohn MM, Tung J, Sun HY, Kumar P, Baker B. Microrna regulation of plant innate immune receptors. Proc Natl Acad Sci USA, 2012, 109(5): 1790-1795.
[46] Zhai JX, Jeong DH, De Paoli E, Park S, Rosen BD, Li YP, González AJ, Yan Z, Kitto SL, Grusak MA, Jackson SA, Stacey G, Cook DR, Green PJ, Sherrier DJ, Meyers BC. Micrornas as master regulators of the plant NB-LRR defense gene family via the production of phased, transacting sirnas. Genes Dev, 2011, 25(23): 2540-2553.
[47] Shivaprasad PV, Chen HM, Patel K, Bond DM, Santos BA, Baulcombe DC. A microrna superfamily regulates nucleotide binding site-leucine-rich repeats and other mRNAs. Plant Cell, 2012, 24(3): 859-874.
[48] Fei QL, Xia R, Meyers BC. Phased, secondary, small interfering rnas in posttranscriptional regulatory networks. Plant Cell, 2013, 25(7): 2400-2415.
[49] Tian D, Traw MB, Chen JQ, Kreitman M, Bergelson J. Fitness costs of r-gene-mediated resistance in Arabidopsis thaliana. Nature, 2003, 423(6935): 74-77.
(责任编委: 张根发)
Evolution of plant NBS encoding disease resistance genes
Sha Luo
Department of Horticulture, College of Agronomy, Jiangxi Agricultural University, Nanchang 330045, China
NBS (nucleotide-binding site) genes are a major class of disease resistance (R) genes in plants. Studies on their evolutionary pattern, structure characteristics and functional regulation have been always paid much attentions. NBS genes exist in a various plants by different copy numbers and low expression levels. They encode proteins containing conserved NBS domain and C-terminal leucine-rich repeats (LRRs). The NBS genes have frequent sequence exchanges among homologs and consequently show extensive diversity and poor synteny. Two types of NBS genes are distinguished based on their frequency of sequence exchanges. In this review, we summarize the latest progress of plant NBS encoding genes in terms of structure, number, evolutionary pattern, sequence diversity and distribution in genome, providing some insights into the further research on NBS genes in plant.
NBS encoding gene; evolutionary pattern; sequence diversity; synteny
2014-06-19;
2014-09-28
国家自然科学基金项目(编号:31360050)资助
罗莎,博士,讲师,研究方向:植物抗病基因进化。E-mail: rosalycake@163.com
10.3724/SP.J.1005.2014.1219
时间: 2014-10-8 10:03:44
URL: http://www.cnki.net/kcms/detail/11.1913.R.20141008.1003.002.html
