选择性反应监测技术在蛋白质组学研究中的进展及应用
2014-05-08单亦初张丽华张玉奎
单亦初, 张丽华, 张玉奎
(中国科学院大连化学物理研究所,中国科学院分离分析化学重点实验室,国家色谱研究分析中心,辽宁大连116023)
定量蛋白质组学研究受到越来越多的关注。其中,鸟枪法技术和SRM技术是定量蛋白质组学领域中应用最为广泛的两种技术。鸟枪法具有很高的分析通量,一次运行即可定量数千个蛋白质;但其缺点是无法对蛋白质的多肽信号进行连续性和选择性的检测,因此目标蛋白质信号容易受到噪音和其他非目标组分的干扰,检测灵敏度低,并且难以实现对低丰度肽段和蛋白质的准确定量。通常情况下,人们感兴趣的不同样品中的差异蛋白质以及疾病标志物的含量较低。选择性反应监测(SRM)技术尽管分析通量较低,但是该技术通过选择性检测特定母离子和子离子来排除非目标组分的干扰,增强了检测灵敏度和定量准确度,特别适合于低丰度蛋白质的准确定量。因此,近年来基于该技术研究疾病标志物及验证差异蛋白质等已成为定量蛋白质组学的重要策略之一。SRM技术通常使用三重四极杆质谱来实现,具体原理如图1所示[1]。本文对该技术的进展及其应用进行了综述,并对未来可能的发展方向进行了展望。

图1 选择性反应监测(SR M)的技术原理[1]Fig.1 Principle of selective reaction monitoring(SR M)technology[1]
1 SR M技术进展
SRM技术与传统的基于鸟枪法技术的蛋白质组定量方法相比,由于有效提高了对离子的选择性,在一定程度上提高了对中、低丰度蛋白质的检测能力。然而由于蛋白质组样品复杂度高、动态范围宽,进一步提高检测灵敏度仍然是一个重要的研究目标。由于SRM技术需要对肽段进行连续扫描,因而对复杂样品分析所需的时间较长,为此近年来研究者也致力于提高SRM技术的分析通量。此外,通过发展新方法提高蛋白质定量的准确度也是该领域的研究热点之一。最后,随着SRM技术逐渐应用于大规模的蛋白质组定量研究,相关数据的处理也变得越来越重要。为此人们也在不断发展相关的软件和数据库。本文从分析通量、检测灵敏度、定量方法以及相关软件资源4个方面,对近期SRM技术的研究进展进行了综述。
1.1 分析通量
基于质谱的SRM技术与传统的定向蛋白质组学技术(如Western blot)相比,分析通量有所提高,并且不受抗体的制约。然而为缩短生物标志物发现周期,需要改进数据采集或者处理方法,进一步提高SRM技术的分析通量。
Kiyonami等[2]率先提出了一种新的数据采集方法——智能选择反应监测(intelligent selected reaction monitoring,iSRM)。该方法通过设定主要和次要母子离子对,在预先设定的洗脱时间窗口内对主要母子离子对进行连续监测以准确定量每个多肽;此外,同时采用数据依赖模式监测6~8个次要离子对。当主要母子离子对信号强度达到一定阈值时,触发次要母子离子对的采集,实现对肽段的定性确认。以酵母的胰蛋白酶酶解产物的分析为例,其检测限低至10 amol水平,通量达到每分钟100母子离子对。该方法在保证SRM的选择性和准确性的前提下,可以在固定的采集时间内分析更多的肽段,从而提高了目标蛋白质组分析的选择性、准确度和通量。在此基础上,毛心丽等[3]建立了依赖色谱保留时间的iSRM方法,并对不同复杂程度的蛋白质样品进行了分析。该方法与不依赖色谱保留时间的iSRM方法相比,可以显著提高肽段及蛋白质的鉴定数目,目标肽段和蛋白质的鉴定数目分别从312和217提高到449和280。
近期AB Sciex公司推出了SWATH(sequential window acquisition of all theoretical fragment-ion spectra)技术。该技术采取非数据依赖采集方法,连续运行32个25 Da宽的母离子分离窗口,并对每个窗口内的母离子进行全碎裂。利用碎片离子谱图库来挖掘共碎裂多肽二级谱中特定多肽的特征碎片离子,进而实现了对多肽的同时鉴定和定量。Gillet等[4]通过实验验证发现该方法的定量动态范围可达到4个数量级。与SRM技术相比,两者在定量准确性及一致性方面相近,但SWATH技术可在一次运行中同时实现蛋白质样品的全谱鉴定和定量,具有更高的分析通量。
1.2 检测灵敏度
由于采取两次质量选择过滤,SRM技术有较高的灵敏度。与传统的基于质谱的蛋白质组学技术相比,SRM技术在分析复杂生物样本时为靶向式获取目标离子,减少了其他离子的干扰,因此与经典技术相比其灵敏度可提高一个数量级[1]。然而由于蛋白质组样品具有很宽的动态范围,对低丰度蛋白质的检测仍然存在挑战。为此,人们通过发展样品分级技术,如亲和富集、高丰度蛋白质去除以及高灵敏度的质谱接口等,进一步提高了SRM技术的灵敏度。
Whiteaker等[5]提出了 SISCAPA(stable isotope standards and capture by antipeptide antibodies)方法。采取多重免疫策略,使用一个蛋白质的至多5条肽段对动物进行免疫,从而产生相应的多肽抗体;然后利用抗体对目标蛋白质多肽进行富集,并结合稳定同位素稀释的方法,使用SRM技术进行质谱检测和定量。利用该方法可以检测到血浆中质量浓度低于100 ng/mL的蛋白质。
Hossain等[6]发展了一种多毛细管入口/双电动离子通道接口(multi-capillary inlet/dual electrodynamic ion funnel interface),并结合SRM 技术实现了蛋白质的定量分析。与传统的SRM接口相比,采用该接口可以通过提高离子传输效率,显著提高检测灵敏度。对于血浆蛋白质样品,不仅峰强度平均提高了70倍,检测限降低了10倍,而且还提高了分析的重现性,无须免疫去除高丰度蛋白质即可实现血浆中40~80 ng/mL蛋白质的检测。此外,Rafalko等[7]利用芯片等电聚焦对目标多肽进行选择性富集后,采用SRM技术对血浆中的前列腺特异性抗原进行了检测,其定量限可低至1~2.5 ng/mL。
1.3 SR M相关定量方法
SRM的定量分析从方法上讲一般分为相对定量分析和绝对定量分析,其中相对定量方法包括无标记和标记定量方法。最初通常使用无标记定量方法[8]进行SRM定量。为了消除液相色谱分离和质谱检测重复性不够高引起的定量误差,进一步提高定量的准确度和精密度,人们发展了多种同位素标记定量方法,包括代谢同位素标记方法(如[15N]硫酸铵标记)[9],化学同位素标记方法(如同位素编码的亲和标签标记(ICAT))[10]以及酶解辅助稳定同位素标记方法(如[18O]水标记)[11]。
Zhang等[12]比较了稳定同位素稀释法、标记参照多肽法及无标方法在蛋白质组定量方面的性能。结果表明,与其他两种方法相比稳定同位素稀释法具有较高的精密度;在识别蛋白质表达的显著性差异方面,3种方法具有相似的性能;与稳定同位素稀释法相比,标记参照多肽法及无标方法实验成本较低,具有较高的性价比。
在使用SRM技术进行绝对定量时,通常采用同位素标记的肽段作为内标,定量结果可能会受到样品处理过程的影响。为此,Huillet等[13]发展了标记蛋白质和SRM技术相结合的方法。在对心血管疾病生物标志蛋白质进行定量时,使用同位素标记的蛋白质作为内标,有效避免了上游样品处理或者不完全酶解引入的实验误差,提高了定量的准确度和精密度。
1.4 SR M相关软件和数据库
采用SRM技术时需要对目标肽段的母子离子对和仪器设置进行选择,以提高蛋白质定量的准确度和灵敏度。为此,人们发展了多种软件和数据库。
Reiter等[14]发展了 SRM 数据分析工具mProphet;通过准确计算目标多肽鉴定的错误率,以及将数据的相关特征引入统计模型来提高SRM技术的选择性和灵敏度,实现了对大规模SRM实验的自动化处理和统计学验证。Brusniak等[15]开发了开源软件ATAQS(Automated and Targeted Analysis with Quantitative SRM),可支持目标蛋白质组学研究流程中的所有步骤,并且提供了应用程序接口,允许在SRM流程的每一步骤加入新的算法。Cham等[16]基于从发表的文章中收集到的SRM 母子离子对开发了MRMaid在线数据库,从而节省了寻找母子离子对所需的时间。该数据库包含了重现母子离子对所需的一切信息,并通过简单的基于网络的数据表格提交母子离子对,使得研究者可以很容易地获取相关信息。Huttenhain等[17]建立了一个含有5 568个N-糖基化位点的数据库,应用于对临床相关N-糖基化蛋白质作为候选生物标志物的可行性进行多重评估,使用该数据库选择合适的SRM方法,对120个人血浆样品进行了癌症相关的N-糖基化蛋白质分析。结果表明,浓度范围跨度超过5个数量级的糖蛋白质可被定量,使用该数据库可以对候选生物标志物进行并行的、有效的、一致的和灵敏的评估。
2 SR M技术的应用
由于SRM技术具有高选择性、高灵敏度及高通量等优势,因此在生物标志物的验证、蛋白质翻译后修饰及生物工程等方面得到了广泛的应用。
2.1 在疾病生物标志物验证中的应用
发现并验证疾病特异性的生物标志物是蛋白质组学研究中的一个重要领域。基于鸟枪法的蛋白质组学分析方法已经被广泛应用于生物标志物的发现;SRM技术对于进一步验证发现的候选生物标志物发挥着重要作用。
Lehnert等[18]使用 iTRAQ(isobaric tags for relative and absolute quantitation)方法对帕金森病人及正常人的脑脊液进行分析,发现了16种表达存在差异的蛋白质。通过SRM进行验证,发现了其中两种蛋白质在病人和正常人间存在显著性差异,有望作为帕金森病的生物标志物。Wang等[19]使用SRM技术定量分析了癌症细胞株中变异与正常Ras蛋白质的差异,并进一步应用于肿瘤组织中变异蛋白质的鉴定和定量,该方法有望应用于癌症的临床诊断。Hüttenhain等[20]利用SRM 技术对血浆和尿液中的癌症相关蛋白质进行了定量:在血浆中检测了182个蛋白质,其浓度动态范围达到5个数量级,最低质量浓度低于10 ng/mL;在尿液中检测到了408个蛋白质,其质量浓度范围与血浆类似。通过对34个生物标志物的监测表明SRM技术可以实现重现性定量;这些数据为研究者对目标蛋白质的分析提供了参考数据。Percy等[21]发展了一种对未处理血浆中的 142种高、中丰度(44~31 ng/mL)蛋白质同时进行选择性反应检测的方法,这些蛋白质与多种非传染性疾病如心血管疾病和糖尿病等相关。Tang等[22]发展了一种候选血清生物标志物的快速验证方法,使用聚丙烯酰胺凝胶电泳对血浆样品进行分级,并对每个组分进行液相色谱分离和SRM监测。该方法无须使用稳定同位素标记多肽,定量检测限可以达到200 pg/mL。他们[23]还使用异种移植的小鼠模型识别了人卵巢癌的血浆生物标志物,并利用SRM方法对候选的生物标志物进行了定量。结果表明,氯离子胞内通道1以及过氧化物还原酶6在卵巢癌病人血浆中的表达显著上调。
Thingholm 等[24]结合 iTRAQ 和 SRM 技术对Ⅱ型糖尿病及正常人肌细胞中的蛋白质进行了鉴定和定量,对其中差异表达的蛋白质进行了识别和验证,结果发现腺苷脱氨酶的丰度在糖尿病人肌细胞中显著下调。IJsselstijn等[25]使用SRM 技术对阿尔茨海默病症状出现前及正常人血浆中的载脂蛋白质含量进行了定量比较,结果表明两者之间无显著性差异。说明该蛋白质不是阿尔茨海默病早期诊断生物标志物,与文献[26]报道结果相反。Pannee等[27]使用SRM方法对人脑脊液中的β淀粉样肽42以及β淀粉样肽40和β淀粉样肽38进行了定量检测。使用同位素标记的肽段作为内标,通过对正常人和病人样品进行分析评估该方法对疾病的诊断能力。结果表明对β淀粉样肽42的定量限低至62.5 pg/mL,RSD小于10%。β淀粉样肽42含量的降低与疾病相关,这与酶联免疫吸附测定(ELISA)方法得到的结果相似;使用β淀粉样肽42与β淀粉样肽40的比值可以更好地对病人进行区分,该方法有望应用于临床诊断。Elschenbroich等[28]结合鸟枪法,基于生物信息学的分类筛选以及SRM技术对恶性及良性上皮性卵巢肿瘤病人腹水中的蛋白质进行了鉴定,并对筛选出的部分差异蛋白质进行了选择性反应监测定量。Addona等[29]将准确包含质量扫描(accurate inclusion mass screening,AIMS)与SRM相结合,发展了一种蛋白质生物标志物发现和验证的流程,并将其应用于心血管疾病候选标志物的寻找。
2.2 其他应用
除了在疾病生物标志物验证这一主要应用领域,SRM技术也逐渐被应用到磷酸化、糖基化、乙酰化等翻译后修饰的研究。Eissler等[30]将无标记的SRM质谱技术应用于磷酸酶和激酶的反应动力学研究,对酶活性进行测量的动态范围达到4个数量级,RSD小于4%。还可以对多种底物或者同一个蛋白质上的不同位点进行同时分析;与传统方法相比,具有更高的通量。Zhao等[31]使用Endo F3酶对糖基化多肽进行部分去糖基化,进而对其进行质谱碎裂和鉴定,然后使用SRM技术对核心岩藻糖化的多肽进行位点的特异性定量。Zhang等[32]利用SRM技术在阿尔茨海默病中定量特异的组蛋白乙酰化水平,并使用Western blot进行了验证。
此外,SRM技术也被应用于生物工程方面的研究。Redding-Johanson等[33]利用 SRM 技术测量了工程化大肠杆菌中蛋白质的表达水平,发现两个甲羟戊酸途径蛋白可能会影响倍半萜烯产量;通过对相关基因进行优化显著提高了蛋白质的表达水平,并且将倍半萜烯的产量提高了3倍。Pandhal等[34]结合基于概率的贝叶斯模型和SRM技术,对工程化大肠杆菌中代谢网络相关蛋白质进行定量分析,并通过增加异柠檬酸裂合酶的表达提高了乙醛酸循环的通量,将糖基化蛋白质的产率提高了300%。
最近,SRM技术也被应用于对细胞信号通路的定量分析。Zhao等[35]以固有免疫应答为信号通路模型,使用SRM技术对低丰度信号通路蛋白质进行定量,以干扰素调节因子3转录因子为例阐述了对高响应肽段的鉴定,方法优化以及评估过程如图2所示。在此基础上他们[36]使用定量10重稳定同位素稀释-SRM方法研究了细胞内双链RNA刺激引起的固有免疫应答的激活模式,观察到NF-kappa B和IRF3信号通路的迅速激活,其中IRF3的激活属于瞬态响应,而NF-kappa B的激活属于延迟的二级放大效应,该结果有助于进一步深入揭示固有免疫应答的机理。
3 结论与展望
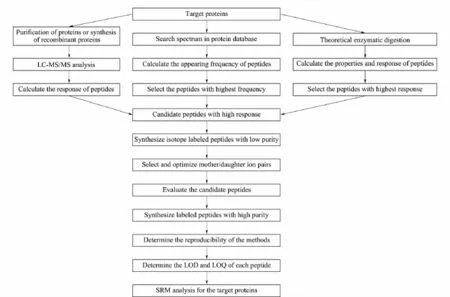
图2 SR M技术的实验流程[35]Fig.2 Experimental workflow of SR M technology[35]
由于SRM技术具有较高的准确度和灵敏度,因此在低丰度肽段和蛋白质的鉴定和定量方面受到了越来越多的关注。然而目前该技术在分析通量和动态范围等方面仍有待进一步提高。例如,SRM技术尚不能对具有宽动态范围的血样品中的蛋白质进行大规模定量。解决这一问题有赖于发展更先进的样品预处理方法,降低样品的复杂程度。同时,SRM技术在灵敏度及特异性方面的改进也将有利于实现复杂样品中超低含量蛋白质的定量分析。此外,iSRM技术的发展和完善必将有助于进一步提高SRM技术的分析通量。该技术有望在生物标志物发现和验证、疾病临床诊断、个性化用药指导等领域发挥重要作用。
[1] Gallien S,Duriez E,Domon B J.Mass Spectrom,2011,46(3):298
[2] Kiyonami R,Schoen A,Prakash A,et al.Mol Cell Proteomics,2011,10(2),DOI:10.1074/mcp.M110.002931
[3] Mao X L,Wei J Y,Niu M,et al.Chinese Journal of Chromatography(毛心丽,卫军营,牛明,等.色谱),2012,30(2):170
[4] Gillet L C,Navarro P,Tate S,et al.Mol Cell Proteomics,2012,11(6),DOI:10.1074/mcp.O111.016717
[5] Whiteaker J R,Zhao L,Abbatiello S E,et al.Mol Cell Proteomics,2011,10(4),DOI:10.1074/mcp.M110.005645
[6] Hossain M,Kaleta D T,Robinson E W,et al.Mol Cell Proteomics,2011, 10(2), DOI:10.1074/mcp. M000062-MCP201
[7] Rafalko A,Dai S J,Hancock W S,et al.J Proteome Res,2012,11(2):808
[8] Choi S,Kim J,Yea K,et al.Anal Biochem,2010,401:196
[9] Picotti P,Bodenmiller B,Mueller L N,et al.Cell,2009,138:795
[10] Jovanovic M,Reiter L,Picotti P,et al.Nat Methods,2010,7:837
[11] Zhao Y,Jia W,Sun W,et al.J Proteome Res,2010,9:3319
[12] Zhang H X,Liu Q F,Zimmerman L J,et al.Mol Cell Proteomics,2011,10(6),DOI:10.1074/mcp.M110.006593
[13] Huillet C,Adrait A,Lebert D,et al.Mol Cell Proteomics,2012,11(2),DOI:10.1074/mcp.M111.008235
[14] Reiter L,Rinner O,Picotti P,et al.Nat Methods,2011,8(5):430
[15] Brusniak M Y K,Kwok S T,Christiansen M,et al.BMC Bioinformatics,2011,12:78
[16] Cham J A,Bianco L,Barton C,et al.J Proteome Res,2010,9(1):620
[17] Huttenhain R,Surinova S,Ossola R,et al.Mol Cell Proteomics,2013,12(4):1005
[18] Lehnert S,Jesse S,Rist W,et al.Exp Neurol,2012,234(2):499
[19] Wang Q,Chaerkady R,Wu J A,et al.P Natl Acad Sci USA,2011,108(6):2444
[20] Hüttenhain R,Soste M,Selevsek N,et al.Sci Transl Med,2012,4(142):142ra97
[21] Percy A J,Chambers A G,Yang J,et al.Biochim Biophys Ac-ta,2013,DOI:10.1016/j.bbapap.2013.06.008
[22] Tang H Y,Beer L A,Barnhart K T,et al.J Proteome Res,2011,10(9):4005
[23] Tang H Y,Beer L A,Chang-Wong T,et al.J Proteome Res,2012,11(2):678
[24] Thingholm T E,Bak S,Beck-Nielsen H,et al.Mol Cell Proteomics,2011,10(9),DOI:10.1074/mcp.M110.006650
[25] IJsselstijn L,Dekker L J,Koudstaal P J,et al.J Proteome Res,2011,10(4):2006
[26] Thambisetty M,Simmons A,Velayudhan L,et al.Arch Gen Psychiatry,2010,67(7):739
[27] Pannee J,Portelius E,Oppermann M,et al.J Alzheimers Dis,2013,33(4):1021
[28] Elschenbroich S,Ignatchenko V,Clarke B,et al.J Proteome Res,2011,10(5):2286
[29] Addona T A,Shi X,Keshishian H,et al.Nat Biotechnol,2011,29(7):635
[30] Eissler C L,Bremmer S C,Martinez J S,et al.Anal Biochem,2011,418(2):267
[31] Zhao Y,Jia W,Wang J F,et al.Anal Chem,2011,83(22):8802
[32] Zhang K L,Schrag M,Crofton A,et al.Proteomics,2012,12(8):1261
[33] Redding-Johanson A M,Batth T S,Chan R,et al.Metab Eng,2011,13(2):194
[34] Pandhal J,Ow S Y,Noirel J,et al.Biotechnol Bioeng,2011,108(4):902
[35] Zhao Y X,Brasier A R.Methods,2013,61(3):313
[36] Zhao Y X,Tian B,Edeh C B,et al.Mol Cell Proteomics,2013,12(6),DOI:10.1074/mcp.M112.023465
