长江中下游两种典型水稻土微生物对砷污染的响应
2014-04-28类成霞蒋瑀霁陈长青中国科学院南京土壤研究所土壤与农业可持续发展国家重点实验室江苏南京20008宁波市农业科学研究院浙江宁波5040南京农业大学应用生态研究所江苏南京20095
汪 峰,类成霞,蒋瑀霁,陈长青,孙 波*(.中国科学院南京土壤研究所,土壤与农业可持续发展国家重点实验室,江苏 南京 20008;2.宁波市农业科学研究院,浙江 宁波 5040;.南京农业大学应用生态研究所,江苏 南京 20095)
长江中下游两种典型水稻土微生物对砷污染的响应
汪 峰1,2,类成霞3,蒋瑀霁1,陈长青3,孙 波1*(1.中国科学院南京土壤研究所,土壤与农业可持续发展国家重点实验室,江苏 南京 210008;2.宁波市农业科学研究院,浙江 宁波 315040;3.南京农业大学应用生态研究所,江苏 南京 210095)
基于温室盆栽实验,利用磷脂脂肪酸分析(PLFA)方法研究了As污染、土壤类型和水稻品种对水稻抽穗期土壤微生物群落的影响.结果表明,水稻土土壤中微生物生物总量(总PLFAs)以及微生物组成均不同程度的受到了As污染、土壤类型及水稻品种的影响;As污染增加了土壤总PLFAs,黄泥田的土壤总PLFAs、真菌/细菌(F/B)、革兰氏阳性细菌/阴性细菌(G+/G-)、腐生真菌/丛枝菌根真菌(SF/AMF)显著高于红泥田;在黄泥田中,种植籼稻品种的土壤总PLFAs较杂交稻和粳稻品种平均减少30.0%,而在红泥田中平均增加24.8%.偏Mantel检验和冗余分析(RDA)表明土壤pH值、DOC、NH4+和As浓度是驱动水稻土微生物群落演变的主控因子,其中受As影响较大的特征PLFAs包括20:0、i17:1ω9c、18:1ω9c、cy17:0和18:3ω6c;ABT预测模型显示水稻品种和土壤类型对总PLFAs、F/B、G+/G-和SF/AMF指标的贡献相对较大,而As处理的影响较小.
砷;水稻品种;红泥田;黄泥田;磷脂脂肪酸
砷在地表系统中含量丰富并广泛分布,影响了环境质量[1-2].近年来,随着工业化、城市化和农业集约化,大量砷污染物进入农田系统,特别是影响了东南亚地区水稻的安全生产,威胁了人体健康[3-5].与旱地作物相比,栽种水稻的淹水厌氧环境提高了水稻籽粒对砷的吸收效率,导致砷成为对水稻最具威胁的有毒污染物[6].我国南方是水稻的主产区,面对日益增加的农田砷污染风险,需要提出土壤-作物系统的综合管理措施,阻控砷污染对水稻安全生产的影响.
土壤微生物参与了砷元素地球化学循环中的还原、氧化和甲基化过程,影响了土壤质量和植物健康[7-9].土壤微生物对砷污染的耐性不同,不同的砷污染状况可以改变土壤微生物的种群结构,从而影响其生态功能.目前,建立指示土壤砷污染的土壤微生物指标,揭示不同气候、土壤和作物条件下砷污染对土壤微生物结构和功能的影响机制已经成为国际研究热点[10-14].在稻田系统中,水稻品种和土壤类型一方面直接影响土壤微生物的生存环境条件,另一方面也影响了土壤砷的形态和有效性,因此对土壤微生物群落的变化产生多重交互影响.植物主要通过输入有机物质驱动了土壤微生物群落的变化,不同植物的基因型显著影响了微生物群落的结构[15-18].对水稻的研究也表明,根际微生物的组成显著受水稻品种的影响,不同籼稻品种的根际细菌群落结构极为相似,但在籼稻与粳稻品种之间存在显著差异[16].土壤类型通过影响土壤微生物的生存环境条件(如pH值、Eh等)也改变了微生物群落的分布和结构[19-20].另一方面,水稻品种和土壤类型同时也影响了砷的生物有效性[21].然而土壤类型、水稻品种和砷污染三者对水稻土微生物群落变化的交互作用规律和影响机制仍不清楚,而这是预测砷污染下水稻土微生物结构变化、建立微生物功能调控措施的理论基础.
磷脂脂肪酸(PLFA)是活体微生物细胞膜的重要组分,不同类群的微生物通过不同的生化过程合成不同的PLFAs,因此PLFAs可作为分析微生物量和微生物群落结构变化的标记物[22]. PLFA图谱已被广泛应用于研究不同污染物胁迫下微生物群落的变化[14,23-25].本研究针对我国长江中下游水稻主产区的两种典型水稻土(江苏的黄泥田和江西的红泥田),选择9个不同的水稻品种(杂交稻、粳稻和籼稻各3个),设置As污染条件下不同水稻品种的温室盆栽实验,利用PLFA分析方法研究As污染对土壤中微生物群落结构的影响,分析土壤类型、水稻品种和As污染对土壤中微生物群落结构变化的交互作用,揭示驱动微生物群落变化的主控因子,为制定砷污染稻田的土壤管理对策提供理论依据.
1 材料与方法
1.1 试验设计
在长江下游地区的江苏常熟(31°36′N, 120°35′E)和中游地区的江西鹰潭(28°12′N, 116°57′E),分别采集了黄泥田水稻土(YP)和红泥田水稻土(RP).黄泥田水稻土发育于湖积物,红泥田水稻土发育于第四纪红黏土母质,分别属于铁聚和简育水耕人为土[26].
采集500kg无污染稻田的表层土壤(0~15cm),在去除石块和根系等杂质后,自然风干,磨碎过筛(5mm)后备用.供试水稻土的基本理化性质如表1所示,两种水稻土均为酸性土,黄泥田水稻土的有机质、阳离子交换量、全钾和全砷含量约为红泥田水稻土的2倍.称取7kg风干土壤置于外径36cm、高30cm的PVC塑料盆钵中,按二级土壤环境质量标准(30mg/kg)的1.5倍加入Na3AsO4溶液[21,27],并设空白对照,即黄泥田加As处理(YP-As)及对照(YP-CK)、红泥田加As处理(RP-As)及对照(RP-CK)4个处理,保持80%的田间持水量平衡3个月备用.水稻盆栽实验于2010年6~10月在南京中山植物园的温室大棚中进行.
供试水稻为我国南方水稻产区的常见品种,包括3个籼型杂交稻品种(Hybrid):德农2000 (DN)、天协6号(TX)、冈优118(GY),3个粳稻品种(Japonica):晚粳9707(WJ)、宁粳1号(NJ)、南粳32(NanJ),3个籼稻品种(Indica):中育1号(ZY)、特三矮2号(TSA)、浙1500(Zhe).精选供试水稻的饱满种子,经过杀菌发芽然后在育秧盘中育秧,于三叶一心期移栽到PVC盆中(6月22日),每种水稻重复3盆,每盆3穴,每穴3株.水稻移栽前一周(6月16日)除草并淹水3~5cm;施入基肥,其中CO(NH2)2:0.15g/kg, Ca(H2PO4)2:0.04g/kg,K2SO4: 0.10g/kg(相当于150kgN/hm2, 40kg P/ha2, 100kgK/ha2),并于分蘖期追施尿素0.075g/kg.移栽后全生育期保持盆钵土面上2~3cm左右水层[21,27].于2010年8月30日采集处于水稻抽穗期0~15cm耕层土壤样品.具体方法为:在每盆3株水稻中间,将灭菌的20mL注射器(已剪去尖头)压到表层土壤中,同时抽动活塞,待注射器内形成土柱后拔出,用活塞推出注射器中土柱样品.每盆取3次,混合土样并去除植物残根后分为2份.1份土样风干后用于基本理化性质分析;另一份放入4℃冰箱保存,在2周内用于测定硝态氮、铵态氮和微生物组成.
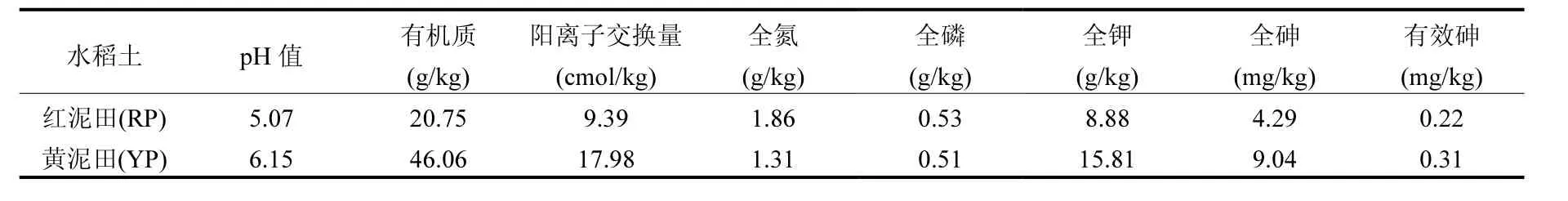
表1 供试土壤的基本理化性质Table 1 Physico-chemical properties of the studied soils
1.2 环境因子的测定
土壤pH值采用玻璃电极测定(水土比2.5:1)[28];土壤有效砷的含量用0.5mol/L的NaHCO3震荡浸提(水土比10∶1),双道原子荧光光度计测定;土壤氨态氮和硝态氮用1.0mol/L KCl溶液浸提,流动分析仪测定;可溶性有机碳(DOC)和可溶性有机氮(DON)的提取和测定参考Jones和Willett法[29],利用岛津TOC-5050A总有机碳仪测定滤液中的DOC和总氮(TN)含量,总氮减去无机氮量即为DON的量.根系生物量和籽粒产量在水稻收获后完成测定.
1.3 磷脂脂肪酸分析
磷脂脂肪酸的提取采用修改的Bligh-Dyer法[30-31].PLFAs组成用MIDI Sherlock 微生物鉴定系统(MIDI, Newark, DE, USA)分析.以19:0脂肪酸甲酯(FAME)作为内标加入样品,按照峰面积比值定量分析PLFA的含量.将i15:0、a15:0、i16:0、i17:0、a17:0划分为革兰氏阳性细菌(G+),将16:l ω7c、17:lω8c、18:1ω7c、cy17:0划分为革兰氏阴性细菌(G-),G+和G-之和计为细菌生物量;以16:1ω5c作为丛枝菌根真菌(AMF)的标记,以18:1ω9c和18:2ω6,9c作为腐生真菌(SF)的标记,AMF和SF之和计为真菌生物量;以10Mel6: 0、10Me17:0和10Me18:0作为放线菌的标记[32].
1.4 数据处理及统计分析
在进行多元排序分析之前对土壤PLFAs图谱进行降噪处理.首先删除PLFA测定中的不确定数据,即3个重复样品只检出1次的土壤样品的测定值;然后将PLFAs的含量测定值转化为相对丰度值,获得物种分布表格,在对物种和环境因子数据标准化后,进行降趋势对应分析(DCA)和冗余分析(RDA).
聚类推进树分析(ABT)分析能够较好地预测和解释不同类型的环境变量对微生物群落分布的相对贡献率[33-34],本研究中利用ABT分析3个因子(土壤类型、水稻品种、砷污染处理)对土壤中活性微生物群落组成的相对重要性.
单因素和多因素方差分析(ANOVA)用统计软件SPSS16.0(SPSS Inc., Chicago, Illinois)完成;DCA、RDA和ABT分析在R-2.13.2(http: //www.R-project.org)中完成,其中DCA和RDA利用“vegan”软件包,ABT使用“gbmplus”软件包.
2 结果
2.1 土壤微生物生物量
土壤中提取的PLFAs总量可以用于定量土壤微生物的生物量.本研究中不同处理下土壤总PLFAs的平均值为49.88nmol/g干土,变幅为25.89~85.10nmol/g干土(图1).
As污染、水稻品种和土壤类型显著影响了(P< 0.01)土壤总PLFAs.与对照相比,As污染总体上增加了土壤总PLFAs,但在不同的土壤和水稻品种条件下其增幅不同.在黄泥田(YP)和红泥田(RP)中,As污染使土壤总PLFAs分别增加了24.8%和6.6%;在YP中所有水稻品种As污染处理中总PLFAs均呈增加趋势,而在RP中只有4个水稻品种(TX、NanJ、ZY、TSA)表现为上升趋势(图1).不同水稻土中,种植的水稻品种对总PLFAs的影响不同.在YP中,种植籼稻品种的土壤PLFAs总量较杂交稻和粳稻品种平均减少30.0%,而在RP中平均增加24.8%.黄泥田水稻土的PLFAs总量显著高于红泥田,在对照处理(无As污染)和As污染条件下,YP的总PLFAs分别比RP高13.3%和34.6%.
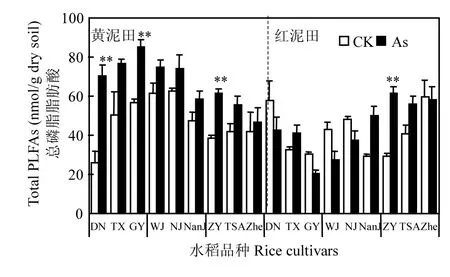
图1 不同水稻品种、土壤类型和As处理中磷脂脂肪酸总量的变化Fig.1 Total PLFAs for soil microbial biomass as affected by rice cultivar, soil type and As treatments
不同类群微生物生物量占土壤PLFAs总量的比例顺序为:细菌>真菌>放线菌,细菌中革兰氏阳性细菌(G+)比例较大.不同类群微生物生物量同样受As污染、水稻品种和土壤类型的影响,其变化趋势与土壤总PLFAs的变化相似(表2).As污染显著增加了黄泥田中不同微生物类群的生物量,而在红泥田中As污染的影响不显著.在不同的水稻品种中,对照处理下红泥田中种植杂交稻DN的土壤细菌和真菌生物量最高,比种植粳稻NanJ分别高出1.0和1.6倍(表2).黄泥田大部分类群的微生物生物量较高,尤其是在As污染下,YP的真菌和放线菌生物量比RP高2倍(表2).在As污染处理下,黄泥田中种植GY后土壤细菌、G+和G-生物量最高,而红泥田中最低,说明品种和土壤类型有交互作用.
2.2 土壤微生物群落结构
不同处理下共检测出64种PLFA类型,检测到的脂肪酸碳原子个数从9到20,包括饱和的、不饱和的、分支的和环状的PLFAs;其中有18种PLFAs在所有土壤样品中普遍存在,占PLFAs类型的82.3%,但其相对丰度存在差异.土壤优势PLFAs主要由16:0、18:1ω9c(SF)、i15:0(G+)、18:0、16:1ω7c(G-)和a15:0(G+)构成,占样品PLFAs类型的50.0%.
As污染、水稻品种和土壤类型显著影响了土壤PLFAs图谱.a14:0和17:0 2OH仅在种植了DN的土壤中检出;a17:1和20:4ω6c仅存在于RP中,而i10:0、10Me19:0和9:0仅存在于YP中;a17:1ω9c仅存在于无砷污染的土壤,i20:0和9:0 3OH仅存在于砷污染土壤.
真菌/细菌(F/B)、革兰氏阳性细菌/阴性细菌(G+/G-)和腐生真菌/丛枝菌根真菌(SF/AMF)可以表征土壤微生物群落的结构特征,这些比值均受As污染、水稻品种和土壤类型的综合影响(图2).总体上,黄泥田的F/B、G+/G-和SF/AMF显著高于红泥田.
对于黄泥田,在种植杂交稻品种处理中,As污染提高了F/B;但在种植籼稻的处理中,As污染降低了F/B.对于红泥田,在种植杂交稻和粳稻(DN和NanJ除外)的处理中,As污染使F/B明显下降,而在种植籼稻ZY的处理中,As污染增加了F/B.
与对照相比,As污染处理大多降低了土壤G+/G-.对于黄泥田,未污染条件下种植DN的土壤G+/G-明显低于其他8个水稻品种;As污染条件下,种植杂交稻和粳稻(除NanJ)比种植籼稻的G+/G-低.对于红泥田,在未污染处理中,种植TX、GY、NanJ、ZY的G+/G-较高;在As污染条件下种植DN、TSA和Zhe的G+/G-较低.
与对照相比,As污染处理大多降低了黄泥田的SF/AMF(ZY、Zhe除外),但在红泥田中变化不显著.在黄泥田中,未污染处理下,种植WJ和TSA的SF/AMF值最高;As污染处理下,种植DN和NanJ的SF/AMF相对较低.以上表明土壤微生物群落是受As污染、水稻品种和土壤类型等因素综合影响的结果.
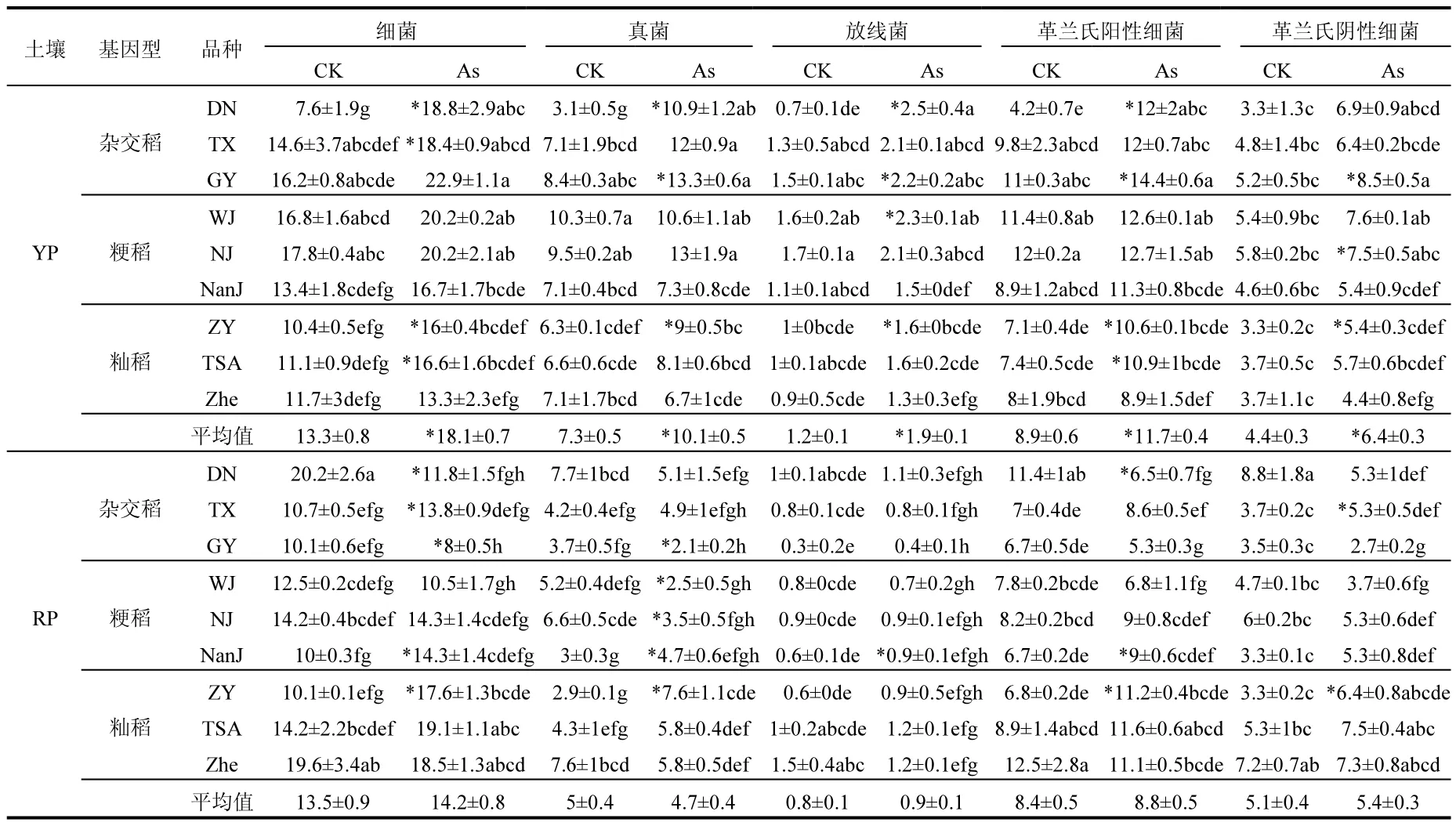
表2 不同水稻品种、土壤类型和As处理中细菌、真菌、放线菌、革兰氏阳性细菌和革兰氏阴性细菌的PLFAs量Table 2 The amounts of bacterial PLFAs, fungal, actinobacterial, Gram-positive bacterial and Gram-negative bacterial PLFAs (nmol g−1dry weight) as affected by rice cultivar, soil type and As treatments
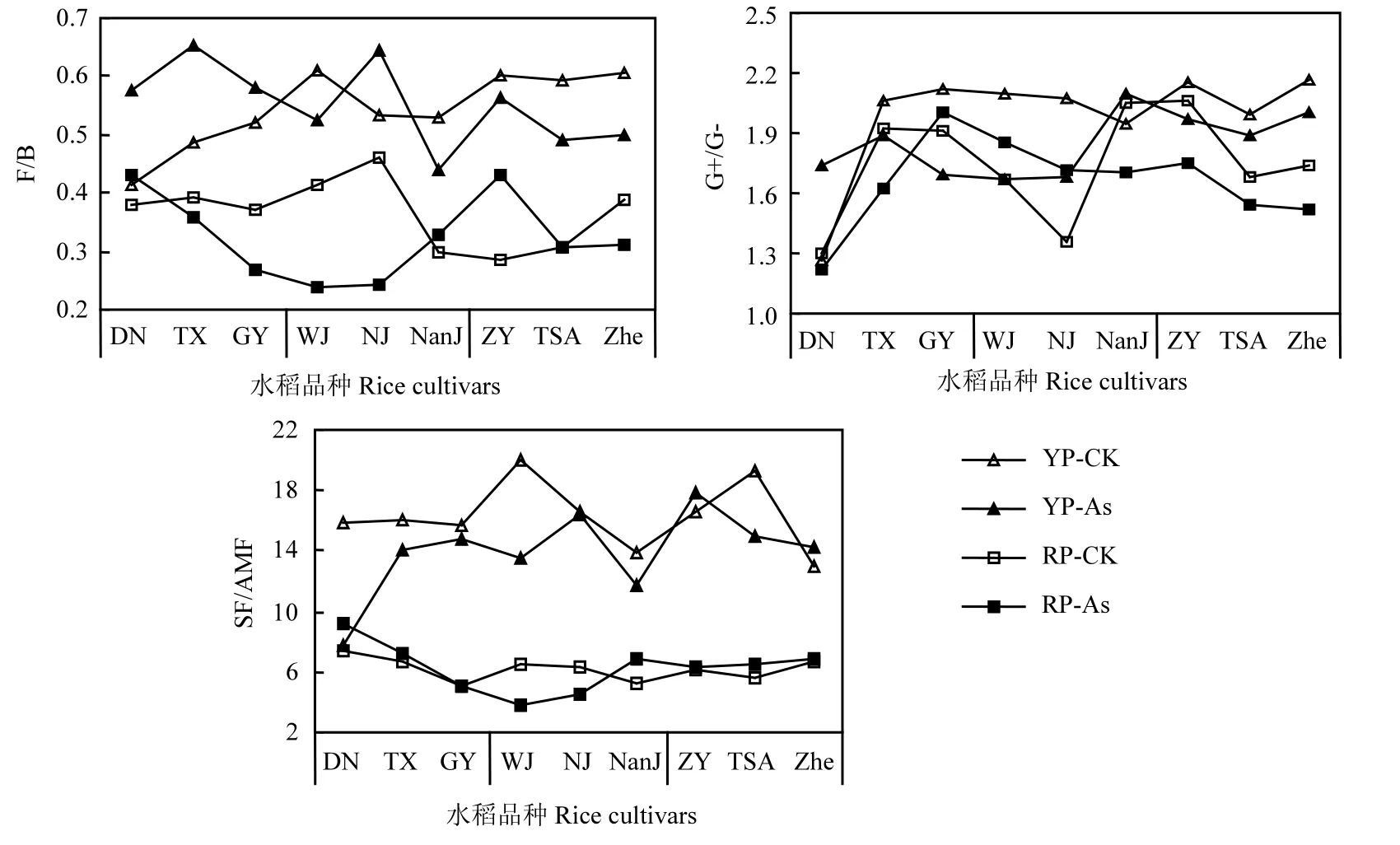
图2 不同水稻品种、土壤类型和As处理中真菌/细菌(A)、革兰氏阳性细菌/革兰氏阴性细菌(B)和腐生真菌/丛枝菌根真菌(C)变化Fig.2 Changes in ratios of F/B (A), G+/G-bacteria (B) and SF/AMF (C) as affected by rice cultivar, soil type and As treatments
2.3 微生物群落与环境因子的关系
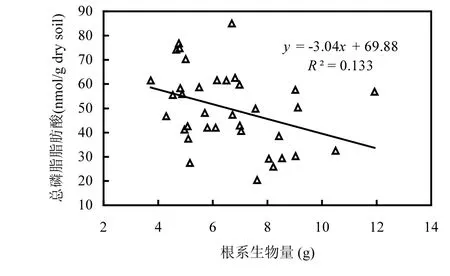
图3 水稻根系生物量与土壤微生物总磷脂脂肪酸之间的皮尔森相关性Fig.3 Pearson correlation between rice root biomass and soil total PLFAs
相关分析表明,水稻根系生物量不仅与土壤总PLFAs呈显著线性相关(图3),也与细菌、放线菌及G-细菌PLFAs有显著负相关关系(P<0.05).偏Mantel分析表明土壤pH值、As、DOC和NH4+含量对土壤中的微生物群落组成有显著的影响(P<0.05),而 DON、C/N、NO3-和根系生物量对微生物群落的影响不显著(表3).生物与环境相关分析(BIOENV)结果也表明解释土壤微生物群落结构特征的最佳环境因子是土壤pH(r = 0.706),余下依次为组合因子pH和NH4+(r = 0.629);pH、DOC和NH4+(r = 0.604); pH值、As、DOC和NH4+(r = 0.540).
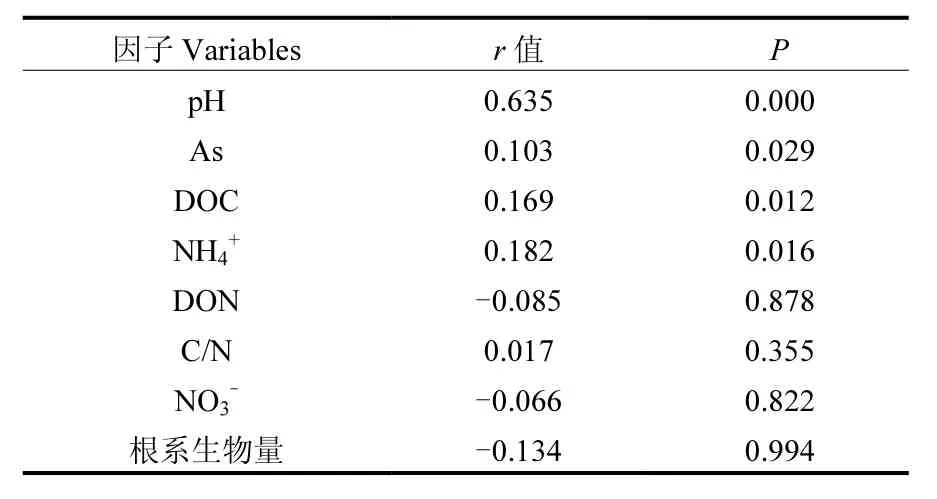
表3 偏Mantel检验环境因子和微生物群落结构相关性的显著性Table 3 The significance of correlations between environmental variables and microbial community structures assessed using partial Mantel test
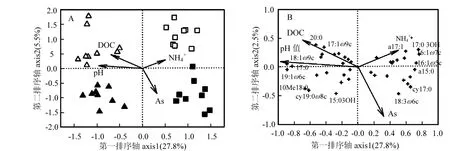
图4 不同水稻品种、土壤类型和As处理中PLFAs图谱的冗余排序分析(A)和PLFAs的载荷因子分析(B)Fig.4 Redundancy analysis (RDA) ordination biplot of the PLFAs pattern (A) from different soil samples under rice cultivar, soil type and As treatments and loadings of the individual PLFAs (B) from the RDA of the PLFA data
通过排序方法能够直观地反映出群落结构与环境因子之间关系.通过DCA分析PLFAs图谱得到每个轴的长度均<3,说明基于线性模型的冗余分析(RDA)能够较好地进行PLFAs图谱分析.第一排序轴和第二轴分别解释了27.8%和 5.5%的总变异(图4).从图中可以看出第一排序轴的正轴方向分布着红泥田的样品,负轴方向分布着黄泥田样品;As污染的土壤与对照样品按第二轴区分开来.土壤pH和DOC与第一轴呈负相关关系,而NH4+与其呈正相关;As浓度与第二轴呈负相关关系(图4A).单一PLFAs载荷分析表明土壤pH主要影响的磷脂脂肪酸类型是18:1ω9c(SF)、19:1ω6c、17:0、16:1ω5c(AMF)、a15:0(G+)、16:1ω7c(G-)和i17:0 3OH;NH4+影响的主要类型是a17:1、10Me 18:0(放线菌)和cy19:0ω8c;As和DOC影响的主要是20:0、i17:1ω9c、18:1ω9c(SF)、cy17:0(G-)和18:3ω6c.以上结果表明土壤pH、DOC和NH4+和As污染均显著影响土壤微生物群落结构.
2.4 土壤类型、砷污染和水稻品种对土壤微生物群落的相对贡献
ABT模型能够较好地预测不同类型环境变量对某一微生物指标的相对贡献率.ABT分析结果(图5)表明,水稻品种对土壤总PLFAs的影响最大(52%),其次是土壤类型(30%)和As污染(18%).影响F/B和SF/AMF变化的相对贡献率顺序均表现为:土壤类型>水稻品种>As污染,对G+/G-变化的相对贡献率大小为:水稻品种>土壤类型>As污染.综上所述,土壤类型和水稻品种对微生物群落结构影响较大,而As污染对其影响相对较小.
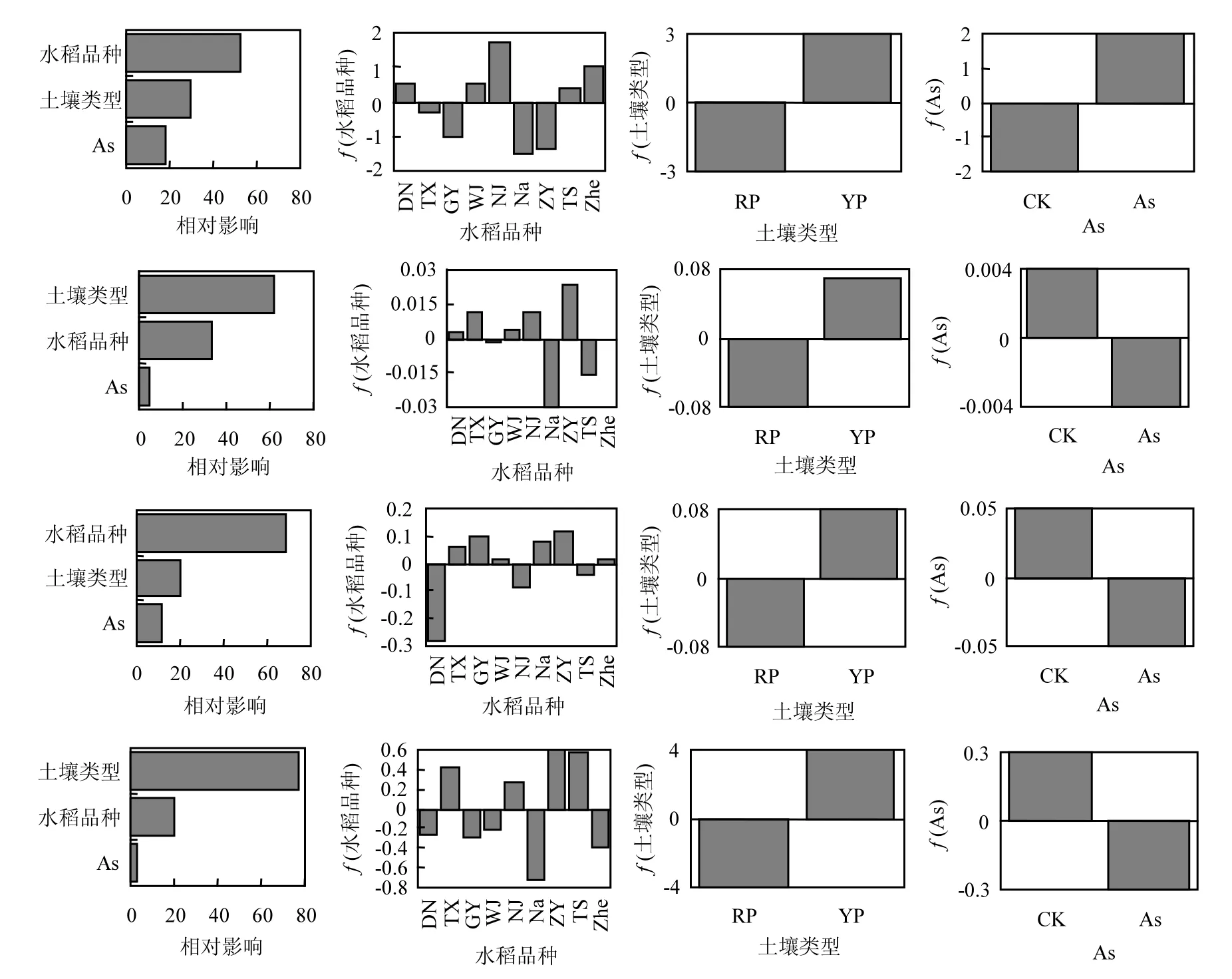
图5 基于ABT模型预测水稻品种、土壤类型和As浓度对土壤微生物总PLFAs (A)、F/B (B)、G+/G-(C)和SF/AMF (D)的相对重要性Fig.5 Relative importance and partial dependency plots of predictors for total PLFAs (A), F/B (B), G+/G-(C) and SF/ AMF (D) by aggregated boosted tree (ABT) model using rice cultivar, soil type and As concentration as preditors
ABT预测模型显示9个水稻品种对总PLFAs、F/B、G+/G-和SF/AMF的影响表现不一致,例如DN、WJ、NJ、TSA、Zhe对微生物量表现为正向作用,其余4个品种表现为负向作用;土壤类型对以上4种微生物指标的影响表现一致,黄泥田对土壤PLFA总量、F/B、G+/G-和SF/AMF有正向作用,而红泥田是负向作用;As污染对微生物量具有正向作用,但对F/B、G+/G-和SF/AMF均表现为负向作用.
3 讨论
3.1 土壤微生物群落对As污染的响应
由于不同微生物对As胁迫下的耐受力不同,微生物生物量、群落组成、代谢活性和生物学功能均不同程度地受到As污染的影响[10-13].本研究结果表明了土壤中的活体微生物群落结构在As作用下发生改变(图4),这与以上的报道一致.然而As污染条件下增加了土壤PLFAs总量(图1),与以往的研究结果不同.Edvantoro等研究表明,As污染(34~2941mg/kg)条件下微生物量碳、真菌数量及呼吸熵剧烈下降,但细菌的数量变化不大[11];Ghosh等[12]发现不同提取态的As(其中NaHCO3提取态0.5~6.1mg/kg)与微生物生物量和土壤呼吸显著负相关.重金属对微生物的抑制作用程度与土壤性质、植被、污染物浓度以及微生物类群等因素有关[11].本研究中导致As污染增加微生物量的原因可能是,As添加量为土壤质量二级标准的1.5倍(45mg/kg),有效As含量范围为1.77~4.01mg/kg,因此As污染水平较低;水稻土中部分微生物对低浓度As污染有较高的耐受力,甚至以As作为其生长的能源[35],因此低浓度的As促使适应性较强的微生物迅速大量积累,致使土壤微生物量有所上升,土壤微生物群落结构发生了变化.
本文研究结果显示7种特定PLFAs与土壤中有效As浓度存在极显著的相关性(P<0.01),其中cy17:0、18:3ω6c和i17:1ω9c的含量随As浓度的升高而增加,而10:0、10:0 3OH、11:0和17:0随As浓度的升高而减少.已有的研究表明革兰氏阴性菌的典型脂肪酸cy17:0在重金属污染的土壤中相对含量会上升[36];cy17:0/16:1ω7c和i17:0+i15: 0/a17:0+a15:0的值反映着外界环境条件对微生物的胁迫作用[37-38],本研究表明了微生物在重金属污染物存在的条件下能合成更多的反式脂肪酸,使 i17:0+i15:0/a17:0+a15:0的值下降,而促进cy17:0的前体16:1ω7c合成cy17:0,使cy17:0/16:1ω7c的值上升.尽管低浓度的As污染促使部分耐受菌迅速积累成为优势菌群导致土壤微生物总量有所增加,但微生物群落结构的改变仍然可能给土壤养分转化等功能带来不利影响[10].
3.2 As污染下土壤类型对土壤微生物群落的影响
不同类型土壤中的微生物类群有着较大的差异,由于耐受力不同导致不同类型土壤中微生物对污染物的响应不一致.对于本研究中两种不同水稻土壤,微生物指标如土壤总PLFAs、F/B、G+/G-和SF/AMF对As污染的响应存在显著差异(图1和图2);相同土壤类型的样品中PLFAs图谱更为相似(图4),因此土壤类型影响微生物对As响应的方向.
土壤性质中的pH值、DOC和NH4+含量是驱动微生物群落结构变化的关键因子(表3和图4).土壤pH在区域尺度(从国家到洲际)上驱动了土壤细菌多样性的变化[20,39].在阔叶林系统中,土壤F/B随着pH的增加(3.0~7.2)而略微升高,归属AMF的16:1ω5c和归属G+的16:1ω7c与pH呈正相关[40].本文的研究结果也表明,水稻土pH不仅与F/B(r2=0.854,P<0.01)存在显著相关,也与G+/G-(r2=0.366,P<0.05)和SF/AMF(r2=0.823,P< 0.01)有较高的相关性;水稻土中19种PLFAs与pH极显著相关(P<0.01),但其中的16:1ω5c和16:1ω7c与pH值均呈负相关,这可能是水稻土与森林土壤在其他性质(如有机质)方面的差异所致.
可溶性有机碳(DOC)主要来源于植物根系分泌物中可溶性糖、有机酸和氨基酸[27].前期研究表明[27],As污染下黄泥田中DOC的平均含量(39.27mg/kg)高于红泥田(33.32mg/kg),不同水稻土中DOC与土壤有效As含量呈显著负相关,但与土壤pH呈显著正相关.本研究发现DOC与真菌PLFA(r2=0.371,P<0.05)、F/B(r2=0.708, P< 0.01)、SF/AMF(r2=0.601,P<0.01)呈显著正相关,说明在水稻土中DOC的变化可以预测真菌群落的变化.而在湿地土壤中,由于土壤碳含量较高导致对微生物群落结构影响不大,而土壤pH显著影响了土壤微生物组成[41].因此,土壤pH和活性有机质含量在As污染下的变化共同影响了土壤微生物的变化.
3.3 As污染下水稻品种对土壤微生物群落的影响
种植不同品种水稻的土壤中微生物对As污染的响应也存在着差异.作物通过根系向土壤中分泌大量的糖类、氨基酸、维生素等有机物质,影响了土壤微生物数量和组成;植物的选择作用也影响了土壤中与植物共生(如根瘤菌)和非共生的细菌组成[15,42].此外,作物基因型也影响了根际微生物群落结构[16-18].本研究中水稻品种影响了总PLFAs、F/B、G+/G-和SF/AMF(图5),不但种植杂交稻、粳稻和籼稻的土壤微生物群落之间存在着差异,同一基因型中不同品种之间的差异也较大.这与Hardoim等[16]的研究不同,他们发现不同籼稻品种的根际土壤中细菌群落相似,而籼稻与粳稻之间存在较大差异.可能原因是本研究中选择的9个水稻品种之间在生长状况和根系分泌物方面的差异更大,水稻籽粒产量和根茎叶的生物量结果已经表明存在显著差异[21].
不同水稻品种的根系分泌物数量和组成存在较大的差异.研究表明,水稻品种PI312777和辽粳9号的根系分泌物中所含的碳水化合物总量、葡萄糖、氨基酸等明显不同[43].耐低磷水稻品种与低磷敏感型品种根系分泌物中检出的有机酸的种类和数量均有不同[44].种植9个水稻品种后,土壤中溶解性碳氮含量存在显著差异在一定程度上反映了不同水稻品种根系分泌物的不同[27].作者的前期研究表明,9个水稻品种对As的吸收和积累的效率差异明显[21],因此不同水稻品种对As的耐受力不一致导致地上生物量存在差异.长期野外控制实验表明,植物产量和多样性共同影响了土壤微生物的生物量、活性和组成[45].因此,As污染下不同品种水稻分泌物的数量和组成共同变化,这些变化同时影响了土壤微生物生物量及群落组成.
针对不同类型的水稻土,可以筛选和种植合适水稻品种,同时配合土壤性质改良,从而有效地调控土壤微生物结构,应对As污染对土壤微生物功能的影响.由于利用单一的PLFA法研究土壤微生物的群落变化存在一定的局限性[46],未来需要联合高通量测序和功能基因芯片等高分辨率的微生物分子生态学方法,深入研究水稻不同生育期土壤微生物结构的变化,同时分析土壤微生物养分转化功能的变化,筛选水稻土污染的敏感监测指标.
4 结语
针对长江中下游水稻主产区的两种典型水稻土,结合温室盆栽模拟实验和PLFA分析方法,研究了在不同土壤类型及水稻品种条件下,土壤微生物群落对低浓度As污染的响应.研究表明土壤总PLFAs、细菌、真菌、放线菌、革兰氏阳性细菌和革兰氏阴性细菌PLFAs均不同程度的受到了As污染、土壤类型及水稻品种的影响;不同样品间微生物群落结构也存在着差异,pH值、DOC和NH4+和As浓度是驱动微生物变化的主要因素;ABT预测模型揭示了低浓度的As处理对微生物群落影响相对较小,而水稻品种和土壤类型的相对贡献率大.
[1] Mandal B K, Suzuki K T. Arsenic round the world: a review [J]. Talanta, 2002,58(1):201-235.
[2] Tamaki S, Frankenberger W T. Environmental biochemistry of arsenic [J]. Reviews of Environmental Contamination and Toxicology, 1992,124:79-110.
[3] Fendorf S, Michael H A, van Geen A. Spatial and temporal variations of groundwater arsenic in South and Southeast Asia [J]. Science, 2010,328(5982):1123-1127.
[4] Khan M A, Stroud J L, Zhu Y G, et al. Arsenic bioavailability to rice is elevated in Bangladeshi paddy soils [J]. Environmental Science and Technology, 2010,44(22):8515-8521.
[5] Zhu Y G, Williams P N, Meharg A A. Exposure to inorganic arsenic from rice: A global health issue? [J]. Environmental Pollution, 2008,154(2):169-171.
[6] Su Y H, McGrath S P, Zhao F J. Rice is more efficient in arsenite uptake and translocation than wheat and barley [J]. Plant and Soil, 2010,328(1/2):27-34.
[7] Cai L, Liu G H, Rensing C, et al. Genes involved in arsenic transformation and resistance associated with different levels of arsenic-contaminated soils [J]. BMC Microbiology, 2009,9:
[8] Ye J, Rensing C, Rosen B P, et al. Arsenic biomethylation by photosynthetic organisms [J]. Trends in Plant Science, 2012, 17(3):155-162.
[9] Jia Y, Huang H, Zhong M, et al. Microbial arsenic methylation in soil and rice rhizosphere [J]. Environmental Science and Technology, 2013,47(7):3141-3148.
[10] Xiong J, Wu L, Tu S, et al. Microbial communities and functional genes associated with soil arsenic contamination and the rhizosphere of the arsenic-hyperaccumulating plant Pteris vittata L [J]. Appl. Environ. Microbiol., 2010,76(21):7277-7284.
[11] Edvantoro B B, Naidu R, Megharaj M, et al. Changes in microbial properties associated with long-term arsenic and DDT contaminated soils at disused cattle dip sites [J]. Ecotoxicology and Environmental Safety, 2003,55(3):344-351.
[12] Ghosh A K, Bhattacharyya P, Pal R. Effect of arsenic contamination on microbial biomass and its activities in arsenic contaminated soils of Gangetic West Bengal, India [J]. Environment International, 2004,30(4):491-499.
[13] Lorenz N, Hintemann T, Kramarewa T, et al. Response of microbial activity and microbial community composition in soils to long-term arsenic and cadmium exposure [J]. Soil Biology and Biochemistry, 2006,38(6):1430-1437.
[14] Turpeinen R, Kairesalo T, Häggblom M M. Microbial community structure and activity in arsenic-, chromium- and copper-contaminated soils [J]. FEMS Microbiology Ecology, 2004,47(1):39-50.
[15] Philippot L, Raaijmakers J M, Lemanceau P, et al. Going back to the roots: the microbial ecology of the rhizosphere [J]. Nature Reviews Microbiology, 2013,11(11):789-799.
[16] Hardoim P R, Andreote F D, Reinhold-Hurek B, et al. Rice rootassociated bacteria: insights into community structures across 10 cultivars [J]. FEMS Microbiol. Ecol., 2011,77(1):154-164.
[17] Aira M, Gómez-Brandón M, Lazcano C, et al. Plant genotype strongly modifies the structure and growth of maize rhizosphere microbial communities [J]. Soil Biol. Biochem., 2010,42(12): 2276-2281.
[18] van Overbeek L, van Elsas J D. Effects of plant genotype and growth stage on the structure of bacterial communities associated with potato (Solanum tuberosum L.) [J]. FEMS Microbiology Ecology, 2008,64(2):283-296.
[19] Ranjard L, Dequiedt S, Chemidlin Prevost-Boure N, et al. Turnover of soil bacterial diversity driven by wide-scale environmental heterogeneity [J]. Nature Communications, 2013,4: 1434.
[20] Griffiths R I, Thomson B C, James P, et al. The bacterial biogeography of British soils [J]. Environ. Microbiol., 2011,13(6): 1642-1654.
[21] Ye X X, Sun B, Yin Y L. Variation of As concentration between soil types and rice genotypes and the selection of cultivars for reducing As in the diet [J]. Chemosphere, 2012,87(4):384-389.
[22] Malik S, Beer M, Megharaj M, et al. The use of molecular techniques to characterize the microbial communities in contaminated soil and water [J]. Environment International, 2008,34(2):265-276.
[23] Pennanen T, Frostegard A, Fritze H, et al. Phospholipid fatty acid composition and heavy metal tolerance of soil microbial communities along two heavy metal-polluted gradients in coniferous forests [J]. Applied and Environmental Microbiology, 1996,62(2):420-428.
[24] Feris K, Ramsey P, Frazar C, et al. Differences in hyporheic-zone microbial community structure along a heavy-metal contamination gradient [J]. Applied and Environmental Microbiology, 2003,69(9):5563-5573.
[25] Frostegard A, Tunlid A, Baath E. Changes in microbial community structure during long-term incubation in two soils experimentally contaminated with metals [J]. Soil Biology and Biochemistry, 1996,28(1):55-63.
[26] 中国科学院南京土壤研究所土壤系统分类课题组. 中国土壤系统分类检索 [M]. 3版.合肥:中国科学技术大学出版社2001:1-275.
[27] 类成霞,陈长青,蒋瑀霁,等.水稻品种和砷污染对土壤溶解性有机碳氮的影响 [J]. 土壤学报, 2013,50(1):157-164.
[28] 鲁如坤.土壤农业化学分析方法 [M]. 北京:中国农业科技出版社, 2000.
[29] Jones D, Willett V. Experimental evaluation of methods to quantify dissolved organic nitrogen (DON) and dissolved organic carbon (DOC) in soil [J]. Soil Biol. Biochem., 2006,38(5):991-999.
[30] Zhong W, Gu T, Wang W, et al. The effects of mineral fertilizer and organic manure on soil microbial community and diversity [J]. Plant and Soil, 2010,326(1/2):511-522.
[31] Brant J B, Sulzman E W, Myrold D D. Microbial community utilization of added carbon substrates in response to long-term carbon input manipulation [J]. Soil Biol. Biochem., 2006,38(8): 2219-2232.
[32] Zelles L. Fatty acid patterns of phospholipids and lipopolysaccharides in the characterisation of microbial communities in soil: a review [J]. Biology and Fertility of Soils, 1999,29(2):111-129.
[33] Ge Y, He J-z, Zhu Y-g, et al. Differences in soil bacterial diversity: driven by contemporary disturbances or historical contingencies? [J]. The ISME Journal, 2008,2(3):254-264.
[34] De'ath G. Boosted trees for ecological modeling and prediction [J]. Ecology, 2007,88(1):243-251.
[35] Wolfe-Simon F, Blum J S, Kulp T R, et al. A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus [J]. Science, 2011,332(6034):1163-1166
[36] Frostegard A, Tunlid A, Baath E. Phospholipid Fatty-Acid Composition, Biomass, and Activity of Microbial Communities from 2Soil Types Experimentally Exposed to Different Heavy-Metals [J]. Applied and Environmental Microbiology, 1993, 59(11):3605-3617.
[37] Macdonald L M, Paterson E, Dawson LA, et al. Defoliation and fertiliser influences on the soil microbial community associated with two contrasting Lolium perenne cullivars [J]. Soil Biol. Biochem., 2006,38(4):674-682.
[38] McKinley V L, Peacock A D, White D C. Microbial community PLFA and PHB responses to ecosystem restoration in tallgrass prairie soils [J]. Soil Biol. Biochem., 2005,37(10):1946-1958.
[39] Fierer N, Jackson R B. The diversity and biogeography of soil bacterial communities [J]. Proceedings of the National Academy of Sciences of the United States of America, 2006,103(3):626-631.
[40] Bååth E, Anderson T H. Comparison of soil fungal/bacterial ratios in a pH gradient using physiological and PLFA-based techniques [J]. Soil Biol. Biochem., 2003,35(7):955-963.
[41] Hartman W H, Richardson C J, Vilgalys R, et al. Environmental and anthropogenic controls over bacterial communities in wetland soils [J]. Proceedings of the National Academy of Sciences of the United States of America, 2008,105(46):17842-17847.
[42] Dini-Andreote F, Elsas JD. Back to the basics: The need for ecophysiological insights to enhance our understanding of microbial behaviour in the rhizosphere [J]. Plant and Soil, 2013, 373(1/2):1-15.
[43] Kong C H, Wang P, Zhao H, et al. Impact of allelochemical exuded from allelopathic rice on soil microbial community [J]. Soil Biol. Biochem., 2008,40(7):1862-1869.
[44] 李德华,向春雷,姜益泉,等.低磷胁迫下水稻不同品种根系有机酸分泌的差异 [J]. 中国农学通报, 2005,21(11):186-188.
[45] Zak D R, Holmes W E, White D C, et al. Plant diversity, soil microbial communities, and ecosystem function: Are there any links? [J]. Ecology, 2003,84(8):2042-2050.
[46] Frostegård Å, Tunlid A, Bååth E. Use and misuse of PLFA measurements in soils [J]. Soil Biol. Biochem., 2011,43(8):1621-1625.
Response of microbial community to arsenic contamination in two major type of paddy soils in the middle and lower reaches of the Yangtse River.
WANG Feng1,2, LEI Cheng-xia3, JIANG Yu-ji1, CHEN Chang-qing3, SUN Bo1(1.State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;2.Ningbo Academy of Agricultural Sciences, Ningbo 315040, China;3.Institute of Applied Ecology, Nanjing Agricultural University, Nanjing 210095, China). China Environmental Science, 2014,34(11):2931~2941
In a pot experiment, the effects of As contamination, soil type and rice cultivar on the composition of soil microbial communities in rice heading stage were unalyzal using phospholipid fatty acid (PLFA) technique. The results unveiled that the amount of total PLFAs and microbial compositions were to some extent affected by arsenic contamination, soil types and rice cultivars. The As contaminated soils had a higher amount of total PLFAs than the uncontaminated soils. Yellow Paddy soils (YP) had higher total PLFAs, fungi to bacteria ratio (F/B), Gram-negative bacteria to Gram-positive bacteria ratio (G+/G-) and saprotrophic fungi to arbuscular mycorrhizal fungi ratio (SF/AMF) than in Red Paddy soils (RP). Total PLFAs associated with indica plants vs. those associated with japonica and hybrid plants decreased by an average of 30.0% in YP, but increased by 24.8% in RP. Partial Mantel tests and redundancy analysis (RDA) showed that soil pH, DOC, NH4+and As concentration were the dominant factors in shaping soil microbial communities. The individual PLFAs including 20:0, i17:1ω9c, 18:1ω9c, cy17:0 and 18:3ω6c were easily affected by As contamination. Aggregated boosted tree (ABT) analysis suggested that soil types and rice cultivars exerted more influence than As concentration on the variation of parameters of total PLFAs, F/B, G+/G-and SF/AMF.
arsenic;rice genotype;red paddy soil;yellow paddy soil;phospholipid fatty acid (PLFA)
Q938
A
1000-6923(2014)11-2931-11
汪 峰(1982-),男,安徽潜山人,助理研究员,博士,研究方向为微生物分子生态学.发表论文13篇.
关于《中国环境科学》网上投稿的通知
《中国环境科学》编辑部
2014-02-28
中国科学院战略性先导科技专项(XDB15030200);国家“973”项目(2011CB100506);农业部公益性行业科研专项(200903015)
* 责任作者, 研究员, bsun@issas.ac.cn
《中国环境科学》编辑部为提高稿件处理的网络化水平和采编工作的效率,及时让作者了解稿件的处理情况,自2008年3月1日起已开通网上投稿查稿系统,请登陆网址: http://www.zghjkx.com.cn点击“作者在线投稿”进行注册后再按要求投稿,点击“作者在线查稿”进行查询.本刊不再接受纸件投稿和电子版稿件的E-mail投稿.特此通知.
