黄河口邻近水域中华哲水蚤现场食物组成的分子生物学检测*
2014-04-17黄有松徐东晖陈洪举林森杰刘光兴
黄有松,徐东晖,2,陈洪举,3,张 寰,4,林森杰,刘光兴,3**
(中国海洋大学1.环境科学与工程学院;2.海洋环境学院;3.海洋环境与生态教育部重点实验室,山东 青岛266100;4.Department of Marine Sciences,University of Connecticut,Groton,CT 06340,USA;5.厦门大学近海海洋环境科学国家重点实验室,海洋生物多样性与全球变化实验室,福建 厦门361005)
作为海洋生态系统中最丰富的后生动物以及浮游植物等的主要摄食者,桡足类在海洋食物网中具有重要的生态意义,其摄食对海洋生态系统的物质循环和能量流动具有重要影响[1-2]。准确测定自然海区中桡足类的食物组成及摄食率等是定量理解它们与其它海洋生物之间的营养关系、有害藻华的生消、水体中有机物的垂直通量以及近海碳循环等过程的关键,也是建立可靠生态动力学模型,提供准确海洋渔业预测的重要步骤[3]。
中华哲水蚤(Calanussinicus)广泛分布于西北太平洋陆架海区,是该水域的特征性桡足类[4],同时,也是我国渤、黄、东海等近岸水域浮游动物的优势种和关键种[5-11]。有关中华哲水蚤的食物组成已有诸多报道[12-19],这些关于中华哲水蚤摄食的研究主要使用培养法、基于色素测定和镜检鉴定的肠道内含物分析法以及脂肪酸标记物法等,由于种种局限(培养法脱离现场水体环境,受到瓶颈效应影响;色素测定受限于色素降解,且只能检测有色素食物;显微镜检法需要专门的形态分类知识,费时耗力,准确性受样品形态影响很大;脂肪酸法受限于其表征食物种类时的不稳定性等)[20-21],所得数据的准确度离得知桡足类的现场(insitu)摄食食性还相差较远。
分子生物学技术在研究摄食[22-23]及寄生[24]等营养关系中具有明显的优势,可避开许多其它方法的缺点和局限。近些年来,已有学者开始探索将分子生物学技术应用于桡足类的摄食习性分析中[20],并且通过一系列的实验证明了分子生物学方法是研究桡足类摄食和水域食物网营养物质传递的有效手段[25-26]。本文使用PCR的分子生物学技术,以核糖体小亚基基因(18S rDNA)为目标序列,通过非桡足类(non-copepod)引物的运用,研究了黄河口邻近水域中华哲水蚤的现场食物组成。本研究期望通过优化桡足类摄食的分子生物学研究方法,为分析桡足类的现场食性提供新的认识,并为深入理解桡足类在海洋食物网中的作用提供方法支持和科学依据。
1 材料与方法
1.1 样品采集及现场处理
浮游动物样品于2011年5月在黄河口邻近水域(37°58′30.20″N,119°33′37.70″E,水深16m)采用浅水Ⅰ型浮游生物网(网口直径为50cm,筛绢孔径为0.505 mm)进行垂直拖网采得。为避免拖网时间过长以及拖网速度过快可能造成的误差(时间过长可能会使高密度的目标桡足类在网底管处存在非自然状态下的摄食行为,拖网速度过快可能会刺激桡足类排便)[27],拖网速度控制在0.5m/s左右。样品采集后立即使用中性鲁哥氏液(Uterm9hl’s solution)[28]固定,并保持鲁哥氏液的终浓度为2%。
1.2 中华哲水蚤的室内处理
现场固定的浮游动物样品带回实验室后,在体视显微镜(Lecia,S8APO)下将中华哲水蚤个体挑出,并置于0.22μm滤膜过滤的现场海水中(加入中性鲁哥氏液,使终浓度为2%)暂存。DNA提取前对样品进行清洗、去除附肢、镜检等处理,以确保在清洗之后中华哲水蚤的体表没有其它生物或者碎屑附着。中华哲水蚤个体在挑选、清洗和去除附肢等DNA提取前处理的过程中采用无菌技术和无菌操作。
1.3 总基因组DNA的提取
使用 Axygen研磨棒(Fisher Scientific)对样品进行充分研磨。待样品研磨至匀浆后加入400μL DNA提取缓冲液[29](1%SDS,100mmol·L-1EDTA pH 8.0和200μg·mL-1蛋白酶K),55℃温育2d进行细胞裂解,使用CTAB方法进行基因组DNA(以下简称DNA)的提取,最终将所获取的DNA溶解在20~50μL 10mmol/L Tris-Cl(pH 8.0)中,并按照Zhang等报道的方法对提取的DNA进行纯化[29],获取的DNA提取储存于-20℃。
1.4 PCR扩增与基因克隆
为了得到桡足类以外的尽可能多的其它真核生物(潜在食物)类群的序列,本实验使用18Snon-copepod引物(18Snon-copepod F2 和 non-copepod R2)(见表1)对提取的DNA进行PCR扩增。25μL反应体系包含2.5μL 10× PCR Buffer(含 Mg2+),2μL 20mmol/L的dNTPs,5μmol/L的正反向引物各0.5,0.2μL的ExTaq酶(Takara),1μL的DNA模板。反应条件为:94℃预变性60s;94℃变性30s,68℃退火和延伸70s,10个循环;94℃变性15s,58℃退火30s,72℃延伸40s,25个循环;最后72℃延伸10min。

表1 本研究中所使用的引物序列Table 1 Information of primer sequences used in this study
另外,使用真核生物通用引物18ScomF1和comR1(见表1)对中华哲水蚤基因组DNA进行扩增,获取中华哲水蚤18SrDNA序列,扩增条件为94℃预变性1min;95℃变性20s,56℃退火30s,72℃延伸45s,共30个循环;最后72℃延伸10min。
PCR产物使用 Agarose Gel DNA Purification Kit v2.0(Takara)进行纯化。回收后进行PCR产物末端加A、连接、转化、培养、蓝白斑筛选、质粒DNA提取与酶切等一系列基因克隆过程,具体方法与步骤参见[30]。随机挑选一定数目的阳性克隆进行序列测定。
1.5 序列比对和分析
对所获原始序列进行手动检查,并去掉载体和引物序列,得到引物真正扩增出的序列。基于BLAST[31]在NCBI的GenBank数据库中寻找同源序列信息。使用Clustal X v2.1[32]将所匹配到的最相似序列(代表最佳匹配类群或种类)与实验所获取序列进行比对,然后使用 MEGA 5.0[33]构建邻接树(Neighbor Joining tree,N-J tree)。所用核苷酸替代模型为 Kimura 2-parameter模型,Gamma值由jModelTest[34]计算获得。自举值(Bootstrap value)为1 000,其它参数设置均为默认值。
另外,基于NCBI的分类浏览器命名法(Taxonomy Browser nomenclature),以所获取序列BLAST时匹配到的前几条(一般为5条左右)序列为基准,对每条BLAST时与GenBank数据库中最佳匹配序列的相似性大于94%的序列推定了其所能确定到的最低分类阶元。
2 研究结果
2.1 中华哲水蚤的分子生物学鉴定
对中华哲水蚤除进行形态鉴定以外,使用真核生物核糖体小亚基通用引物(18ScomF1和comR1)扩增其DNA。所得1.7kb序列与GenBank数据库已登记的中华哲水蚤序列(GU969174)相似度达到99%~100%。这也从18SrDNA序列水平上证实了实验所用目标桡足类为中华哲水蚤。
2.2 中华哲水蚤食物18SrDNA序列的扩增
使用18Snon-copepod F2和non-copepod R2扩增中华哲水蚤现场样品的总DNA中非中华哲水蚤来源的DNA(中华哲水蚤的食物等其它种类的DNA),所得基因序列丰富多样,来源于不同生物类群(见表2),表明中华哲水蚤可能有着广泛的食物谱。
基于18Snon-copepod引物扩增出的PCR克隆文库共获取32条序列,其中7条为中华哲水蚤来源。剩余的25条非中华哲水蚤来源的序列分属于硅藻、甲藻、隐藻、绿藻、水螅水母类、栉水母类、软体动物和Ichthyosporea等8个门类。其中,水母类(栉水母类、水螅水母类)占有显著优势地位,各类群所占比例见图1。

表2 2011年5月黄河口邻近水域中华哲水蚤样品中扩增出的潜在食物Table 2 Taxonomic distribution of potential diets retrieved frominsitufixed Calanussinicus sampled in Yellow River Estuary and adjacent waters in May,2011
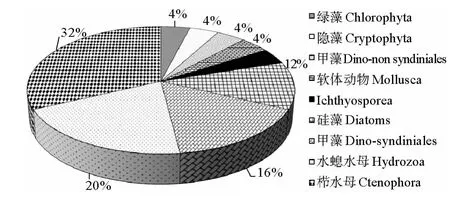
图1 用18Snon-copepod引物扩增中华哲水蚤样品所得非中华哲水蚤来源序列中各生物类群及所占比例Fig.1 Lineages and corresponding proportions of non-Calanussinicus origin 18SrDNA sequences amplified frominsitufixed Calanussinicus
在检测出的生物类群中,占最优势地位的是栉水母类(32.0%)。8条栉水母来源序列中有5条(YRE-I-15~19)序列可确定的最低分类阶元为目,属于栉水母门Typhlocoela纲中的球栉水母目(Cydippida)(相似度为98%~99%),从亲缘关系可以看出(见图2),这5条序列很可能来自同一个属,甚至是同一种。其余3条栉水母序列(YRE-I-12~14)与上述5条序列以及GenBank中的已知同源序列相似度较低。其中,序列YRE-I-14的最低分类阶元可确定到球栉水母目中的Mertensiidae科,YRE-I-13的最低分类阶元到栉水母门触手纲(Tentaculata),进一步的分类阶元不能确定,而序列YRE-I-12因与已知序列的相似度较低(90%),故未进行最低分类阶元的推定(见表2)。具有第二优势的序列来源于水螅水母类,其中3条为水螅水母纲花水母(Anthomedusae)来源,另外2条来源于软水母(Leptomedusae)。样品中水母类(栉水母类和水螅水母类)来源的序列所占比例达52.0%。
甲藻来源的5条序列处于第三优势地位,其中4条(YRE-I-07~10)很可能来源于寄生性的甲藻Syndiniales(见表2),系统发育关系分析显示,它们以较高的自举值(85)和已知的Syndiniales类群聚在一起。其中YRE-I-09与 YRE-I-10可确定的最低分类阶元到Syndiniales中的Euduboscquella属(这2条序列与Euduboscquellasp.JN934989 的相似度为 96%)。序列YRE-I-08因与已知序列的相似度较低(93%),故未进行可确定的最低分类阶元的推定。序列YRE-I-07可确定的最低分类阶元为Syndiniales中的另一个寄生性类群Ichthyodinium属。第5条甲藻序列(YRE-I-06)可确定的最低分类阶元为甲藻纲(Dinophyceae),可能来源于裸甲藻目(Gymnodiniales)中的裸甲藻科(Gymnodiniaceae)。
此外还检测到硅藻的序列,占总非中华哲水蚤序列的12.0%,它们均来源于中心硅藻纲(Coscinodisco-phyceae)海链藻目(Thalassiosirales)海链藻科(Thalassiosiraceae)海链藻属(Thalassiosira)。剩余4条序列中有2条分别隶属于绿藻门(Chlorophyta)石莼纲(Ulvophyceae)石莼目(Ulvales)石莼科(Ulvaceae)石莼属(Ulva)(与UlvarigidaAJ005414的相似度为99%)和软体动物(与RissoellarissoaformisFJ917214的相似度为85%)。另外的2条序列分别来源于隐藻和Ichthyosporea,一种介于原生生物和真菌之间的寄生生物。
3 讨论
浮游动物与其食物之间的营养关系是海洋生源要素循环的重要影响因子,理解这类营养关系有助于提高和完善对生态系统、气候和渔业预测及管理模型等的认识[20]。在传统测定方法(如解剖法和肠道色素分析法)受到局限时,分子生物学手段提供了一个能够准确认识浮游动物现场食物组成的新方法。
以往基于镜检法的中华哲水蚤摄食研究表明,渤海中华哲水蚤成体[13]和桡足幼体[14]的食物组成类似,均是以圆筛藻为代表的硅藻占绝对优势,推测中华哲水蚤为植食性桡足类,并且对圆筛藻表现出明显的摄食选择性。也有研究指出,厦门海域的中华哲水蚤也营植食性生活,以硅藻为主要食物,但是对饵料基本没有选择性摄食[12]。需要注意的是,基于镜检的肠道内含物分析法常常会因为方法本身局限性(例如:圆筛藻具有厚硅质外壳,不易消化,更容易残留在消化道内而被检出,而甲藻等其它类群却因为桡足类的消化产生出很多难以辨认的残留,有些种类例如纤毛虫等甚至没有残余留下)造成误差。类似情况也发生在使用扫描电镜分析中华哲水蚤食物的研究中。虽然通过可辨认的生物成分表明台湾东北部海域中华哲水蚤的食物组成是以当地浮游植物中的硅藻为主,但是有很大部分是不可辨认的食糜、海雪和浮游植物的残留[18]。另外,Liu等基于脂肪酸组成研究了胶州湾中华哲水蚤的食物组成,认为中华哲水蚤除了浮游植物之外还可能摄食有机碎屑、细菌及小型桡足类等[19]。
与以往研究中中华哲水蚤摄食食物的种类和范围相比,基于18Snon-copepod引物对黄河口邻近水域中华哲水蚤食物的分子生物学分析检测出属于8个门的生物,揭示了多样化的潜在食物类群(见图2)。这一结果拓展了对于中华哲水蚤食物范围的认识。同时,多样化生物类群的检出也显示出分子生物学技术在桡足类摄食研究中的强大效用和优势。
3.1 水母——中华哲水蚤可能的食物类群

图2 黄河口邻近水域中华哲水蚤现场样品使用18S通用引物和non-copepod引物所获取的序列与在GenBank中匹配到的同源序列比对后构建的邻接树(N-J树)Fig.2 Neighbor joining tree of 18SrDNA sequences we obtained by 18Snon-copepod primers and their top-hit sequences from GenBank
基于18Snon-copepod引物的分子生物学分析检测到了包括栉水母和水螅水母在内的水母类来源的序列。其中,栉水母来源的序列属于栉水母门中的球栉水母目(相似度为98%~99%)(见表2)。同步的大面调查资料显示,黄河口及其邻近水域分布的栉水母仅记录到球栉水母目中的球形侧腕水母(Pleurobrachia globosa)一种,在浅水I和浅水II型浮游生物网采集的浮游动物样品中均有发现[35]。马喜平和高尚武对渤海水母类的研究表明,渤海有2种栉水母——球形侧腕水母和兜水母(Bolinopsis)中的瓜水母(Boroecucumis)[36]。作者在GenBank数据库中没有检索到瓜水母的18SrDNA序列信息,这可能是栉水母来源的序列匹配的最相近种类中没有瓜水母的原因。
水螅水母来源的序列主要属于花水母和软水母。花水母和软水母是黄河口及其邻近水域[35]和渤海[37]常见的水螅水母,其中,八斑芮氏水母(Rathkeaoctopunctata)和和平水母属(Eirene)是渤海水母类的优势种属[36]。特别是八斑芮氏水母,同步的大面积调查资料显示,调查区域内八斑芮氏水母可占到水母总生物量的95%以上[35]。对渤海水母类的研究也表明,八斑芮氏水母的密度在5月份可占到水母类总密度的80%以上[38],且主要出现在莱州湾。由此可见,中华哲水蚤潜在的食物类群——水螅水母和栉水母在调查时间和区域内均具有丰富的分布,意味着中华哲水蚤可能和它们有着很高的遭遇率。
一般认为,水母是水域生态系统重要的摄食者,是浮游植物、浮游动物、鱼类卵和仔稚鱼的主要消费者之一。关于八斑芮氏水母等与桡足类的营养关系研究中皆是八斑芮氏水母摄食桡足类的报道[39-40]。其它种类的水母(杯水母(Phialidiumlomae,P.gregarium)、萨氏水母(Sarsiapsinceps)、囊水母(Euphysatentaculata)、半口壮丽水母(Aglaurahemistoma)、海月水母(Aureliaaurita))等的摄食研究中也发现它们对桡足类的摄食[40-46],并且有研究指出桡足类是某些水母种类的主要食物来源[45]。本文基于分子生物学技术的分析在中华哲水蚤的潜在食物类群中检测出栉水母类和水螅水母类,而且这2个类群在所有非中华哲水蚤来源的生物类群中占有绝对优势地位(52.0%)。除此之外,作者在青岛近海的中华哲水蚤(2004年1月和2010年6月)、以及胶州湾的太平洋纺锤水蚤(2010年9月)样品中均检出数量丰富的水母类来源的序列(占潜在食物类群18SrDNA文库的比例分别为44.4%、41.2%和68.4%)。潜在食物类群中水母类的普遍检出和丰富比例意味着中华哲水蚤对这2类水母可能存在很强的摄食作用。
中华哲水蚤是一种杂食性桡足类。基于胃肠内含物电镜分析的研究表明,其主要摄食以硅藻为主的浮游植物,还有少量的原生动物以及细菌等[18]。另外,基于脂肪酸组成分析的研究表明,中华哲水蚤除主要摄食浮游植物之外,还可能摄食有机碎屑、细菌及小型桡足类等[19]。尽管如此,以前的研究从未在其动物性饵料中发现水螅水母和栉水母类生物,迄今为止,作者也未发现有关浮游桡足类对水母类生物摄食的报道。基于摄食者和食物大小关系的考虑,作者认为中华哲水蚤对栉水母和水螅水母类中某些种类的摄食可能发生在卵到浮浪幼虫阶段。据此推测,桡足类可能在控制水母暴发、影响海洋生态系统平衡中具有重要的潜在作用。
3.2 寄生性甲藻类群与中华哲水蚤的关系
Syndiniales是甲藻中的一大类群,其中许多均为寄生性种类。它们具有广泛的宿主,包括其它甲藻、纤毛虫、放射虫、鱼卵以及桡足类在内的甲壳动物等[47-48]。5条甲藻来源的序列中4条属于寄生性的Syndiniales,其中3条隶属于其中的Euduboscquella属,1条可能来源于Ichthyodinium属。作为Syndiniales中的一个类群,Euduboscquella属的研究较少,在已被描述的种类中,多是它们遗传多样性和系统发育关系的研究[49],未见有其与桡足类营养关系的报道。Ichthyodinium属则经常被发现寄生于鱼卵的卵黄囊内[50]。
有许多生物与桡足类形成共生关系[51],Syndiniales便是其中一个类群。较传统方法,分子生物学方法的使用让作者发现了更多与桡足类有关联的生物类群,这些类群可能与桡足类形成共生关系,而与桡足类有着复杂的营养关系。虽然作者以研究桡足类的食物为目的,但认为基于序列分析检出的Syndiniales这2个属(Euduboscquella和Ichthyodinium)的种类与桡足类真实的营养关系尚不能判定为简单的摄食与被摄食的关系,而需要借助其他手段进行更加深入地研究。
3.3 大型藻类在中华哲水蚤食物类群中的出现
通常,浮游食物网中有机物总是从小个体生物向较大个体生物依次传递,“小个体”的浮游动物对“大个体”的大型水生植物摄食的研究报道相对较少。曾有研究将大型水生植物Thalassiatestudinum的碎屑作为饵料,在实验室条件下验证了汤氏纺锤水蚤(Acartia tonsa)对这种海草碎屑的摄食[52],但未见自然海区中有桡足类对大型水生植物摄食的报道。然而,曾有学者观察到并且使用分子生物学手段证明了近岸潮间带池塘中营混合营养的纤毛虫Strombidiumoculatum和S.stylifer对大型绿藻中浒苔(Ulva)属种类的摄食[53],发现纤毛虫可以摄食绿藻释放的游动孢子细胞。作者在样品中检测到绿藻来源的序列,属绿藻门石莼属。石莼一般生长在海湾内中低潮带的岩石上或石沼中,广泛分布于我国黄渤海沿岸[54],在潮间带的低、中、高潮带均有分布[55]。石莼的生活史存在世代交替,随环境变化,藻体能释放大量孢子。基于实验结果,本文认为中华哲水蚤可能是石莼属藻类孢子或配子细胞的潜在摄食者,虽然也有可能摄食DNA未被完全降解的绿藻碎屑。
传统认识中,大型藻类和海草等的归宿是进入到碎屑食物链或被大型底栖生物所摄食[56],进入到底栖食物链。本研究基于分子生物学检测的结果与McManus等[53]关于纤毛虫对大型藻类的摄食研究类似,展示了一个别样的营养关系,表明大型藻类可以进入到浮游食物网中。作为一种繁殖策略,在有外界压力胁迫或处于有利扩散条件时,绿藻可快速产出大量的孢子细胞。有研究表明大型石莼属绿藻Ulvalactuca在生长季节可以释放其生物量20%~60%的游动孢子细胞[57]。对于潮间带池塘或近岸水域中的浮游生物,这种大型植物孢子的释放可能给它们提供了周期性的重要食物来源。
致谢:感谢本课题组的房静和李自尚等同学协助采集样品。
[1]Turner J.The importance of small planktonic copepods and their roles in pelagic marine food webs[J].Zoological Studies,2004,43(2):255-266.
[2]Zhang H,Finiguerra M,Dam H G,et al.An improved method for achieving high-quality RNA for copepod gene transcriptomic studies[J].Journal of Experimental Marine Biology and Ecology,2013,446:57-66.
[3]Verity P G,Smetacek V,Smayda T J.Status,trends and the future of the marine pelagic ecosystem [J].Environmental Conservation,2002,29(2):207-237.
[4]Uye S.Why doesCalanussinicusprosper in the shelf ecosystem of the Northwest Pacific Ocean?[J].ICES Journal of Marine Science,2000,57(6):1850-1855.
[5]陈清潮.中华哲水蚤的繁殖、性比率和个体大小的研究 [J].海洋与湖沼,1964,6(3):272-288.
[6]毕洪生,孙松,高尚武,等.渤海浮游动物群落生态特点Ⅱ桡足类数量分布及变动 [J].生态学报,2001,21(02):177-185.
[7]李超伦,王荣,孙松.南黄海鳀产卵场中华哲水蚤的数量分布及其摄食研究 [J].水产学报,2003,27(S1):55-63.
[8]Li C L,Sun S,Wang R,et al.Feeding and respiration rates of a planktonic copepod (Calanussinicus)oversummering in Yellow Sea Cold Bottom Waters[J].Marine Biology,2004,145(1):149-157.
[9]Xu Z L,Chen B J.Seasonal distribution ofCalanussinicus(Copepoda,crustacea)in the East China Sea[J].Acta Oceanologica Sinica,2007,26(3):150-159.
[10]Xu Z L,Ma Z L,Wu Y M.Peaked abundance ofCalanussinicusearlier shifted in the Changjiang River(Yangtze River)Estuary:a comparable study between 1959,2002and 2005[J].Acta Oceanologica Sinica,2011,30(3):84-91.
[11]马静,陈洪举,刘光兴.2007年夏季黄河口及其邻近水域浮游动物的群落特征 [J].中国海洋大学学报:自然科学版,2012,42(5):74-80.
[12]李少菁.厦门几种海洋浮游桡足类的食性与饵料成分的初步研究[J].厦门大学学报:自然科学版,1964,11(03):93-109.
[13]杨纪明.渤海中华哲水蚤摄食的初步研究 [J].海洋与湖沼,1997,28(4):376-382.
[14]杨纪明.渤海中华哲水蚤桡足幼体摄食的初步研究 [J].海洋水产研究,1998,19(2):1-4.
[15]Zhang G T,Li C L,Sun S,et al.Feeding habits ofCalanussinicus(Crustacea:Copepoda)during spring and autumn in the Bo-hai Sea studied with the herbivore index [J].Scientia Marina,2006,70(3):381-388.
[16]孙军,宋书群,徐兆礼,等.东海米氏凯伦藻水华中中华哲水蚤的选择性摄食 [J].海洋与湖沼,2007,38(6):536-541.
[17]Huo Y Z,Wang S W,Sun S,et al.Feeding and egg production of the planktonic copepodCalanussinicusin spring and autumn in the Yellow Sea,China[J].Journal of Plankton Research,2008,30(6):723-734.
[18]Chen M R,Samba K A,Hwang J S.Diet of the copepodCalanus sinicusBrodsky,1962 (Copepoda,Calanoida,Calanidae)in northern coastal waters of Taiwan during the northeast monsoon period[J].Crustaceana,2010,83(7):851-864.
[19]Liu M T,Li C L,Sun S.Seasonal variation in fatty acid composition of seston and the copepodCalanussinicus(Brodsky,1962)in Jiaozhou Bay and its trophic implications[J].Chinese Journal of Oceanology and Limnology,2011,29(6):1164-1173.
[20]Nejstgaard J,Frischer M,Raule C,et al.Molecular detection of algal prey in copepod guts and fecal pellets[J].Limnology and O-ceanography:Methods,2003,1:29-38.
[21]Boling W,Sinclair G,Wawrik B.Identification of calanoid copepod prey species via molecular detection of carbon fixation genes[J].Marine Biology,2012,159(5):1165-1171.
[22]Symondson W O C.Molecular identification of prey in predator diets[J].Molecular Ecology,2002,11(4):627-641.
[23]King R A,Read D S,Traugott M,et al.Molecular analysis of predation:a review of best practice for DNA-based approaches[J].Molecular Ecology,2008,17(4):947-963.
[24]Guo Z L,Liu S,Hu S,et al.Prevalent ciliate symbiosis on copepods:high genetic diversity and wide distribution detected using small subunit ribosomal RNA gene[J].PLoS ONE,2012,7(9):844-847.
[25]Troedsson C,Frischer M E,Nejstgaard J C,et al.Molecular quantification of differential ingestion and particle trapping rates by the appendicularianOikopleuradioicaas a function of prey size and shape[J].Limnology and Oceanography,2007,52(1):416-427.
[26]Durbin E G,Casas M C,Rynearson T A.Copepod feeding and digestion rates using prey DNA and qPCR [J].Journal of Plankton Research,2012,34(1):72-82.
[27]B mstedt U,Gifford D J,Irigoien X,et al.Feeding[M].∥ Harris R,Wiebe P,Lenz J,et al.eds.ICES zooplankton methodology manual.London:Academic Press,2000.
[28]Uterm hl H.Zur vervollkommnung der quantitativen phytoplankton methodik mitt[J].Int Verein Limnol,1958,9:1-38.
[29]Zhang H,Lin S J.Development of acob-18SrRNA gene real-time PCR assay for quantifyingPfiesteriashumwayaein the natural environment [J].Applied and Environmental Microbiology,2005,71(11):7053-7063.
[30]Zhuang H,Bhattacharya D,Lin S J.Phylogeny of dinoflagellates based on mitochondrial cytochrome b and nuclear small subunit rDNA sequence comparisons[J].Journal of Phycology,2005,41(2):411-420.
[31]Altschul S F,Madden T L,Schffer A A,et al.Gapped BLAST and PSI-BLAST:A new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
[32]Larkin M A,Blackshields G,Brown N P,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[33]Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].ICES Molecular Biology and Evolution,2011,28(10):2731-2739.
[34]Posada D.jModelTest:phylogenetic model averaging[J].Molecular Biology and Evolution,2008,25(7):1253-1256.
[35]李自尚.春季黄河口及其邻近水域浮游动物群落特征与粒径谱的初步研究 [D].青岛:中国海洋大学,2012.
[36]马喜平,高尚武.渤海水母类生态的初步研究——种类组成、数量分布与季节变化 [J].生态学报,2000,20(4):533-540.
[37]毕洪生,孙松,高尚武,等.渤海浮游动物群落生态特点Ⅲ.部分浮游动物数量分布和季节变动 [J].生态学报,2001,21(4):513-521.
[38]孙松,张芳,李超伦,等.黄海小型水母的分布特征 [J].海洋与湖沼,2012,43(03):429-437.
[39]Zelickman E A,Gelfand V I,Shifrin M A.Growth,reproduction and nutrition of some Barents Sea hydromedusae in natural aggregations[J].Marine Biology,1969,4(3):167-173.
[40]Matsakis S,Conover R J.Abundance and feeding of medusae and their potential impact as predators on other zooplankton in Bedford Basin(Nova Scotia,Canada)during Spring[J].Canadian Journal of Fisheries and Aquatic Sciences,1991,48(8):1419-1430.
[41]Larson R J.Daily ration and predation by medusae and ctenophores in Saanich Inlet,B.C.,Canada[J].Netherlands Journal of Sea Research,1987,21(1):35-44.
[42]Colin S P,Costello J H,Graham W M,et al.Omnivory by the small cosmopolitan hydromedusaAglaurahemistoma[J].Limnology and Oceanography,2005,50(4):1264-1268.
[43]Omori M,Ishii H,Fujinaga A.Life history strategy ofAurelia aurita(Cnidaria,Scyphomedusae)and its impact on the zooplankton community of Tokyo Bay [J].ICES Journal of Marine Science,1995,52(3-4):597-603.
[44]Ishii H,Tanaka F.Food and feeding ofAureliaauritain Tokyo Bay with an analysis of stomach contents and a measurement of digestion times[J].Hydrobiologia,2001,451(1-3):311-320.
[45]Lo W T,Purcell J E,Hung J J,et al.Enhancement of jellyfish(Aureliaaurita)populations by extensive aquaculture rafts in a coastal lagoon in Taiwan [J].ICES Journal of Marine Science,2008,65(3):453-461.
[46]陈丽华,陈钢,李少菁,等.厦门港球型侧腕水母(Pleurobrachia globosa)的摄食研究 [J].厦门大学学报:自然科学版,2003,42(2):228-232.
[47]Coats D W.Parasitic life styles of marine dinoflagellates [J].Journal of Eukaryotic Microbiology,1999,46(4):402-409.
[48]Stentiford G D,Shields J D.A review of the parasitic dinoflagellatesHematodiniumspeciesandHematodinium-likeinfections in marine crustaceans[J].Diseases of Aquatic Organisms,2005,66(1):47-70.
[49]Bachvaroff T R,Kin S,Guillou L,et al.Molecular diversity of the syndinean genusEuduboscquellabased on single-cell PCR analysis[J].Applied and Environmental Microbiology,2012,78(2):334-345.
[50]Skovgaard A,Meneses I,Anglico M M.Identifying the lethal fish egg parasiteIchthyodiniumchabelardi as a member of Marine Alveolate Group I[J].Environmental Microbiology,2009,11(8):2030-2041.
[51]Ohtsuka S,Horiguchi T,Hanamura Y,et al.Symbiosis of planktonic copepods and mysids with epibionts and parasites in the North pacific:diversity and interactions [J].New Frontiers in Crustacean Biology,2011,1-14.
[52]Roman M R.Utilization of detritus by the copepod,Acartiatonsa[J].Limnology and Oceanography,1984,39(5):949-959.
[53]McManus G,Zhuang H,Lin S J.Marine planktonic ciliates that prey on macroalgae and enslave their chloroplasts[J].Limnology and Oceanography,2004,49(1):308-313.
[54]李秀辰,谷晓华,张国琛,等.石莼属海藻的环境增殖及生物质能开发潜力 [J].水产科学,2011,30(12):789-793.
[55]Zeng C K.Common seaweeds of China[M].Beijing:Science Press,1983.
[56]Vanderklift M A,Kendrick G A.Contrasting influence of sea urchins on attached and drift macroalgae[J].Marine Ecology Progress Series,2005,299:101-110.
[57]Niesenbaum R A.The ecology of sporulation by the macroalgaUlvalactucaL.(chlorophyceae)[J].Aquatic Botany,1988,32(1-2):155-166.
