2012~2013年山东省禽源大肠杆菌中质粒介导喹诺酮类药物耐药基因的检测
2014-04-13孔令超李志国张瑞华焦玉祥马明杰郭翠平朱岩丽谢之景姜世金
孔令超,李志国,张瑞华,焦玉祥,马明杰,郭翠平,朱岩丽,谢之景,姜世金
(山东农业大学动物医学院 山东省动物生物工程与疾病防治重点实验室,泰安 271018)
·简报·
2012~2013年山东省禽源大肠杆菌中质粒介导喹诺酮类药物耐药基因的检测
孔令超,李志国,张瑞华,焦玉祥,马明杰,郭翠平,朱岩丽,谢之景,姜世金
(山东农业大学动物医学院 山东省动物生物工程与疾病防治重点实验室,泰安 271018)
为研究近年来山东省禽源致病性大肠杆菌中质粒介导喹诺酮类药物耐药(plasmid-mediated quinolone resistance,PMQR)基因的基因型分布,及其对喹诺酮类抗生素的耐药性的影响,分别采用针对qnrA、qnrB、qnrC、qnrD、qnrS、oqxA、oqxB与qepA 8个耐药基因的通用引物,对93株2012~2013年分离自山东省的禽源大肠杆菌进行PCR检测,并对其进行了5种喹诺酮类药物的药敏试验。结果表明山东省禽源大肠杆菌对5种喹诺酮类抗生素均产生了较高耐药性(50.54%~86.30%);PMQR基因携带率达到60.21%(56/93),其中26.88%(25/93)的菌株携带2种PMQR基因,1.07%(1/93)的菌株携带3种PMQR基因;qnrA、qnrB、qnrC、qnrD与qepA基因未被检测到,qnrS、oqxA和oqxB基因在山东省禽源致病性大肠杆菌中分布较为广泛,其检出率依次为22.58%(21/93)、40.86%(38/93)和24.73%(23/93)。
禽源致病性大肠杆菌;药敏试验;质粒介导喹诺酮类药物耐药基因;PCR
喹诺酮类药物是一类广谱高效抗菌药物,在人医和兽医临床中被广泛应用。已投入使用或即将进入兽医领域的药物有10多种,主要有两类,一类从人医用移植转化而来,如诺氟沙星、环丙沙星、氧氟沙星、培氟沙星、洛美沙星等;另一类是动物专用品种,己批准上市的兽医专用喹诺酮类药物有德国拜耳公司生产的恩诺沙星、美国雅培公司生产的沙拉沙星和二氟沙星、美国辉瑞公司生产的单诺沙星、瑞士罗氏公司生产的麻保沙星、日本武田制药生产的倍诺沙星以及日本大日本制药生产的奥比沙星。近年来细菌对喹诺酮类药物耐药率逐年上升,引起了人们的高度关注。喹诺酮类药物的耐药机制主要是引起细菌中染色体介导药物作用靶位的改变、外膜通透性的改变以及外排泵过度表达[1]。
1998年,Martinez-Martinezd等[2]首次在一株肺炎克雷伯氏菌中存在的一个可介导喹诺酮类耐药的质粒pMG252上发现了qnrA基因,进一步研究发现qnr基因可在不同细菌中迅速水平传播,其表达的Qnr蛋白能够与喹诺酮类药物以及喹诺酮类药物的作用靶位相互作用,保护细菌的DNA解旋酶不被喹诺酮类药物所抑制,从而产生耐药。随后,在中国、土耳其、美国、澳大利亚、德国、法国、韩国、意大利、日本等国家均发现了携带 qnr 基因的临床分离大肠杆菌[3-11]。目前发现的qnr 基因包括 qnrA、qnrB、qnrC、qnrD和qnrS,均为质粒所携带,其中qnrA、qnrB和qnrS的一级结构非常相似[2,12-15]。
近年来,引起细菌耐药的外排泵机制受到研究者的高度关注。目前已经发现了3种质粒介导的喹诺酮类转运蛋白:OqxA和OqxB和QepA[16,17]。外排泵oqxA和oqxB基因可介导细菌对喹诺酮、喹乙醇、氯霉素的耐药,二者是在猪源大肠杆菌的耐药质粒POLA52中发现的[16]。外排泵qepA基因则于2002年在日本兵库县病人尿液样本中分离的大肠杆菌携带的耐药质粒pHPA中被发现,它可以介导对氨基糖苷类药物、氟喹诺酮类药物和广谱内酰胺酶类药物的多重耐药[17]。
为了解质粒介导喹诺酮类药物耐药(plasmid mediated quinolone resistance,PMQR)基因近年来在山东省禽源大肠杆菌中的流行和分布特点,本研究对2012~2013年间分离自山东省的93株禽源致病性大肠杆菌进行了5种喹诺酮类药物的药敏试验,并对qnrA、qnrB、qnrC、qnrD、qnrS、oqxA、oqxB与qepA 八个耐药基因进行了PCR 检测,为临床合理用药提供科学依据。
1 材料与方法
1.1 菌株93株禽源致病性大肠杆菌为2012~2013年分离自山东省临沂、泰安、济宁、聊城、德州、枣庄6个市的11个规模化养禽场的病死禽,由山东省动物生物工程与疾病防治重点实验室鉴定并保存。
1.2 试剂营养琼脂购自杭州天和微生物试剂有限公司;凝胶回收试剂盒购自美国OMEGA公司;pMD18-T载体、T4 DNA ligase、Taq酶和限制性内切酶等购自大连宝生物有限公司;质粒提取试剂盒、DNA Marker购自北京全式金生物技术有限公司;氧氟沙星、诺氟沙星、环丙沙星、洛美沙星、培氟沙星药敏片购自北京天坛药物生物技术开发公司。
1.3 PCR扩增引物参照参考文献[17-20]中的引物序列,由上海生工生物工程有限公司合成分别针对qnrA、qnrB、qnrC、qnrD、qnrS、oqxA、oqxB与qepA基因的通用引物(见表1),用于大肠杆菌PMQR基因型的检测。
1.4 药敏试验采用WHO推荐的纸片法(K-B法)对93株禽源致病性大肠杆菌进行5种常用喹诺酮类抗生素(诺氟沙星、氧氟沙星、环丙沙星、培氟沙星和洛美沙星)的药敏试验。
1.5 基因扩增与测序以上述93株禽源大肠杆菌菌体为模板,进行PCR扩增,反应体系(25 μL)如下:ddH2O 16.2 μL、10×Buffer (Mg2+free) 2.5 μL、MgCl2(25 mol/L) 2 μL、dNTP (2.5 mol/L) 2 μL、引物F 1 μL、引物R 1 μL、菌液0.1 μL、Taq DNA聚合酶0.2 μL。PCR扩增程序:94℃预变性10 min;94℃变性50 s,根据每对引物的不同选择不同的退火温度退火45 s,72℃延伸60 s,32个循环;72℃延伸10 min。0.8%琼脂糖凝胶电泳检查PCR产物。
1.6 基因测序随机抽取部分PCR阳性样品,将其PCR 产物纯化后连接到pMD18-T载体上,转化DH5α大肠杆菌进行阳性克隆的筛选,阳性克隆送上海生工生物工程有限公司进行序列测定。
2 结果与讨论
2.1 普通药敏纸片试验结果依据美国临床实验室国家标准化管理委员会(CLSI2008)标准判定敏感率、中介率、耐药率情况,药敏试验统计结果如表2。

表1 引物序列Table 1 The nucleotide sequence of the primers in this study
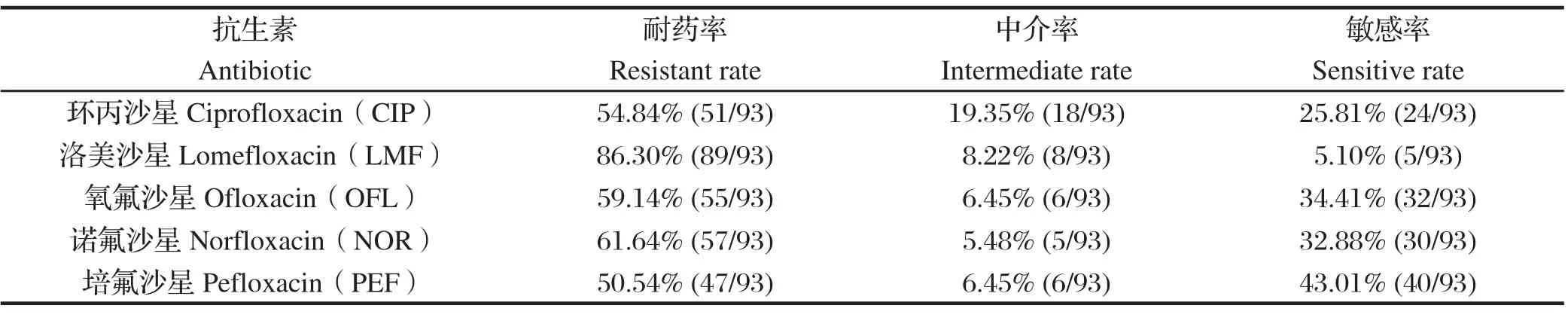
表2 93 株大肠杆菌对5种喹诺酮类药物的药敏试验结果Table 2 The susceptibility test results of the 93 E. coli isolates to 5 kinds of quinolones
由表2可知山东省禽源致病性大肠杆菌对喹诺酮抗生素已普遍产生耐药性。93株大肠杆菌对洛美沙星的耐药率最高,达到86.30%(80/93),而对诺氟沙星、氧氟沙星、环丙沙星、培氟沙星的耐药率则依次降低,分别为61.64%、59.14%、54.84%和50.54%。
2.2 耐药基因的PCR扩增结果及产物分析经PCR检测以及PCR阳性产物的克隆测序与分析,确定93株致病性大肠杆菌中有56株检出产PMQR的耐药基因,检出阳性率为60.21%;其中30株仅携带1种PMQR基因,占所有菌株的32.26%;25株携带2种PMQR基因,占所有菌株26.88%;1株携带3种PMQR基因,占所有菌株的1.07%。93株禽源大肠杆菌仅检测到了qnrS、oqxA和oqxB基因,检出率依次为22.58%(21/93)、40.86%(38/93)和24.73%(23/93),而未检出qnrA、qnrB、qnrC、qnrD与qepA基因型耐药基因。93株禽源致病性大肠杆菌中携带PMQR基因的情况见表3。

表3 93株大肠杆菌中产PMQR基因携带情况Table 3 Prevalence of PMQR genes among the 93 E. coli isolates
11个家禽场中分离到的禽源致病性大肠杆菌中携带PMQR基因的情况(见表4)。其中qnrS基因在不同地区分离菌株中的检出率为16.67%~33.33%,其中聊城市检出率最高,枣庄市检出率最低;oqxA基因在不同地区分离菌株中的检出率为33.33%~50.00%,临沂市检出率最高,聊城市检出率最低;oqxB基因在不同地区分离菌株中的检出率为16.67%~33.33%,临沂市检出率最高,聊城市检出率最低。结果说明这3种PMQR基因在山东省各个养禽场中分布已经相当普遍。3种耐药基因在地域分布上没有明显的规律可循,推测这可能和不同地区用药习惯性相关。

表4 11个养禽场大肠杆菌中产PMQR基因携带情况Table 4 Prevalence of PMQR genes among the E. coli isolates from 11 poultry-farms
2.3 耐药表型与耐药基因符合率结果对93株禽源致病性大肠杆菌对5种喹诺酮类抗生素的耐药表型与携带PMQR基因的基因型之间的符合率进行了分析:符合率=(PMQR基因检出阳性菌株数/耐药菌株数)×100%。结果表明耐药基因型与耐药表型之间符合率最高的为oqxA基因对培氟沙星,为80.85%;其次为oqxA型对环丙沙星、氧氟沙星与诺氟沙星,其符合率分别为74.51%、69.09%和66.67%;符合率最低的qnrS型对洛美沙星,为26.25%(见表5)。

表5 携带PMQR基因的大肠埃希菌的耐药表型与基因的符合率Table 4 Coincidence rate of antimicrobial resistant phenotype and PMQR genes in E. coli isolates
qnrS、oqxA、oqxB这3种PMQR基因在对喹诺酮类药物敏感和耐药禽源致病性大肠杆菌中的检出率结果表明,含与不含这3种PMQR基因的禽源致病性大肠杆菌对喹诺酮类抗菌药物敏感率差异无统计学意义,显示耐药表型与PMQR基因型之间无明显规律可循,这是因为细菌抗喹诺酮类药物的耐药机制比较复杂,而PMQR基因主要引起细菌对喹诺酮类药物的低水平耐药。对洛美沙星等喹诺酮类药物耐药的菌株的耐药基因型与耐药表型之间符合率较低,推断可能有其他其他耐药基因或机制的存在。
近年来,PMQR基因引起的细菌对喹诺酮类药物的耐药性引起了研究者的广泛关注[2-17]。PMQR基因可在不同菌株甚至不同菌种之间迅速水平传播,将会加快喹诺酮类药物耐药性在细菌间的传播,从而导致大肠杆菌对喹诺酮类药物的耐药性进一步增强。PMQR基因常与其他多种耐药基因共同整合,缩小了临床医生治疗相关细菌感染时选药或联合用药的空间[21,22]。目前,PMQR基因的广泛存在已经在世界范围内得到证实,各种基因的阳性率从1%~36%不等[23,24]。
家禽大肠杆菌病是兽医上常见的重要细菌病之一,其发病率高、致死率高、经济损失大、治疗难度大,因此对禽源大肠杆菌进行PMQR基因检测具有重意义。苏晶等[25]曾对2004~2011年分离自山东省的230株禽源致病性大肠杆菌进行了qnrA、qnrB和qnrS 三种基因的PCR检测,结果未检出qnrA和qnrB基因,qnrS基因检出率为16.52%(38/230)。本研究利用PCR方法对93株大肠杆菌中的8种PMQR基因进行检测及测序分析,结果仅检测到了qnrS、oqxA和oqxB基因,检出率依次为22.58%(21/93)、40.86%(38/93)和24.73%(23/93)。60.21%(56/93)的禽源致病性大肠杆菌中检出PMQR基因,其中30株仅携带1种PMQR基因,25株携带2种PMQR基因,1株携带3种PMQR基因,显示山东省禽源大肠杆菌中qnrS基因的携带率呈上升趋势,而且PMQR基因型越来越呈现多样性分布,需要在生产中加以高度重视。
喹诺酮类药物是人工合成的具有4-喹诺酮环结构的药物,其主要抗菌机制是通过抑制细菌的脱氧核糖核酸旋转酶(DNA解旋酶)和拓扑异构酶Ⅳ,干扰细菌DNA的复制而引起细菌死亡。目前,细菌尤其是大肠杆菌对喹诺酮类药物产生的耐药问题已经十分突出,主要耐药机制是细菌染色体上编码DNA解旋酶与拓扑异构酶的基因发生突变,导致喹诺酮类药物失去作用靶位、细菌细胞膜通透性的改变以及相关的外排泵系统过度表达,引起对喹诺酮类药物的高水平耐药[1]。这些耐药基因和机制可能是耐药基因型与耐药表型之间符合率较低的重要原因,进一步说明这些耐药菌株中可能还存在其他的对喹诺酮类药物的耐药基因或机制。
[1] 康德, 陈福祥, 倪语星, 等. 质粒qnr介导的肠杆菌科细菌对喹诺酮类药物耐药机制研究进展[J]. 中国抗生素杂志, 2009, 34(1): 7 -11.
[2] Martinez-Martinez L, Pascual A, Jacoby G A. Quinolone resistance from a transferable plasmid[J]. Lancet, 1998, 351(9105): 797-799.
[3] Wang M G, Tran J H, Jacoby G A, et al. Plasmidmediated quinolone resistance in clinical isolates of Escherichia coli from Shanghai, China[J]. Antimicrob Agents Chemother, 2003, 47(7): 2242-2248.
[4] Poirel L, Pitout J D D, Calvo L, et al. In vivo selection of fluo-roquinolone-resistant scherichia coli isolates expressing plasmid-mediated quinolone resistance and expanded- spectrumbeta-actamase[J]. Antimicrob Agents Chemother, 2006, 50(4): 1525-1527.
[5] Wang M G, Sahm D F, Jacoby G A, et al. Emerging plasmid-mediated quinolone resistance associated with the qnr Gene in Klebsiella pneumoniae clinical isolates in the United States [J]. Antimicrob Agents Chemother, 2004, 48(4): 1295-1299.
[6] Rodriguez-Martinez J M, Poirel L, Pascual A, et al. Plasmid-mediated quinolone resistance in Australia[J]. Microb Drug Resist, 2006, 12(2): 99-102.
[7] Jonas D, Biehler Y, Hartung D, et al. Plasmid-mediated quinolone resistance in isolates obtained in German intensive care units[J]. Antimicrob Agents Chemother, 2005, 49(2): 773-775.
[8] Nordmann P, Poirel L. Emergence of plasmid-mediated resistance to quinolones in Enterobacteriaceae[J]. Pathol Biol, 2006, 54 (1) : 7-9.
[9] Jeong J Y, Yoon H J, Kim E S, et al. Detection of qnr in clinical isolates of Escherichia coli from Korea[J]. Antimicrob Agents Chemother, 2005, 49(6) : 2522-2524.
[10] Perilli M, Forcella C, Celenza G, et al. Evidence for qnrB1 and aac(6’)-Ib-cr in CTX-M-15-producing uropathogenic Enterobacteriaceae in an Italian teaching hospital[J]. Diagn Microbiol Infect Dis, 2009, 64 (1): 90-93.
[11] Ode T, Saito R, Kumita W, et al. Analysis of plasmidmediated multidrug resistance in Escherichia coli and Klebsiella oxytoca isolates from clinical specimens in Japan[J]. Int J Antimicrob Agents, 2009, 34(4): 347-350.
[12] Jacoby G A, Walsh K E, Mills D M, et al. qnrB, another plasmid-mediated gene for quinolone resistance[J]. Antimicrob Agents Chemother, 2006, 50(4) : 1178-1182.
[13] Hata M, Suzuki M, Matsumoto M, et al. Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b[J]. Antimicrob Agents Chemother, 2005, 49(2): 801-803.
[14] Wang M, Guo Q, Xu X, et al. New plasmid-mediated quinolone resistance gene, qnrC, found in a clinical isolate of Proteus mirabilis[J]. Antimicrob Agents Chemother, 2009, 53(5): 1892-1897.
[15] Cavaco L M, Hasman H, Xia S, et al. qnrD, a novel gene conferring transferable quinolone resistance in Salmonella enterica serovar Kentucky and Bovismorbificans strains of human origin[J]. Antimicrob Agents Chemother, 2009, 53(2): 603-608.
[16] Yamane K, Wachino J, Suzuki S, et al. Plasmid-mediated qepA gene among Escherichia coli clinical isolates from Japan[J]. Antimicrob Agents Chemother, 2008, 52(4): 1564-1566.
[17] Kim H B, Wang M, Park C H, et al. oqxAB encoding a multidrug efflux pump in human clinical isolates of Enterobacteriaceae[J]. Antimicrob Agents Chemother, 2009, 53(8): 3582-3584.
[18] Guerra B, Helmuth R, Thomas K, et al. Plasmidmediated quinolone resistance determinants in Salmonella spp. isolates from reptiles in Germany[J]. J Antimicrob Chemother, 2010, 65(9): 2043-2045.
[19] Gay K, Robicsek A, Strahilevitz J, et al. Plasmidmediated quinolone resistance in non-Typhi serotypes of Salmonella enterica[J]. Clin Infect Dis, 2006, 43(3): 297-304.
[20] Liu J H, Deng Y T, Zeng Z L, et al. Coprevalence of plasmid-mediated quinolone resistance determinants QepA, Qnr, and AAC(6')-Ib-cr among 16S rRNA methylase RmtB-producing Escherichia coli isolates from pigs[J]. Antimicrob Agents Chemother, 2008, 52(8): 2992-2993.
[21] Wiegand I, Luhmer-Becher I, Wiedemann B. In vitro studies of oral Ciprofloxacin(CIP) doses: influence ofplasmid encoded floquinolone resistance determinant qnr[M]. The forty-fourth Interscience Conference on Antimicrobial Agents and Chemotherapy, Washington DC, 2004: Abstract A2-448.
[22] Hooper D C. Growing family of PMQR[M]. The fortyfifth Interscience Conference on Antimicrobial Agents and Chemotherapy, Washington D C, 2005: Abstract C1-1343.
[23] Ode T, Saito R, Kumita W, et al. Analysis of plasmidmediated multidrug resistance in Escherichia coli and Klebsiella oxytoca isolates from clinical specimens in Japan[J]. Int J Antimicrob Agents, 2009, 34(4): 347-350.
[24] Baudry P J, Nichol K, DeCorby M, et al. Mechanisms of resistance and mobility among multidrug-resistant CTXM-producing Escherichia coli from Canadian intensive care units: the 1st report of QepA in North America[J]. Diagn Microbiol Infect Dis, 2009, 63(3): 319-326.
[25] 苏晶, 杨芳芳, 孙慧, 等. 山东省禽源致病性大肠杆菌质粒介导喹诺酮类药物耐药基因的检测[J]. 山东农业科学, 2012, 44(9): 5-8.
DETECTION OF PLASMID-MEDIATED QUINOLONE RESISTANCE GENES IN AVIAN ESCHERICHIA COLI ISOLATES FROM SHANDONG PROVINCE IN 2012-2013
KONG Ling-chao, LI Zhi-Guo, ZHANG Rui-hua, JIAO Yu-xiang, MA Ming-jie, GUO Cui-ping, ZHU Yan-li, XIE Zhi-jing, JIANG Shi-jin
(Shandong Province Key Laboratory of Animal Biotechnology and Disease Control and Prevention, College of Veterinary Medicine, Shandong Agriculture University, Taian 271018, China )
To investigate the distribution of plasmid-mediated quinolone resistance (PMQR) genes and their effect on the bacterial resistance to the quinolone antibiotics in recent years, qnrA, qnrB, qnrC, qnrD, qnrS, oqxA, oqxB and qepA genes of 93 avian E. coli strains isolated from Shandong Province were detected individually in PCR, and the resistance to Quinolones (Ciprofloxacin, Lomefl oxacin, Ofl oxacin, Norfl oxacin, Pefl oxacin) was detected in antimicrobial susceptibility test. Among 93 E. coli isolates tested, 47 (50.54%) to 80 (86.30%) strains were resistant to fi ve Quinolones respectively, and 56 (60.21%) strains were positive to PMQR genes. In addition, 25 isolates (26.88%) harbored two kinds of PMQR genes and 1 isolates (1.07%) harbored three kinds of PMQR genes. The positive rates of qnrS, oqxA and oqxB gene were 22.58% (21/93), 40.86% (38/93) and 24.73% (23/93) , respectively. None of qnrA, qnrB, qnrC, qnrD and qepA genes was detected.
Avian pathogenic Escherichia coli; drug susceptibility test; PMQR genes; PCR
S852.612
B
1674-6422(2014)04-0056-07
2014-02-14
山东省现代农业产业技术体系家禽创新团队(SDAIT-13-011-15)
孔令超,男,硕士研究生,预防兽医学专业
姜世金,E-mail: sjjiang@sdau.edu.cn
